高脂饮食诱导胰岛素抵抗小鼠血浆microRNA表达谱及其与TLR4的关系①
马 科 朱晓波 徐志伟 常晓彤
(河北北方学院临床检验诊断学重点实验室,张家口075000)
高脂饮食诱导胰岛素抵抗小鼠血浆microRNA表达谱及其与TLR4的关系①
马 科②朱晓波 徐志伟 常晓彤
(河北北方学院临床检验诊断学重点实验室,张家口075000)
目的:筛选高脂饮食诱导的胰岛素抵抗小鼠,以及应用Toll样受体4(Toll-like receptor4,TLR4)抑制剂TAK-242干预的胰岛素抵抗小鼠血浆差异表达的微小RNA(microRNA,miRNA),探讨胰岛素抵抗、TLR4和差异表达miRNAs的关系。方法:血浆标本来自前期实验的3组小鼠,即以普通基础饲料喂养(Low fat diet,LFD)的正常对照组、给予TAK-242的高脂饮食组(Treated high fat diet,HFD-T)和单纯高脂饮食组HFD-C,应用小鼠miRNA芯片检测血浆miRNA表达谱,筛选差异表达的miRNAs;采用实时荧光定量PCR方法验证芯片结果。应用生物信息学方法,以TLR4及其信号传导通路为中心,预测差异表达miRNAs的靶基因,并对靶基因分别进行GO和KEGG富集分析,以判定差异miRNAs主要影响的生物学功能或者通路。结果:miRNA基因芯片筛查结果显示,单纯高脂饮食组与正常饮食组比对,筛选出差异显著的miRNAs共计 185种,其中6种miRNAs表达上调,179种miRANs表达下调;给予TAK-242的高脂饮食组与正常饮食组比对,筛选出差异显著的miRANs 共计171种,均表达下调;给予TAK-242的高脂饮食组与单纯高脂饮食组比对,筛选出差异表达的miRNAs共计13种,均为下调。生物信息学分析结果显示,以TLR4为中心挖掘与其互作的蛋白质,共发现有10种蛋白;在TAK-242给予的高脂饮食组与单纯高脂饮食组中差异表达倍数均在1 000以上的4种miRNAs (mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p),可在TLR4的互作蛋白质或Toll样受体信号通路中找到其作用的靶基因,发现这些靶基因74%属于生物过程基因,其中转录调控因子占82%。实时荧光定量PCR检测mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p水平,变化趋势与芯片结果一致。结论:胰岛素抵抗发生时,血浆miRNAs表达谱存在改变,此变化与TLR4及其信号通路相关。该研究结果丰富了胰岛素抵抗发生的机制,为寻找胰岛素抵抗血浆miRNAs诊断标志物提供了实验依据。
高脂饮食;胰岛素抵抗;Toll样受体4;微小RNA表达谱
肥胖被认为是21世纪公共健康的最大威胁,肥胖患者忍受生活质量和期望值的降低,并提高了2型糖尿病、心血管疾病、肝的脂肪变性和癌症的发病风险[1,2]。
肥胖相关的胰岛素抵抗是糖尿病等多种疾病起始进程的早期事件,与血脂障碍、高血压、葡萄糖耐受不良和内皮组织功能紊乱相关。大量研究表明,肥胖、胰岛素抵抗患者呈现低水平的炎症反应,慢性炎症是导致胰岛素抵抗的一个重要原因[3]。Toll样受体4(Toll-like receptor 4,TLR4)是模式识别受体Toll样受体家族的一个成员,在病原识别和固有免疫反应中发挥重要作用[4]。最近研究发现,TLR4不仅参与应答病原生物入侵的天然免疫,许多无菌的炎症导致的疾病,包括肥胖的发生、发展及预后,同样依赖于TLR4信号[5]。因此,TLR4在肥胖相关的胰岛素抵抗中的作用值得深入研究。
微小RNA(microRNA,miRNA)是一类进化上高度保守的单链非编码RNA分子,通常由19~22个核苷酸构成。其主要通过抑制靶基因的翻译过程,在转录后水平负性调控基因表达。近几年,国内外学者相继报道miRNAs可以影响体内的多种代谢过程,包括糖脂代谢、胰岛素分泌、细胞分化和炎症反应[6-8]。一些miRNAs已被确定在胰岛素作用的靶组织(肝脏、脂肪组织、骨骼肌)中具有重要的生理作用[9]。进一步的研究证实miRNAs不仅存在于细胞内,在细胞外也可稳定存在,且在不同病理状态下,某些血浆miRNAs水平发生特异性改变,推测血浆miRNAs检测可能成为疾病早期诊断的新一代生物学标志物[10]。
那么,高脂饮食诱导胰岛素抵抗时,血浆miRNAs表达谱是否发生改变、此改变是否与TLR4信号相关,尚不清楚。为此,我们通过已建立的高脂饮食诱导的胰岛素抵抗小鼠模型和TLR4抑制剂TAK-242干预的胰岛素抵抗小鼠模型,筛选了胰岛素抵抗状态下血浆差异表达的miRNAs和行TLR4抑制剂TAK-242干预的胰岛素抵抗小鼠血浆miRNAs表达谱的变化,探讨TLR4、miRNAs与高脂饮食诱导胰岛素抵抗的关系。
1 材料与方法
1.1 材料
1.1.1 血浆标本 来自前期实验的3组小鼠,即以普通基础饲料喂养(Low fat diet,LFD)的正常对照组、给予TAK-242的高脂饮食组(Treated high fat diet,HFD-T)和单纯高脂饮食组HFD-C。前期实验简要概述:选取出生21 d的雄性C57BL/6小鼠36只,随机分为2组,正常对照组12只,以普通基础饲料喂养(Low fat diet,LFD),高脂饮食实验组24只,给予高脂饲料喂养(High fat diet,HFD)。待2组小鼠体重出现显著差异时,进行葡萄糖耐量实验(Glucose tolerance test,GTT)和胰岛素耐量实验(Insulin tolerance test,ITT),观察小鼠胰岛素抵抗的发生。小鼠胰岛素抵抗模型建立后,观察TLR4抑制剂TAK-242对小鼠胰岛素抵抗的作用。LFD组小鼠继续以基础饲料喂养(即LFD对照组);HFD组小鼠随机分为2组,每组12只小鼠,均继续以高脂饲料喂养,其中一组HFD小鼠腹腔注射TLR4抑制剂TAK-242,以0.5 mg TAK-242/kg体重的剂量给予,每周2次(即给予TAK-242的高脂饮食实验组,HFD-T组);另一组HFD小鼠作为单纯高脂饮食的胰岛素抵抗空白对照组(HFD-C组),只给予等体积的溶媒——二甲基亚砜(DMSO)。给予小鼠TLR4抑制剂 5个月,进行GTT和ITT后,处死小鼠。采用EDTA-K2抗凝管收集血液标本,立即分离血浆。
1.1.2 主要仪器及试剂 TGL-16B型高速台式离心机(上海飞鸽);Fresco 21型低温高速离心机、NanoDrop 2000分光光度计(Thermo公司);Agilent 2100 生物分析仪、Agilent G2505C扫描仪、Agilent G2545A 杂交炉(安捷伦公司);97型PCR仪(ABI公司);Rotor-Gene Q实时荧光定量PCR仪为德国QIAGEN公司产品。
miRNA检测试剂(美国安捷伦公司):主要包括总RNA提取试剂mirVanaTMRNA Isolation Kit (Applied Biosystem p/n AM1556 );Gene Expression Wash Pack;miRNA complete Labeling and Hyb Kit(24×);miRNA Spike In Kit;Micro Bio-Spin 6--(Bio-RAD);Package 20 backings,8 HD arrays per slide;Package 20 backings,4 arrays per slide;实验使用芯片为Agilent Mouse miRNA(8*60K,Design ID为046065)。
实时荧光定量PCR检测试剂(北京天根生化科技有限公司产品):miRNA提取试剂盒;逆转录试剂FastQuant RT Kit(with gDNase);实时荧光定量PCR试剂盒SuperReal PreMix Plus(SYBR Green I)。
1.2 方法
1.2.1 Agilent miRNA芯片检测血浆miRNA表达谱 采集LFD正常对照组(C-1)、给予TAK-242的高脂饮食组HFD-T(T-1)和单纯高脂饮食组HFD-C(F-1)小鼠血浆。将每组小鼠血浆标本混合,每个标本样本量不小于1 ml,运用Agilent miRNA芯片进行检测。
血浆标本用mirVanaTMRNA Isolation Kit提取总RNA后,经NanoDrop ND-2000分光光度仪定量,使用Agilent Bioanalyzer2100检验RNA完整性。RNA检验合格后参照芯片标准流程,进行样本的标记、芯片的杂交以及洗脱处理。首先将总RNA去磷酸化、变性,用Cyanine-3-CTP(Cy3)标记;然后标记好的溶液经纯化后进行芯片杂交;经洗脱后利用Agilent Scanner G2505C扫描得到原始图像。原始图像使用Feature Extraction软件处理提取原始数据,再利用Genespring软件进行quantile标准化处理。分析比较正常低脂饲料组(C-1)、单纯高脂组(F-1)、应用TAK-242处理组(T-1)数据。在筛选差异miRNA之前,先进行探针过滤,比较的每组标本中应至少有一组100%标记为Detected的探针才能进行后续分析。利用t检验的P值和倍数Flod change值进行差异miRNA筛选,筛选标准为上调或下调倍数变化值≥2.0并且P值≤0.05(此部分实验由北京康普森生物科技有限公司完成)。
1.2.2 茎环-逆转录实时荧光定量PCR验证芯片结果
1.2.2.1 茎环-逆转录实时荧光定量PCR引物的设计与合成 选取芯片中显著差异表达的miRNA进行验证。在miRbase数据库(http://www.mirbase.org)中获得mmu-miR-3095-3p(MIM-AT0014912)、mmu-miR-5113(MIMAT0020621)、mmu-miR-709(MIMAT0003499)和mmu-miR-335-3p(MIMAT0004704)的基因序列,在Genbank中获得内参U6(ENSMUSG00000095132)的序列,采用Primer 5.0软件设计引物,并在NCBI中分析引物的特异性。逆转录所使用的茎环引物、PCR上下游引物均由宝锐通生物科技(北京)有限公司合成。茎环引物序列mmu-miR-3095-3p:5′GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACAAA-AGC3′;mmu-miR-5113:5′GTCGTATCCAGTGCAG-GGTCCGAGGTATTCGCACTGGATACGACACAGGA3′;mmu-miR-709 :5′GTCGTATCCAGTGCAGGGTCCG-AGGTATTCGCACTGGATACGACTCCTCC3′;mmu-mi-R-335-3p:5′GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACGGTCAG3′。实时荧光定量PCR引物U6上游引物:5′ATTGGAACGATACAGAGAAGATTAGC3′;U6下游引物:5′TGGAA-CGCTTCACGAATTTG3′;mmu-miR-3095-3p上游引物:5′TGGAGAGAGAGCTTTTGCTTTTGT3′;mmu-miR-5113上游引物:5′AGGAGACAGAGGAGGAG-AGAGATC3′;mmu-miR-709上游引物:5′AGAGGAGGCAGAGGCAGGAG3′;mmu-miR-335-3p上游引物:5′TTGCTCCTGACCCTGACCG3′;通用下游引物:5′CGCAGGGTCCGAGGTATTC3′。
1.2.2.2 逆转录cDNA的合成 采用miRNA提取试剂盒进行miRNAs的抽提;FastQuant RT Kit(with gDNase)进行逆转录。去除基因组DNA的反应体系:在预处理无核酸酶的EP管中,加入5×gDNA Buffer 2 μl、RNA样本1 μl、RNase-Free-ddH2O 7 μl,混匀后简短离心,置于42℃孵育3 min,然后置于冰上放置。逆转录反应体系:10×Fast RT Buffer 2 μl、RT Enzyme 1 μl、Stem loop primers 2 μl,U6 Reverse primer 2 μl,RNase-Free ddH2O 3 μl,混匀后将此体系加入到基因组DNA去除的反应液中,充分混匀,42℃孵育15 min;95℃孵育3 min后放于冰上,得到的cDNA产物稀释15倍用于实时荧光定量PCR实验。
1.2.3 SYBR Green I实时荧光定量PCR检测差异表达的miRNAs 同一个样本设3个重复孔,同时设阴性对照孔。20 μl PCR反应体系:2×SuperReal PreMix Plus 10 μl、Forward primer 0.6 μl、Reverse primer 0.6 μl、cDNA模板1 μl、RNase-free ddH2O 7.8 μl。反应条件:95℃预变性15 min;95℃变性10 s,60℃ 退火20 s,72℃延伸27 s,共40个循环。通过溶解曲线检测样本的特异性,通过同一样本3个复孔检测的变异系数评估实验的可重复性。
1.2.4 数据的处理 实时荧光定量PCR数据采用2-ΔΔCT法进行分析,用来验证血浆miRNAs表达差异,其中ΔCt= Ct(目的基因)-Ct( U6内参基因),ΔΔCt=ΔCt(实验组)-ΔCt(对照组)。
1.2.5 靶基因预测与功能显著性分析 应用miRNA生物信息学软件TargetScan数据库对筛选出的差异miRNA进行靶基因预测。基于GO(Gene Ontology)数据库,根据靶基因的基因功能富集程度,进行GO显著性分析;基于KEGG(Kyoto Encyclopedia of Gene and Genomes)数据库,根据靶基因在信号通路中的富集程度,进行pathway显著性分析,以判定差异miRNA主要影响的生物学功能和通路。

2 结果
2.1 血浆miRNA表达谱检测及分析 采集LFD正常对照组(C-1)、给予TAK-242的高脂饮食组HFD-T(T-1)和单纯高脂饮食组HFD-C(F-1)小鼠血液分离血浆。将每组小鼠血浆标本混合,每个标本样本量不小于1 ml,运用Agilent miRNA芯片进行检测。
2.1.1 血浆样本总RNA质检结果 应用NanoDro-pND-2000检测样本浓度,利用Agilent bioanalyel 2100电泳检测RNA完整性。样品总RNA的浓度、纯度如表1所示,本实验成功提取3例血浆总RNA,浓度及纯度均达到后续芯片实验要求。
2.1.2 芯片实验检测结果 芯片检测结果显示,单纯高脂饮食实验组F-1与基础饲料对照组C-1比对,筛选出差异显著的miRNA共计185个,其中mmu-miR-335-3p、mmu-miR-5113、mmu-miR-3095-3p、mmu-let-7b-5p、mmu-let-7c-5p和mmu-miR-223-3p为上调,其余179个均为下调(详细结果略),提示高脂饮食诱导的胰岛素抵抗小鼠血浆miRNA表达谱发生改变。以给予TAK-242的T-1作为实验组,以C-1作为对照比对,筛选出差异显著的miRNA共计171个,均为下调(详细结果略),表明已产生胰岛素抵抗的高脂饮食小鼠,在给予TLR4抑制剂TAK-242 干预5个月后,血浆miRNA表达谱与基础饲料喂养的对照组同样出现差异。当以T-1作为实验组,F-1作为对照组,筛选出差异显著的miRNA共计13个,均为下调(表2,根据差异倍数降序排列),由此可见,给予TLR4抑制剂TAK-242的高脂饮食组T-1与单纯高脂饮食组F-1对比,有13个miRNA存在明显差异,这些miRNA表达谱的改变可能与TAK-242的干预有关。将上述数据导入Mev 4.6得到聚类图,其中绿色代表下调,红色代表上调,如图1所示。
表1 标本RNA质量评估结果
Tab.1 Results of sample RNA quality assessment
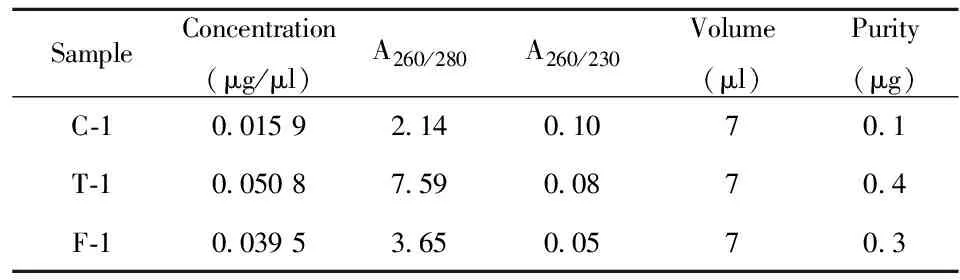
SampleConcentration(μg/μl)A260/280A260/230Volume(μl)Purity(μg)C⁃100159214010701T⁃100508759008704F⁃100395365005703
Note:C-1.Low fat diet;T-1.High fat diet with TAK-242;F-1.High fat diet without TAK-242.
2.2 实时荧光定量PCR验证芯片结果 T-1与F-1差异表达倍数显著的mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p均可在以TLR4为中心挖掘的互作蛋白质或Toll样受体信号通路中找到其作用的靶基因。故针对此4种miRNAs进行了实时荧光定量PCR验证,采用U6作为内参进行归一化。结果显示,该4种miRNAs表达均下调,与芯片检测结果一致,见图2。
表2 HFD-T和HFD-C组血浆标本差异表达的miRNAs(T-1 vs F-1)
Tab.2 Summary of miRNAs differential expressed in plasma samples with HFD-T and HFD-C(T-1 vs F-1)

miRNAs_nameFoldchange(abs)RegulatedtrendAccessionNommu⁃miR⁃39603918404downMIMAT0019336mmu⁃miR⁃3095⁃3p20071172downMIMAT0014912mmu⁃miR⁃223⁃3p1744065downMIMAT0000665mmu⁃miR⁃511315781564downMIMAT0020621mmu⁃miR⁃70914738173downMIMAT0003499mmu⁃miR⁃636613585627downMIMAT0025110mmu⁃let⁃7c⁃5p13025131downMIMAT0000523mmu⁃let⁃7b⁃5p12563236downMIMAT0000522mmu⁃miR⁃3107⁃5p12104552downMIMAT0014943mmu⁃miR⁃335⁃3p10606154downMIMAT0004704mmu⁃miR⁃624326571746downMIMAT0024863mmu⁃miR⁃1224⁃5p24811573downMIMAT0005460mmu⁃miR⁃510920247705downMIMAT0020617
Note:Selection criteria is Fold change≥2.0 andP≤0.05.
2.3 差异表达的miRNAs的生物信息学分析
2.3.1 差异表达的miRNA靶基因预测 TLR4抑制剂TAK-242的应用,影响了小鼠血浆miRNA的表达谱。为此,我们针对给予TAK-242的高脂饮食组(T-1)与单纯高脂饮食组(F-1)差异表达的miRNA,以TLR4为中心寻找以TLR4信号通路上下游基因及TLR4互作蛋白基因为靶基因的miRNA。
利用在线分析工具STRING(http://string.embl.de),以TLR4为中心挖掘与其互作的蛋白质,共发现有10种蛋白,分别为:Birc2、Birc3、Traf3、Traf6、Ticam1、Ly96、Myd88、Ube2n、Map3k7和Ikbkg。其中,Birc3、Traf3、Traf6、Map3k7和Ikbkg是mmu-miR-5113和mmu-miR-335-3p的靶基因,此两种miRNA在TAK-242给予的高脂饮食组与单纯高脂饮食组中差异表达倍数均在1 000以上(图3)。
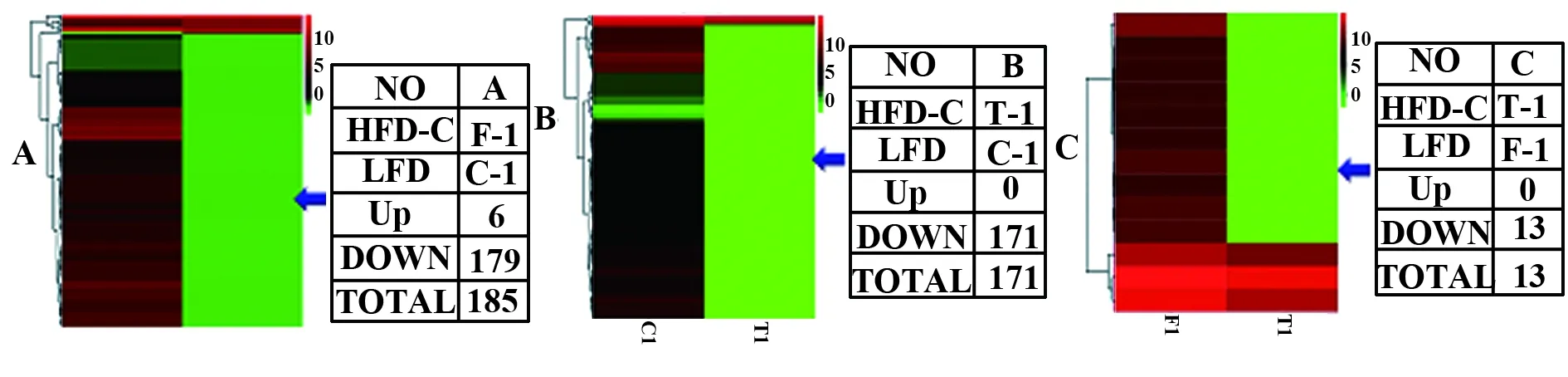
图1 三组血浆标本差异表达miRNAs的聚类分析Fig.1 Cluster analysis of expressed miRNAs in 3 plasma samplesNote:Columns represent samples and rows represent miRNAs(black,green and red correspond to unchanged,down-regulated and upregulated,respectively).A.Left panel stands for LFD sample;right panel stands for HFD-C sample;B.Left panel stands for LFD sample;right panel stands for HFD-T sample;C.Left panel stands for HFD-C sample;right panel stands for HFD-T sample.
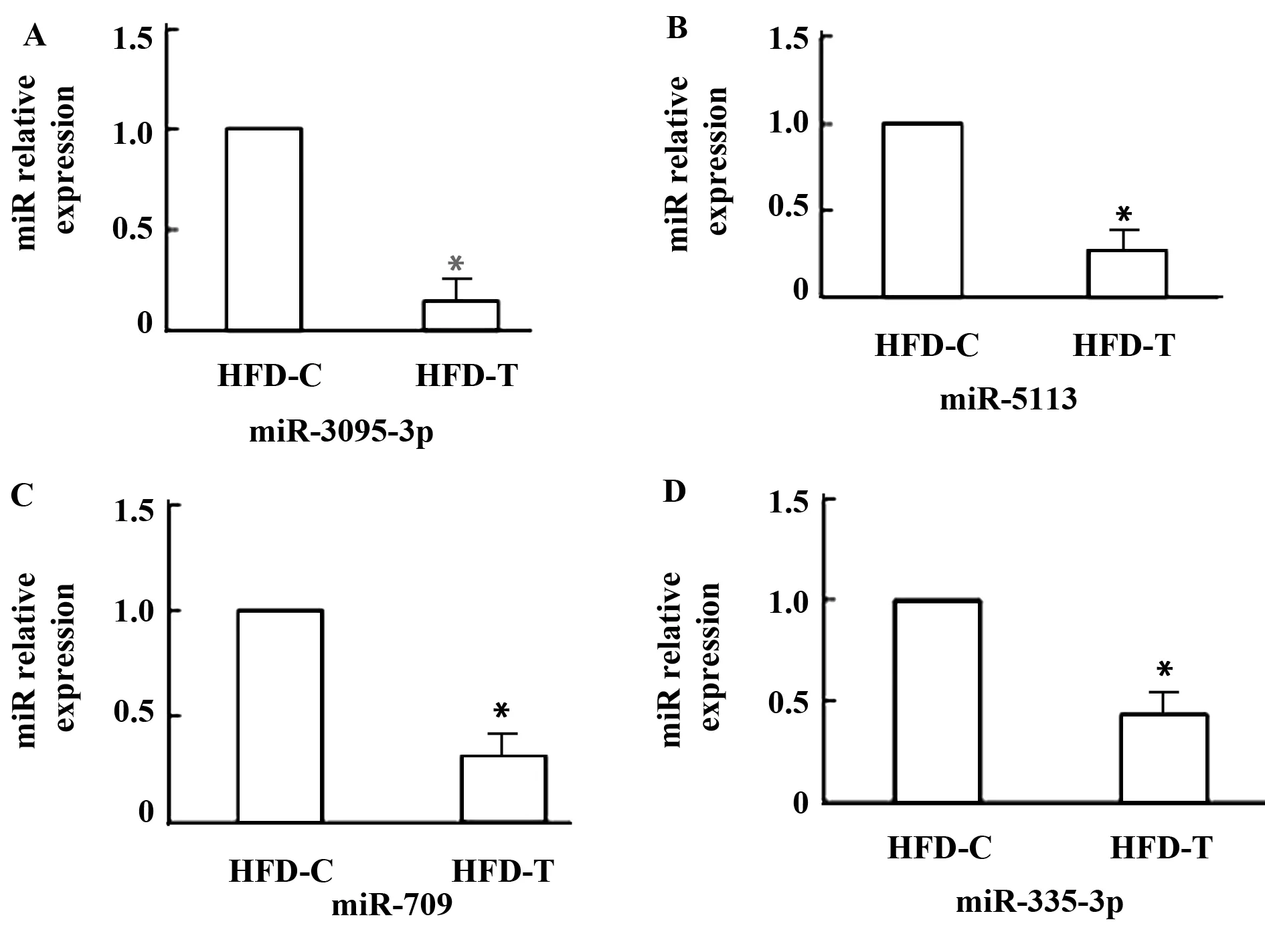
图2 实时荧光定量PCR验证miRNAs芯片结果(n=15)Fig.2 Validation of miRNAs microarray data via qRT-PCR(n=15)Note:The relative expression value of miR-3095-3p,miR-5113,miR-709 and miR-335-3p were normalized to U6 RNA level.*.P<0.01 vs HFD-C group.
利用KEGG在线分析工具,以TLR4(K0160)查找其所在的Toll样受体信号通路,其中T-1与F-1差异表达的微小RNA,mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p作用的靶基因见表3。
由此可见,T-1与F-1差异倍数较为显著的mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p可在以TLR4为中心挖掘的互作蛋白质基因或Toll样受体信号通路中找到其作用的靶基因。TAK-242作为TLR4的抑制剂,可以影响以TLR4信号通路上下游基因及TLR4互作蛋白基因为靶基因的miRNA差异表达。
2.3.2 预测靶基因的功能及分析 对F-1与C-1、T-1与C-1、T-1与F-1差异miRNA预测的靶基因分别进行GO和KEGG富集分析,判定差异miRNA主要影响的生物学功能或者通路。
基因本体(Gene ontology,GO)是一个在生物信息学领域中广泛使用的本体,分为生物过程(Biological process),细胞组分(Cellular component)和分子功能(Molecular function)三个组分。GO分析结果表明,F-1与C-1差异miRNA预测的靶基因属于生物过程中的共计11 325个,属于细胞组分的共计12 865个,属于分子功能的共计11 624个。T-1与C-1差异miRNA预测的靶基因属于生物过程中的共计11 234个,属于细胞组分的共计12 758个,属于分子功能的共计11 526个。T-1与F-1差异miRNA预测的靶基因属于生物过程中的共计53 515个,属于细胞组分的共计6 066个,属于分子功能的共计5 515个。

图3 与TLR4相互作用的分子信号通路网络Fig.3 Molecular signal-pathway networks interacted with TLR4Note:The main framework of this pathway network include of Birc2,Birc3,Traf3,Traf6,Ticam1,Ly96,Myd88,Ube2n,Map3k7 and Ikbkg.Birc3,Traf3,Traf6,Map3k7 and Ikbkg were regulated by mmu-miR-5113 and/or mmu-miR-335-3p.
KEGG又称京都基因与基因组百科全书,是用于基因组破译方面的数据库。应用KEGG分析使细胞和有机体能够在计算机上完整的表达和演绎,并让计算机利用基因信息对更高层次和更复杂细胞活动和生物体行为做出计算推测。KEGG富集分析显示F-1与C-1差异miRNA预测的靶基因的信号通路共计4 858个,T-1与C-1差异miRNA预测的靶基因的信号通路共计4 827个,T-1与F-1差异miRNA预测的靶基因的信号通路共计2 282个。见表4,将T-1与F-1差异表达的miRNA:mmu-miR-5113和mmu-miR-335-3p的靶基因Birc3、Traf3、Traf6、Map3k7和Ikbkg,以及表3中T-1与F-1差异倍数较为显著的mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p在Toll样受体信号通路中找到的靶基因进行GO和KEGG分类,发现74%属于生物过程基因(其中转录调控因子占82%)。
表3 在Toll样受体信号通路中差异表达的miRNAs的靶基因(F-1 vs T-1)
Tab.3 Targeted genes of expressed miRNAs in Toll like receptor signaling pathways(F-1 vs T-1)

SystematicnameFoldchange(abs)Targetgenesmmu⁃miR⁃3095⁃3p20071172OPNAB2mmu⁃miR⁃511315781564TOLLIPTRAMIRAK4AKTTRAF3STAT1TAB2mmu⁃miR⁃70914738173Rac1TOLLIPCD14TLR5IRAK1FADDAKTIRF3STAT1mmu⁃miR⁃335⁃3p10606154CD14TIRAPTRAMTLR5IRAK1TBK1OPNCASP8
表4 靶基因的GO分析
Tab.4 Gene ontology analysis of targeted genes
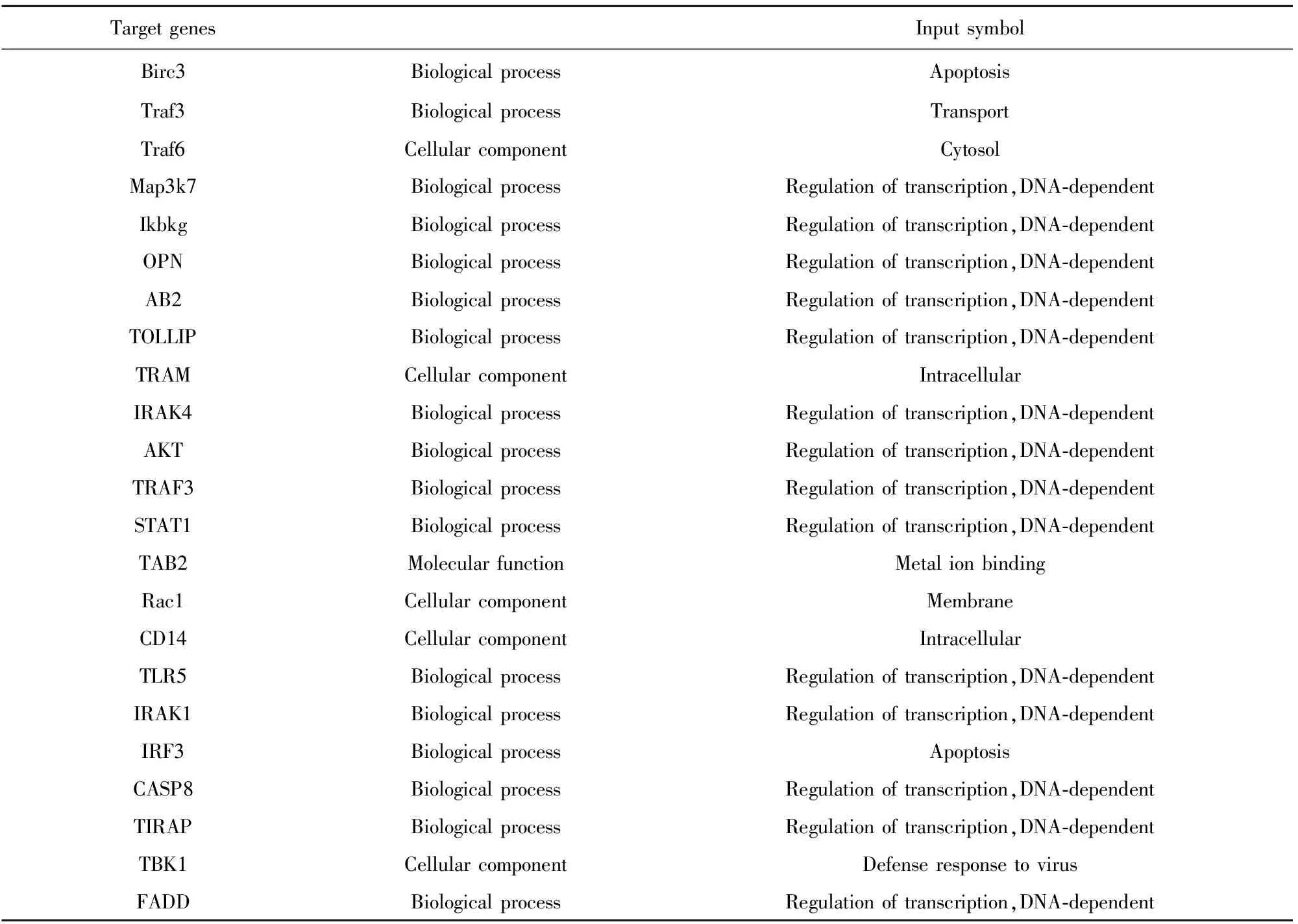
TargetgenesInputsymbolBirc3BiologicalprocessApoptosisTraf3BiologicalprocessTransportTraf6CellularcomponentCytosolMap3k7BiologicalprocessRegulationoftranscription,DNA⁃dependentIkbkgBiologicalprocessRegulationoftranscription,DNA⁃dependentOPNBiologicalprocessRegulationoftranscription,DNA⁃dependentAB2BiologicalprocessRegulationoftranscription,DNA⁃dependentTOLLIPBiologicalprocessRegulationoftranscription,DNA⁃dependentTRAMCellularcomponentIntracellularIRAK4BiologicalprocessRegulationoftranscription,DNA⁃dependentAKTBiologicalprocessRegulationoftranscription,DNA⁃dependentTRAF3BiologicalprocessRegulationoftranscription,DNA⁃dependentSTAT1BiologicalprocessRegulationoftranscription,DNA⁃dependentTAB2MolecularfunctionMetalionbindingRac1CellularcomponentMembraneCD14CellularcomponentIntracellularTLR5BiologicalprocessRegulationoftranscription,DNA⁃dependentIRAK1BiologicalprocessRegulationoftranscription,DNA⁃dependentIRF3BiologicalprocessApoptosisCASP8BiologicalprocessRegulationoftranscription,DNA⁃dependentTIRAPBiologicalprocessRegulationoftranscription,DNA⁃dependentTBK1CellularcomponentDefenseresponsetovirusFADDBiologicalprocessRegulationoftranscription,DNA⁃dependent
3 讨论
肥胖相关的2型糖尿病和心脑血管疾病的迅速攀升已成为最严重的世界健康问题之一,而毋庸置疑,肥胖诱导的胰岛素抵抗是导致许多有害代谢异常的重要环节,但其潜在的分子机制尚不清楚。一个可能的机制是肥胖与炎症应答提高有关,炎症加剧了胰岛素抵抗,此过程与天然免疫信号转导通路的活化密切相关[3]。
近十余年,关于天然免疫系统的研究最主要的进展之一,就是一组高度保守的受体——Toll样受体(Toll-like receptors,TLRs)的发现。TLR4是TLRs家族的一个成员,通过活化前炎症事件的级联信号,起始对病原生物的应答。脂多糖(Lipopolysacch-aride,LPS)是革兰氏阴性细菌的外膜成分,为TLR4的有效配体。当配体结合了TLR4和其共同受体CD14和MD-2,通过接合分子TIRAP、MyD88、IRAK和TRAF6的级联,活化MAPKs和NF-κB信号通路[4]。近期研究发现,TLR4信号通路与胰岛素抵抗存在诸多联系,TLR4与下游信号MAPKs和NF-κB在胰岛素抵抗过程中发挥着重要作用。来源于营养和代谢的LPS(代谢性内毒素血症)以及饱和的非酯化脂肪酸(Non-esterified fatty acid,NEFA)均可活化TLR4[11-13]。
为了探讨肥胖相关的胰岛素抵抗作用机制,首先建立了高脂饮食诱导的胰岛素抵抗小鼠模型。接着给予胰岛素抵抗小鼠TLR4特异性抑制剂TAK-242进行干预。前期结果提示,出现胰岛素抵抗的小鼠,在给予TLR4抑制剂5个月后,TLR4蛋白的表达部分受到抑制,使之对葡萄糖的调节能力和对胰岛素的敏感性均有所改善,提示TAK-242部分改善了高脂导致的炎症反应,抑制TLR4有利于提高胰岛素的敏感性,改善胰岛素抵抗状态(论文待发表)。
受体是信号通路的重要门户,因此受体的调节对其信号通路至关重要。已知miRNAs是一类内源性的、非编码的短链RNA分子,其通过与靶mRNA的3′非翻译区(3′untranslated region,3′UTR)结合抑制靶基因的翻译表达,在多种生理过程中执行着重要的调控作用[14]。来自肥胖相关的2型糖尿病的激素敏感组织(肝脏、肌肉、脂肪组织)的研究,揭示有几种miRNA表达的改变,推测miRNA在相关疾病中发挥着重要作用[15,16]。有研究发现miRlet-7家族可以负性调控TLR4,当miRlet-7i过表达时,TLR4表达则减少[17,18]。此外,在小鼠腹腔巨噬细胞中发现TLR4是let-7e的靶基因,let-7e高表达可以明显降低TLR4水平;当TLR4 mRNA的3′UTR区与let-7e结合部位的基因序列发生突变时,这种负性调控就会消除[19]。最近研究发现,外周血中也能检测到miRNAs,而且发现许多疾病患者血浆中都有其特征性的miRNAs表达谱[20]。那么,高脂饮食诱导胰岛素抵抗时,血浆miRNAs表达谱是否发生改变、此改变是否与TLR4信号相关呢?
microRNA基因芯片筛查结果显示,单纯高脂饮食组与正常饮食组比对,筛选出差异显著的miRNAs共计 185种,其中6种miRNAs表达上调,179种miRANs表达下调,说明高脂饮食诱导的胰岛素抵抗小鼠血浆miRNA表达谱发生改变。给予TLR4抑制剂的高脂饮食组与正常饮食组比对,筛选出差异显著的miRANs 共计171种,均表达下调,表明已产生胰岛素抵抗的高脂饮食小鼠,在给予TLR4抑制剂TAK-242后,血浆miRNA表达谱与正常饮食组也存在差异。当给予TAK-242的高脂饮食组与单纯高脂饮食组比对,筛选出差异表达的miRNAs共计13种,均为下调,说明此13种miRNAs表达的改变可能与TLR4蛋白表达的部分抑制有关。
进一步的生物信息学分析结果显示,以TLR4为中心挖掘与其互作的蛋白质,共发现有10种蛋白;在TAK-242给予的高脂饮食组与单纯高脂饮食组中差异表达倍数均在1 000以上的4种miRNAs (mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709、mmu-miR-335-3p),可在TLR4的互作蛋白质或Toll样受体信号通路中找到其作用的靶基因。将这些靶基因进行功能富集分析和信号转导通路富集分析(GO和KEGG分类),发现74%属于生物过程基因,其中转录调控因子占82%。提示TAK-242作为TLR4的抑制剂,可以影响以TLR4信号通路上下游基因及TLR4互作蛋白基因为靶基因的miRNA差异表达。已有文献报道,hsa-miR-335在胃癌复发患者血浆中表达升高[21];mmu-miR-709可能参与了人胚肾细胞的凋亡[22]和脂肪细胞的分化[23];mmu-miR-5113在与系统性红斑狼疮相关的极化巨噬细胞中表达下调[24]。目前关于mmu-miR-3095-3p、mmu-miR-5113、mmu-miR-709和mmu-miR-335-3p在胰岛素抵抗中的作用,以及与TLR4信号通路的相关性未见报道。进一步课题组将在不同标本中验证这4种差异表达miRNAs,并对其进行深入的功能分析,确定这些差异表达的miRNA在TLR4信号通路中的作用及在胰岛素抵抗中扮演的角色。
综上,胰岛素抵抗发生时,血浆miRNAs表达谱存在改变,此变化与TLR4及其信号通路相关。该研究结果丰富了胰岛素抵抗发生的机制,为进一步深入研究TLR4、miRNAs在胰岛素抵抗中的作用、及为寻找胰岛素抵抗血浆miRNAs诊断标志物提供了实验依据。
[1] Cao H.Adipocytokines in obesity and metabolic disease[J].J Endocrinol,2014,220(2):T47-T59.
[2] Honors MA,Kinzig KP.Diet-induced obesity and insulin resistance spur tumor growth and cancer cachexia in rats bearing the Yoshida sarcoma[J].Nut Cancer,2014,66(5):872-888.
[3] Könner AC,Brüning JC.Toll-like receptors:linking inflammation to metabolism[J].Trends Endocrinol Metab,2011,22(1):16-23.
[4] 常晓彤,辇晓峰,王振辉.Toll样受体信号转导途径研究进展[J].生理科学进展,2011,42(5):340-346.
[5] Jialal I,Kaur H,Devaraj S.Toll-like receptor status in obesity and metabolic syndrome:a translational perspective[J].J Clin Endocrinol Metab,2014,99(1):39-48.
[6] Zhao Z,Ma X,Hsiao TH,etal.A high-content morphological screen identifies novel microRNAs that regulate neuroblastoma cell differentiation[J].Oncotarget,2014,5(9):2499-2512.
[7] Chakraborty C,Doss CG,Bandyopadhyay S,etal.Influence of miRNA in insulin signaling pathway and insulin resistance:micro-molecules with a major role in type-2 diabetes[J].Wiley Interdiscip Rev RNA,2014,5(5):697-712.
[8] Rebane A,Akdis CA.MicroRNAs:Essential players in the regulation of inflammation[J].J Allergy Clin Immunol,2013,132(1):15-26.
[9] Kim E.Insulin resistance at the crossroads of metabolic syndrome:systemic analysis using microarrays[J].Biotechnol J,2010,5(9):919-929.
[10] Rome S.Are extracellular microRNAs involved in type 2 diabetes and related pathologies?[J].Clin Biochem,2013,46(10-11):937-945.
[11] Razolli DS,Moraes JC,Morari J,etal.TLR4 expression in bone marrow-derived cells is both necessary and sufficient to produce the insulin resistance phenotype in diet-induced obesity[J].Endocrinology,2015,156(1):103-113.
[12] Liang H,Hussey SE,Sanchez-Avila A,etal.Effect of lipopolysaccharide on inflammation and insulin action in human muscle[J].PLoS One,2013,8(5):1-8.
[13] Hussey SE,Liang H,Costford SR,etal.TAK-242,a small-molecule inhibitor of Toll-like receptor 4 signalling,unveils similarities and differences in lipopolysaccharide-and lipid-induced inflammation and insulin resistance in muscle cells[J].Biosci Rep,2012,33(1):37-47.
[14] Pillar N,Yoffe L,Hod M,etal.The possible involvement of microRNAs in preeclampsia and gestational diabetes mellitus[J].Best Pract Res Clin Obstet Gynaecol,2015,29(2):176-182.
[15] Herrera BM,Lockstone HE,Taylor JM,etal.Global microRNA expression profiles in insulin target tissues in a spontaneous rat model of type 2 diabetes[J].Diabetologia,2010,53(6):1099-1109.
[16] Herrera BM,Lockstone HE,Taylor JM,etal.MicroRNA-125a is over-expressed in insulin target tissues in a spontaneous rat model of Type 2 Diabetes[J].BMC Med Genomics,2009,doi:10.1186/1755-8794-2-54.
[17] Chen XM,Splinter PL,O′Hara SP,etal.A cellular micro-RNA,let-7i,regulates Toll-like receptor 4 expression and contributes to cholangiocyte immune responses against Cryptosporidium parvum infection[J].J Biol Chem,2007,282(39):28929-28938.
[18] Satoh M,Tabuchi T,Minami Y,etal.Expression of let-7i is associated with Toll-like receptor 4 signal in coronary artery disease:effect of statins on let-7i and Toll-like receptor 4 signal[J].Immunobiology,2012,217(5):533-539.
[19] Androulidaki A,Iliopoulos D,Arranz A,etal.The kinase Akt1 controls macrophage response to lipopolysaccharide by regulating microRNAs[J].Immunity,2009,31(2):220-231.
[20] Mo MH,Chen L,Fu Y,etal.Cell-free circulating miRNA biomarkers in cancer[J].J Cancer,2012,3:432-448.
[21] Yan Z,Xiong Y,Xu W,etal.Identification of hsa-miR-335 as a prognostic signature in gastric cancer[J].PLoS One,2012,7(7):1-10.
[22] Tang R,Li L,Zhu D,etal.Mouse miRNA-709 directly regulates miRNA-15a/16-1 biogenesis at the posttranscriptional level in the nucleus:evidence for a microRNA hierarchy system[J].Cell Res,2012,22(3):504-515.
[23] Chen H,Mo D,Li M,etal.miR-709 inhibits 3T3-L1 cell differentiation by targeting GSK3β of Wnt/β-catenin signaling[J].Cell Signal,2014,26(11):2583-2589.
[24] Xiao P,Dong C,Yue Y,etal.Dynamic expression of microRNAs in M2b polarized macrophages associated with systemic lupus erythematosus[J].Gene,2014,547(2):300-309.
[收稿2016-05-23]
(编辑 张晓舟)
Plasma microRNA expression profile in mice with high fat diet-induced insulin resistance and its relationship with TLR4
MA Ke,ZHU Xiao-Bo,XU Zhi-Wei,CHANG Xiao-Tong.
Key Laboratory of Clinical Diagnostics,Hebei North University,Zhangjiakou 075000,China
Objective:To screen the plasma microRNAs(miRNAs)of differential expression in a high fat diet-induced insulin resistance in mouse models;further investigations on the mice with insulin resistance treated by TLR4 inhibitors TAK-242,and to study the changes of plasma miRNAs expression profile and the relationship among TLR4,miRNAs and high fat diet-induced insulin resistance.Methods:The plasma samples were from 3 mouse groups of previous study,namely,the control group with general basic diet(low fat diet,LFD),TLR4 inhibitors TAK-242 treatment group with a high fat diet (HFD-T) and the high fat diet control group(HFD-C).The differential expressed miRNAs was screened by expression profiling of plasma miRNAs,which was detected using mouse miRNA microarray.The quantitative Real-Time PCR(qRT-PCR) was used to verify the results of microarray.The target genes of differential expressed miRNAs were predicted in TLR4 signaling pathway using bioinformatics methods,and the GO and KEGG database molecular annotation system were used to investigate the main effects of the miRNAs targeted genes on the biological functions or signal pathway.Results:The screening results of miRNA microarray chip showed that,comparing miRNAs expression between HFD group and LFD control group,185 miRNAs were significant in the high fat diet group,including 6 up-regulated and 179 down-regulated miRANs.A significant difference of miRANs was also found between HFD-T group and LFD control group,the total number of differential expression miRNAs was 171,and all of them were down-regulated.Comparing miRNAs expression between HFD-C group and HFD-T group,13 miRNAs were significant in HFD-T group,all of them were down-regulated.Bioinformatics analysis results showed that a total of 10 interaction proteins with TLR4 were predicted;the difference of mmu-miR-3095-3p,mmu-miR-5113,mmu-miR-709 and mmu-miR-335-3p expression levels was more than 1 000 times between HFD-C group and HFD-T group,and their target genes can be found in TLR4-interaction protein or Toll like receptor signaling pathway;GO and KEGG analysis showed 74% of these target genes belonged to the biological processes genes,and the transcription factors accounted for 82%.The expression of mmu-miR-3095-3p,mmu-miR-5113,mmu-miR-709 and mmu-miR-335-3p detected by qRT-PCR exhibited the similar patterns of down regulation to those shown in microarray results.Conclusion:When insulin resistance occurs,there is a change in plasma miRNAs expression profile,this change is associated with TLR4 and its signaling pathways.The finding enrichs the possible mechanisms of insulin resistance and provides a basis for finding miRNAs diagnostic markers for early diagnosis of insulin resistance.
High fat diet;Insulin resistance;Toll like receptor 4;miRNAs expression profile
10.3969/j.issn.1000-484X.2016.12.004
①本文为河北省自然科学基金资助项目(C2011405015)和河北北方学院创新人才培育项目(CXRC1316)。
马 科(1977年-),男,硕士,主要从事脂代谢障碍相关疾病的分子机制与临床应用方面的研究。
及指导教师:常晓彤(1968年-),女,硕士,教授,主要从事内分泌的分子免疫学研究,E-mail:changxt1212@vip.sina.com。
R392.11
A
1000-484X(2016)12-1745-08
②河北北方学院附属第一医院检验科,张家口075000。

