XRCC1基因Arg280His多态与肺癌易感性关系的Meta分析
谭晓刚 王若天 姚舒洋 支修益
(首都医科大学宣武医院胸外科 首都医科大学肺癌中心,北京 100053)
· 肺癌精准治疗 ·
XRCC1基因Arg280His多态与肺癌易感性关系的Meta分析
谭晓刚 王若天 姚舒洋 支修益*
(首都医科大学宣武医院胸外科 首都医科大学肺癌中心,北京 100053)
目的 综合评价XRCC1 Arg280His多态与肺癌易感性的关系。方法 检索中国学术期刊文献数据库和Pubmed,查找有关XRCC1 Arg280His多态与肺癌易感性的关系的病例对照研究,并用Meta分析的方法分析XRCC1 Arg280His多态与肺癌易感性的关系,然后进行亚组分析、敏感性分析和文献的发表偏倚检验。结果 本次Meta分析共纳入14篇文献,累计病例数5 711例,对照6 202例,显性模型下His/His+Arg/His基因型相对于Arg/Arg基因型的OR值为0.97,95%可信区间为 0.78~1.22,无统计学意义关系。结论XRCC1 Arg280His多态与肺癌易感性无统计学意义关系。
XRCC1 Arg280His; 多态; 肺癌; Meta分析
肺癌是影响人类健康的主要肿瘤之一,每年约有超过一百万人死于肺癌[1]。最近的研究[2]显示个体间的基因多态可能会造成不同个体对肺癌易感性的差异。X射线损伤修复的交叉互补基因(X-ray repair cross complementing group 1,XRCC1)是一种DNA损伤修复基因,其基因型的不同会导致个体DNA损伤修复能力的改变,从而影响肿瘤发生的危险性[3]。到目前为止,共有14个XRCC1的Arg280His多态[即Ex9+16 G>A (rs25489)]与肺癌易感性关系的病例对照实验,但结果不一致。因而笔者通过Meta分析的方法分析已经存在的数据,以综合评价XRCC1的Arg280His多态与肺癌易感性之间的关系。
1 材料与方法
1.1 文献检索
首先使用关键词包括“XRCC1”,“Arg280His”,“lung cancer”,“polymorphism”,“肺癌”, “多态”,检索CNKI和Pubmed数据库,以得到XRCC1 Arg280His多态和肺癌易感性的关系的所有病例对照研究。最后检索日期为2016年8月5日。同时笔者也采用了文献追溯及人工筛选的方法。纳入文献的标准:1)采用的病例-对照研究设计;2)有各种基因型频率的数据。文献排除标准:1)病例组为肺癌患者,对照组为健康人群;2)对照组研究资料的基因型频率分布满足Hardy-Weinberg平衡(HWE)。
1.2 数据提取
提取的数据包括:国家、作者、种族来源、发表时间、病例组和对照组中XRCC1 Arg280His基因型数据,年龄的匹配情况,病例和对照特征, SNP的检测方法等。2个研究人员独立提取数据,最后所有项目都要达到一致。
1.3 统计学方法
对各研究的原始数据进行整理,以OR值和95%CI作为每个研究结果的研究效应测定指标。首先对各研究结果进行异质性检验来决定Meta分析的模型,如果异质性显著,使用D-L随机效应模型进行统计合并;如果异质性不显著,使用M-H固定效应模型进行数据合并[4]。合并OR值的显著性用Z检验来计算[5]。
文献敏感性分析是根据各研究中显性模型下Arg/His+His/His基因型及Arg/Arg基因型的数据,用依次减少1篇文献的方法来进行。发表偏倚的检验使用Egger线性回归的方法进行[6]。所用的软件为STATA 9。所有的P值都是双尾检验。
2 结果
2.1 纳入文献及特征
通过设定的关键词及纳入标准,在中国学术期刊文献数据库中找到了2篇相关文献,在Pubmed上找到了14篇相关文献,因而共有16篇符合本研究的文献纳入标准。但Han等[7]的文献数据与Kang等[8]的文献数据重复故排除,王娜(Wang)在2篇文献[9-10]中使用了同一批样本,故采用样本数量较多的文献[9],因而在本Meta分析中共纳入14篇文献(表1),它们都在文献中提到或经过计算其对照组基因型分布,基因型数据符合Hardy-Weinberg平衡。本Meta分析累计病例5 711例,对照6 202例。纳入的文献中有11篇文献在文中提到了病例组和对照组性别、年龄相匹配,另有3篇没有提及。关于SNP的检测方法,纳入的文献分别采用了经典的RFLP的方法、 Taqman的方法及Chip的方法等进行检测。这些文献中有2篇文献显示Arg280His多态和肺癌易感性呈负相关,1篇文献显示Arg280His多态和肺癌易感性呈正相关,其余11篇没有统计学意义关系。
表1 Meta分析中引用文献的特征和病例对照中基因型分布
Tab. 1 The characteristics of citation in Meta analysis and the genotype distribution in case control

AuthorPublishedyearEthnics(Country)CasesArg/ArgArg/HisHis/HisControlsArg/ArgArg/HisHis/HisMatchedDNAsourceDetectedmethodHWEChang[11]2008Caucasian(America)86251242515YBloodChipYDeRuyck[12]2007Caucasian(Belgium)1054096140YBloodRFLPYHao[13]2006Asian(China)848169790420410YBloodRFLPYHung[14]2005Caucasian(European)1901181618961906YBloodTaqmanYKim[15]2010Asian(Korea)107333180331NBloodChipYLi[16]2008Asian(China)26679574724YBlood+tissueRFLPYMisra[17]2003Caucasian(Finland)260472260420YBloodTaqmanYRatnasinghe[18]2001Asian(China)83203177320YBloodTaqmanYSchneider[19]2005Caucasian404402562600NBloodReal⁃timeYShen[20]2005Asian(China)7630581281YSputumReal⁃timeYVogel[21]2004Caucasian(Denmark)229261241280YBloodReal⁃timeYYin[22]2007Asian(China)190462179594YBloodRFLPYWang[9]2012Asian(China)13653201846111YBloodRFLPYKang[8]2015Asian(China)10087231098219YBloodRFLPY
HWE:Hardy-Weinberg equilibrium;Y:yes;N:No.
2.2 Meta分析结果
笔者首先在显性模型下对各文献数据整体上进行异质性检验,由于存在异质性(P=0.000),因而采用随机效应模型D-L法进行数据合并。显性模型下,His/His+Arg/His基因型相对于Arg/Arg基因型合并OR值为0.97(95%CI: 0.78~1.22),OR值经Z检验(Z=0.23,P=0.820),没有统计学意义(图1)。同时笔者也进行了显性模型下亚组分析,亚洲人His/His+Arg/His基因型相对于Arg/Arg基因型OR为0.99(95%CI:0.69~1.43),也没有统计学意义(采用随机效应模型合并),欧洲人His/His+Arg/His基因型相对于Arg/Arg基因型OR为 0.98(95%CI:0.84~1.15),也没有统计学意义(采用固定效应模型合并)。
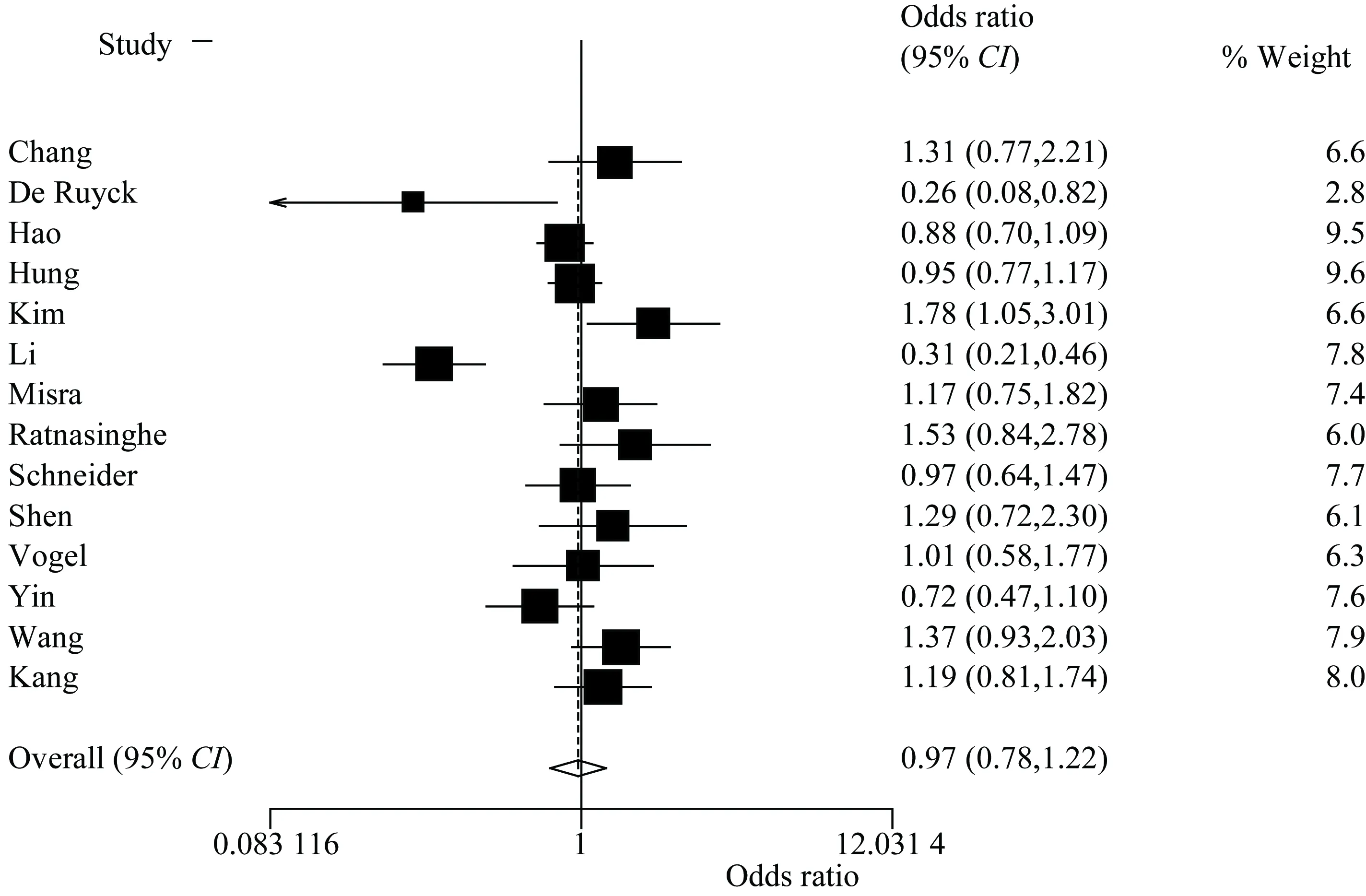
图1 显性模型下His/His+Arg/His基因型相对于Arg/Arg基因型肺癌易感性OR值的整体Meta分析
Fig.1 Overall Meta analysis of His/His+Arg/His genotype in the dominant model with
respect to the susceptibility of lung cancer to Arg/Arg genotypeOR(odd ratio)
另外,笔者也分析了其他模型下OR值的合并情况。在等位基因模型下,His等位基因相对于Arg 等位基因OR值为1.02(95%CI:0.83~1.25)(采用随机效应模型合并),等位基因模型下,亚洲人亚组OR值为1.05(95%CI:0.76~1.47)(采用随机效应模型合并),欧洲人亚组OR值为0.99(95%CI:0.86~1.15)(采用固定效应模型合并);两组人群均没有统计学意义,说明XRCC1 Arg280His多态与肺癌易感性的关系没有统计学意义(表2)。由于His/His基因型的人数很少,所以没有进行隐性模型的分析。
表2 不同基因模型下整体及亚组Meta分析结果OR值及可信区间
Tab.2OR(odd ratio) value andCI(confidence interval) of the whole and subgroup Meta analysis under different genetic models

ModelAllCaucasiansubgroupAsiansubgroupDominantgeneticmodel(His/His+Arg/HisvsArg/Arg)OR=0.97(95%CI:0.78-1.22)OR=0.98(95%CI:0.84-1.15)OR=0.99(95%CI:0.69-1.43)Allelegeneticmodel(HisvsArg)OR=1.02(95%CI:0.83-1.25)OR=0.99(95%CI:0.86-1.15)OR=1.05(95%CI:0.76-1.47)
2.3 敏感性分析和发表偏倚的评估
在显性模型下对文献进行敏感性分析显示Meta分析结果稳定,任意1篇文献去除后Meta分析结果均无明显变化。笔者更进一步使用了Egger’s检验来分析文献发表偏倚,其中t=0.25,P=0.81,显示没有发表偏倚存在。
3 讨论
本Meta分析总结了14个病例对照研究,累计病例5 711例,对照6 202例。本Meta分析揭示了XRCC1 Arg280His多态位点在显性模型中His/His+Arg/His基因型相对于Arg/Arg基因型的肺癌易感性的OR值为0.97 ,XRCC1 Arg280His多态与肺癌易感性的相关性没有统计学意义关系。同时进行的其他模型分析与亚组分析也没有发现XRCC1 Arg280His多态与肺癌易感性相关性具有统计学意义关系。
人类XRCC1基因有17个外显子,大小为31.9 KB,位于染色体19q13.2,编码633个氨基酸。XRCC1基因是一个BER损伤修复途径重要组成部分,能修复电离辐射和烷基化合物引起的碱基损伤和单链断裂损伤。尽管没有任何已知的酶活动,XRCC1可能作为一个支架蛋白,为BER的不同功能发挥协调作用[23]。XRCC1 Arg280His多态位于XRCC1的APE (apurinic/apyrimidinic endonuclease)结合区域,其氨基酸的改变可能影响DNA修复能力,从而影响肿瘤的易感性[24]。但由于肿瘤是一种多因素诱导、多基因参与、多步骤发展的疾病,有很多混杂因素,而XRCC1 Arg280His多态只是具有微小的效应,这可能导致合并OR值没有统计学意义。
XRCC1 Arg280His多态与其他肿瘤的易感性关系的研究也有被报道。如Yang等[25]研究了子宫平滑肌瘤与XRCC1 Arg280His多态的关系,发现XRCC1His等位基因多态会增加子宫平滑肌瘤风险。García-Quispes等[26]分析了XRCC1 Arg280His多态与甲状腺癌的易感性研究,结果显示XRCC1 Arg280His多态会增加甲状腺癌风险。另外,Sangrajrang等[27]发现XRCC1 Arg280His多态在泰国妇女中会降低乳腺癌的易感性,这可能和不同的肿瘤位置和遗传背景有关。邓小丽等[28]发现中国人中XRCC1 Arg280His多态与胃癌易感性没有统计学意义关系,Applebaum等[29]发现美国人中XRCC1 Arg280His多态与头颈癌风险没有统计学意义的关系,这与本文Meta分析结果一致,说明XRCC1 Arg280His多态可能只是一个微效基因多态。
笔者进行了在显性模型下的敏感性分析,没有发现哪篇文献对合并OR值有明显的影响,说明Meta分析的结果比较稳定。笔者通过Egger’s检验也没有发现文献发表偏倚,但合并OR值可能受到一些因素的影响。尽管可考虑的因素都用来评估XRCC1 Arg280His多态与肺癌的相关性,笔者的Meta分析存在着源发表文献的局限性。首先,人口统计学的参数不是非常配对,比如在有些研究中没有按照年龄配对,其次,不同的种族中遗传背景不同,这可能一定程度影响XRCC1 Arg280His多态与肺癌相关性的结果。
综上所述,笔者的Meta分析显示XRCC1 Arg280His多态与肺癌易感性没有统计学意义的关系,但XRCC1 Arg280His可能是一个具有微效作用的多态。
[1] Guilbert J J. The world health report 2002-reducing risks, promoting healthy life[J]. Educ Health (Abingdon),2003, 16(2):230.
[2] Tomescu D, Kavanagh G, Ha T,etal. Nucleotide excision repair gene XPD polymorphisms and genetic predisposition to melanoma[J]. Carcinogenesis,2001,22(3):403-408.
[3] Thompson L H,West M G. XRCC1 keeps DNA from getting stranded[J]. Mutat Res, 2000, 459(1) : 1-18.
[4] Lau J, Ioannidis J P, Schmid C H. Quantitative synthesis in systematic reviews [J]. Ann Intern Med,1997, 127(9):820-826.
[5] Petitti D B. Meta-analysis, decision analysis, and cost-effectiveness analysis [M]. New York: Oxford University Press, 1994.
[6] Egger M, Davey Smith G, Schneider M, et al. Bias in meta-analysis detected by a simples, graphical test[J]. BMJ, 1997, 315(7109):629-634.
[7] Han J C, Zhang Y J, Li X D.Association between polymorphisms in the XRCC1 gene and the risk of non-small cell lung cancer[J].Genet Mol Res, 2015,14(4):12888-12893.
[8] Kang S, Ma Y, Liu C,et al.Assoication of XRCC1 gene polymorphisms with risk of non-small cell lung cancer[J].Int J Clin Exp Pathol, 2015, 1(4):4171-4176.
[9] 王娜,吴拥军,周晓蕾,等. 河南省人群五种基因多态性与肺癌易感性的关系[J]. 卫生研究, 2012,41 (2):251-256.
[10]王娜,吴拥军,吴逸明,等.CYP1A1和XRCC1基因多态性与肺癌易感性[J]. 郑州大学学报:医学版,2012,47(1):27-30.
[11]Chang J S, Wrensch M R, Hansen H M, et al. Base excision repair genes and risk of lung cancer among San Francisco Bay Area Latinos and African-Americans[J]. Carcinogenesis,2009, 30(1):78-87.
[12]De Ruyck K, Szaumkessel M, De Rudder I, et al. Polymorphisms in base-excision repair and nucleotide-excision repair genes in relation to lung cancer risk[J]. Mutat Res,2007, 631(2):101-110.
[13]Hao B, Miao X, Li Y, et al. A novel T-77C polymorphism in DNA repair gene XRCC1 contributes to diminished promoter activity and increased risk of non-small cell lung cancer[J]. Oncogene,2006, 25(25): 3613-3620.
[14]Hung R J, Brennan P, Canzian F, et al. Large-scale investigation of base excision repair genetic polymorphisms and lung cancer risk in a multicenter study[J].J Natl Cancer Inst,2005, 97(8):567-576.
[15]Kim I S, Lee G W, Kim D C,et al. Polymorphisms and haplotypes in the XRCC1 gene and the risk of advanced non-small cell lung cancer[J]. J Thorac Oncol,2010,5(12):1912-1921.
[16]Li M, Yin Z, Guan P, et al. XRCC1 polymorphisms, cooking oil fume and lung cancer in Chinese women nonsmokers[J]. Lung Cancer,2008, 62(2):145-151.
[17]Misra R R, Ratnasinghe D, Tangrea J A, et al. Polymorphisms in the DNA repair genes XPD, XRCC1, XRCC3, and APE/ref-1, and the risk of lung cancer among male smokers in Finland[J]. Cancer Lett,2003, 191(2):171-178.
[18]Ratnasinghe D, Yao S X, Tangrea J A, et al. Polymorphisms of the DNA repair gene XRCC1 and lung cancer risk[J]. Cancer Epidemiol Biomarkers Prev,2001, 10(2):119-123.
[19]Schneider J, Classen V, Bernges U,et al. XRCC1 polymorphism and lung cancer risk in relation to tobacco smoking[J]. Int J Mol Med,2005, 16(4):709-716.
[20]Shen M, Berndt S I, Rothman N, et al. Polymorphisms in the DNA base excision repair genes APEX1 and XRCC1 and lung cancer risk in Xuan Wei, China[J]. Anticancer Res,2005, 25(1B):537-542.
[21]Vogel U, Nexo B A, Wallin H, et al. No association between base excision repair gene polymorphisms and risk of lung cancer[J]. Biochem Genet,2004, 42(11-12):453-460.
[22]Yin J, Vogel U, Ma Y, et al. The DNA repair gene XRCC1 and genetic susceptibility of lung cancer in a northeastern Chinese population[J]. Lung Cancer,2007, 56(2):153-160.
[23]Ladiges W C. Mouse models of XRCC1 DNA repair polymorphisms and cancer[J]. Oncogene,2006,25(11):1612-1619.
[24]Hung R J, Hall J, Brennan P, et al. Genetic polymorphisms in the base excision repair pathway and cancer risk: a HuGE review[J]. Am J Epidemiol,2005, 162(10):925-942.
[25]Yang Y, Zhai X D, Gao L B, et al. Genetic polymorphisms of DNA repair gene XRCC1 and risk of uterine leiomyoma[J]. Mol Cell Biochem,2010, 338(1-2):143-147.
[26]García-Quispes W A, Pérez-Machado G, Akdi A, et al. Association studies of OGG1, XRCC1, XRCC2 and XRCC3 polymorphisms with differentiated thyroid cancer[J].Mutat Res,2011,709-710:67-72.
[27]Sangrajrang S I, Schmezer P, Burkholder I, et al. Polymorphisms in three base excision repair genes and breast cancer risk in Thai women[J].Breast Cancer Res Treat,2008,111(2):279-288.
[28]邓少丽, 陈鸣,陈伟,等. DNA修复基因多态性与胃癌易感性的关系[J]. 分子诊断与治疗杂志,2010,2(6):371-374.
[29]Applebaum K M, McClean M D, Nelson H H, et al. Smoking modifies the relationship between XRCC1 haplotypes and HPV16-negative head and neck squamous cell carcinoma[J]. Int J Cancer,2009, 124(11):2690-2696.
编辑 陈瑞芳
Meta analysis of the association between the polymorphism ofXRCC1 Arg280His and lung cancer susceptibility
Tan Xiaogang, Wang Ruotian, Yao Shuyang, Zhi Xiuyi*
(DepartmentofThoracicSurgery,XuanwuHospital,CapitalMedicalUniversity,LungCancerCenterofCapitalMedicalUniversity,Beijing100053,China)
Objective To evaluate the association betweenXRCC1 Arg280His and lung cancer susceptibility. Methods To collect the reports of case-control studies on the association betweenXRCC1 Arg280His and lung cancer susceptibility, we searched CNKI and Pubmed, then evaluated the association ofXRCC1 Arg280His and lung cancer susceptibility by Meta analysis. Then we performed subgroup analysis and sensitivity analysis and publication bias test. Results There were 14 studies with 5 711 cases and 6 202 controls enrolled in this Meta-analysis. The pooledORof His/His+Arg/His genotype compared to Arg/Arg genotype under dominant model was 0.97 without statistically significant association (95%CI: 0.78-1.22). Conclusion TheXRCC1 Arg280His polymorphism was not statistically significantly associated with the lung cancer susceptibility.
XRCC1 Arg280His; polymorphism; lung cancer; Meta analysis
国家自然科学基金青年科学基金项目(81101773),北京市自然科学基金(7132106),首都医科大学基础-临床科研合作基金(14JL25,16JL67)资助。This study was supported by National Natural Science Foundation of China(81101773), Natural Science Foundation of Beijing(7132106), Capital Medical University Foundation-Clinical Research Cooperation Fund(14JL25,16JL67).
时间:2016-12-14 20∶19
http://www.cnki.net/kcms/detail/11.3662.r.20161214.2019.036.html
10.3969/j.issn.1006-7795.2016.06.008]
R 734.2
2016-10-03)
*Corresponding author, E-mail: xiuyizhi2015@163.com

