茶树CsSMT基因的克隆、原核表达及植物超表达载体构建
刘声传,鄢东海,周 雪,曹 雨,莫 雪,胡伊然,周玉锋
(贵州省农业科学院茶叶研究所,贵州 贵阳 550006)
茶树CsSMT基因的克隆、原核表达及植物超表达载体构建
刘声传,鄢东海,周 雪,曹 雨,莫 雪,胡伊然,周玉锋*
(贵州省农业科学院茶叶研究所,贵州 贵阳 550006)
为了发掘利用茶树(Camelliasinensis)富硒关键酶硒代半胱氨酸甲基转移酶(Selenocysteine methyltransferase, SMT)基因CsSMT,基于宁州2号转录组测序结果,通过RACE克隆该基因的cDNA全长,并通过原核表达和蛋白质印迹(Western blotting)验证该基因,然后构建超表达载体,转化至根癌农杆菌。结果表明:该基因cDNA全长为1277 bp,开放阅读框(Open reading frame,ORF)为1 050 bp,与NCBI公布的茶树该基因ORF序列有8个点突变,且缺少GTCGTC序列。CsSMT基因编码349个氨基酸,其氨基酸序列与毛果杨、野生大豆、黄芪的SMT以及川桑的同型半胱氨酸-S-甲基转移酶2(Homocysteine-S-methyltransferase 2, HMT2)的氨基酸序列有较高的相似性。目标蛋白分子量约38 kDa,为水溶性蛋白,与预测结果相符。成功构建CsSMT基因的超表达载体,并获得携带CsSMT的农杆菌菌株。
茶树;硒;CsSMT;原核表达;超表达
硒是人体必需的微量元素,对人体具有抗氧化、抗克山病、抗节骨病、抗心脏病、抗甲状腺机能减退及抗癌等保健功能[1]。虽未证明硒是植物的必须营养元素,但有研究报道[2-3]植物适量吸收累积硒可促进其生长、抗氧化胁迫和抗病虫害等。据地质学家考证[4]:我国72 %的地区属于缺硒地区,2/3的人口存在不同程度的硒摄入量不足。一些植物可吸收有毒的无机硒,转换成对人体安全保健的天然有机硒。茶为当今世界上最受欢迎的一种天然健康饮料,茶树(Camelliasinensis)叶片具有富集硒的功能,可为人体提供硒源[5]。但高浓度的硒对动植物有毒害作用,故很有必要研究茶树等农作物的硒代谢,挖掘关键酶基因,培育或改良富硒作物。
甲基硒代半胱氨酸(Methylselenocysteine,MeSeCys)是一种氨基酸衍生物,具抗癌功能。花菜(Brassicaoleracea)、葱属(Allium)、黄芪(Astragalusbisulcatus)和茶树等一些植物可合成这种非蛋白质硒氨基酸,减少硒对蛋白质的掺入,解除硒对这些植物的毒害[6-8]。硒代半胱氨酸甲基转移酶(Selenocysteine methyltransferase, SMT)是植物硒代谢途径中的一个关键酶,催化硒代半胱氨酸进行甲基化反应生成MeSeCys。最先从黄芪中克隆到AbSMT基因[9],此后在花菜[10]、茶树[11]等作物中相继克隆了该基因。AbSMT转拟南芥(Arabidopsisthaliana)[6]和烟草(Nicotianatabacum)[12]使宿主MeSeCy含量显著增加,表明利用该基因,通过遗传工程可在相关作物中合成MeSeCys,以及减少硒的毒害。2008年已从茶树中克隆了CsSMT,但序列的准确性及品种间的差异性有待验证,该基因体外表达研究尚少。转录组测序技术的快速发展,为高效发掘克隆重要基因带来新途径。为此,本研究基于转录组测序,结合RT-PCR与RACE克隆技术,从茶树叶片中克隆到该基因的cDNA全长,进行生物信息学分析,原核表达及蛋白质印迹(Western blotting)验证,构建植物超表达载体,为进一步研究其酶学特性和调控茶树硒代谢的机理奠定基础。
1 材料与方法
1.1 供试材料
供试品种:宁州2号(NZ2),采自国家种质杭州茶树圃。
菌株、载体及试剂:Qiagen RNeasy Plant Mini Kitaccording试剂盒购自Qiagen公司。TaKaRaTaqTM酶、TaKaRa LATaq酶、rTaq酶、PrimeSTAR@HS (Premix)、dATP、E.coliDH5α 感受态细胞、E.coli感受态细胞JM109、感受态细胞BL21T1R、PrimeScriptII 1st Strand cDNA Synthesis Kit、T-Vector pMD20、DNA Ligation Kit、SMARTTMRACE cDNA Amplification Kit试剂盒、原核表达载体pCold TF、根癌农杆菌(Agrobacteriumtumefaciens) LBA4404 Electro-Cells、植物超表达载体pRI101-AN、NdeI酶和BamH I酶均购自TaKaRa公司。AxyPrep DNA凝胶回收试剂盒购自Axygen公司。
1.2 目的基因cDNA的全长克隆
基于笔者前期茶树耐旱机理研究的NZ2的转录组测序结果(http://www.ncbi.nlm.nih.gov/bioproject/297732)。经BLASTx比对,将获得缺5'端的CsSMT基因EST片段。设计5′ 扩增引物,进行5′ RACE PCR扩增,获得缺失片段,利用DNAstar软件,拼接获得全长序列。引物设计均使用Primer Premier 5.0软件,设计反向嵌套引物CsSMT5′端第1轮扩增引物 TGCAGCAACCTCCGACAAGGGAC和CsSMT5′端第2轮扩增引物GCAATGGCAGCGCATTCAAGCAA。引物合成、测序均由上海华津生物有限公司完成。
采用Qiagen试剂盒(Qiagen,德国)提取茶叶总RNA,采用RACE试剂盒扩增5′,AxyPrep DNA试剂盒回收目的片段,连接到pMD18-T载体,转化,菌落PCR鉴定,测序[13]。采用NCBI的ORF Finder寻找目的基因ORF序列,以此设计引物扩增ORF,CsSMT全长扩增引物:正向引物GGGTGTTATGTTCTTGGTG和反向引物TGTCAGTTAATCAGTGGGAT,利用PrimeScriptII 1st Strand cDNA Synthesis Kit合成cDNA第1链,连接至T-Vector pMD20,转化E.coliDH5α感受态细胞,菌落PCR鉴定、测序方法同RACE 5′扩增。
1.3 目的基因生物信息学分析
BLASTx比较分析目的基因大小、序列方向、同源性,采用NCBI的ORF Finder程序分析目的基因的开放阅读框(Open reading frame, ORF),用DNAstar查阅ORF并翻译成氨基酸序列,编码产物的理化性质采用Protparam分析,ProtScale程序预测疏水性/亲水性,采用TMHMM2.0程序预测跨膜结构域、SignalP4.1程序分析信号肽及TargetP1.1程序分析亚细胞定位,蛋白磷酸化位点采用NetPhos2.0程序分析,用ClustalX软件对目的基因编码的氨基酸序列与其他物种进行同源比对,利用Mega5.05软件对目的基因的氨基酸与其他物种的氨基酸序列进行系统发育分析。序列分析所用各种工具网址见表1。

表1 对目的基因进行生物信息学分析所用的主要工具
1.4 CsSMT基因原核表达
将带有目的基因的重组质粒T-Vector pMD20经NdeI和BamH I双酶切,同时,pCold TF经NdeI和BamH I双酶切。然后,利用DNA Ligation Kit将CsSMT基因和pCold TF片段在16 ℃下过夜连接,热转化至E.coli感受态细胞JM109中,涂布平板,37 ℃过夜培养,菌落PCR鉴定,测序。
选择经测序验证正确的重组pCold TF表达载体1.0 μl转入感受态细胞BL21T1R中。取35 μl转化液涂布于LB (Amp100 μg/mL)平板,37 ℃过夜培养。分别挑取单菌落至2 mL LB (Amp 100 μg/mL)液体培养基中,37 ℃过夜培养。取培养菌液100 μl,添加至含5 mL LB (Amp 100 μg/mL)培养基的玻璃管中,37 ℃培养至OD600 nm值约为0.6,15 ℃静置15 min,添加100 mmol/L IPTG 50 μl进行诱导,15 ℃继续培养22 h。取集菌后2 OD相当的菌体加入320 μl PBS悬浊后进行超声波破碎,对菌体破碎液进行离心分离(12 000 r/min,5min)。取各抽提液(全蛋白、上清、沉淀)8 μl(浓度相当于0.05 OD),加入2 μl 5×SDS Loading Buffer,95 ℃加热10 min,以12.5 % polyacrylamide gel进行SDS-PAGE 电泳。电泳结束后,CBB-R250染色,使用脱色液脱色。
1.5 CsSMT基因原核表达Western blotting验证
Western blotting样品电泳对照:取各抽提液(全蛋白、上清、沉淀)8 μl(浓度相当于0.05 OD),加入2 μl 5×SDS Loading Buffer,95 ℃加热10 min,使用彩色Marker及组氨酸标签Marker,12.5 %聚丙烯酰胺凝胶进行SDS-PAGE电泳。
Western Blotting:将PVDF膜、滤纸分别剪切成与凝胶相同大小,使用转膜缓冲液处理后,按滤纸、PVDF膜、凝胶和滤纸的顺序依次放在转膜仪电极板之间,开始转膜。转膜后,将PVDF膜置于含1.5 % BSA的10 mL Blocking buffer中,4 ℃平放过夜封闭。使用稀释后的Penta-His Antibody溶液5 mL,进行1 h抗体反应1 次。TBST 缓冲液(20 mL)洗涤2
次,TBS缓冲液冲洗3次。使用稀释后的HRP-Rabbit Anti-Mouse IgG 抗体溶液5 mL,进行1 h抗体反应2次。TBST 缓冲液(20 mL)洗涤2次,TBS 缓冲液冲洗3次。1 mL TrueBlue Peroxidase Substrate显色1 min。
1.6 植物超表达载体的构建及农杆菌转化
将带有目的基因的重组质粒T-Vector pMD20经NdeI和BamH I双酶切,同时,pRI101-AN经NdeI和BamH I双酶切。然后,利用DNA Ligation Kit将CsSMT基因和pRI101-AN片段在16 ℃下过夜连接,得到重组质粒pRI101-AN。转化感受态细胞E.coliDH5α,菌落PCR鉴定,测序。
农杆菌转化:筛选阳性重组质粒pRI101-AN,根癌农杆菌LBA4404 Electro-Cells,涂布LB平板(含Kan和Str),30 ℃培养48 h。挑单克隆菌落转LB液体培养基培养,菌液PCR验证。
2 结果与分析
2.1 目的基因cDNA全长克隆
经BLASTx比对,获得了缺5′端的、长度为720 bp的CsSMT基因EST片段。以EST序列为模板进行5′ RACE扩增,获得缺失片段约500 bp(图1 A)。将克隆得到的5′端序列与原有EST序列拼接,初步获得目的基因全长1277 bp。通过对ORF的cDNA克隆测序,获得ORF扩增后全长1114 bp(图1 B)。进一步BLASTx比对,最终得到目的基因ORF全长1 050 bp,其编码349个氨基酸,且与拼接结果相同。
NZ2的CsSMT的cDNA序列与NCBI上公布的祁门种的CsSMT序列(DQ480337.1)比对结果显示,2个品种CsSMT的相似性为98.4 %,在起始密码子32 bp后,NZ2的CsSMT缺少GTCGTC。此外,在ORF内有8个单碱基变异,在ORF外也存在点突变(图2)。

A.CsSMT的5′RACE扩增产物;B.CsSMT的开放阅读框扩增产物A.amplification products of 5′RACE for CsSMT; B.amplification products of ORF of CsSMT 图1 CsSMT目的基因扩增结果Fig.1 RACE-amplification of target gene of CsSMT

图2 2个茶树品种的CsSMT 的cDNA序列比对Fig.2 The cDNA sequence alignment of CsSMT in two tea cultivars
2.2 目的基因编码产物的理化性质
CsSMT基因编码产物分子量为37 937.8 Da,理论等电点为5.00,分子式为C1661H2637N457O528S15。不稳定指数为37.54(不稳定指数>40时不稳定),表明此蛋白稳定。脂肪指数为91.46,总平均亲水性为-0.139,表明该蛋白为亲水性蛋白。此外,经ProtScale程序分析,氨基酸序列多数位于亲水区域,该蛋白为亲水蛋白(图3A)。经TMHMM2.0分析,编码产物均位于细胞膜表面,属非跨膜蛋白(图3B)。TargetP1.1分析表明,该蛋白没有定位于叶绿体和线粒体。通过软件SignalP4.1信号肽分析发现,该蛋白的信号肽分值(S值)均值<0.5(S信号肽平均值>0.5表示存在信号肽,为分泌蛋白),表明该蛋白无信号肽(图3C)。利用NetPhos 2.0分析表明,该蛋白存在丝氨酸磷酸化(pSer)、苏氨酸磷酸化(pThr)和酪氨酸磷酸化(pTyr)3种主要的氨基酸磷酸化位点,酸化位点共24个(pSer: 15,pThr: 4,pTyr: 5),以丝氨酸磷酸化为主(图3D)。
2.3 氨基酸多序列的相似性和同源性
利用BLASTp将目的基因编码产物氨基酸序列与其他植物比对,结果显示CsSMT氨基酸序列与以下植物同源性较高:毛果杨(Populustrichocarpa)SMT(84.8 %)、野生大豆SMT (Glycinesoja)(78.7 %)、黄芪SMT(76.3 %)、花菜SMT(72.4 %)、川桑(Morusnotabilis)(Homocysteine S-methyltransferase 2, HMT2)(80.3 %)、拟南芥(Arabidopsisthaliana)HMT2(74.0 %)、毛果杨HMT3(69.0 %)、蒺藜苜蓿(Medicagotruncatula)HMT(67.2 %)和树棉花(Gossypiumarboreum)HMT3(64.8 %)。经氨基酸序列对比可知,不同物种SMT和HMT的两末端以及约在114 aa至129 aa之间多态性较高,其他位置存在多个保守域(图4)。

A.亲水性/疏水性;B.跨膜结构域;C.跨信号肽;D.磷酸化位点A. hydrophilicity or hydrophobicity; B. transmembrane region; C. signal peptide; D. phosphorylation图3 目的基因编码产物理化性质的预测结果Fig.3 Potential physicochemical characteristics of the target gene encoding protein

图4 茶树CsSMT及其直系同源基因编码氨基酸序列的比对Fig.4 Protein sequence alignment of CsSMT and orthologous genes

图5 茶树CsSMT与其他植物同源蛋白的系统进化Fig.5 Phylogenetic relationship of CsSMT in tea plant with the homologous proteins from other plants
经氨基酸序列的同源性比对与系统进化分析表明,茶树SMT与毛果杨、野生大豆、黄芪的SMT以及川桑的HMT2亲缘关系较近,但与花菜SMT及其他植物的HMTs亲缘关系较远(图5)。
2.4 目的蛋白表达及Western blotting验证
SDS-PAGE显示pCold TF质粒载体约在52 kDa有1条明显的外源蛋白条带,重组质粒pCold TF在90 kDa左右有1条明显的外源蛋白条带(图6 AB)。除去52 kDa的融合表达标签,可得出实际诱导的茶树SMT蛋白约为38 kDa,与理论值基本一致,且只有少量融合蛋白以包涵体的形式存在。Western blotting验证表明,带有目的基因的重组质粒pCold TF在低温表达系统中有表达,目的蛋白约38 kDa,基本为可溶性表达(图6 C)。
2.5 超表达载体的构建及其农杆菌转化
利用NdeI和BamH I双酶切T-Vector pMD20和pRI101-AN质粒,将CsSMT基因连接于含有CaMV35S强启动子的pRI101-AN超表达载体上(图7A)。筛选阳性重组质粒pRI101-AN,转化根癌农杆菌,PCR检测超表达载体构建结果(图7B)显示,约在2200 bp有1条明显的条带,除去约1100 bp的融合表达标签序列,可得出实际表达的目的基因约为1100 bp,与克隆结果一致。说明,超表达载体的构建及其农杆菌转化成功,可用于下一步的转基因研究。
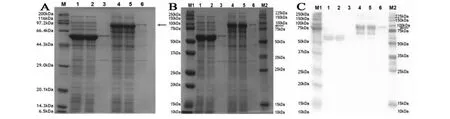
A为SDS-PAGE分析,B为Western blotting 样品电泳对照,C为PVDF膜图。1为pCold TF全细胞,2为pCold TF上清,3为pCold TF沉淀,4为CsSMT融合蛋白全细胞,5为CsSMT融合蛋白上清,6为CsSMT融合蛋白沉淀,M为markerA.SDS-PAGE analysis; B.Electrophoresis of Western blotting samples for control; C. PVDF film; 1.Whole cell with pCold TF; 2. pCold TF supernatant; 3.pCold TF precipitate; 4.Whole cell with CsSMT fusion proteins; 5.Supernatant of CsSMT fusion proteins; 6.Precipitate of CsSMT fusion proteins; M. Marker图6 CsSMT融合蛋白的SDS-PAGE及Western blotting分析Fig.6 Analysis of expressed CsSMT fusion proteins through SDS-PAGE and Western blotting

A.构建的超表达载体;B.农杆菌转化的PCR检测结果;M:markerA.Construction of overexpress vector; B. PCR examination result of its Agrobacterium transformation; M.Marker图7 CsSMT基因超表达载体构建示意图与农杆菌转化PCR检测结果Fig.7 Sketch map of overexpressed CsSMT plasmids and PCR examination result of its Agrobacterium transformation
3 结论与讨论
转录组测序及生物信息学的快速发展,为茶树基因克隆及功能分析等提供了有利条件。本研究从茶树中克隆了CsSMT基因的cDNA全长序列,分析该基因的生物信息学,并通过原核表达验证该基因,为后续酶学研究和遗传性状改良奠定了基础;成功构建CsSMT基因的超表达载体及其农杆菌转化菌株,可用于后续茶树等其他作物转基因研究。Brummell[14]通过转SMT基因已获得可富集MeSMT的西红柿 (Solanumlycopersicum)。
本研究克隆的CsSMT的ORF全长与NCBI公布的该基因有8个单碱基变异,在起始密码子32 bp后,NZ2的ScSMT缺少GTCGTC。这些突变位点可能可用于富硒茶树品种选育的分子标记。如魏艳丽[15]研究发现,茶树咖啡碱合成酶(Tea caffeine synthase,TCS)基因TCS1的1个点突变与咖啡碱含量显著相关。CsSMT基因中是否存在硒含量相关的功能突变位点,以及如何调控CsSMT的表达还需要进一步验证。
经生物信息学分析,CsSMT编码产物结构稳定,为亲水蛋白,不具跨膜结构和信号肽,不定位在叶绿体和线粒体,分子量约为38 kDa。原核表达及Western blotting验证表明,该蛋白为亲水蛋白,分子量约为38 kDa。Sors[16]等认为,SMT在拟南芥的胞液中发挥催化功能、定位于胞液中、不具跨膜结构,本研究结果与其一致。
本研究还发现,茶树SMT其他植物HMT的氨基酸序列同源性显著,SMT可能与HMT具有相似的结构和功能,其编码基因可能起源于同一基因。Zhao等[17]通过比较拟南芥、水稻(Oryzasativa)和玉米(Zeamays)等7种单子叶植物这2个基因的序列发现,SMT与HMT2具有很高的同源性,认为SMT和HMT起源于同一基因。已有研究表明,SMT和HMT催化甲基转移时以硫甲基蛋氨酸(S-methylmethionine)为甲基转移供体,但体外试验证明,SMT和HMT分别选择含硒(Se)域和含硫(S)域的底物为甲基受体[18]。一定程度证明SMT富集硒的专一性。茶树等木本植物中有关SMT是否具有富集硫的研究尚未见报道。植物硒代谢过程复杂,涉及一系列基因[19]。本研究只对茶树富硒关键酶基因SMT进行了初步分析与超表达载体构建,而一些研究已证明ATP硫化酶(ATP sulfurylase,APS)和 胱硫醚-g-合成酶 (Cystathionine-g-synthase,CgS)也是一些植物硒富集的关键酶。如APS×SMT双转基因植物富集的MeSMT含量是对照的8倍,是SMT转基因植物的2倍[20]。超表达CgS的芥菜(Brassicajuncea)富集的硒约为对照的40 %[21]。因此,还需挖掘植物硒富集的候选功能基因,理解植物硒的代谢调控机理,培育超富集硒植物用于硒污染修复,培育硒的累积量对作物、人体安全的优良富硒农作物。
[1]Ellis D R, Salt D E. Plants,selenium and human health[J]. Current Opinion in Plant Biology, 2003, 6(3):273-279.
[2]Pilon-Smits E A H, Hwang S, Lytle C M, et al. Overexpression of ATP sulfurylase in Indian mustard leads to increased selenate uptake, reduction, and tolerance[J]. Plant Physiology, 1999, 119: 123-132.
[3]Lyons G H, Genc Y, Soole K, et al. Selenium increases seed production inBrassica[J]. Plant Soil, 2009, 318: 73-80.
[4]刘声传,鄢东海,魏 杰. 茶树硒代半胱氨酸甲基转移酶基因生物信息学分析[J]. 西南农业学报, 2013, 26(6): 2221-2226.
[5]于 振, 李建科, 黄瑞蕊, 等. 陕西汉阴茶园土壤硒含量及茶叶硒富集能力[J]. 食品科学技术学报, 2013, 31(4): 15-18.
[6]Ellis D R, Sors T G, Brunk D G, et al. Production of Se-methylselenocysteine in transgenic plants expressing selenocysteine methyltransferase[J]. BMC Plant Biology, 2004, 4(1): 11-22.
[7]Pilon-Smits E A H, Quinn C F. Selenium metabolism in plants[M]//Cell biology of metals and nutrients. Springer Berlin Heidelberg, 2010: 225-241.
[8]Cakir Ö, Turgut-Kara N, Ari N. Selenium metabolism in plants: molecular approaches[J]. Advances in Selected Plant Physiology Aspects, Intech, Crotia, 2012: 209-232.
[9]Neuhierl B, Thanbichler M, Lottspeich F, et al. A family of Smethylmethionine dependent thiol/selenol methyltransferases: Role in selenium tolerance and evolutionary relation[J]. Journal of Biological Chemistry, 1999, 274(9): 5407-5414.
[10]Lyi S M, Heller L I, Rutzke M, et al. Molecular and biochemical characterization of the selenocysteine Se-methyltransferase gene and Se-methylselenocysteine synthesis in broccoli[J]. Plant Physiology, 2005, 138(1): 409-420.
[11]Zhu L, Jiang C J, Deng W W, et al. Cloning and expression of selenocysteine methyltransferase cDNA fromCamelliasinensis[J]. Acta Physiologiae Plantarum, 2008, 30(2): 167-174.
[12]McKenzie M J, Hunter D A, Pathirana R, et al. Accumulation of an organic anticancer selenium compound in a transgenicSolanaceousspecies shows wider applicability of the selenocysteine methyltransferase transgene from selenium hyperaccumulators[J]. Transgenic Research, 2009, 18(3): 407-424.
[13]刘声传. 茶树对干旱胁迫和复水响应的生理、分子机理[D]. 北京: 中国农业科学院, 2015:47-53.
[14]Brummell D A, Watson L M, Pathirana R, et al. Biofortification of tomato (Solanumlycopersicum) fruit with the anticancer compound methylselenocysteine using a selenocysteine methyltransferase from a selenium hyperaccumulator[J]. Journal of Agricultural and Food Chemistry, 2011,59(20): 10987-10994.
[15]魏艳丽. 茶树AMP脱氨酶基因的克隆及咖啡碱合成酶基因(TCSl)的cSNP分析[D]. 合肥: 安徽农业大学, 2013:29-35.
[16]Sors T G, Ellis D R, Salt D E. Selenium uptake, translocation, assimilation and metabolic fate in plants[J]. Photosynthesis Research, 2005, 86(3): 373-389.
[17]Zhao D Y, Sun F L, Zhang B, et al. Systematic comparisons of orthologous selenocysteine methyltransferase and homocysteine methyltransferase genes from seven monocots species[J]. Notulae Scientia Biologicae, 2015, 7(2): 210-216.
[18]Ranocha P, Bourgis F, Ziemak M J, et al. Characterization and functional expression of cDNAs encoding methionine-sensitive and-insensitive homocysteine S-methyltransferases fromArabidopsis[J]. Journal of Biological Chemistry, 2000, 275(21):15962-15968.
[19]Lin J, Peng T, Jiang L, et al. Comparative genomics reveals new candidate genes involved in selenium metabolism in prokaryotes[J]. Genome biology and Evolution, 2015, 7(3): 664-676.
[20]LeDuc D L, AbdelSamie M, Montes-Bayo′n M, et al. Overexpressing both ATP sulfurylase and selenocysteine methyltransferase enhances selenium phytoremediation traits in Indian mustard[J]. Environmental Pollution, 2006, 144(1): 70-76.
[21]Huysen T, Terry N, Pilon-Smits E A H. Exploring the Selenium phytoremediationpotential of transgenic Brassica juncea overexpressing ATP sulfurylase or cystathionine-g-synthase[J]. International Journal of Phytoremediation, 2004, 6(2):111-118.
(责任编辑 刘忠丽)
Cloning and Prokaryotic Expression of Selenocysteine Methyltransferase Gene from Tea Plant and Its Plant Overexpression Vector Construction
LIU Sheng-chuan, YAN Dong-hai, ZHOU Xue, CAO Yu, MO Xue, HU Yi-ran, ZHOU Yu-feng*
(Guizhou Tea Research Institute, Guizhou Academy of Agricultural Sciences, Guizhou Guiyang 550006, China)
To explore the selenocysteine methyltransferase (SMT) geneCsSMT, which plays an essential role inCamelliasinensisleaves enriching selenium, based on transcriptome sequencing results ofCamelliasinensiscv. ‘Ningzhou 2’, the whole cDNA was obtained by RACE cloning. The target gene was identified by prokaryotic expression and western blotting, integrated into overexpression vector, and then transformed toAgrobacteriumtumefaciens. The results showed that the whole cDNA contained an open reading frame (ORF) of 1050 bp. In comparison toCsSMTORF published in NCBI, it had eight point mutations and missed GTCGTC. In the present study,CsSMTencoded 349 amino acids, and shared high similarity with SMT fromPopulustrichocarpa,Glycinesoja, andAstragalusbisulcatusas well as homocysteine-S-methyltransferase 2 (HMT2) inMorusnotabilis. CsSMT was a water-soluble protein and its molecular weight was approximately 38 kDa, which was consistent with the putative results. The authors successfully constructed the overexpression vector ofCsSMTand gained a strain ofA.tumefacienswith the target gene.
Tea plant; Selenium;CsSMT; Prokaryotic expression; Overexpression
1001-4829(2016)07-1540-07
10.16213/j.cnki.scjas.2016.07.008
2015-12-07
贵州省农业科学院研究生科研创新基金项目“茶树富硒作用关键酶基因CsSMT的克隆与超表达研究”[黔农科合(创新基金)2011006];贵州省体改项目“现代高效农业园区茶叶栽培与加工技术集成示范”[黔科合Z字[2013]4008];贵州省农业攻关项目“贵定鸟王茶种质资源的保护及开发利用”[黔科合NY字[2011]3046];贵州省农业动植物育种专项“黔茶1号区域适应性高效栽培研究与示范”[黔农育专字(2012)022号],“贵定鸟王种扩繁和栽培技术集成与示范”[黔农育专字(2015)003]
刘声传(1981-),男,副研究员,博士,从事茶树资源与育种研究,E-mail: gtscliu@sohu.com, *为通讯作者。
S571.1
A

