福建柏遗传多样性ISSR分析
李单琦,谢德金,王汉琪,周少卿,荣俊冬,何天友,郑郁善
(福建农林大学 a.艺术园林学院;b.工业原料林研究所,福建 福州 350002)
福建柏遗传多样性ISSR分析
李单琦a,谢德金b,王汉琪b,周少卿b,荣俊冬b,何天友b,郑郁善b
(福建农林大学 a.艺术园林学院;b.工业原料林研究所,福建 福州 350002)
采用ISSR分子标记技术,对我国濒危植物福建柏的7个种群进行遗传多样性分析。结果显示:17个引物共扩增出202个位点,多态性位点167个,占总位点的82.67%;引物UBC849可以将24份福建柏样本完全鉴别出来;7个福建柏种群的多态位点百分率在17.82%~54.95%之间;群体总观测等位基因数Na为1.821 8,有效等位基因数Ne为1.431 3,Nei指数为0.258 2,Shannon指数为0.394 0;种群内平均Na指数为1.372 0,平均Ne指数为1.289 2,平均Nei指数为0.160 0,平均Shannon指数为0.203 5;基因分化系数Gst为32.41%,种群间基因流Nm为0.878 0;7个种群遗传相似度在0.668 3~0.806 9之间,遗传距离在0.214 5~0.404 3之间;利用UPGMA法进行聚类,将泉州市、福州市和龙岩市种群分为第一类,将南平市、三明市和漳州市种群分为第二类,将宁德市种群分为第三类。
福建柏;ISSR;遗传多样性;聚类分析
福建柏Fokienia hodginsii 又称建柏,为裸子植物门柏科纲福建柏属的单一种植物[1]。从福建柏叶片中提取出的精油,具有杀灭蚊虫作用[2]。福建柏是一种古老的孑遗植物,也是我国珍贵的濒危物种,被列为国家Ⅱ级保护树种,为福建省乡土树种[3]。现阶段福建柏研究主要集中在群落研究、造林研究、育苗研究、种源研究等方面,对其遗传多样性仅从过氧化物同工酶角度进行粗略的分析[4],为了研究福建柏致濒机理以及提出切实可行的保护策略,有必要从DNA分子水平上对其遗传多样性进行深入研究。
ISSR DNA分子标记技术(简单重复序列inter simple sequence repeat)[5]最早于1994年由加拿大蒙特利尔大学的Zietkiewicz教授等人提出[6]。这种标记根据广泛存在于植物中的微卫星序列(SSR)[7],在其3′或5′端加上2~4个非重复随机的核苷酸序列作为PCR反应引物[8],引起特定位点退火并对间隔不大的重复序列间DNA片段进行PCR扩增[6],再根据谱带类型对各样品的遗传多样性等进行分析[9]。ISSR标记以其引物设计简单、产物具有较好的稳定性与多态性[10]、重复性好[11]、DNA用量较小、安全性高[12]、技术难度和复杂度较低、耗时少、成本低等特点已广泛应用于我国特有濒危植物的研究[13-16]。本实验对福建省7个福建柏种群的24个样本进行ISSR分子标记研究,旨在揭示福建柏遗传多样性水平,研究其致濒原因,为福建柏种质资源保护提供科学依据。
1 材料与方法
1.1 实验材料
本实验所采用的福建柏样品于2013年3月至4月在福建省内各福建柏天然林、人工林和古树进行采集。7个福建柏种群信息详见表1。

表1 福建柏种群信息Table 1 The population information of Fokienia hodginsii
1.2 实验方法
1.2.1 DNA提取与检测
福建柏基因组DNA采用天根试剂盒提取,并在胡迪科等人[17]的基础上加以改进;提取产物分别经1.5%的琼脂糖凝胶电泳和NANODROP 2000 C分光光度计分析质量和浓度,于-20℃保存。
1.2.2 扩增体系
对影响ISSR-PCR反应体系的各因素进行梯度实验筛选,最终确定福建柏ISSR-PCR反应体系为:总体积为20 mL,其中10´PCR buffer 2 mL,Mg2+浓度 1.8 mmol·L-1,dNTPs 浓度 0.25 mmol·L-1,Taq酶浓度0.25 U,引物浓度0.7 mmol·L-1,模板DNA用量50 ng,最后用灭菌蒸馏水补齐至20 mL[18]。扩增体系为:94 ℃预变性5 min,接着94 ℃变性30 s、48.6 ℃退火40 s、72 ℃延伸2 min,此3步进行40个循环,最后72 ℃延伸7 min,反应完毕后于4 ℃环境保存[19]。
1.2.3 数据处理
对24份福建柏样品DNA进行ISSR-PCR扩增,根据凝胶成像分析仪观察电泳图上是否有条带,将同一位点上有条带的记为1,无条带的记为0,对每一引物扩增结果建立二元数据矩阵,统计总位点数和多态性位点数并构建指纹图谱;运用POPGENE 32基因软件对此0、1矩阵进行分析,分析指标主要有:多态位点百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Shannon多样性指数、基因多样性指数(I)、遗传分化系数(Gst)、种群总基因多样性(Ht)、种群内基因多样性(Hs)、基因流(Nm)、遗传距离与遗传相似度。根据种群间遗传距离,运用UPGMA法对福建柏各种群间亲缘关系进行聚类分析,构建树状聚类图。
2 结果与分析
2.1 引物筛选结果
对100条ISSR引物进行筛选,最终确定17条条带清晰、明亮度好、多态性丰富的引物进行福建柏ISSR研究,同时对每一引物筛选最佳退火温度。筛选出的17条引物占所有引物的17%,对供试的24份福建柏材料共扩增出202个重复性好、清晰稳定的位点,其中多态性位点167个,占总位点的82.67%;各个引物扩增出的位点在9~16个之间,平均11.88个位点;多态性位点在5~14个之间,平均9.82个;各个引物检测出的DNA片段大小范围在200~4 500 bp之间,说明福建柏有较长的基因组;各引物扩增结果的多态位点百分数在50.00%~100%之间,平均为82.09%。结果显示,17条ISSR引物均能扩增出高多态性的位点,由此显示了福建柏具有非常丰富的多态性(详细数据见表2)。
2.2 指纹图谱的构建
种质资源鉴别是ISSR分子标记技术的重要作用之一,根据引物扩增结果显示的特殊条带,可以快速有效地进行种质资源鉴别,因此筛选出最少的引物以获得最大的鉴别能力,对于种质资源鉴别尤为重要。通过对17个引物扩增结果进行仔细筛选比较后发现,引物UBC849可以将供试的24份福建柏样本完全鉴别出来,表现出较好的鉴别能力,扩增结果见图1。将扩增结果进行数字转化(有条带记为1,无条带记为0),形成特有的分子身份证编码,为福建柏构建一个基于ISSR分析的DNA指纹图谱(见表3)。
2.3 福建柏遗传多样性分析
多态位点百分率(PPB)指标对于衡量植物种
群间遗传变异水平的高低非常重要,若一个植物种群的PPB值高则说明该种群对于当前环境有较强的适应能力,可以继续繁衍下去;若PPB值低则说明其环境适应能力较弱,有灭绝的可能性,需要及时对其采取适当的保护措施。由表4可知,7个福建柏种群多态位点百分率有较大差异,PPB值在17.82%~54.95%之间,平均为37.20%。其中多态位点百分率最高的为泉州市种群,其次为三明市种群,说明这两个种群福建柏适应性强,遗传多样性丰富,遗传基础较宽,是良种选育的最佳种源地;多态位点百分率最低的为漳州市种群,其次为南平市种群,说明这两个种群的福建柏适应环境能力较差,需要加强保护,否则会导致灭绝。

表2 引物检测结果Table 2 Test results of primers

图1 引物UBC849扩增结果Fig.1 The ampli fi cation results by primer UBC849
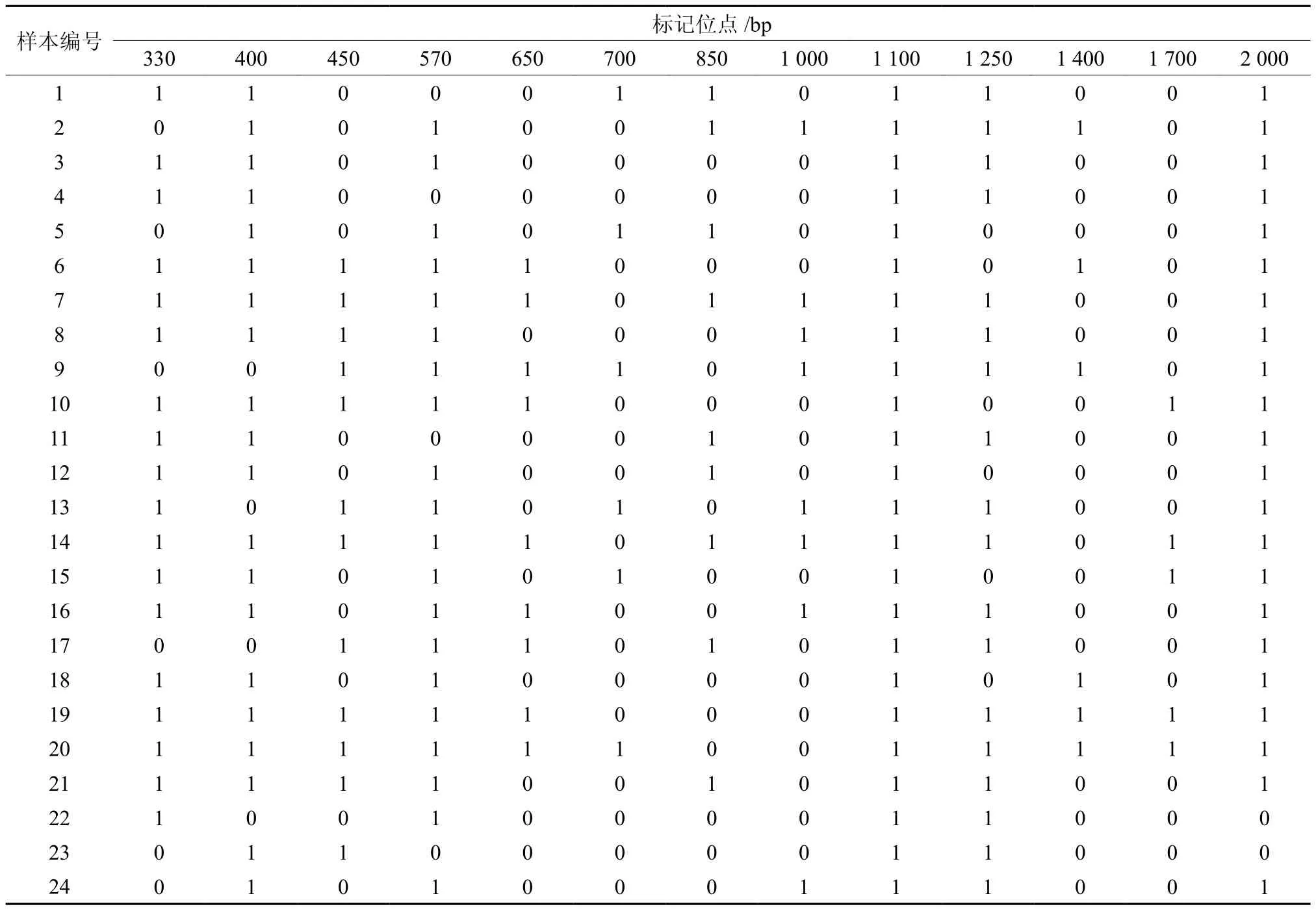
表3 24份福建柏样本ISSR身份证编码Table 3 The ISSR ID code of 24 samples of Fokienia hodginsii
利用POPGENE 32软件分别对7个福建柏种群的遗传多样性进行总体和单独分析,结果显示:7个种群总体观测等位基因数为1.821 8,有效等位基因数为1.431 3,Nei遗传多样性指数为0.258 2,Shannon信息指数为0.394 0,显示了7个福建柏种群间丰富的遗传多样性;7个群体的总遗传多样性为0.258 2,种群内遗传多样性为0.174 5,种群间分化水平为0.324 1,表明在总遗传多样性变异中,有32.41%的变异存在于种群间,有67.59%的变异存在于种群内;种群间基因流为0.878 0,表明各种群间存在较小的基因交流,有增加种群间遗传变异的可能性[20]。
由表4、图2可知,7个种群的Na指数在1.178 2~1.545 9之间,平均为1.372 0;Ne指数在1.787 2~1.356 3之间,平均为1.289 2;Nei指数在0.089 1~0.205 2之间,平均为0.160 0;Shannon指数在0.123 5~0.305 7之间,平均为0.203 5。种群间遗传多样性由大到小排列为泉州市>三明市>福州市>宁德市>龙岩市>南平市>漳州市。
2.4 种群间亲缘关系
由表5可知,7个种群遗传相似度在0.668 3~0.806 9之间,平均为0.765 5,遗传距离在0.214 5~0.404 3之间,平均为0.289 5;其中亲缘关系最近的是福州市与龙岩市种群,亲缘关系最远的是泉州市和宁德市种群。

图2 福建柏7种群遗传多样性比较Fig. 2 The comparison among 7 populations of F’s genetic diversity

表5 7个种群间遗传相似度(对角线上方)和遗传距离(对角线下方)Table 5 Genetic identity (above diagonal) and genetic distance (below diagonal) among 7 populations
采用UPGMA法构建基于遗传距离的聚类图(见图3),7个种群可以分为三类:第一类包括泉州市、福州市和龙岩市种群;第二类包括南平市、三明市和漳州市种群;第三类包括宁德市种群。

图3 7个福建柏种群的聚类树状图Fig. 3 Dendrogram of 7 populations of Fokienia hodginsii
3 讨 论
植物遗传多样性是其所携带遗传信息的总和,是植物种群生存、发展进化的基础。遗传多样性水平可以反映出一个植物群体在其生长环境中基因的丰富程度,也决定其对于环境变化的适应能力。因此对植物遗传多样性进行研究,可以清楚地了解到不同地区种群的适应性与生长发展趋势,对植物种质资源的良种选育工作以及濒危植物的致濒机理研究等具有重要意义。
多态位点百分率(PPB)指标是衡量植物种群对环境适应能力的重要指标之一,本研究对福建省的7个福建柏种群的PPB进行研究发现,各种群间有较大差异,表明不同地区环境因素对福建柏生长有较大影响,其中环境适应力最差的为漳州市和南平市种群,因此应及时对其进行加强保护,防止在自然选择过程中被淘汰。
本研究结果表明,福建柏种群间和种群内均具有较高的遗传多样性[21],7个种群总体观测等位基因数为1.821 8,有效等位基因数为1.431 3,Nei遗传多样性指数为0.258 2,Shannon信息指数为0.394 0;种群内观测等位基因指数平均为1.372 0,有效等位基因指数平均为1.289 2,Nei指数平均为0.160 0,Shannon指数平均为0.203 5[22]。由此表明,导致福建柏濒危并非由于遗传多样性的缺乏,而是受到其他因素影响。
一个植物种群的遗传结构在很大程度上决定了其进化潜力,而遗传结构主要通过种群间分化水平体现的,若一个植物种群间遗传分化系数较高,说明需要保护较多的种群,反之则仅需保护较少种群。福建省内的福建柏种群间分化水平为0.324 1,表明遗传变异主要存在于种群内,只需要对部分种群进行保护。
导致种群间出现遗传分化的主要原因是自然选择的作用,而基因流是对抗自然选择的重要因素。一般认为,当植物种群间基因流大于1时,可以防止近交衰退而保持较高的遗传多样性;若基因流小于1,则会引起种群间遗传分化[23]。福建柏7个种群的基因流为0.878 0,小于1,表明种群间存在一定的遗传分化。
综上所述,导致福建柏成为濒危植物的主要原因是人为对其生长环境的破坏,导致其居群规模不断减小,生境片段化,使种群间基因交流减少,增加了近交和遗传漂变作用。因此,需要及时采取相应保护措施,抑制福建柏种群的衰败趋势。首先,应该对现有的福建柏资源进行就地保护,建立保护区,禁止乱砍乱伐,保护其生长环境;其次,对福建柏生态学特性进行研究,制定科学的保护方法;第三,加强人工繁育力度,运用组织培养或扦插等方式进行大量培育;第四,加大种群间基因交流,将各种群采集的大量种子和幼苗进行交换种植,人为增加基因交流,以便更好地保持、提高福建柏遗传多样性水平。
[1]郑万钧.中国树木志:第一卷[M].北京:中国林业出版社,1983: 344-345.
[2]卢钰铎,彭映辉,扶巧梅,等.福建柏精油对蚊虫的生物活性[J].农药学学报,2014,16(3):293-299.
[3]国家林业局,农业部.国家重点保护野生植物名录(第一批)[R].北京:1999.
[4]肖 晖.不同产地福建柏过氧化物同工酶的变异类型研究[J].福建林业科技,2003,30(4):35-37.
[5]Litt M, Luty J A. A hypervariable microsatellite revealed by in vitro amplification of a dinucleotide repeat within the cardiac muscle actin gene [J]. American journal of human genetics, 1989,44(3): 397-401.
[6]Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction ampli fi cation[J]. Genomics,1994,20(2):176-183.
[7]秦 玥,刘梦培,傅大立,等.华仁杏SSR标记的筛选与评价[J].经济林研究,2013,31(3):72-76.
[8]宋晓兵,彭埃天,刘景梅,等.ISSR分子标记及其在中国南方果树上的应用[J].广东农业科学,2009,(7):199-201.
[9]王 绪,邓俭英,方锋学.ISSR分子标记技术及其在园艺作物中的应用[J].广西农业科学,2007,38(4):371:374.
[10]Fang D Q, Roose M L. Identification of closely related Citrus cultivars with inter-simple sequence repeat markers [J]. Theor.Appl. Genet., 1997, (95): 408-417.
[11]Blair M W, Panaud O,et al.Inter-simple sequence repeat (ISSR)amplification for analysis of microsatellite motif sequence and fingerprinting in rice (Oryza sativaL.) [J]. Theor. Appl.Genet.,1999,(98):780-792.
[12]张青林,罗正荣.ISSR及其在果树上的应用[J].果树学报,2004, 21(1): 54-58.
[13]Femandez M E, Figueiras A M, Benito C. The use of ISST and RAPD markers for detecting DNA polymorphism genotype identi fi cation and genetic diversity among barely cultivars with known origin [J]. Theor. Appl. Genet.,2002,104:845-851.
[14]何天友.短葶山麦冬的遗传多样性分析及ISSR指纹图谱构建[D].福建:福建农林大学,2011:4.
[15]廖文芳,夏念和,邓云飞,等.华木莲的遗传多样性研究[J].云南植物研究,2004,26(1):58-64.
[16]金则新,李钧敏.珍稀濒危植物夏蜡梅遗传多样性的ISSR分析[J].应用生态学报,2007,18(2):247-253.
[17]胡迪科.雷公藤遗传多样性的ISSR和SRAP分析[D].福建:福建农林大学,2011:18-19.
[18]王弦云,朱晓敏,王 勤,等.杜仲ISSR-PCR反应体系的建立与引物筛选及其在遗传多样性研究中的应用[J].经济林研究,2013,31(1):30-34.
[19]田 华,张党权,谭晓风,等.细叶桉ISSR-PCR体系的建立与优化[J].经济林研究,2009,27(1):13-16.
[20]陈小勇.植物的基因流及其在濒危植物保护中的作用[J].生物多样性,1996,4(2):97-98.
[21]张党权,田 华,谢耀坚, 等.桉树4个种遗传多样性的ISSR分析[J].中南林业科技大学学报,2010,30(1):12-17.
[22]王湘莹,何 钢,王晓明, 等.金银花不同品种间遗传变异的ISSR分析[J].中南林业科技大学学报,2013,33(11):77-82.
[23]Wright S. Evolution in Mendelian population [J]. Genetics, 1931,16: 97-159.
ISSR analysis on genetic diversity of Fokienia hodginsii
LI Dan-qia, XIE De-jinb, WANG Han-qib, ZHOU Shao-qinb, RONG Jun-dongb, HE Tian-youb, ZHENG Yu-shanb
(a.College of Arts & Landscape Architecture; b. Industrial Raw Material Forest Institute, Fujian Agriculture and Forestry University,Fuzhou 350002, Fujian, China )
This paper studied the genetic diversity of 7 populations of endangered plants Fokienia hodginsii by using ISSR markers technique. The results showed that 17 primers ampli fi cation 202 loci, 167 polymorphic loci, accounting for 82.67%. The 24 samples could be identi fi ed ef fi ciently by primer UBC849. The percentage of polymorphic loci was between 17.82% and 54.95%, at species level the observed number of alleles was 1.8218, the effective number of alleles was 1.4313, Nei’s genetic diversity was 0.2582, Shannon’s index was 0.3940, at population level they were 1.3720, 1.2892, 0.1600 and 0.2035. The gene differentiation coef fi cient was 32.41%and gene fl ow was 0.8780. The genetic identity was between 0.6683 and 0.8069, the genetic distance was between 0.2145 and 0.4043.Through cluster analysis by UPGMA, the 7 populations of Fokienia hodginsii were classi fi ed into 3 groups, the fi rst group including populations of Quanzhou city, Fuzhou city and Longyan city, the second group including populations of Nanpin city, Sanming city and Zhangzhou city, the last group was population of Ningde city.
Fokienia hodginsii; ISSR; genetic diversity; clustering analysis
S791.43
A
1673-923X(2016)05-0063-05
10.14067/j.cnki.1673-923x.2016.05.012
2015-02-02
国家科技支撑计划课题(2011BAI01B06)
李单琦,硕士研究生 通讯作者:郑郁善,教授;E-mail:zys1960@163.com
李单琦,谢德金,王汉琪,等. 福建柏遗传多样性ISSR分析[J].中南林业科技大学学报,2016, 36(5): 63-67, 78.
[本文编校:谢荣秀]

