靶向敲除绒山羊lnc15479的CRISPR/ Cas9构建及活性验证
冯帅帅,雒志新,闫 强, 徐 坤,王善禾,邵斯旻,张智英, 王 昕
(西北农林科技大学 动物科技学院,陕西杨凌 712100)
靶向敲除绒山羊lnc15479的CRISPR/ Cas9构建及活性验证
冯帅帅,雒志新,闫 强, 徐 坤,王善禾,邵斯旻,张智英, 王 昕
(西北农林科技大学 动物科技学院,陕西杨凌 712100)
为研究生长期和休止期lnc15479的差异表达在陕北白绒山羊毛囊周期性发育中的作用及功能,利用CRISPR/Cas9技术,在lnc15479的上、下游各设计1个sgRNA靶位点,构建CRISPR/cas9表达载体;并基于SSA修复机制,分别构建含有红色和绿色荧光标记的双荧光报告载体系统。通过将表达载体和报告载体共同转染 HEK293T细胞,检测该CRISPR/Cas9系统的工作效率。结果表明,lnc15479的CRISPR/Cas9表达载体成功构建,根据红色和绿色荧光表达情况及细胞计数的方法,该系统的工作效率约为20%。研究结果为进一步分析lnc15479的功能提供技术支持。
CRISPR/Cas9;绒山羊;lncRNA
羊绒由于其纤维具有轻、柔、暖等特点,被誉为“纤维宝石”和“软黄金”,在畜牧业生产中具有重要的经济价值。羊绒是由次级毛囊产生的,其生长是一个动态循环过程,包括生长期、退行期和休止期[1]。长链非编码RNAs(long noncoding RNA,lncRNAs)是一类本身不编码蛋白、转录本长度超过200 nt的非编码RNA,能够在表观遗传调控、转录调控及转录后调控等多种水平上调控基因的表达[2- 3]。前期对陕北白绒山羊羊绒生长期和休止期皮肤组织的RNA-seq结果分析表明,lnc15479在生长期和休止期显著差异表达,q-PCR结果表明该lncRNA在生长期的表达量比休止期高13.98倍(未发表数据),推测其可能对羊绒的周期性生长具有调控作用。
CRISPR/Cas9(Clustered regulatory interspaced short palindromic repeat/Cas9)是继ZFN、TALEN之后的第3代基因组定点编辑技术,来源于简单的细菌免疫系统,经过人为改造后,可在真核细胞中实现高度灵活且特异的基因组编辑。CRISPR/Cas9系统可通过直接合成、体外转录和载体表达等多种方式引入与靶序列碱基配对的特异性向导RNA(Single guide RNA,sgRNA)序列,从而引导Cas9蛋白结合到靶序列处,行使DNA切割功能,然后利用细胞的非同源性末端连接或同源重组修复机制对断裂的DNA进行插入、缺失、修复或替换[4],进而实现对多基因、多位点的打靶,甚至是基因组中大片段的删除。CRISPR/Cas9技术目前广泛应用于各种动物[5-8]、植物[9-11]、微生物[12]和病毒[13-14]的基因组编辑研究。
由于lncRNA不具有编码的外显子,短片段的缺失或插入可能不改变lncRNA的功能,因此,传统方法很难实现对lncRNA的敲除。CRISPR/Cas9 技术具有易操作、设计简单、能够特异地靶向目标序列,实现大片段的靶向敲除,本研究旨在利用CRISPR/Cas9基因编辑技术建立lncRNA的靶向敲除系统,为后续研究lnc15479在羊绒周期性生长调控中的作用机制提供技术平台。
1 材料与方法
1.1 材 料
1.1.1 细胞株和质粒 HEK293T细胞和DH5α感受态细胞均为陕西省农业分子生物学重点实验室保存;质粒PX330、pcDNA3.1、pPRIME-CMV-dsRed-recipent和p156RRL-EF1a-GFP-U3H1SatA购自Addgene公司。
1.1.2 材料和试剂 陕北白绒山羊皮肤组织样由榆林学院提供,限制性内切酶(BamHⅠ、EcoRⅠ、XhoⅠ、ApaⅠ、NotⅠ、XmaⅠ、NarⅠ、KpnⅠ)、T4DNA连接酶、TaqDNA聚合酶、buffer、dNTPs均购自NEB公司,蛋白酶K购自Calbiochem公司,氨苄青霉素、琼脂糖、Tris、SDS、EDTA购自上海生工生物工程有限公司,蛋白胨、酵母提取物购自Sigma公司,质粒提取试剂盒、胶回收试剂盒购自Promega公司,胶纯化试剂盒购自QIAGEN公司,Sofast转染试剂购自厦门太阳马生物工程有限公司。
1.1.3 引物合成和测序 所用引物序列见表1,引物合成及测序均由英潍捷基(上海)贸易有限公司完成。

表1 引物序列信息
1.2 方 法
利用在线软件(http://crispr.mit.edu)在绒山羊lnc15479的上、下游各设计1个sgRNA靶位点,分别记为Target1(ATTAGCCCTTAACCTCCCCC)和Target2(CAGGGGAGTCTGGTCAACCT),构建敲除lnc15479(1 083 nt)的CRISPR/Cas9系统,并在HEK293T细胞中验证其活性。
1.2.1 构建表达载体 msgRNA-2 设计含有sgRNA靶位点Target1的引物F1和R1,并且在引物两端引入BamHⅠ和EcoRⅠ的酶切位点。以pX330质粒载体为模板,分别以F1和R1为上、下游引物进行PCR,琼脂糖凝胶电泳后进行胶回收,得到含有双酶切位点BamHⅠ和EcoRⅠ的Target1.sgRNA序列;同样的方法以F2和R2为引物得到含有双酶切位点XhoⅠ和XbaⅠ的Target2.sgRNA序列。PCR体系:10×buffer 5 μL,dNTPs 4 μL,模板1 μL,DNA聚合酶0.5 μL,引物(F+R) 1 μL(10 μmol/L),补水至50 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,共36个循环;最后72 ℃延伸10 min,4 ℃保存。取PCR产物2 μL进行琼脂糖凝胶电泳检测。
以pcDNA3.1为母载体,分别进行双酶切(BamHⅠ/EcoRⅠ)、(EcoRⅠ/XhoⅠ)、(XhoⅠ/XbaⅠ)后,将上一步得到的目的片段与载体pcDNA3.1于4 ℃过夜连接,连接产物转化至DH5α感受态细胞,氨苄抗性平板筛选,挑取单克隆、摇菌、提取质粒。测序验证含有CD40.shRNA[15]、Target1.sgRNA和Target2.sgRNA序列的载体,命名为msgRNA-2(pcDNA3.1(+)-U6-Target1.sgRNA-CD40.shRNA-Target 2.sg RNA)(图1)。
1.2.2 构建双荧光报告载体 以pPRIME-CMV-dsRed-recipent为模板,F3和R3为引物进行PCR扩增,得到含双酶切位点XmaⅠ和NarⅠ的CMV-dsRed序列;以p156RRL-EF1a-GFP-U3H1SatA为模板,F4和R4为引物进行PCR扩增,胶回收得到含有酶切位点XbaⅠ的EF1a + 5′GFP,同样的方法以F5和R5为引物得到含有酶切位点KpnⅠ的3′GFP序列。PCR条件:10×buffer 5 μL,dNTPs 4 μL,模板1 μL,DNA聚合酶0.5 μL,引物(F+R)1 μL,补水至50 μL。扩增程序:94 ℃预变性5 min;94 ℃变性30 s,62 ℃退火30 s, 72 ℃延伸30 s,共30个循环;最后72 ℃延伸10 min,4 ℃保存。

图1 CRISPR/Cas9表达载体示意图
以EF1a + 5′GFP和3′GFP为模板,F4和R5为引物,利用重叠PCR扩增得到带有双酶切位点XbaⅠ和KpnⅠ的EF1a-GFP完整序列。重叠PCR条件:10×buffer 5 μL,dNTPs 4 μL,模板(EF1a + 5′GFP和3′GFP)1 μL,DNA聚合酶0.5 μL,引物(F4+R5)1 μL,补水至50μL。扩增程序同“F3和R3扩增程序”。
以pcDNA3.1为母载体,分别利用双酶切(XmaⅠ/NarⅠ)和(XbaⅠ/KpnⅠ)进行胶回收,4 ℃过夜连接,将CMV-dsRed和EF1a-GFP连接到载体pcDNA3.1。连接产物转化至DH5α感受态细胞,氨苄抗性平板筛选,挑取单克隆,按照质粒提取试剂盒说明书提取质粒并测序验证,得到载体pcDNA3.1-CMV-DsRed-EF1a-GFP.BS.with repeats。
分别以等量的F6和R6、F7和R7为引物,利用PCR(单链退火形成双链)拟合得到含有双酶切位点NotⅠ和BamHⅠ的Target1序列和Target2序列。PCR条件:引物(F+R) 2 μL,10×buffer 1 μL,补水至10 μL。程序:37 ℃ 30 min,95 ℃ 5 min,-1 ℃/s降温至25 ℃。
以质粒pcDNA3.1-CMV-DsRed-EF1a-GFP.BS.with repeats为载体骨架,双酶切(NotⅠ/BamHⅠ)后连接、转化,氨苄抗性平板筛选,挑取单克隆,提取质粒并测序验证,最终分别得到含有Target1和Target2序列的双荧光报告载体,命名为pcDNA3.1-CMV-DsRed-EF1a-Target1.GFP.BS.with repeats和pcDNA3.1-CMV-DsRed-EF1a-Target2.GFP.BS.with repeats。报告载体工作原理见图2。
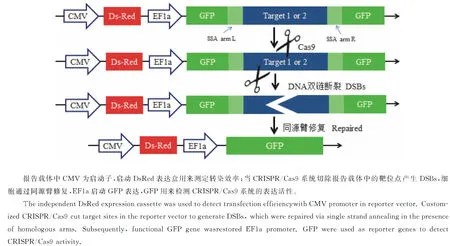
图2 双荧光报告载体原理
1.2.3 细胞培养 HEK293T细胞培养条件:DMEM高糖培养基(含φ=10%胎牛血清)、L-glutamine、丙酮酸钠。37 ℃、φ=5% CO2恒温培养。
1.2.4 在HEK293T细胞中检测CRISPR/Cas9活性 将HEK293T细胞在12孔板上培养至融合度达60%~70%左右时,采用Sofast转染试剂推荐体系,将CRISPR/Cas9表达载体分别与双荧光报告载体共转染到HEK293T细胞中,同时转染无sgRNA的空CRISPR/Cas9表达载体和报告载体为空白对照。转染10 h后更换培养液。48 h后,在倒置荧光显微镜下检测荧光表达情况。红色荧光检测转染效率,绿色荧光检测CRISPR/Cas9的工作效率。
将CRISPR/Cas9表达载体和对应的报告载体共转染至HEK293T细胞,CRISPR/Cas9系统表达的嵌合型sgRNA引导Cas9酶作用于报告载体的靶点,产生DSB,刺激细胞利用单链退火的机制修复报告载体,修复过程将删除靶序列,同时也恢复GFP基因的开放阅读框,细胞表现为GFP阳性。因此,HEK293T细胞共转染表达载体和报告载体后,在荧光显微镜下通过细胞计数的方法观察GFP阳性细胞的比率,间接反应CRISPR/Cas9系统切割靶序列的效率。
2 结果与分析
2.1 CRISPR/Cas9真核表达载体及报告载体的构建
构建的表达载体msgRNA-2分别利用BamHⅠ和EcoRⅠ、XhoⅠ和XbaⅠ进行双酶切鉴定,分别得到5 660和105 bp、5 653和112 bp的目的条带(图3-A和3-B),经测序进一步验证,与预期结果一致,表明表达载体构建成功。由于相对应的双荧光报告载体插入的目的片段只有20 bp,因此对其进行测序验证(图3-C),经序列比对后,结果正确,表明报告载体构建成功。
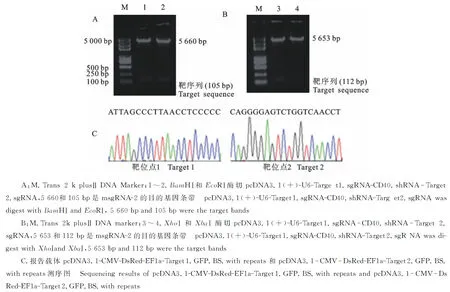
图3 CRISPR/Cas9表达载体与报告载体的鉴定
2.2 CRISPR/Cas9在哺乳细胞中的活性检测
将构建好的表达载体和报告载体共转染HEK293细胞后,通过在荧光显微镜下观察红色和绿色荧光来判断CRISPR-cas9系统是否工作。红色荧光检测转染效率,绿色荧光指示载体工作效率。转染24 h后,荧光显微镜下观察到细胞中绿色荧光微弱,但已经可以观察到绿色荧光。继续培养至48 h后,试验组均检测到绿色荧光表达(图4),证明CRISPR/Cas9在HEK293T细胞中工作。通过对绿色荧光细胞进行计数,与对照组细胞相比较,该系统的工作效率约为20%。
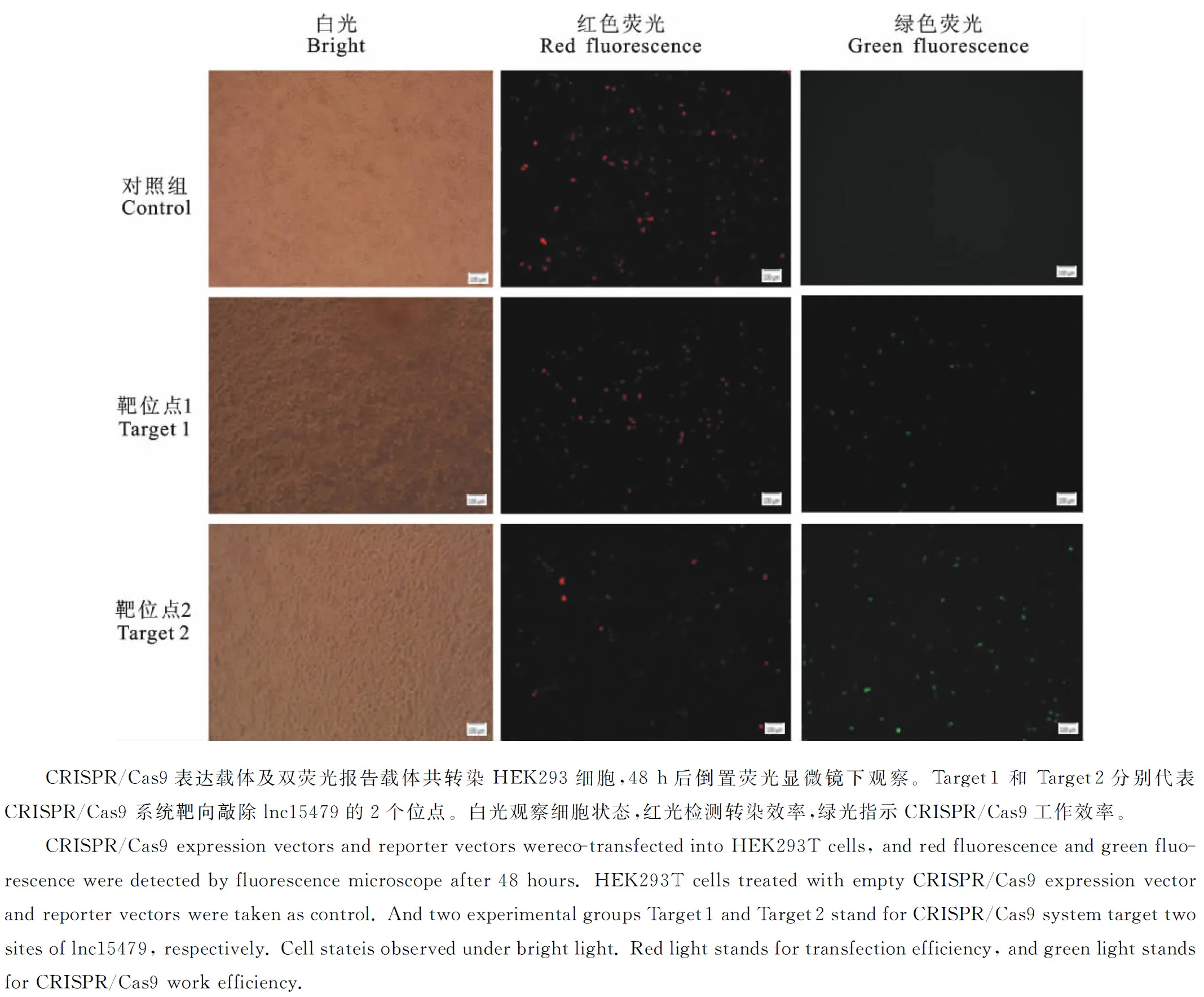
图4 CRISPR/Cas9转染HEK293T 细胞后的荧光表现
3 讨 论
陕北白绒山羊羊绒的生长具有明显的周期性变化,研究表明,多种调控因子参与这一过程,而且非编码RNA对调控毛囊细胞的周期性发育、平衡毛囊的正常生长和毛发纤维形态等具有重要作用[16-19]。在哺乳动物细胞中,除1.5%~2.0%的基因编码蛋白外,其他的都转录为非编码RNA,包括长链非编码RNA(long noncoding RNAs, lncRNAs)、miRNA、piRNA及siRNA等。研究表明[20],以miRNA为代表的非编码RNA参与绒山羊的毛囊发育及羊绒的周期性生长过程,而关于lncRNAs调控毛囊发育的研究鲜见报道。由于许多lncRNAs仅在特定的发育阶段出现[21],或具有组织特异性和细胞特异性[22]。前期对羊绒生长期和休止期皮肤组织通过RNA-seq技术获得一些差异表达的lncRNA[23]。lnc15479作为羊绒生长期和休止期有极显著差异的lncRNA,在羊绒生长期的表达量显著高于休止期,对羊绒的周期性生长可能具有一定的调控作用,但作用机制目前仍不清楚。因此,本研究拟通过CRISPR/Cas9技术实现对大片段lncRNA的敲除,为进一步研究lncRNA调控羊绒周期性生长的机制奠定基础。
RNA干扰(RNAi)技术是通过外源或内源的双链RNA(double-stranded RNA,dsRNA)特异性地引起基因表达沉默,干扰序列可能随机整合到基因组,而且整合位置对细胞的影响不确定,且对照较难控制,因此采用此方法无法实现对lncRNA的特异性敲除,进而研究其功能。CRISPR/Cas9系统是一个由蛋白和核酸组成的蛋白核酸复合物,利用sgRNA对特定的DNA序列进行识别,进而介导Cas9蛋白对靶点序列进行切割[24]。CRISPR/Cas9系统的优势在于设计简单、可针对不同的靶点设计sgRNA;而且CRISPR/Cas9 系统能够实现多位点、多基因的打靶。可通过直接合成、体外转录和载体表达等多种方式引入多个sgRNA,同时介导Cas9对不同靶序列的切割,进而实现多基因多位点打靶,甚至是基因组中大片段的删除。研究[25]表明CRISPR/Cas9可以结合内源性RNA,在活体细胞中通过内源性RNA追踪进行基因编辑。Cong等[26]和Mali等[24]发现位于同一条染色体上的2个sgRNA对DNA双链进行操作时,在获得定点突变的同时也存在DNA片段删除的情况。Fujii等[27]成功获得约10 kb的DNA片段删除小鼠,并且成功繁育出下一代。但是关于CRISPR-cas9特异性敲除lncRNA的研究尚未见报道。本研究正是基于CRISPR/Cas9系统的优点,成功构建靶向lnc15479进行大片段敲除的CRISPR/Cas9系统,工作效率达到20%左右,为进一步研究lnc15479的功能提供技术支持。
Reference:
[1]GUISE J M,SEGEL S Y,LARISON K,etal.STORC safety initiative: a multicentre survey on preparedness & confidence in obstetric emergencies [J].Quality&SafetyinHealthCare,2010,19(6):e41.
[2]MERCER T R,DINGER M E,MATTICK J S.Long non-coding RNAs:insights into functions [J].NatureReviewsGenetics,2009,10(3):155-159.
[3]WILUSZ J E,SUNWOO H,SPECTOR D L.Long noncoding RNAs:functional surprises from the RNA world [J].Genes&Development,2009,23(13):1494-1504.
[4]DOUDNA J A,CHARPENTIER E.Genome editing the new frontier of genome engineering with CRISPR-Cas9[J].Science,2014,346(6213):1258096.
[5]PARIKH B A,BECKMAN D L,PATEL S J,etal.Detailed phenotypic and molecular analyses of genetically modified mice generated by CRISPR-Cas9-mediated editing [J].PloSOne,2015,10(1):e0116484.
[6]IRION U,KRAUSS J,NUSSLEIN-VOLHARD C.Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system [J].Development,2014,141(24):4827-4830.
[7]REN X J,YANG ZH H,XU J,etal.Enhanced specificity and efficiency of the CRISPR/Cas9 system with optimized sgRNA parameters inDrosophila[J].CellReports,2014,9(3):1151-1162.
[8]LI W J,YI P SH,OU G SH.Somatic CRISPR-Cas9-induced mutations reveal roles of embryonically essential dynein chains inCaenorhabditiseleganscilia [J].TheJournalofCellBiology,2015,208(6):683-692.
[9]ENDO M,MIKAMI M,TOKI S.Multigene knockout utilizing off-target mutations of the CRISPR/Cas9 system in rice [J].PlantCellPhysiology,2015,56(1):41-47.
[10]JIANG W ZH,ZHOU H B,BI H H,etal.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification inArabidopsis,tobacco,Sorghumand rice [J].NucleicAcidsResearch,2013,41(20):e188.
[11]JOHNSON R A,GUREVICH V,FILLER S,etal.Comparative assessments of CRISPR-Cas nucleases’ cleavage efficiency in planta [J].PlantMolecularBiology,2015,87(1/2):143-156.
[12]VAN PIJKEREN J P,BRITTON R A.Precision genome engineering in lactic acid bacteria [J].MicrobialCellFactories,2014,13(Suppl1):S10.
[13]YUEN K S,CHAN CH P,WONG N H M,etal.CRISPR/Cas9-mediated genome editing of Epstein-Barr virus in human cells [J].JournalofGeneralVirology,2015,96( Pt 3) :626-636.
[14]SEEGER C,SOHN J A.Targeting hepatitis B virus with CRISPR/Cas9[J].MolecularTherapy-NucleicAcids,2014,3(12):e216.
[15]ZHANG L,ZHANG T,WANG L,etal.In vivo targeted delivery of CD40 shRNA to mouse intestinal dendritic cells by oral administration of recombinantSacchromycescerevisiae[J].GeneTherapy,2014,21(7):709-714.
[16]FATICA A,BOZZONI I.Long non-coding RNAs:new players in cell differentiation and development [J].NatureReviewsGenetics,2014,15(1):7-21.
[17]JAVE-SUAREZ L F,WINTER H,LANGBEIN L,etal.HOXC13 is involved in the regulation of human hair keratin gene expression [J].JournalofBiologicalChemistry,2002,277(5):3718-3726.
[18]MARDARYEV A N,AHMED M I,VLAHOV N V,etal.MicroRNA-31 controls hair cycle-associated changes in gene expression programs of the skin and hair follicle [J].FasebJournal,2010,24(10):3869-3881.
[19]BOTCHKAREVA N V.MicroRNA/mRNA regulatory net- works in the control of skin development and regeneration [J].CellCycle,2012,11(3):468-474.
[20]SU R,FU SH Y,ZHANG Y J,etal.Comparative genomic approach reveals novel conserved microRNAs in Inner Mongolia cashmere goat skin and longissimus dorsi [J].MolecularBiologyReports,2015,42(5):989-995.
[21]DINGER M E,AMARAL P P,MERCER T R,etal.Long noncoding RNAs in mouse embryonic stem cell pluripotency and differentiation [J].GenomeResearch,2008,18(9):1433-1445.
[22]PRASANTH K V,SPECTOR D L.Eukaryotic regulatory RNAs:an answer to the ‘genome complexity’ conundrum[J].Genes&Development,2007,21(1):11-42.
[23]郭 杨.羊绒周期性生长特异性lncRNAs的筛选[D].陕西杨凌:西北农林科技大学,2015.
GUO Y.Specificity lncRNAs screening of cashmere cyclical growth[D].Yangling Shaanxi:Northwest A&F University(in Chinese with English abstract).
[24]MALI P,YANG L,ESVELT K M,etal.RNA-guided human genome engineering via Cas9 [J].Science,2013,339(6121):823-826.
[25]NELLES D A,FANG M Y,O’CONNELL M R,etal.Programmable RNA tracking in live cells with CRISPR/Cas9 [J].Cell,2016,165(2):488-496.
[26]CONG L,RAN F A,COX D,etal.Multiplex genome engineering using CRISPR/Cas systems [J].Science,2013,339(6121):819-823.
[27]FUJII W,KAWASAKI K,SUGIURA K,etal.Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease [J].NucleicAcidsResearch,2013,41(20):e187.
(责任编辑:顾玉兰 Responsible editor:GU Yulan)
Construction and Active Verification of lnc15479 Targeted Knockout System on Cashmere Goat by CRISPR/Cas9 Technology
FENG Shuaishuai, LUO Zhixin, YAN Qiang, XUN Kun, WANG Shanhe,SHAO Simin, ZHANG Zhiying and WANG Xin
(College of Animal Science and Technology, Northwest A&F University, Yangling Shaanxi 712100, China)
In order to understand the function of lnc15479 in hair follicle periodic development, two sgRNA target sites in the upstream and downstream of lnc15479 were designed and the CRISPR/Cas9 expression vector was constructed. The double fluorescent reporter vector based on SSA repair mechanism was then constructed to detect the cleavage efficiency of CRISPR-cas9. The expression and report vectors were then co-transfected HEK293T cells to detect the work efficiency of CRISPR/Cas9. The results showed that CRISPR/Cas9 vector was successfully constructed and its cleavage efficiency was about 20% from the red and green fluoresence. The results would provide technical support for the further study on lnc15479.
CRISPR/Cas9; Cashmere goat; LncRNA
FENG Shuaishuai, male, master student. Research area:biotechnology and animal breeding. E-mail:972223216@qq.com
WANG Xin, female, professor. Research area:biotechnology and animal breeding. Email:wxwza@126.com
2016-05-19
2016-05-30
国家自然科学基金(31201769,31472068)。
冯帅帅,男,硕士研究生,研究方向为生物技术与动物育种。E-mail: 972223216@qq.com
王 昕,女,教授,研究方向为生物技术与动物育种。E-mail: wxwza@126.com
日期:2016-10-20
Q812
A
1004-1389(2016)10-1442-07
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20161020.1653.006.html
Received 2016-05-19 Returned 2016-05-30
Foundation item National Natural Science Foundation of China (No.31201769,31472068).

