CRTC3基因多态性及基因型组合与秦川牛生长性状的关联分析
徐怀超,昝林森,2*,王洪宝,2,宁 越
(1.西北农林科技大学动物科技学院, 杨凌 712100; 2. 国家肉牛改良中心, 杨凌 712100)
CRTC3基因多态性及基因型组合与秦川牛生长性状的关联分析
徐怀超1,昝林森1,2*,王洪宝1,2,宁 越1
(1.西北农林科技大学动物科技学院, 杨凌 712100; 2. 国家肉牛改良中心, 杨凌 712100)
旨在探讨CRTC3基因作为秦川牛生长性状候选基因的可能性,寻找与秦川牛生长相关的分子标记。本研究采用PCR-RFLP方法检测395头健康秦川牛CRTC3基因的多态性,分析其多态位点不同基因型及组合基因型与秦川牛生长性状的关联性。结果发现,CRTC3基因扩增序列区间存在2个SNPs位点(位于外显子区域的G66478C和位于内含子区域的C91297T)。关联性分析表明,在本试验所选取的395头秦川牛群体中, G66478C位点GC基因型个体在体斜长方面极显著高于CC型个体均值(P<0.01),且在胸深方面显著高于CC型个体均值(P<0.05)。在C91297T位点,CT基因型个体均值在体斜长、腰高、尻长和胸深方面显著高于TT基因型个体均值(P<0.01)。CRTC3基因的优势基因型组合CC-TT的个体在腰高、尻长上极显著高于CC-CT基因型组合的个体均值(P<0.01),且基因型组合CC-TT的个体在胸深、胸围显著高于CC-CT组合的个体均值(P<0.05)。综上,可以尝试将CRTC3基因作为影响秦川牛生长性状的候选基因用于标记辅助选择,为秦川牛选育工作提供科学依据。
CRTC3;生长性状;基因型组合;分子标记
生长性状是畜牧业生产中一个重要的经济性状,是衡量育种价值及经济效益的重要指标,寻找相关的分子标记可以缩短对生长性状的选育年限[1]。CRTC3(CREB regulated transcription coactivator 3)是最近发现在葡萄糖和脂质代谢中发挥重要作用的一个蛋白因子,CRTC3属于CREB共活化因子家庭。CRTCs是一个主要调节CREB转录因子活性的蛋白质家族,2003年首先由诺华制药公司的科研人员利用基因组高通量筛选发现。CRTCs首次发现时被命名TORC(Transducer of regulated CREB)[2],CRTCs基因被证实存在于果蝇、鼠及人类中。哺乳动物的CRTCs蛋白家族有3个成员,CRTC1、CRTC2和CRTC3。3种亚型在N端同源性高,其高度保守的卷曲螺旋结构域(Coiled-coil domain) 能与CREB的碱性亮氨酸拉链结构域(Basic leucine zipper,bZIP)相结合,提示其对CREB依赖的转录调控可能具有重要作用[3]。
CRTC3是环磷腺苷反应元件结合物协同激活物家族一个新发现的重要成员。研究证实,CRTC3在棕色脂肪里高度表达,且能够通过调控脂肪组织里的儿茶酚胺信号通路来影响脂肪的代谢和促进肥胖的发生发展[4]。CRTC3也能通过增加PPARγ和PGC-lα的表达来诱导棕色脂肪组织的UCP1表达增加,从而促进其脂肪组织的代谢活性,消耗更多的能量[5]。此外,CRTC3在肌肉组织中也表现出较强的代谢活性。在肌细胞里,CRTC3与CREB的互作强有力的刺激PGC-7amRNA转录活性,从而促进线粒体呼吸链和三羧酸循环下游目的基因的表达,其结果是增加了线粒体代谢活动。敲除CRTC3大鼠的能量消耗和氧化代谢明显增加,对胰岛素敏感性明显增加,并且出现脂肪组织减少和体重减轻的现象[6]。Y.Song等研究发现CRTC3基因突变小鼠表现出较强的脂肪分解和脂肪酸氧化的能力[7]。基于CRTC3的分子结构和生物学特性以及在老鼠和人类中发现的生物学功能,说明它很有可能是一种能影响动物生长性状的调节因子。
鉴于CRTC3基因在能量代谢和脂质代谢过程中的重要作用,我们推测其在畜禽生长性状方面可能发挥直接或间接的作用,但是目前尚未见到相关报道。因此,本研究拟以CRTC3基因为目标基因,采用PCR-RFLP方法,分析不同标记基因型及基因型组合对秦川牛生长性状的影响,以期为秦川牛分子育种提供新的理论方法和技术路径。
1 材料与方法
1.1 试验材料
血样采自陕西省良种肉牛繁育中心、陕西省秦川牛保种厂和西北农林科技大学良种肉牛繁育中心3个场区,选择395头同等饲养条件下的18~24月龄健康的秦川牛,对每头牛颈静脉采血10 mL,ACD抗凝(V(ACD)∶V(血液)=1∶6),置于-80 ℃保存。同时采集试验牛的8个生长性状指标(体斜长、体高、腰高、尻长、腰角宽、胸深、胸围和坐骨端宽)。
1.2 基因组DNA的提取及检测
采用常规的酚-氯仿提取法[8]提取秦川牛血样基因组DNA,提取的DNA 用TE缓冲液处理后,采用0.5%琼脂糖凝胶电泳检测DNA质量,紫外分光光度计测定DNA浓度,然后-20 ℃保存备用。
1.3 CRTC3基因PCR扩增、测序
根据GenBank公布的牛CRTC3序列(GenBank AC_000178),利用Primer 5.0软件设计引物(表1),引物均由上海生工生物工程技术服务有限公司合成。
PCR反应体系30.0 μL:含有核酸染料的dNTPs、TaqDNA聚合酶、10×Buffer的Mix 15.0 μL,ddH2O 11.8 μL,上游、下游引物各(10 pmol·μL-1)0.6 μL、模板DNA(50 ng·μL-1)2.0 μL。PCR反应程序:95 ℃预变性 5 min;94℃ 35 s,退火30 s (退火温度见表1),72 ℃ 30 s,37个循环;72 ℃延伸10 min;4 ℃保存。PCR扩增产物用10 g·L-1琼脂糖凝胶电泳检测。在扩增好的PCR产物中,每种随机选取20个样本使用凝胶回收试剂盒回收纯化,送至上海生物工程股份有限公司进行测序,测序结果使用DNAMAN软件进行对比分析,寻找突变位点。
1.4 PCR-RFLP分析
经测序比对后,取含有突变位点的PCR扩增产物10 μL,依照G66478C和C91297T顺序,分别加入限制性内切酶Eco47 III (TaKaRa, Dalian, China)和ApaL I (TaKaRa, Dalian, China) 各1.0 μL,2.0 μL的10×Buffer,加入去离子水至20 μL,将反应体系分别置于37、65、37和37 ℃恒温箱中4 h,然后用2%的琼脂糖凝胶电泳检测,凝胶成像系统照相分析。对切开的不同条带的DNA样品扩增送至上海生物工程股份有限公司进行测序,验证酶切效果。
表1CRTC3基因2个SNPs位点引物信息
Table 1 Primer information of 2 SNPs inCRTC3 gene

SNPs突变区域Mutationregion引物序列(F/R)(5'-3')Primersequence退火温度/℃Tm扩增片段大小/bpFragmentsizeG66478CExon6TTTATGGATGATTGTAAATCGGGAGAAAGAGGTGGGAGGGAAGG67424C91297TIntron13AGTGTGCTGAGGAGGGAGGTGGACAGTGCGGATTAGAAGGCGTG66843
1.5 数据统计分析
根据基因型统计分析结果计算2个SNPs位点的基因型和等位基因频率,并进行Hardy-Weinberg平衡适应性检验。通过SHEsis在线软件计算连锁不平衡相关系数γ2。利用SPSS (19.0) 软件中的GLM分析模型对秦川肉牛生长性状的影响。分析结果用“平均数±标准误”表示(P<0.05为差异显著水平)。分析模型:
Yij=μ+Gi+eij
其中,Yij为个体表型值,μ为群体均值,Gi为标记基因型效应,еij为随机误差。
2 结 果
2.1 CRTC3基因型分析
通过测序分析发现,CRTC3基因扩增序列存在2处SNPs,分别命名为G66478C和C91297T。G66478C是错义突变且突变导致了丝氨酸到苏氨酸的改变,C91297T是位于内含子区域的突变。
对SNP位点所在序列扩增后进行PCR-RFLP分型。具体酶切效果如图1所示。对G66478C所在序列酶切分型结果为CC型(424 bp),GC型 (163、261、424 bp)。对C91297T所在序列酶切分型结果为CC型(843 bp),CT型(246、597、 843 bp),TT型(246、597 bp)。将G66478C和C91297T突变位点不同基因型对应的DNA样品进行PCR扩增,送至上海生物工程股份有限公司进行测序,经检验发现酶切正确(图2)。
从群体遗传学角度分别分析这2个SNPs的基因型频率和等位基因频率,并进行卡方检验检测Hardy-Weinberg平衡状态。表2结果显示,在G66478C和C91297T处,CC为优势基因型,C是G66478C和C91297T的优势基因。卡方检验表明,G66478C和C91297T属于Hardy-Weinberg不平衡状态(P<0.01)。

A. G66478C酶切图;B. C91297T酶切图A. The RFLP map of G66478C; B. The RFLP map of C91297T 图1 SNP位点RFLP酶切图Fig.1 RFLP patterns of SNP loci

A.G66478C不同基因型的验证图;B. C91297T不同基因型的验证图A.The verification of different genotypes at G66478C; B. The verification of different genotypes at C91297T图2 CRTC3基因SNPs位点测序验证图Fig.2 The verification of CRTC3 at SNPs
2.2 CRTC3基因SNP位点与秦川牛生长性状的关联分析
对395头秦川牛的8个生长性状分别与CRTC3基因2个SNPs位点不同基因型进行关联分析。表3结果显示,G66478C位点GC基因型个体均值在体斜长方面极显著高于CC型个体均值(P<0.01),GC基因型个体均值在胸深方面显著高于CC型个体均值(P<0.05)。C91297T位点CT基因型个体均值在体斜长、腰高、尻长、胸深方面极显著高于TT型个体均值(P<0.01);CT基因型个体均值在体高、腰角宽、坐骨端宽方面显著高于其他两种不同基因型个体均值(P<0.05)。
表2CRTC3位点的基因型和等位基因分布频率
Table 2 Genotype and allele frequency ofCRTC3

位点Site等位基因频率Allelefrequency基因型频率Genotypefrequency卡方检验χ2G66478CGGGCCCGC00.180.880.090.913.97C91297TTTCTCCTC0.100.190.710.200.8062.02
表3CRTC3基因各SNP位点不同基因型和秦川牛生长性状关联分析
Table 3 Associations betweenCRTC3 SNP genotypes and growth traits of Qinchuan cattle

位点Site基因型Genotype体斜长/cmBL体高/cmWH腰高/cmHH尻长/cmRLG66478CGC(71)135.89±0.74A121.20±0.65124.10±0.4042.70±0.24CC(348)130.09±1.63B117.97±1.42123.93±0.8641.92±0.53P0.0010.3290.0710.079C91297TCC(280)135.19±0.81a120.90±0.70124.99±0.4243.53±0.26ACT(75)139.58±2.13A123.70±1.85a125.71±1.12A44.10±0.69ATT(40)131.70±1.56Bb118.22±1.35b120.89±0.82B40.72±0.05BP0.0030.0180.0010.000位点Site基因型Genotype腰角宽/cmHW胸深/cmCD胸围/cmCC坐骨端宽/cmPBWG66478CGC(71)39.36±0.3259.98±0.40a165.48±1.0719.18±0.20CC(348)38.93±0.7157.55±0.88b163.92±2.3618.82±0.45P0.0570.0130.1480.063C91297TCC(280)37.31±0.51b59.79±0.43162.90±1.82b19.20±0.22CT(75)39.16±0.56a62.43±1.14A163.98±2.01b19.52±0.59aTT(40)39.27±0.70a58.43±0.83B170.87±2.48a17.96±0.43bP0.0160.0010.0110.012
同列相同字母表示差异不显著,不同小写字母表示差异显著P<0.05,不同大写字母表示差异极显著P<0.01,下表同
Data marked with different superscripts in the same row differ significantly (Capital letters.P<0.01; Small letters.P<0.05). BL. Body length; WH. Wither height; HH. Hip height; RL. Rump length; HW. Hip width; CD. Chest depth; CC. Chest circumference;PBW. Pin bone width.The same as below
2.3 CRTC3基因突变位点的连锁不平衡分析及单倍型分析
运用PHASE软件对2个突变位点进行连锁不平衡分析。统计得到r2值为0.005(小于0.33),表明这2个SNPs位点连锁性不强。研究表明,当SNP位点处于弱的连锁性时,基因型组合比单位点标记在分子标记方面更能利于相关性状的关联分析[9-10]。表4所示单倍型频率大于3%的有4种,分别命名为 Hap1~Hap4,单倍型频率分别为0.668、0.149、0.135和0.047(表4)。
2.4 CRTC3基因型组合与秦川牛生长性状的关联分析
CRTC3基因型组合与秦川牛8个生长性状的关联分析结果如表5所示(基因型组合频率个体数小于5%的不参与统计分析)。分析结果表明,基因型组合CC-TT个体均值在腰高、尻长指标上极显著高于CC-CT基因型组合的个体均值(P<0.01),且基因型组合CC-TT个体在胸深、胸围显著高于CC-CT组合的个体均值(P<0.05)。在3种基因型组合中CC-TT型秦川牛生长性状的数值均是最优的。因此,在实际育种生产中可加强CC-TT组合个体的选择强度,不断提高其在群体当中的比例,以加快秦川肉牛生产性能的改良和提高。
表4CRTC3基因单倍型分析
Table 4 Haplotypes ofCRTC3 gene and their frequencies

单倍型HaplotypeG66478CC91297T频率Frequency数量NumberHap1CC0.668528Hap2CT0.149118Hap3GC0.135107Hap4GT0.04737
表5 秦川牛CRTC3基因不同基因型组合生长性状的平均值
Table 5 The average values of different genotype combinations in growth traits inCRTC3 of Qinchuan cattle
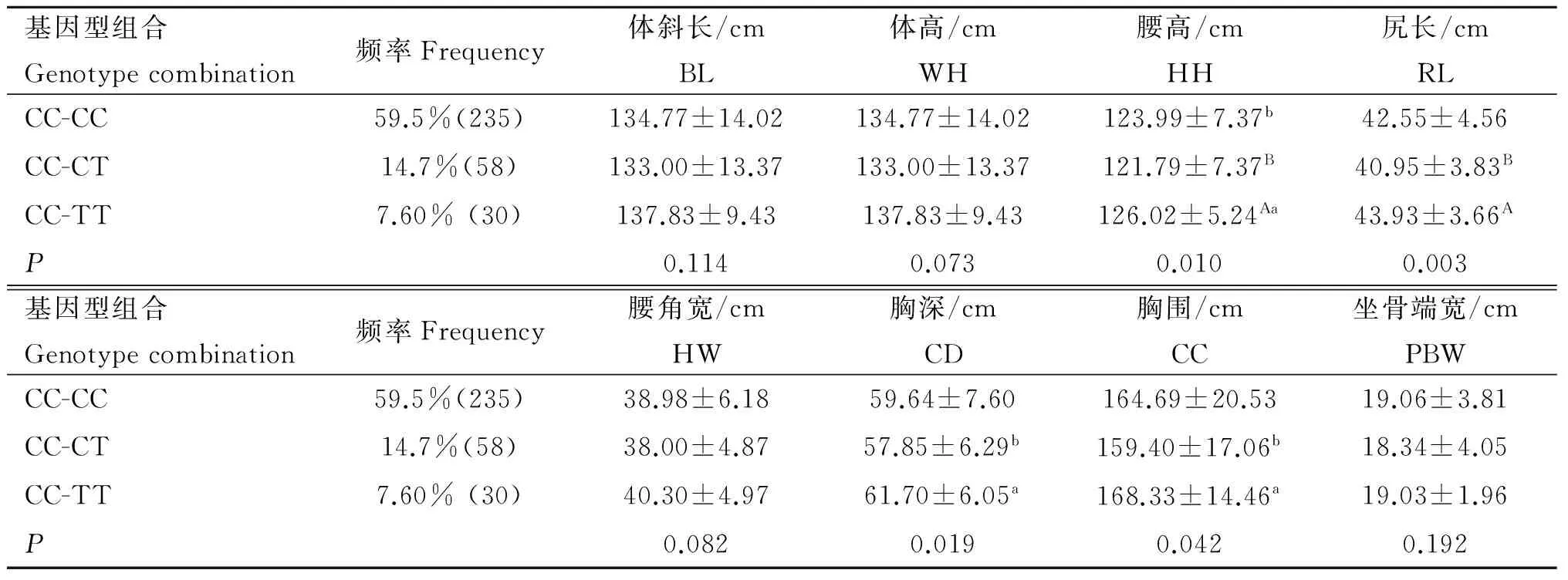
基因型组合Genotypecombination频率Frequency体斜长/cmBL体高/cmWH腰高/cmHH尻长/cmRLCC-CC59.5%(235)134.77±14.02134.77±14.02123.99±7.37b42.55±4.56CC-CT14.7%(58)133.00±13.37133.00±13.37121.79±7.37B40.95±3.83BCC-TT7.60%(30)137.83±9.43137.83±9.43126.02±5.24Aa43.93±3.66AP0.1140.0730.0100.003基因型组合Genotypecombination频率Frequency腰角宽/cmHW胸深/cmCD胸围/cmCC坐骨端宽/cmPBWCC-CC59.5%(235)38.98±6.1859.64±7.60164.69±20.5319.06±3.81CC-CT14.7%(58)38.00±4.8757.85±6.29b159.40±17.06b18.34±4.05CC-TT7.60%(30)40.30±4.9761.70±6.05a168.33±14.46a19.03±1.96P0.0820.0190.0420.192
3 讨 论
单倍型是同一染色体上能够共同遗传的多个基因座上等位基因的组合。D.Fallin等研究发现,在种群中单倍型比单个位点能够更有效的得到传递,近年来在家畜多态性分析中单倍型分析得到越来越多的应用,研究者多利用单倍型分析来研究数量性状位点(QTL)与家畜生产性能的关系[11]。与家畜生产性能相关的大多数性状都是由多基因或是某一基因的多个位点共同调控的,所以在试验中分析某一基因中单个位点的多态性往往不能准确得出基因或等位基因与性状的真实相关性,需要同时加强对多个位点的综合效应进行探讨,分析结果的可靠性才更加有效[12]。桂林生等研究发现SIRT1和SIRT2基因的基因型组合对秦川牛眼肌面积和肌间脂肪有显著影响[13]。张松研究发现CD36和KEAP1基因突变位点的基因型组合对秦川牛的体尺性状有显著影响[14]。
作为我国著名的五大地方黄牛之一的秦川牛,存在生长发育较慢,后躯肌肉欠充实等明显缺陷[15]。目前,通过传统的育种方法来改善牛的生长性状是一件长期而艰巨的工作。研究人员已经发现的生长性状相关的基因分子标记可以加快家畜育种工作进程,并缩短育种时间。目前,针对秦川牛的遗传改良仅集中于基因单个位点的多态性研究[16-18],鉴于基因单倍型分析的优势以及在畜禽上的逐步应用,本试验挑选与机体脂肪代谢和能量相关的CRTC3基因,研究其多态性及单倍型组合与秦川牛生长性状的关联性。
CRTC3作为cAMP-CREB信号转录通路的新成员,在脂肪、肝和骨骼肌等胰岛素敏感组织中与环磷腺苷反应元件结合物(CREB)协同激活调控糖和脂类的代谢[19]。相关研究表明,CRTC3基因的多态性位点在墨西哥裔美国人中与肥胖有关[7]。此外,Z.Ou等的研究表明,在中国汉族人群中CRTC3基因多态性rs11635252位点与超重有关[20]。本试验通过PCR-RFLP对395头秦川牛CRTC3基因进行了多态性分析,发现了2个多态位点。经过与GenBank上牛CRTC3基因比对发现:G66478C的突变导致了丝氨酸到苏氨酸的改变。通过基因分型在C91297T发现3种基因型,而G66478C只有两种基因型。这可能与试验中选择的群体过小或者在进化过程中自然和人为定向选择有关。相对于外显子区域的突变,越来越多的研究表明内含子区域的突变也能够对性状产生显著的影响。虽然内含子的突变不会对编码的氨基酸序列发生影响,但其可能对顺式作用原件的功能产生影响或者改变细胞内mRNA的可变剪切,进而对基因的功能或者表达造成影响[21-22]。研究表明绵羊MSTN基因内含子突变对绵羊的生长发育造成显著的影响[23]。本试验中,位于内含子区域的C91297T位点上的突变可显著影响秦川牛的部分生长。通过CRTC3基因不同基因型组合与秦川牛生长性状的关联分析发现,CRTC3基因型组合CC-TT在腰高、尻长上极显著高于CC-CT基因型组合的个体均值,在胸深、胸围方面显著高于CC-CT组合。因此在本试验群体中,CC-TT可被视为一种最为优秀的合并基因型。
4 结 论
本研究发现CRTC3基因在秦川牛中存在多态性且与生长性状显著关联。G66478C位点GC基因型个体在体斜长方面极显著高于CC型,GC基因型个体在胸深方面显著高于CC型个体。C91297T位点CT基因型个体均值在体斜长、腰高、尻长、胸深方面极显著高于TT基因型个体均值;CT基因型个体均值在体高、腰角宽和坐骨端宽方面显著高于其他两种基因型个体均值。CRTC3基因的优势基因型组合CC-TT在腰高、尻长指标上极显著高于CC-CT基因型组合的个体均值,在胸深、胸围方面显著高于CC-CT组合。可以尝试将CRTC3基因作为影响生长性状的候选基因用于标记辅助选择,为秦川牛选育工作提供科学依据。
[1] CHANG M T,CHENG Y S, HUANG M C. A novel non-synonymous SNP of theCOLXgene and its association with duck reproductive traits[J].MolCellProbes, 2012, 26(5): 204-207.
[2] IOURGENKO V,ZHANG W, MICKANIN C,et al.Identification of a family of cAMP response element-binding protein co-activators by genome-scale functional analysis in mammalian cells[J].ProcNatlAcadSciUSA,2003,100(21): 12147-12152.
[3] CONKRIGHT M D,CANETTIERI G,SCREATON R,et al.TORCs: Transducers of regulated CREB activity[J].MolCell,2003,12(2): 413-423.
[4] BACHMAN E S, DHILLON H, ZHANG C Y, et al.betaAR signaling required for diet-induced thermogenesis and obesity resistance[J]Science,2002, 297(5582):843-845.
[5] THAN T A, LOU H, JI C,et al. Role of cAMP-responsive element-binding protein (CREB)-regulated transcription coactivator 3 (CRTC3) in the initiation of mitochondrial biogenesis and stress response in liver cells[J].JBiolChem,2011, 286(25): 22047-22054.
[6] WU Z, HUANG X, FENG Y, et al. Transducer of regulated CREB-binding proteins (TORCs) induce PGC-1α transcription and mitochondrial biogenesis in muscle cells[J].ProcNatlAcadSciUSA,2006, 103(39): 14379-14384.
[7] SONG Y,ALTAREJOS J, GOODARZI M O, et al. CRTC3 links catecholamine signalling to energy balance[J].Nature, 2010, 468(7326): 933-939.
[8] SAMBROOK J, FRITSCH E F, MANIATIS T. Molecular cloning a laboratory manual[M].Beijing: Science Press, 2002.
[9] AKEY J, JIN L, XIONG M. Haplotypes vs single marker linkage disequilibrium tests: what do we gain?[J].EurJHumGenet, 2001, 9(4): 291-300.
[10] MORRIS R W, KAPLAN N L. On the advantage of haplotype analysis in the presence of multiple disease susceptibility alleles[J].GenetEpidemiol, 2002, 23(3):221-233.
[11] FALLIN D, COHEN A, ESSIOUX L, et al. Genetic analysis of case/control data using estimated haplotype frequencies:Application to APOE locus variation and Alzheimer's disease[J].GenomeRes, 2001,11(1): 143-151
[12] 赵秀华.京海黄鸡IGFBP-1、IGFBP-2和STAT5b基因遗传效应及表达规律的研究[D]. 扬州:扬州大学,2012.
ZHAO X H. Studies on the genetic effects and expression characterization ofIGFBP-1,IGFBP-2 andSTAT5bin Jinghai Yellow chicken[D].Yangzhou:Yangzhou University,2012. (in Chinese)
[13] 桂林生, 昝林森, 王洪宝, 等.SIRT1和SIRT2基因多态性及合并基因型与秦川牛肉用性状的关联分析[J]. 畜牧兽医学报, 2015, 46(10): 1741-1749.
GUI L S,ZAN L S,WANG H B,et al. Effects of single and combined genotypes ofSIRT1 andSIRT2 genes on meat quality traits in Qinchuan cattle[J].ActaVeterinariaetZootechnicaSinica,2015,46(10):1741-1749. (in Chinese)
[14] 张 松. 秦川牛CD36和KEAP1 基因的多态性及其与体尺和肉质性状的相关性分析[D]. 杨凌:西北农林科技大学, 2014.
ZHANG S. Association of polymorphisms of CD36, KEAP1 gene with body measurements and meat quality traits in Qinchuan cattle[D]. Yangling:Northwest A&F University,2014. (in Chinese)
[15] 高建斌,昝林森,杨 宁,等. 秦川牛DKK1基因SNPs检测及其与体尺、肉质性状的关联分析[J]. 畜牧兽医学报,2013,44(3):376-386.
GAO J B,ZAN L S,YANG N,et al. Polymorphisms ofDKK1 gene and its association with body measurement and meat quality traits in Qinchuan cattle[J].ActaVeterinariaetZootechnicaSinica,2013,44(3):376-386. (in Chinese)
[16] 杨彦杰,昝林森,王洪宝. 秦川牛脂联素基因SNPs检测及其与胴体、肉质性状的相关性[J]. 遗传,2009,31(10):1006-1012.
YANG Y J,ZAN L S,WANG H B. SNPs detection ofadiponectingene and its relationship with carcass and meat quality traits in Qinchuan cattle[J].Hereditas(Beijing),2009,31(10):1006-1012. (in Chinese)
[17] TIAN W Q, WANG H C, SONG F B, et al. Association between a single nucleotide polymorphism in the bovine chemerin gene and carcass traits in Qinchuan cattle[J].GenetMolRes, 2011, 10(4): 2833-2840.
[18] XUE M, ZAN L S, GAO L , et al. A novel polymorphism of the myogenin gene is associated with body measurement traits in native Chinese breeds[J].GenetMolRes, 2011, 10(4): 2721-2728.
[19] FU A, SCREATON R A. Using kinomics to delineate signaling pathways: control of CRTC2/TORC2 by the AMPK family[J].CellCycle, 2008, 7(24):3823-3828.
[20] OU Z, WANG G, LI Q, et al. CRTC3 polymorphisms were associated with the plasma level of total cholesterol and the risks of overweight and hypertriglyceridemia in a Chinese Han population[J].MolBiolRep, 2014, 41(1): 125-130.
[21] BACHL J, OLSSON C, CHITKARA N, et al. The Ig mutator is dependent on the presence, position, and orientation of the large intron enhancer[J].ProcNatlAcadSciUSA, 1998, 95(5): 2396-2399.
[22] NOBUYOSHI M, LIN X H, TAKIMOTO Y,et al. Transcription regulation of the PDGF A-chain gene by first intron elements[J].BiochemBiophysResCommun, 1997,230(3): 569-572.
[23] SJAKSTE T,PARAMONOVA N,GRISLIS Z, et al. Analysis of the single-nucleotide polymorphism in the 5′ UTR and part of intron I of the sheepMSTNgene[J].DNACellBiol, 2011, 30(7): 433-444.
(编辑 郭云雁)
Association ofCRTC3 Gene Polymorphisms and Genotype Combination with Growth Traits of Qinchuan Cattle
XU Huai-chao1,ZAN Lin-sen1,2*,WANG Hong-bao1,2,NING Yue1
(1.CollegeofAnimalScienceandTechnology,NorthwestA&FUniversity,Yangling712100,China;2.NationalBeefCattleImprovementCenter,Yangling712100,China)
The aim of this study was to investigate the possibility ofCRTC3 gene as candidate gene for growth performance in Qinchuan cattle, and search the molecular markers related to Qinchuan cattle growth traits. PCR-RFLP method was applied to detect the polymorphisms ofCRTC3 in 395 healthy Qinchuan cattle, and the association of its polymorphisms and genotype combination with growth traits of Qinchuan cattle was also analyzed. The result showed that 2 SNPs were identified, including G66478C in exon and C91297T in intron. At the G66478C locus, the cattle with genotype GC had greater body length and chest depth values than those with genotype CC(P<0.01 andP<0.05, respectively). At the C91297T locus, animals with genotype CT had greater body length, hip height, rump length and chest depth values than those with genotype TT(P< 0.01) . Animals with CC-TT had significantly greater hip height and rump length values than those with CC-CT(P< 0.01), and chest depth and chest circumference values for CC-TT were greater than CC-CT(P<0.05). Therefore, variations in theCRTC3 gene and the genotype combination CC-CT may be useful as molecular markers for growth traits in Qinchuan cattle breeding.
CRTC3; growth traits; genotype combination; molecular markers
10.11843/j.issn.0366-6964.2016.11.005
2016-02-29
国家863计划(2013AA102505);国家科技支撑计划(2015BAD03B04);国家自然科学基金(31272411);国家肉牛牦牛产业技术体系(CARS-38); 陕西省科技统筹创新工程计划(2014KTZB02-02-01)
徐怀超(1989-),男,山东聊城人,硕士生,主要从事动物生物技术研究, E-mail: 727751348@qq.com
*通信作者:昝林森,教授,主要从事秦川肉牛新品系选育、肉牛遗传改良与种质创新等方面的研究,E-mail: zanlinsen@163.com
S823;S813.3
A
0366-6964(2016)11-2184-07

