用于微阵列数据分类的子空间融合演化超网络
王 进,刘 彬,张 军,陈乔松,邓 欣
(重庆邮电大学计算智能重庆市重点实验室,重庆 400065)
用于微阵列数据分类的子空间融合演化超网络
王 进,刘 彬,张 军,陈乔松,邓 欣
(重庆邮电大学计算智能重庆市重点实验室,重庆 400065)
针对传统模式识别方法在学习具有小样本特性的DNA微阵列数据时存在的过拟合问题,本文提出了一种子空间融合演化超网络模型.该模型通过子空间划分、超边全覆盖和子空间融合三种方法降低模型对初始化的依赖,减少了对数据空间的拟合误差,提高了演化超网络的泛化能力.对四个DNA微阵列数据集的实验结果表明,子空间融合演化超网络的识别率和在小样本训练集下的泛化能力均优于参与对比的其他传统模式识别方法.
模式识别;微阵列数据分类;演化超网络;子空间;过拟合
1 引言
DNA微阵列技术的出现为从分子水平研究疾病的发病机理和临床诊断提供了强有力的手段,特别是在临床诊断白血病[1]、结肠癌[2]等恶性肿瘤上具有较高的应用价值.与传统基于形态学信息的癌症诊断方法相比,基于DNA微阵列技术获得的基因表达谱的癌症诊断方法具有更高的准确率和可信度[1].
传统的模式识别方法在学习具有小样本特性的DNA微阵列数据时存在过拟合问题[3],这导致模型分类的泛化能力下降.同时DNA微阵列数据包含着不同基因之间庞大而复杂的并行交互作用,这些基因间的交互作用对我们研究癌症的复杂发展机制有着重要意义.传统模式识别方法[4~7]虽然取得了较好的分类效果,却难以深度挖掘基因之间的相互作用.
超网络(Hypernetwork,HN)是受生物分子网络启发而建立的一种基于超图(Hypergraph)的认知学习模型[8,9].通过演化学习,超网络可以有效获取与分类相关的关键特征,拟合输入模式空间中数据的分布概率,从而表达复杂数据的内在结构和相互之间的关系.因能有效挖掘与癌症分类相关的基因以及基因间的相互作用,演化超网络模型已成功用于DNA微阵列数据分类[10,11],然而该模型的分类效果与泛化能力受超边库初始化质量的影响较大.
针对上述问题,本文将子空间概念引入到演化超网络模型中,提出了一种子空间融合演化超网络(Evolutionary Hypernetworks with Subspace Fusion,SF-HN).通过子空间超边覆盖,弱化模型对超边初始化过程的依赖,提升其在小样本训练集下的泛化能力.为验证子空间融合演化超网络的性能,本文根据部分替代整体思想提出了一种分类器泛化能力评价方法.通过对四个DNA微阵列数据集进行试验,证明了该模型具有更优的准确性和泛化能力.
2 演化超网络
超网络是一种由大量超边组成的概率图模型,通过超边表达模式空间中数据的分布概率[8].超边所连接的顶点数称为超边的阶数(Order),所有超边阶数都为k的超网络称为k阶超网络[12].超网络演化学习通过调整超边库,提高模型与数据在模式空间概率分布的拟合度.超边替代法[11]和梯度下降法[12]是常用的演化学习方法.在分类模式下,超网络通过输入样本X与输出类别Y的联合概率P(X,Y)以及X的分布概率P(X),得到最终的决策输出:
(1)
细粒度演化超网络(Fine-Grain Evolutionary Hypernetwork,FG-HN)[11]将最优类别信息离散化(Optimal Class-Dependent Discretization,OCDD)算法与超网络结合,采用多位二进制来表述特征属性,降低了数据离散化过程中的信息损失.然而FG-HN仍无法解决在学习具有小样本特性的DNA微阵列数据时存在的过拟合问题.
3 子空间融合演化超网络
传统演化超网络只对输入模式中的训练样本集进行学习,处理小样本数据时,其泛化性将受到影响.为了提高模型的泛化能力,本文在FG-HN[11]的基础上提出了一种子空间融合演化超网络.
令S=A1×A2×…×AD表示D维数据空间,Aj(j=1,…,D)表示S中的一个属性域,k维空间Pi=Ai1×Ai2×…Aik(ik≤D)为S的一个子空间.S=P1∪P2∪…∪Pi∪…为空间S的一个子空间划分.在分类过程中,X=A1×A2×…×AD表示特征属性空间,Y表示类别标签空间.对于空间X的数据进行离散化处理,特征Aj的离散区间数为mj,则子空间Pi包含的总数据点为m=mi1×mi2×…×mik,m也称为Pi的秩,空间中的数据点也称为单元格.超边所包含的特征空间可表示为Ei=Ai1×Ai2×…×Aik.将超边看作输入模式空间的子空间,超边库表示特征属性空间X的一个划分X=E1∪E2∪…∪E|L|,其中|L|表示超边总数.
偏斜度SOD(T,P)[13]是衡量子空间划分效果的评价指标,其定义如下:
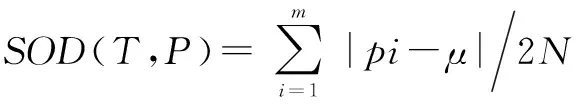
(2)
其中,N为训练集T的样本数,P为子空间,pi为训练集T投影在子空间P的第i个单元格上的样本数,m是子空间的秩,μ=N/m表示平均分布在单元格上的数据点数.SOD(T,P)的取值范围为[0,1],其值越小,数据点的分布越均匀;反之,则分布越集中.
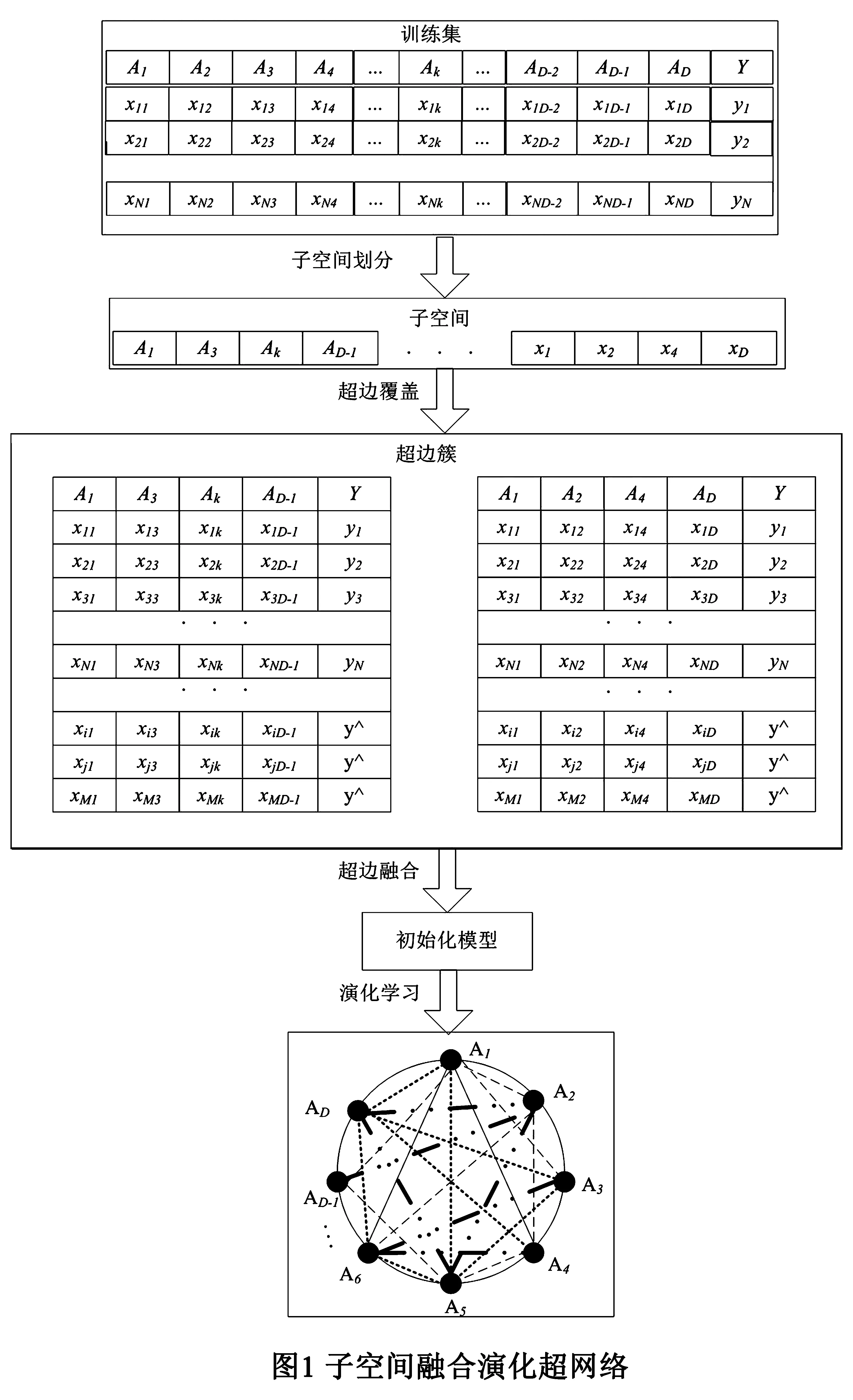
在SF-HN中,首先进行子空间划分,选择样本分布均匀的子空间集合;其次,生成超边并把超边决策范围覆盖到整个子空间;接着融合子超边簇,生成初始化模型;最终通过梯度下降方法对模型进行演化学习,提高模型对输入数据的拟合精度.子空间融合演化超网络流程如图1所示,其中超网络中的每种连线代表一条超边(例如实线表示一条包含顶点A4,A1和A6的3阶超边).3.1 子空间划分算法
超网络是对输入模式空间数据分布概率的拟合,当数据散列分布时,其覆盖的数据空间广,拟合时的误差小.因此在子空间划分算法中,采用训练集在子空间上的偏斜度对子空间的优劣进行评价,并选取样本分布信息较多的子空间集合.
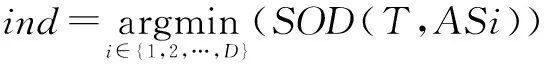
算法1 子空间划分算法
输入:训练集T,超边阶数k;子空间数sn,冗余倍数d,阈值tsod.
输出:划分的子空间集合E.
步骤1num←sn,E←Φ,计算tsod.
步骤2 初始化num*d条阶数为k的超边.
步骤3 将T向每条超边对应的子空间EEi投影,并计算SOD(T,EEi).
步骤4 将所有子空间按SOD值升序排序.
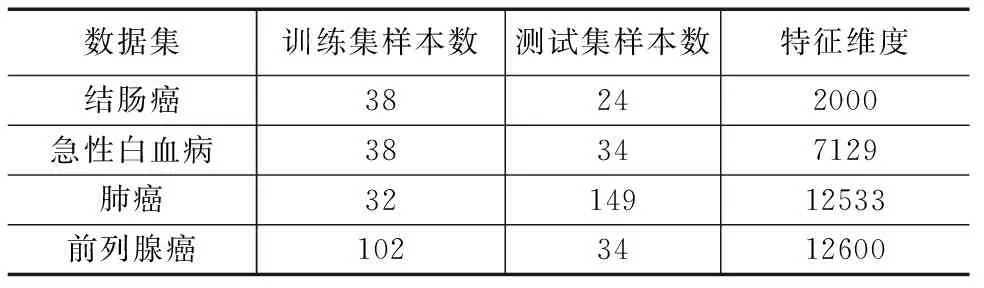
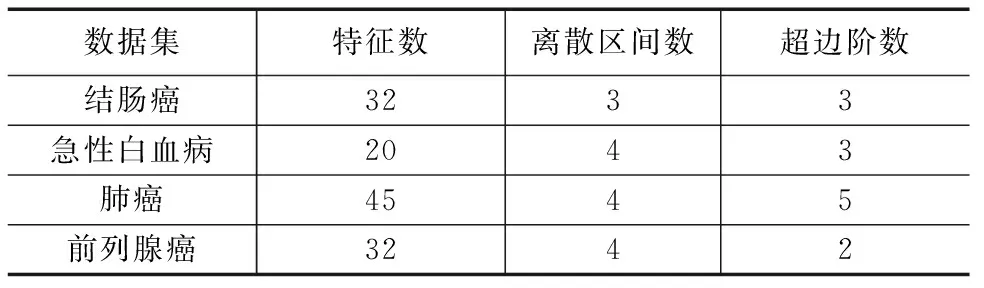
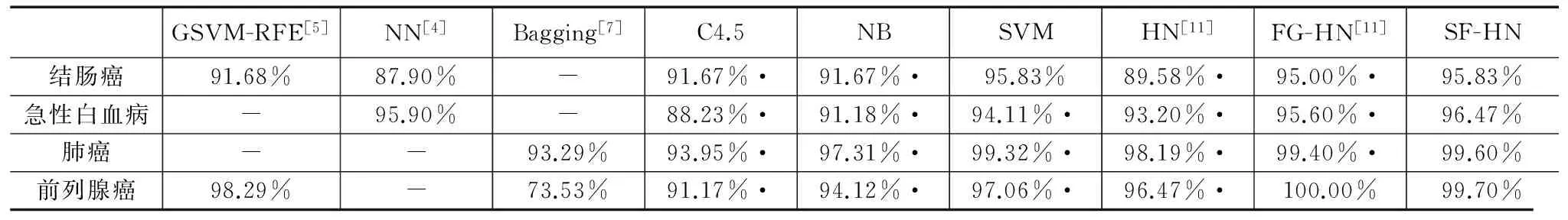
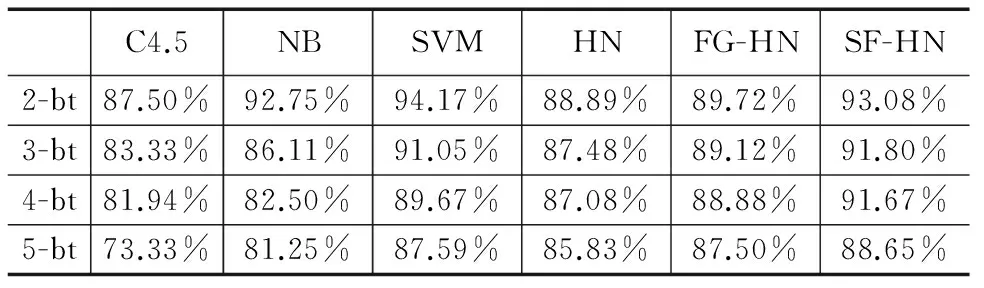
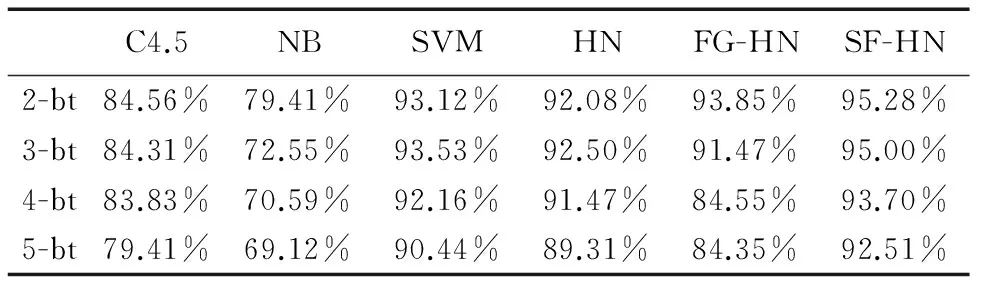
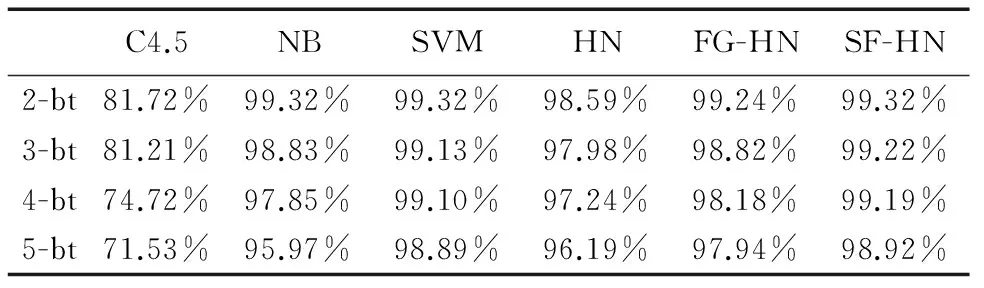
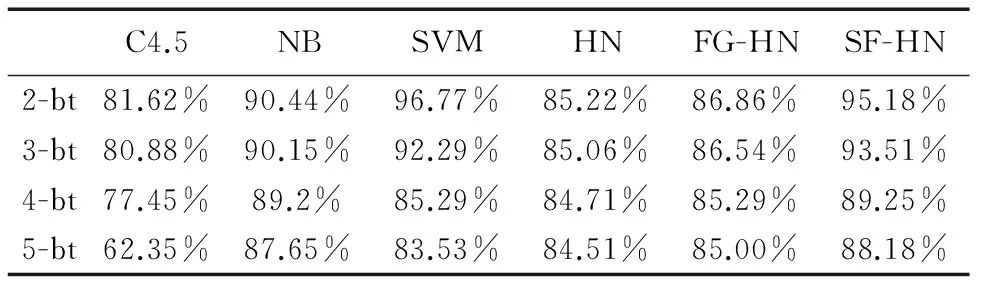
步骤5 若选择前num个子空间的SOD值均小于tsod,则将前num个子空间加入E;否则将满足SOD(T,EEi) 步骤6 若tsum>0,num←tsum,转入步骤2;否则转入步骤7. 步骤7 返回E. 3.2 子空间超边生成算法 子空间超边生成算法通过产生由训练样本映射得到的与训练集完全拟合的映射超边和通过映射超边信息确定类别的预测超边,加入样本关联信息,扩展超边的决策范围,对子空间进行超边全覆盖. 子空间超边生成算法的流程为:将子空间Ei中对应的单元格转化为超边加入到子超边簇LSi中,此时超边不包含类别信息;将训练集T在子超边簇LSi上投影,并确定至少有一个样本映射到对应单元格的超边类别;最后对剩余未知类别信息的超边进行类别预测.由于输入模式空间数据为连续分布,因此对模式空间中的数据点,其类别可由其相邻数据点的类别确定.故对每条未知类别超边,统计其相邻超边的类别,并将包含超边最多的类别赋给待预测类别超边;若不同类别包含的超边数相等,则此超边处在类别分界线上,不对其类别赋值.当无新确定类别的超边时,算法终止. 算法2 子空间超边生成算法 输入:训练集T,子空间Ei. 输出:子超边簇LSi. 步骤1LSi←Φ. 步骤2 子空间Ei中每个单元格fj转化为超边lj并加入到子超边簇LSi,其中超边的类别标签为空. 步骤3 将训练集T在子超边簇LSi上投影. 步骤4 遍历每条超边lj对应的单元格fj,若至少有一个样本映射到fj,则将该超边类别赋为映射到相应单元格中数量最多的样本类别. 步骤5 统计未知类别超边的数量ln,lt←ln. 步骤6 统计每条未知类别超边的相邻超边类别,若不同类别超边数量不等,则将包含超边数量最多的类别赋给该超边. 步骤7 统计未知类别的超边数量ln,若lt≠ln,转入步骤5;否则转入步骤8. 步骤8 返回LSi. 3.3 子空间融合算法 覆盖子空间的子超边簇既包含由训练集映射而成的超边,也包含由映射超边对未知类别超边进行预测扩展而成的超边.子超边簇中由训练集映射而成的超边是对训练集样本分布的零误差拟合,而经预测扩展而成的超边则存在拟合误差,并且不同子空间中预测超边的拟合误差不同.子空间融合算法通过融合不同子空间上的子超边簇,降低预测超边的拟合误差,进而提高模型对训练集的拟合精度.由于偏斜度低的子空间生成的超边簇具有更优的拟合效果,在融合时采用加权集成的方式,将1-SOD(T,Ei)作为子空间对应超边簇的权重. 算法3 子空间融合算法 输入:训练集T,超边簇集合LS. 输出:超边库L. 步骤1L←Φ. 步骤2 遍历每个子超边簇,根据T在每个子超边簇LSi对应子空间Ei的投影,计算SOD(T,Ei),并将LSi中每个超边的权重设为1-SOD(T,Ei). 步骤3 将赋予权重的超边簇加入到L中. 步骤4 返回L. 3.4 子空间融合超网络的演化学习 SF-HN通过将子超边簇融合为一个超边库,拟合模式空间的数据分布.因模型对子空间进行了超边全覆盖操作,无需替代操作,故采用梯度下降演化学习方法[12],通过训练集来控制超边权重的调整方向,调整模型结构,降低融合后模型对未知样本的预测误差.权重变化值Δwj计算公式为: (3) 其中,P(y*|xi)和P*(y*|xi)分别表示样本xi属于类别y*的实际概率和目标概率,y*是超网络对样本xi的分类结果,y是样本xi的真实类别,η是学习速率.I为匹配函数,当超边lj与样本xi匹配时值为1;否则值为0.子空间融合超网络的分类方法与传统超网络的流程[11]相似,唯一的区别在于估计概率时统计超边权重之和而不是数量之和. 算法4 子空间融合超网络演化学习算法 输入:训练集T,子空间数sn;超边阶数k;梯度下降演化代数iternum. 输出:超边库L. 步骤1 根据子空间划分算法,生产包含sn个子空间的子空间集合E. 步骤2 对每个子空间Ei,利用子空间超边生成算法,生成子超边簇LSi.最终得到包含sn个子超边簇的超边簇集合LS. 步骤3 对超边簇LS,利用子空间融合算法,得到初始超边库L. 步骤4t←0. 步骤5 用当前子空间融合超网络模型对训练集分类. 步骤6 对每个错分样本xi,更新与xi匹配的超边lj的权重wj=wj+Δwj,其中通过式(3)计算Δwj. 步骤7t++,若t 步骤8 返回L. 为验证子空间融合演化超网络的分类准确性和泛化性,本文采用结肠癌[2]、急性白血病[1]、肺癌[14]、前列腺癌[15]四个DNA微阵列数据集进行实验验证.数据集的具体信息如表1所示. 4.1 分类性能测试 本文采用OCDD算法[16]对输入数据进行离散化处理,采用信噪比特征基因选择方法[1]对数据进行降维处理.为了验证SF-HN的分类效果,将其与其他文献方法(GSVM-RFE[5],NN[4],Bagging[7])、传统分类方法(C4.5决策树、朴素贝叶斯(Nave Bayes,NB)、支持向量机(Support Vector Machine,SVM))以及HN和FG-HN进行对比.本文的所有实验结果为20次实验的平均值.SF-HN的参数通过训练集5折交叉验证来确定,其参数设置如表2所示.HN和FG-HN采用文献[11]中的实验参数设定,分类算法C4.5、NB、SVM采用Weka机器学习开源项目提供的算法(http://www.cs.waikato.ac.nz/ml/weka/),其输入数据的特征维度与FG-HN相同.此外,本文通过t-检验来测试SF-HN在统计学上是否显著优于C4.5、NB、SVM、HN和FG-HN方法. 通过对完整的训练集进行学习,然后对独立测试集进行测试,所得结果如表3所示.在表3中,“·”表示SF-HN在p<0.01的水平下显著优于对比方法.相对于其它对比分类算法,SF-HN具有较好的分类性能和显著性优势.这主要是由于SF-HN在空间中进行超边覆盖,增加了模型的信息熵,从而更有效地拟合输入模式空间中的数据分布. 表1 数据集信息表 表2 SF-HN的参数设置 表3 不同方法对4个DNA微阵列数据集的测试集分类结果 4.2 泛化性能测试 泛化能力表示分类器通过对训练集的学习,对未知样本的预测能力[17].但在据作者所知的文献中,还缺乏公认的对分类器泛化性能评价的定量指标.机器学习领域通常认为泛化性能好的算法在小样本训练集下仍可获得较高的分类精度.本文采用部分替代整体思想进行泛化性能测试的实验设计,通过拆分原始训练集获得小样本训练集,进而验证不同分类方法在小样本训练集下的泛化性能.泛化性测试的主要流程为:首先是将训练集按原正负类别的比例平均分为n份;之后利用拆分后的每一份数据分别训练分类器并对独立测试集进行测试.对得到的n个独立测试集测试结果求取平均,作为分类器泛化能力评价指标.在本文中,训练集平均划分为n份以n-bt表示. 泛化性能测试中对训练集进行拆分后,训练集中样本数量减少,离散区间数过大将导致数据中同类别样本间的关联概率降低;而阶数过大的超边很难与样本进行匹配.因此对四个数据集,特征选择数设为32,特征最大离散区间数为设为3,HN、FG-HN和SF-HN的阶数分别设定为5、4、3.对每个数据集,采取2-bt、3-bt、4-bt、5-bt泛化性能实验. 泛化性测试结果如表4~7所示.相对于其他方法,在3-bt、4-bt、5-bt设定下SF-HN具有更高的泛化性能.这是因为在分类器的学习过程中,SF-HN通过对超边类别的预测,对子空间进行超边覆盖,在本质上类似于虚拟样本生成,通过增加样本的数量,实现了对数据分布的更优拟合.而在2-bt时,由于某些数据集中正负类别的界限较宽,SVM能够发挥更优的性能.当n-bt中的n增大时,所有方法对独立测试集的识别率随之降低.这是因为随着训练集样本数的减少,关于模式空间描述的信息量相应减少,从而导致分类器对模式空间的描述可信度降低.然而相对其它方法,SF-HN下降趋势最缓慢.这是因为SF-HN通过子超边簇对子空间进行全覆盖,增加了超边对未知样本的匹配概率,避免了超边对其生成样本的过度依赖,不会出现对训练集的过拟合,在小样本数据中具有较高的优势. 表4 结肠癌数据集不同分类器泛化性能测试结果 表5 急性白血病数据集不同分类器泛化性能测试结果 表6 肺癌数据集不同分类器泛化性能测试结果 表7 前列腺癌数据集不同分类器泛化性能测试结果 本文提出了一种子空间融合演化超网络模型.通过将子空间概念引入到演化超网络中,把超边包含的特征看作是输入模式空间的子空间,在子空间进行超边覆盖,减弱了模型对超网络初始化效果的依赖;同时通过超边子空间覆盖和子空间融合,加入样本间的关联信息,提高了模型对未知样本的分类效果和泛化性能.本文根据部分替代整体思想设计了分类器泛化性能测试实验,并提出了评价分类器泛化性能的方法.通过四个DNA微阵列数据集下的对比实验表明,本文方法的识别率和在小样本训练集下的泛化能力均优于其他传统模式识别方法. [1]Golub T R,Slonim D K,Tamayo P,et al.Molecular classification of cancer:class discovery and class prediction by gene expression monitoring[J].Science,1999,286(5439):531-537. [2]Alon U,Barkai N,Notterman D,et al.Broad patterns of gene expression revealed by clustering analysis of tumor and normal colon tissues probed by oligonucleotide arrays[J].Proceedings of the National Academy of Sciences,1999,96(12):6745-6750. [3]Reunanen J.Overfitting in making comparisons between variable selection methods[J].Journal of Machine Learning Research,2003,3:1371-1382. [4]Cho S B,Won H.Cancer classification using ensemble of neural networks with multiple significant gene subsets[J].Applied Intelligence,2007,26(3):243-250. [5]Mundra P A,Rajapakse J C.SVM-RFE with MRMR filter for gene selection[J].IEEE Transactions on Nanobioscience,2010,9(1):31-37. [6]Prasartvit T,Banharnsakun A,Kaewkamnerdpong B,et al.Reducing bioinformatics data dimension with ABC-kNN[J].Neurocomputing,2013,116:367-381. [7]Tan A C,Gilbert D.Ensemble machine learning on gene expression data for cancer classification[J].Applied Bioinformatics,2003,2(3 suppl):75-83. [8]Zhang B T.Hypernetworks:a molecular evolutionary architecture for cognitive learning and memory[J].IEEE Computational Intelligence Magazine,2008,3(3):49-63. [9]Kim S J,Ha J W,Zhang B T.Bayesian evolutionary hypergraph learning for predicting cancer clinical outcomes[J].Journal of Biomedical Informatics,2014,49(6):101-111. [10]Park C H,Kim S J,Kim S,et al.Use of evolutionary hypernetworks for mining prostate cancer data[A].Proceedings of the 8th International Symposium on Advanced Intelligent Systems[C].Springer,2007.702-706. [11]王进,张军,胡白帆.结合最优类别信息离散的细粒度超网络微阵列数据分类[J].上海交通大学学报,2013,47(12):1856-1862. Wang Jin,Zhang Jun,Hu Bai-fan.Optimal class-dependent discretization-based fine-grain hypernetworks for classification of microarray data[J].Journal of Shanghai Jiaotong University,2013,47(12):1856-1862.(in Chinese) [12]Wang J,Huang P L,Sun K W,et al.Ensemble of cost-sensitive hypernetwork for class-imbalance learning[A].Proceedings of IEEE International Conference on Systems,Man,and Cybernetics[C].IEEE,2013.1883-1888. [13]孙焕良,鲍玉斌,于戈.一种基于划分的孤立点检测算法[J].软件学报,2006,17(5):1009-1016. Sun Huan-liang,Bao Yu-bin,Yu Ge.An algorithm based on partition for outlier detection[J].Journal of Software,2006,17(5):1009-1016.(in Chinese) [14]Gordon G J,Jensen R V,Hsiao L L,et al.Translation of microarray data into clinically relevant cancer diagnostic tests using gene expression ratios in lung cancer and mesothelioma[J].Cancer research,2002,62(17):4963-4967. [15]Singh D,Febbo P G,Ross K,et al.Gene expression correlates of clinical prostate cancer behavior[J].Cancer Cell,2002,1(2):203-209. [16]Liu L,Wong K C,Wang Y.A global optimal algorithm for class-dependent discretization of continuous data[J].Intelligent Data Analysis,2004,8(2):151-170. [17]张海,徐宗本.学习理论综述(I):稳定性与泛化性[J].工程数学学报,2008,25(1):1-9. Zhang Hai,Xu Zong-ben.A survey on learning theory (I):stability and generalization[J].Chinese Journal of Engineering Mathematics,2008,25(1):1-9.(in Chinese) 王 进 男,1979年1月出生于重庆,教授.主要研究方向为数据挖掘、机器学习. E-mail:wangjin@cqupt.edu.cn 刘 彬(通信作者) 男,1989年11月出生于河北保定,硕士研究生.主要研究方向为数据挖掘. E-mail:nanfeizhilu@163.com Classification of Microarray Data Using Evolutionary Hypernetworks with Subspace Fusion WANG Jin,LIU Bin,ZHANG Jun,CHEN Qiao-song,DENG Xin (ChongqingKeyLaboratoryofComputationalIntelligence,ChongqingUniversityofPostsandTelecommunications,Chongqing400065,China) In order to solve the over-fitting problem of the traditional pattern recognition approaches under the DNA microarray data with small train samples,a subspace fusion-based evolutionary hypernetwork model is proposed in this paper.With the methods of subspace division,hyperedge coverage,and subspace fusion,the proposed scheme reduces the dependence on the initialization,decreases the fitting error of the data space,and enhances the generalization ability of the evolutionary hypernetwork.The experimental results on four DNA microarray datasets show that the proposed model achieves higher classification accuracy and stronger generalization ability than other compared traditional pattern recognition method. pattern recognition;microarray data classification;evolutionary hypernetwork;subspace;over-fitting 2015-03-11; 2015-06-30;责任编辑:李勇锋 国家自然科学基金(No.61203308,No.61403054);重庆教委科学技术研究项目(自然科学类)(No.KJ1400436);重庆市基础与前沿研究计划项目(No.cstc2014jcyjA40001) TP39 A 0372-2112 (2016)10-2308-06 ��学报URL:http://www.ejournal.org.cn 10.3969/j.issn.0372-2112.2016.10.0044 实验结果与分析
5 结论