全局调控因子CodY在益生菌中的研究进展
冯佳,吴昊,王斌斌,乔建军
(天津大学 化工学院,制药工程系,系统生物工程教育部重点实验室,天津,300072)
全局调控因子CodY在益生菌中的研究进展
冯佳,吴昊,王斌斌,乔建军*
(天津大学 化工学院,制药工程系,系统生物工程教育部重点实验室,天津,300072)
CodY是一个具有一定的保守性的全局转录调控因子,普遍存在于低G-C含量的革兰氏阳性菌中。对枯草芽孢杆菌、乳酸乳球菌等益生菌的研究表明,CodY对菌株的碳代谢、氮代谢、胁迫响应、营养物质的吸收以及自然感受态等具有重要的调控作用。文中对益生菌中调控因子CodY的结构、调控机制和结合位点进行综述,并以枯草芽孢杆菌和乳酸乳球菌等为例介绍CodY的调控作用,为进一步改造菌株,探索CodY潜在的调控功能,以及为其在食品发酵中的应用提供参考。
CodY;调控因子; 革兰氏阳性菌;益生菌
乳酸乳球菌、嗜热链球菌和枯草芽孢杆菌都属于益生菌,具有不产生毒素和致热致敏蛋白、非致病性等特点,可用于食品及发酵工业中。乳酸乳球菌(Lactococcuslactis)属于乳酸菌的乳球菌属,可以产生一种多肽物质——乳酸链球菌素(Nisin)[1],是被FDA唯一公认的一种高效的天然食品防腐剂,并广泛应用于肉制品、鱼类制品、果汁饮料等食物的保鲜和防腐中。Nisin进入人体消化道后,会被蛋白酶降解成游离氨基酸并维持正常的菌落环境。但是乳酸乳球菌在发酵过程中不断产生乳酸,导致pH值降低,不利于Nisin的产生[2],所以乳酸乳球菌的耐酸调控机制成为研究热点。枯草芽孢杆菌是重要的工业生产菌株和遗传改造受体菌株,在工业上广泛用来生产α-淀粉酶、中性蛋白酶、纳豆以及肌苷发酵;在植物病原菌生物防治领域、农业生产、水产养殖、污水处理中也有重要的作用。嗜热链球菌来源于牛乳中,与瑞士乳杆菌等混合发酵生产酸奶和奶酪,可以借助自身合成β-半乳糖苷酶高效利用乳糖为自己的生长提供能量,并且帮助不耐受乳糖的人消化乳糖,但自身合成的蛋白酶的活性很弱[3],只能利用氨基酸和寡肽来补充氮源,所以氨基酸的合成代谢和寡肽的高效摄取对于嗜热链球菌的生长非常重要。
全局转录调控因子CodY普遍存在于低G-C含量的革兰氏阳性菌中,在面对环境变化时,通过改变转录速率调控靶基因的表达水平,对细胞内10%以上的基因进行转录调控,从而对细胞代谢做出迅速的调整,构成一个巨大的代谢调控网络。通过不同的作用机制调控菌体内不同基因的转录,帮助细胞适应营养匮乏的环境。目前,CodY的调控作用主要集中在碳代谢、氮代谢以及自然感受态等过程。本文对全局调控因子CodY的结构、作用机制、结合位点以及在乳酸乳球菌、枯草芽孢杆菌和嗜热链球菌的生物功能的研究进展进行了详细的阐述,为深入了解细菌体内的调控网络以及解决食品和发酵工业中的问题提供有效的参考。
1 全局调控因子CodY的结构
目前研究较为透彻的是枯草芽孢杆菌CodY 的结构,它是1个具有259个氨基酸残基的二聚体蛋白,序列高度保守[4],其结构由C端和N端2部分组成。N端为GAF结构域,是鸟嘌呤三磷酸(Guanosine-5′- Triphosphate,GTP)和分支氨基酸(Branched Chain Amino Acids,BCAAs)2个效应分子的结合位点,这个结构广泛存在于环核苷酸磷酸二酯酶和腺苷酸环化酶中。当BCAAs作用于CodY时,CodY的构型转变并再次折叠,从而形成一个疏水口袋,这一疏水口袋既是BCAAs的结合位点,同时构型的转变会传递给C端,激活CodY和DNA的相互作用。在这一过程中,CodY的核心部分及二聚作用界面不发生变化[5]。C端含1个螺旋-转角-螺旋(“HTH”)结构域,与DNA启动子区域结合,抑制靶基因的转录。每个螺旋含有20个氨基酸且成120角,这种结构有利于螺旋和DNA的相互作用。
通过对CodY的晶体结构的研究,STENZ等[6]归纳出较为简单易懂的平面结构图,如图1,G1、G3和G4是GTP结合位点。通过晶体结构和序列比对发现精氨酸(Arg8),谷氨酸(Glu144),谷氨酰胺(Gln15)和苏氨酸(Thr148)对于CodY二聚体的形成具有关键作用[4]。利用定点诱变和DNA结合实验发现丙氨酸(Ala207),天冬氨酸(Asp208),精氨酸(Arg214)和丝氨酸(Ser215)对C端特异性与DNA结合具有重要作用[7]。
在长期的自然进化中,CodY蛋白序列在不同菌中会发生变异,导致CodY结合不同DNA,改变了CodY调控的基因[8]。
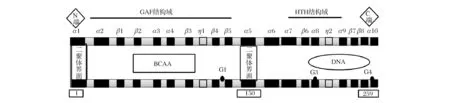
图1 枯草芽孢杆菌CodY二级平面结构图Fig.1 Structure of Bacillus subtilis CodY
2 CodY的调控机制和结合位点
2.1 CodY的效应分子
全局转录调控因子CodY有2个效应分子[9],分别是 GTP[10-11]和BCAAs[5,12-13],其中BCAAs包括异亮氨酸,亮氨酸和缬氨酸,它们的存在能提高CodY与DNA结合的活性。GTP和BCAAs既可协调作用于CodY,也可单独作用于CodY。在不同的菌株中,效应分子对CodY的作用是不同的。例如,在枯草芽孢杆菌中,CodY同时受到GTP和BCAAs的作用,而在肺炎链球菌和乳酸乳球菌中,CodY的活性只受到BCAAs的作用,与GTP没有关系[12],当细胞处于营养丰富的状态下,受CodY调控抑制的基因都处于静止状态,此时高浓度的GTP可以激活CodY的抑制作用[10];在培养基里营养物质发生变化时,细菌会改变CodY调控的靶基因的表达模式,在没有BCAAs结合的情况下CodY会丧失调控大部分依赖于CodY表达的基因的能力。在枯草芽孢杆菌中,由于碳源、氮源或者磷源的缺乏而限制菌体生长时,GTP的浓度下降,CodY就会失去抑制活性,被抑制的基因就会被转录激活,同时,由于胞内BCAAs的浓度也下降,进一步增强了下游基因的表达[9]。
2.2 效应分子的来源和CodY的相互作用
在细菌体内,GTP的来源有两条途径:(1)从细菌生存的环境中获取(2)通过合成途径从5-磷酸核糖经过purCLFMNH和purDEK进行合成。当GTP的水平降低时,会促进一个特殊的反应发生——细胞的严谨反应[14]。严谨反应是广泛应用于细菌的压力响应系统,在营养受到限制时,大量的空载tRNA激活核糖体上的RelA蛋白,由GTP和ATP合成ppGpp或pppGpp调节代谢活动,从而引发细菌的严谨反应,如图2。有研究表明ppGpp或pppGpp对GTP水平的平衡有着关键作用,从而影响CodY调控氨基酸代谢途径。这个过程中,GTP将全局调控因子CodY和细菌严谨反应联系在一起进行氨基酸的代谢调控。
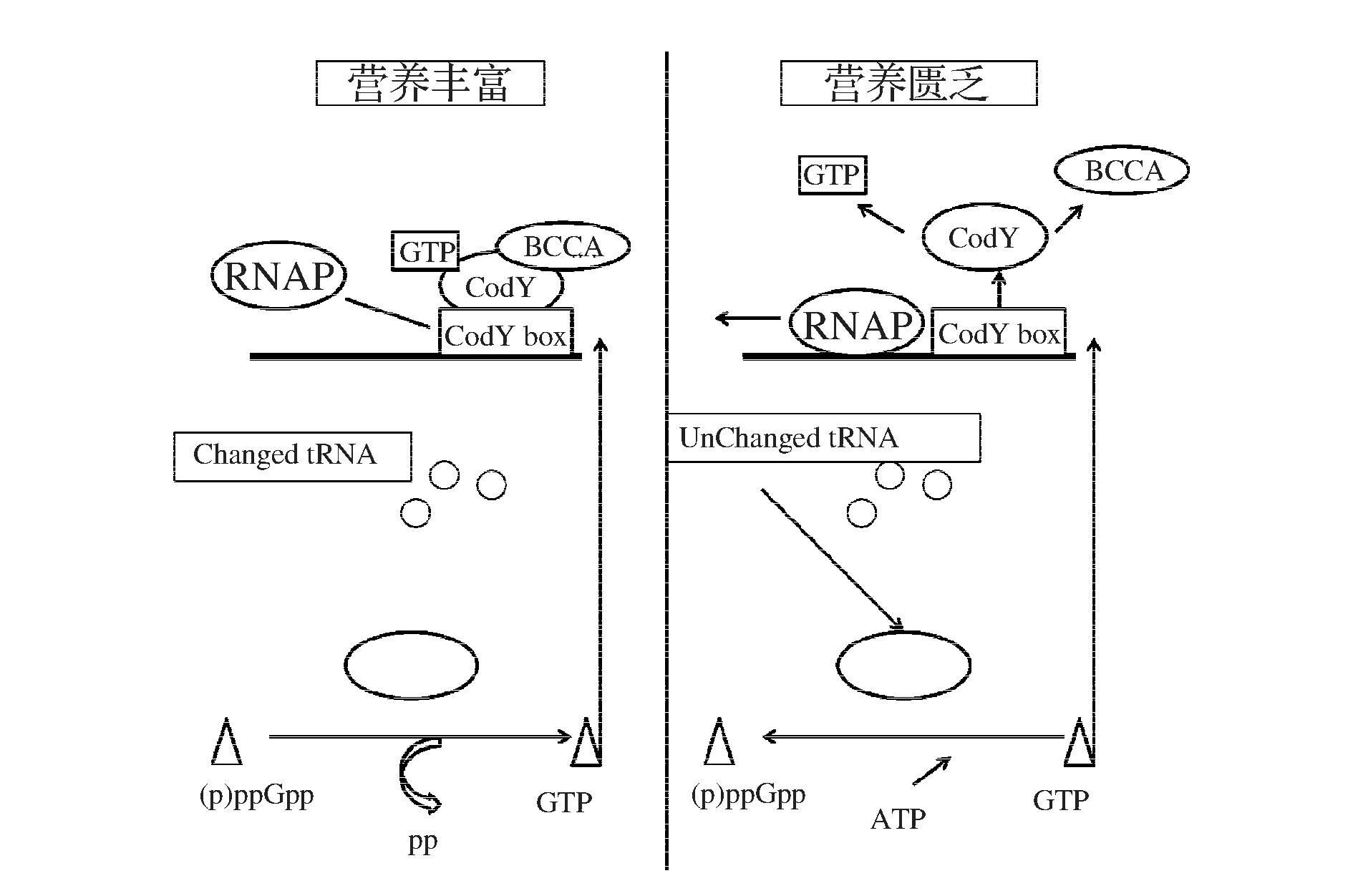
图2 细胞的严谨反应示意图Fig.2 Rigorous reaction of cells
BCAAs既可以通过ilvBHC和leuABCD合成途径进行自身合成,也可以通过吸收并利用环境中的氮源加工得到。研究发现CodY对BCAAs的合成途径也具有调控作用,而BCAAs作为CodY的效应分子,2者相互作用完成CodY对细菌生长的调控作用。
2.3 CodY的作用机制和结合位点
研究发现,CodY至少通过4种不同的机制调控转录过程[15]:(1)通过结合到启动子区域附近发挥正调控,(2)通过结合到启动子区域附近发挥负调控,(3)通过充当RNA聚合酶的路障发挥负调控作用[16],(4)通过干扰正调控因子的作用发挥负调控作用。
研究发现在乳酸乳球菌中,CodY蛋白的结合位点必须是一个15 bp的标准的共同回文序列(AATTTTCNGAAAATT)[17],称为CodY-box,位于目标启动子区域-35区附近。HENGST等[18]发现CodY-box普遍存在于葡萄球菌、李斯特菌、芽孢杆菌和链球菌中,并通过体外足迹法实验证实了这个回文序列在其他革兰氏阳性菌中的保守性。在枯草芽孢杆菌中,CodY调节操纵子区包含重叠的CodY结合位点[19],这2个独立作用的CodY高亲和位点可以抑制基因ybgE的表达,其中位于转录起始位点下游80bp处的位点在调控中发挥主要作用[20]。CodY还可以通过控制sigma因子sigD的表达和活性来直接调控细胞运动和成链[21]。研究表明,CodY可以结合于sigD的第1个基因上游位置的PD3和Pfla/che启动子区,CodY的敲除导致sigD累积量和活性急剧增加,并使得非链状的细胞数量从15%增加到75%,这表明codY是sigD的1个重要的翻译后调控因子。通过对枯草芽孢杆菌突变体的全基因组分析表明,表达受到影响的基因都没有CodY结合位点,而有CodY结合位点的基因的表达在codY缺失菌株中不受影响[15]。这些发现也潜在的解释了CodY通过控制其他调控因子来间接调控基因以及和其他蛋白共同调控相同基因的复杂调控模式。
3 CodY的生物学功能
全局调控因子CodY是作为枯草芽孢杆菌中寡肽通透酶(dppABCDE)操纵子的抑制物被发现的[22],随后发现CodY对芽孢的形成和稳定期早期基因的表达有重要的调控作用[23]。DNA微阵列、染色质免疫沉淀与微阵列(ChIP-chip)、体外DNA结合实验、大规模并行测序(IDAP-Seq)和转录组测序分析(RNA测序)等表明, CodY直接或者间接调控枯草芽孢杆菌中200多个基因的表达[15],调控作用主要表现为基因表达抑制。通过对CodY调控细菌的碳代谢、氮代谢和自然感受态等研究,可以通过基因编辑技术改造CodY,得到基因工程改造菌株,用于食品和发酵工业中。下面以CodY在枯草芽孢杆菌、乳酸乳球菌和嗜热链球菌中的调控作用对CodY的生物学功能进行详细介绍。
3.1 CodY对碳和能量代谢的调控作用
碳代谢是细菌体内能量来源的主要途径,当细胞处于胁迫环境中,细菌会改变代谢方式来增加ATP应对胁迫环境,而CodY作为全局调控因子,对菌体碳代谢的过程具有重要的调控作用。在枯草芽孢杆菌中,调控因子CcpA主要对碳代谢途径具有调控作用,并控制了菌体内10%的基因的表达[24]。当枯草芽孢杆菌处于营养丰富的情况下,ROBERT等[25]发现乳酸激酶基因ackA受CcpA和CodY的共同正调控,且作用是累积的,CodY通过和CcpA共同作用于乳酸激酶和乳酸脱氢酶使丙酮酸转化为乳酸或乙酸,体外实验中CodY结合到ackA启动子区域的两个位点,并激活ackA转录,不同于负调控,影响CodY活性的只有BCAAs一个效应分子,具体调控机制见图3。
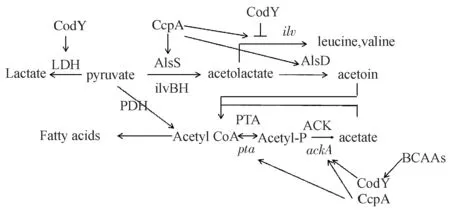
图3 CodY和CcpA共同调控作用机制示意图Fig.3 Common regulation mechanism of CodY and CcpA(AlsS and IlvBH: α-acetolactate synthetase; AlsD: α-acetolactate dehydrogenase; PDH: pyruvate dehydrogenase; PTA, phosphotransacetylase; ACK, acetate kinase; LDH: lactate dehydrogenase. Bold lines with arrow heads indicate positive regulation by CcpA or CodY; bold blunted lines indicate repression of gene expression by CcpA or CodY.)
嗜热链球菌的碳代谢过程主要是乳糖代谢过程,通过β-半乳糖苷酶以及半乳糖代谢途径(Leloir途径)利用乳制品中的乳糖,生成UDP-半乳糖和1-磷酸葡萄糖,后者在磷酸变位酶的催化下生成6-磷酸葡萄糖。研究发现,缺失codY的枯草芽孢杆菌中磷酸变位酶STND_1003、STND_1004和STND_1007的转录显著上调,从而更加充分地利用半乳糖产生乳酸,因此调控因子CodY对半乳糖的调控作用是通过对磷酸变位酶的转录调控实现的。
3.2 CodY对氮代谢的调控作用
全局调控因子CodY在氮代谢方面有重要的调控作用,包括寡肽转运、氨基酸降解途径、胞外蛋白酶以及支链氨基酸的生物合成。
在乳酸乳球菌里,全局调控因子CodY作为蛋白水解的中央调控因子被发现,作为一个全局调控因子[18],对一系列的细胞功能的表达都具有调控作用,包括营养物质运输、大分子降解、氨基酸代谢和肽代谢等[26]。与枯草芽孢杆菌不同的是,乳酸乳球菌的CodY只受BCAAs这一个效应分子的影响,而BCAAs的浓度决定了CodY的调控作用,所以BCAAs对菌体的生长非常重要。研究发现CodY对细胞内氨基酸的合成也具有负调控作用,包括缬氨酸、天冬氨酸、组氨酸、赖氨酸、丝氨酸[26]和谷氨酸[27]的合成。
乳酸乳球菌CodY可以抑制包括寡肽转运蛋白(oppopt)、肽酶(pepC,pepN)[28]和转氨酶(araT,bcaT)[29]相关基因的表达。其中转氨酶(bcaT)在BCAAs的合成代谢中发挥关键作用,支链酮酸在转氨酶的作用下可以合成BCAAs,促进CodY的调控作用。作为枯草芽孢杆菌中的通透酶BcaP的远亲,乳酸乳球菌中的通透酶BcaP 除了对BCAAs有作用外还对蛋氨酸有运输作用[30]。
BELITSKY等[31]发现在基本培养基里枯草芽孢杆菌内在的CodY效应分子池足够去调控一些基因,但是在存在BCAAs和其他氨基酸的培养基里,CodY显示出最高的活性。因此,BCAAs通透酶的活性和BCAAs摄取的效率对CodY的激活起着关键的作用。研究表明,有3个通透酶(BcaP、BraB和BrnQ)作用于异亮氨酸和缬氨酸的高效摄取过程,同时失活这3个通透酶时对CodY活性的影响。更多的研究表明,至少有一个通透酶作用于亮氨酸摄取过程和异亮氨酸及缬氨酸的运输过程。编码寡肽通透酶的基因dtpT,被ScoC直接负调控,而CodY通过抑制ScoC间接激活dtpT基因的表达[32]。同时opp操纵子也负责编码一个寡肽通透酶,与孢子的形成和基因感受态相关。研究证明opp直接被CodY和ScoC共同抑制,而不会被CodY或者ScoC单独影响,这种联合作用保证了当其中一个调控因子失去活性的时候保持对opp的抑制作用,但是作用会减弱[32]。
AprE和NprE是枯草芽孢杆菌中两个重要的胞外蛋白酶,占总胞外蛋白酶活性的95%以上,它们是由几个多效转录因子共同调控表达的,包括AbrB、DegU,、ScoC和 SinR。在营养丰富的培养基中, AprE和NprE仅在指数期后期大量表达。CodY是另一个调控因子ScoC的抑制子,在缺失codY的菌株中,ScoC的过表达很少或不会对AprE和NprE产生去阻遏作用;而在含有CodY的菌株中,缺失scoC对AprE和NprE的表达没有影响,所以CodY通过限制第二抑制子ScoC的合成间接正调控AprE和NprE[33]。GIULIA等[34]通过构建vpr-lacZ或者mpr-lacZ融合菌株使CodY结合位点失活,发现CodY对枯草芽孢杆菌中的胞外蛋白酶(Vpr和Mpr)的表达具有直接负调控作用,同时vpr的启动子不仅受CodY调控同时也是sigma H依赖型启动子。在缺失codY的菌株中,Vpr蛋白表达量显著提高。同时,枯草芽孢杆菌中的CodY对编码肽转运蛋白(Dpp和App)、胞内肽酶(DppA)以及胞内蛋白酶(IspA)的基因都具有负调控作用[35]。
在嗜热链球菌中,调控因子CodY对谷氨酸脱氢酶(gdhA)、氨基酸合成和转运和ABC转运蛋白的转录具有调控作用[38]。
3.3 CodY对胁迫响应的调控作用
在枯草芽孢杆菌中,Com分泌途径是蛋白质分泌途径之一,而Com家族和自然感受态相关,CodY对comK具有负调控作用[36]。研究发现CodY的结合位点也位于乳酸乳球菌中自然感受态相关蛋白基因(coiA、comEA、comEC和comGC)的上游[37],说明在细菌面对胁迫环境时,不仅通过改变碳、氮代谢来应对胁迫,也通过CodY对自然感受相关基因的调控,提高菌体对外源DNA的摄入,来获得有利于应对胁迫环境的基因,使菌株更好地生长。
ONUR等[44]利用凝胶阻滞实验通过碱基替换证实了乳酸乳球菌中CodY-box与CodY的相关性,发现CodY基序位于转录调节基因、细胞分裂机制相关基因(ddl、murCDFG、mutM、papL、parC、r/minF、rexB、truA和yacB)和嘌呤代谢过程相关基因(guaC和purHL)的上游。这说明在菌体面对饥饿环境时,为了适应恶劣的环境,CodY对于细胞的严谨反应和嘌呤代谢等过程具有重要的调控过程。在嗜热链球菌中,CodY可以抑制双组分系统(TCS01)的转录,并调控与胁迫相关基因的转录,如分子伴侣蛋白DnaK以及和氧胁迫、渗透压胁迫相关的基因[38]。
3.4 CodY在一些毒力菌株中的调控作用
调控因子CodY对一些有毒力的菌株也具有调控作用。LOBEL等[39]发现CodY可以激活李斯特菌的鞭毛生物合成相关基因的转录,促进和哺乳动物细胞的细菌性黏附,并首次验证了CodY对细菌的运动性、抗胁迫、毒力相关功能和适应性等生物代谢途径的调控作用。TAYLOR等[40]在小鼠实验中发现relA突变会引起毒力的丧失,缺失codY后减弱了relA突变带来的影响,使毒力功能恢复,说明至少有一个毒力因子是被CodY调控的,而relA突变丧失了减弱GTP浓度使CodY活性降低的能力[41]。在梭状芽胞杆菌、炭疽芽孢杆菌和酿脓链球菌中,调控因子CodY可以间接地激活毒力因子相关基因的转录[42-44]。在蜡样芽孢杆菌中CodY是调控毒力基因的重要因子,同时也是ces操纵子的阻遏蛋白[45]。在金黄色葡萄球菌中,当菌体处于极度营养缺乏状态时,才会利用codY去限制对菌株的损害,缺失codY的菌株会发生毒力基因,蛋白酶等的去阻遏作用,激活下游基因的表达[46]。
在苏云金芽孢杆菌中,调控因子CodY也可以调控苏云金芽孢杆菌孢子的形成、聚-β-羟基丁酸酯的生物合成、菌体生长、基因的自然感受和翻译过程。QI等[47]通过细菌单杂交和凝胶迁移实验发现苏云金芽孢杆菌中自然感受基因comEA基因含有一个保守的10bp大小的CodY结合位点(TTTCAGAAAA),这说明CodY可以直接调控“com”基因。研究发现CodY通过控制调控因子PlcR的信号肽PapR的输入对PlcR调控的毒力基因进行调控,同时发现OppA不是PapR识别的唯一底物结合蛋白,还有其他类似于OppA的蛋白对信号肽进行运输[48]。
4 总结与展望
全局转录调控因子CodY具有非常重要的调控作用,可正向和负向对相关基因的转录进行直接调控或间接调控,从而控制细菌的生长活动。GTP和BCAAs作为CodY的两个效应分子,可以帮助细胞感受营养和能量状态,从而促进CodY对基因表达的调控作用。通过和这两个效应分子结合,CodY发生二聚作用并被激活,从而结合到基因启动子区域的一段共同回文序列上发挥调控作用。作为全局调控因子,CodY对细菌的碳代谢、氮代谢、支链氨基酸代谢、营养物质的吸收、运动功能、趋药性、致病性、大分子的降解、芽孢形成、抗生素的合成、毒力,环境适应以及自然感受态等相关基因都具有调控作用。
细菌体内的CodY和其他调控因子共同发挥作用组成了细菌体内的联合调控网络,如全局调控因子AbrB,基因citB和citZ的抑制子CcpC,碳代谢的调控因子CcpA[25],以及自然感受相关基因ComK[49]。然而,CodY和其他调控因子之间形成的调控网络还需要进一步探究。CodY对CRISPR/Cas系统也具有调节作用[38],但是相关机制还需要进一步探究。由于CodY对菌株自然感受相关基因也具有调控作用,所以通过研究CodY和自然感受之间的调控机制对构建耐受性菌株也具有一定的价值。同时通过对CodY对毒力因子的调控作用机制研究,为定向改造毒力菌株也提供了一定的参考。CodY的深入研究有助于全面了解细菌在面对营养匮乏环境时的作用机制,为利用基因工程改造技术获得适用于食品和发酵工业中的菌株奠定基础。
[1] LI Yuan-li,LU Xin,CHEN Xiao-hong,et al.Lactic acid bacteria as functional starter cultures for the food fermentation industry[J].China Dairy Industry,2006,15(2):67-78.
[2] HUTKINS R W,NANNEN N L.pH homeostasis in lactic acid bacteria[J].Journal of Dairy Science,1993,76(8): 2 354-2 365.
[3] SHAHBAL S,HEMME D,DESMAZEAUD M.High cell wall-associated proteinase activity of someStreptococcusthermophilusstrains (H-strains) correlated with a high acidification rate in milk[J].Le Lait,1991,71(3):351-357.
[4] TOBIAS G,CHRISTIANE W.Intersection of the stringent response and the CodY regulon in low GC Gram-positive bacteria[J].International Journal of Medical Microbiology Ijmm,2013,304(2):150-155.
[5] LING B,MIN S, BI S,et al.Molecular dynamics simulations of isoleucine-release pathway in GAF domain of N-CodY fromBacillussubtilis[J].Journal of Molecular Graphics & Modelling,2013,44(7):232-240.
[6] LUDWIG S,PATRICE F,KATRINE W,et al.The CodY pleiotropic repressor controls virulence in gram-positive pathogens[J].Fems Immunology & Medical Microbiology,2011,62(2):123-139.
[7] JOSEPH P,RATNAYAKE-LECAMWASAM M,SONENSHEIN A L.A Region ofBacillussubtilisCodY protein required for interaction with DNA[J].Journal of Bacteriology,2005,187(12):4 127-4 139.
[8] IRMA L C,SARATH CHANDRA J,JULIO C V.Bacterial regulatory networks are extremely flexible in evolution[J].Nucleic Acids Research,2006,34(12):3 434-3 445.
[9] SHIVERS R P,SONENSHEIN A L.Activation of the Bacillus subtilis global regulator CodY by direct interaction with branched-chain amino acids[J].Molecular Microbiology,2004,53(2):599-611.
[10] KRIEL A,BRINSMADE S R,TSE J L,et al.GTP dysregulation inBacillussubtiliscells lacking (p)ppGpp results in phenotypic amino acid auxotrophy and failure to adapt to nutrient downshift and regulate biosynthesis genes[J].Communications in Agricultural & Applied Biological Sciences,2013,196(1):189-201.
[11] BRINSMADE S R,SONENSHEIN A L.Dissecting complex metabolic integration provides direct genetic evidence for CodY activation by guanine nucleotides[J].Journal of Bacteriology,2011,193(20):5 637-5 648.
[12] PETRANOVIC D,GUEDON E B,DELORME C,et al.Intracellular effectors regulating the activity of theLactococcuslactisCodY pleiotropic transcription regulator[J].Molecular Microbiology,2004,53(2):613-621.
[13] DEN HENGST C D,CURLEY P,LARSEN R,et al.Probing direct interactions between CodY and the oppD promoter ofLactococcuslactis[J].Journal of Bacteriology,2005,187(2):512-521.
[14] ABABNEH Q O,HERMAN J K.RelA inhibitsBacillussubtilismotility and chaining[J].Journal of Bacteriology,2015,197(1):128-137.
[15] BELITSKY B R,SONENSHEIN A L.Genome-wide identification ofBacillussubtilisCodY-binding sites at single-nucleotide resolution[J].Proceedings of the National Academy of Sciences,2013,110(17):7 026-7 031.
[16] BELITSKY B R,SONENSHEIN A L.Roadblock repression of transcription by Bacillus subtilis CodY[J].Journal of Molecular Biology,2011,411(4):729-743.
[17] BELITSKY B R,SONENSHEIN A L.Genetic and biochemical analysis of CodY-binding sites inBacillussubtilis[J].Journal of Bacteriology,2008,190(4):1 224-1 236.
[18] DEN HENGST C D,VAN HIJUM S A,GEURTS J M,et al.TheLactococcuslactisCodY regulon: identification of a conserved cis-regulatory element[J].Journal of Biological Chemistry,2005,280(40):34 332-34 342.
[19] WRAY L V,FISHER S H.Bacillus subtilis CodY operators contain overlapping CodY binding sites[J].Journal of Bacteriology,2011,193(18):4 841-4 848.
[20] BELITSKY B R,SONENSHEIN A L.Contributions of multiple binding sites and effector-independent binding to CodY-mediated regulation inBacillussubtilis[J].Journal of Bacteriology,2011,193(2):473-484.
[21] ABABNEH Q O,HERMAN J K.CodY regulates SigD levels and activity by binding to three sites in the fla/che operon[J].Journal of Bacteriology,2015,197(18):2 999-3 006.
[22] SLACK F J,SERROR P,JOYCE E,et al.A gene required for nutritional repression of theBacillussubtilisdipeptide permease operon[J]. Molecular Microbiology,1995,15(4):689-702.
[23] MOLLE V,NAKAURA Y,SHIVERS R P,et al.Additional targets of theBacillussubtilisglobal regulator CodY identified by chromatin immunoprecipitation and genome-wide transcript analysis[J].Journal of Bacteriology,2003,185(6):1 911-1 922.
[24] MARCINIAK B C,PABIJANIAK M,JONG A D,et al.High- and low-affinity cre,boxes for CcpA binding inBacillussubtilis, revealed by genome-wide analysis[J].Bmc Genomics,2012,13(32):1-15.
[25] ROBERT P S,DINEEN S S,SONENSHEIN A L.Positive regulation ofBacillussubtilisackA by CodY and CcpA:establishing a potential hierarchy in carbon flow[J]. Molecular Microbiology,2006,62(3):811-822.
[26] GUÉDON E,SPERANDIO B,PONS N,et al.Overall control of nitrogen metabolism inLactococcuslactisby CodY, and possible models for CodY regulation in Firmicutes[J].Microbiology,2005,151(12):3 895-3 909.
[27] LAPUJADE P,COCAIGN-BOUSQUET M,LOUBIERE P.Glutamate biosynthesis inLactococcuslactissubsp. lactis NCDO 2118[J].Applied & Environmental Microbiology,1998,64(7):2 485-2 489.
[28] SANZ Y,LANFERMEIJER F C,RENAULT P,et al.Genetic and functional characterization ofdppgenes encoding a dipeptide transport system inLactococcuslactis[J].Archives of Microbiology,2001,175(5):334-343.
[29] CHAMBELLON E,YVON M.CodY-regulated aminotransferases AraT and BcaT play a major role in the growth ofLactococcuslactisin milk by regulating the intracellular pool of amino acids[J].Applied & Environmental Microbiology,2003,69(6):3 061-3 068.
[30] DEN HENGST C D,GROENEVELD M,KUIPERS O P,et al.Identification and functional characterization of theLactococcuslactisCodY-regulated branched-chain amino acid permease BcaP (CtrA)[J].Journal of Bacteriology,2006,188(9):3 280-3 289.
[31] BELITSKY B R.Role of Branched-chain amino acid transport inBacillussubtilisCodY activity[J].Journal of Bacteriology,2015, 197(8):1 330-1 338.
[32] BELITSKY B R,BARBIERi G,ALBERTINI A M,et al.Interactive regulation by theBacillussubtilisglobal regulators CodY and ScoC[J].Molecular Microbiology,2015,97:698-716.
[33] BARBIERI G,ALBERTINI A M,FERRARI E,et al.Interplay of CodY and ScoC in the regulation of major extracellular protease genes ofBacillussubtilis[J].Compte Rendu De Lacademie Des Sciences,2016,22(4):269-270.
[34] GIULIA B,BIRGIT V,DIRK A,et al.CodY regulates expression of theBacillussubtilisextracellular proteases Vpr and Mpr[J].Journal of Bacteriology,2015,197(8):1 423-1 4232.
[35] SLACK F J,SERROR P,JOYCE E,et al.A gene required for nutritional repression of theBacillussubtilisdipeptide permease operon[J].Molecular Microbiology,1995,15(4):689-702.
[36] WIEP KLAAS S,TRAN THU H,HAMOEN L W,et al.Antirepression as a second mechanism of transcriptional activation by a minor groove binding protein.[J].Molecular Microbiology,2007,64(2):368-381.
[37] ONUR E,MICHIEL W,SMID E J,et al.Genome-wide transcriptional responses to carbon starvation in nongrowingLactococcuslactis[J].Applied & Environmental Microbiology,2015,81(7):2 554-2 561.
[38] 陆文伟.全局转录调控因子DodY在嗜热链球菌中的调控机制研究[D].山东:山东大学, 2014.
[39] LOBEL L,HERSKOVITS A A.Systems level analyses reveal multiple regulatory activities of CodY controlling metabolism,motility and virulence inListeriamonocytogenes[J].Plos Genetics,2016,12(2).http://dx.doi.org/10.1371/journal.pgen.1005870.
[40] TAYLOR C M,BERESFORD M,EPTON H A S,et al.ListeriamonocytogenesrelA and hpt mutants are impaired in surface-attached growth and virulence[J].Journal of Bacteriology,2002,184(3): 621-628.
[41] BENNETT H J,PEARCE D M,SARAH G,et al.Characterization of relA and codY mutants ofListeriamonocytogenes: identification of the CodY regulon and its role in virulence[J].Molecular Microbiology,2007,63(5):1 453-1 467.
[42] KRETH J,CHEN Zhi-yun,Ferretti J,et al.Counteractive balancing of transcriptome expression involving CodY and CovRS inStreptococcuspyogenes[J].Journal of Bacteriology,2011,193(16):4 153-4 165.
[43] LI Ji-hong,MA Meng-lin,SARKER M R,et al.CodY is a global regulator of virulence-associated properties for Clostridium perfringens type D strain CN3718[J].Mbio,2013,4(5):00770-13.
[44] VAN S W,CHTEAU A,DILLIES M A,et al.The global regulator CodY regulates toxin gene expression inBacillusanthracisand is required for full virulence[J].Infection & Immunity,2009,77(10):4 437-4 445.
[45] FRENZEL E,DOLL V,PAUTHNER M,et al.CodY orchestrates the expression of virulence determinants in emeticBacilluscereusby impacting key regulatory circuits[J].Molecular Microbiology,2012,85(1):67-88.
[46] NICHOLAS R,DAVID J,RANJAN K,et al.A Spectrum of CodY activities drives metabolic reorganization and virulence gene expression inStaphylococcusaureus[J].Molecular Microbiology,2016,101(3):495-514.
[47] QI Ming-xia,MEI Fei,WANG Hui,et al.Function of global regulator CodY inBacillusthuringiensisBMB171 by comparative proteomic analysis[J].Journal of Microbiology & Biotechnology,2015,25(2):152-161.
[48] SLAMTI L,LEMY C,HENRY C,et al.CodY regulates the activity of the virulence quorum sensor PlcR by controlling the import of the signaling peptide PapR inBacillusthuringiensis[J].Frontiers in Microbiology,2016,6(e1002629).
[49] SMITS W K,TRAN T H,HAMOEN L W,et al.Antirepression as a second mechanism of transcriptional activation by a minor groove binding protein[J].Molecular Microbiology,2007,64(2):368-381.
Advances in research on global regulatory factor CodY of probiotics
FENG Jia, WU Hao, WANG Bin-bin, QIAO Jian-jun*
(Department of Pharmaceutical Engineering, School of Chemical Engineering and Technology, Key Laboratory of Systems Bioengineering, Ministry of Education, Tianjin University, Tianjin 300072, China)
CodY is a highly conserved global regulator of transcription in most Gram-positive bacteria. Studies onBacillussubtilisandLactococcuslactisand other probiotics show that CodY-regulated genes are involved in carbon or nitrogen metabolism, environmental stress responses, absorption of nutrients, genetic competence and other relevant processes. The structure, regulatory mechanisms and binding sites of CodY are summarized in this article, which may provides effective reference for further exploration of the potential regulatory functions of CodY and manufacture and modification of probiotics.
CodY; regulator; Gram-positive bacteria; probiotics
10.13995/j.cnki.11-1802/ts.201610037
硕士研究生(乔建军教授为通讯作者,E-mail:jianjunq@tju.edu.cn)。
2016-04-05,改回日期:2016-05-11

