采用多基因联合方法鉴定福建长乐和福清产区马铃薯Y病毒株系组成
沈林林,邹文超,高芳銮,詹家绥
(福建农林大学植物病毒研究所/福建省植物病毒学重点实验室,福州 350002)
采用多基因联合方法鉴定福建长乐和福清产区马铃薯Y病毒株系组成
沈林林,邹文超,高芳銮,詹家绥
(福建农林大学植物病毒研究所/福建省植物病毒学重点实验室,福州 350002)
【目的】马铃薯Y病毒(Potato virus Y,PVY)是马铃薯生产上危害较为严重的病毒,也是制约马铃薯可持续发展的主要病毒之一。论文旨在开发一套简便、准确、快速的PVY分子鉴定技术,并采用该技术及时查明福建省部分产区PVY病害的发生、分布及PVY株系组成。【方法】采用ELISA方法对采自福建省长乐市、福清市马铃薯种植区疑似受PVY感染的样品进行检测,并根据文献报道的PVY P1、VPg和CP基因保守区设计3对简并引物,对ELISA检测后的阳性样品进行基因扩增、克隆,并将获得的序列进行核苷酸序列一致性、重组位点、基因型分布和系统发育分析。【结果】ELISA检测结果表明,17份样品中有13个样品与PVY抗体呈阳性反应,其他呈阴性反应。13个阳性样品均能成功扩增出3个与P1、VPg和CP基因预期大小一致的特异片段。BLAST比对分析显示P1、VPg和CP基因与文献报道的已知PVY分离物的核苷酸序列一致性分别为72%—99%、85%—99%和88%—99%。P1、VPg和CP 3个基因联合序列分析显示,FQ01分离物与PVYN-Wi株系的核苷酸序列一致性最高,FQ08分离物与PVYE株系的核苷酸序列一致性最高,CL01、CL02、CL05和CL13 4个分离物与PVYNTN-NW株系SYR-I型的核苷酸序列一致性最高,CL03、CL04、FQ02、FQ06、FQ09、FQ11和CL12 7个分离物与PVYNTN-NW株系SYR-II型的核苷酸序列一致性最高。重组分析显示,除CP基因外,长乐市和福清市两个产区的PVY分离物的P1和VPg基因中均检测到显著的重组信号。基因型统计分析结果显示,长乐产区的P1基因为N型(60%)和N×O重组型(40%),VPg基因均为N×O重组型,而CP基因均为O型,而福清产区的P1基因为N型(25%)和N×O重组型(75%),VPg基因为N×O重组型(87.5%)和O型(12.5%),而CP基因除了一个分离物为N型外,其他均为O型。系统发育分析显示分离物FQ01与PVYN-Wi株系聚为一簇,FQ08与PVYE株系聚为一簇,CL01、CL02、CL05和CL13与PVYNTN-NW株系SYR-I型聚为一簇,CL03、CL04、FQ02、FQ06、FQ09、FQ11和CL12与PVYNTN-NW株系SYR-II型聚为一簇,表明在系统发育关系上,分离物FQ01与PVYN-Wi株系最近,FQ08与PVYE株系最近,CL01、CL02、CL05和CL13与PVYNTN-NW株系SYR-I型最近,CL03、CL04、FQ02、FQ06、FQ09、FQ11和CL12与PVYNTN-NW株系SYR-II型最近。【结论】PVY在福建省长乐市、福清市马铃薯种植区普遍存在,重组株系已成为田间的主流株系,且PVYNTN-NW已成为优势重组株系。
马铃薯Y病毒;ELISA;多基因联合序列;系统发育;重组株系
0 引言
【研究意义】马铃薯是仅次于水稻和小麦的全球第三大粮食作物[1],也是世界上种植较为广泛的经济作物之一。中国是世界上马铃薯生产第一大国[2],病毒病是制约马铃薯可持续发展的主要因素之一,它不仅使得马铃薯大幅减产[3],而且还严重影响种薯质量[4]。其中,马铃薯Y病毒(Potato virus Y,PVY)是分布最为广泛且危害最为严重的病毒之一[5-6]。因此,及时、准确地鉴定出PVY株系,有助于了解PVY的发生、分布情况及株系组成,可以为综合防控研究提供理论依据。【前人研究进展】PVY是马铃薯Y病毒科(Potyviridae)马铃薯Y病毒属(Potyvirus)的代表成员[7],其基因组由正单链RNA分子组成,大小约10 kb,包含一个大的开放阅读框(open reading frame,ORF),成熟后最终生成11个多功能蛋白[7-8]。其中,P1蛋白的编码区位于PVY基因组的起始端,编码丝氨基酸蛋白酶(P1 peptidase)。该蛋白酶与RNA结合活力密切有关,并且影响病毒在细胞之间的传播[9];VPg蛋白是一个无序的多功能蛋白,具有稳定病毒基因组结构的功能[10];而CP蛋白在病毒RNA的衣壳过程中起着重要作用,且其序列具有高度的保守性,是检测PVY常用的分子标记之一[4,10]。根据基因组内是否存在重组位点,PVY可以分为PVYC、PVYO、PVYN等非重组株系以及在PVYO和PVYN株系之间不同基因区段重组而成的PVYNTN、PVYN-Wi、PVYN:O等重组株系[11]。除此之外,还有一些不常见的株系,如PVYE[12-13]。其中,PVYN-Wi株系含有两个重组位点,分别位于P1和HC-Pro/P3基因区域;而PVYNTN-NW株系是近年来在研究叙利亚和中国分离物中新发现的重组株系,具有明显的PVYNTN和PVYN-Wi重组株系进一步重组的特征,并且含有SYR-I和SYR-II型两种基因组结构[14-15]。目前,用于马铃薯病毒病检测的方法有传统生物学检测法、电镜观察、血清学检测及分子生物学等,但单独使用以上的任何一种方法均无法实现PVY株系的准确鉴定,因此,实践中需要综合应用多种方法进行分析。近年来,联合多基因系统发育被广泛用于细菌、真菌等的鉴定[16-19]。在PVY株系鉴定中,杨庆东等通过P1和CP基因联合系统发育分析,可以将PVYN:O、PVYN-Wi、PVYO、PVYN和PVYNTN准确区分开,但却无法准确区别PVYN:O与PVYNTN-NW(SYR-I 型)及PVYN-Wi与PVYNTN-NW(SYR-II 型)[20-21]。高芳銮[22]通过系统发育分析与性状关联分析,表明仅联合P1、VPg和CP基因分析可以将PVY的常见株系区分开来。【本研究切入点】福建省具有独特的地理气候优势,属于中国马铃薯南方冬作区。近年来,随着国家马铃薯主粮化战略的实施,福建省种植马铃薯的面积正呈逐年上升的趋势,在春粮作物中占有越来越重要的地位。为及时了解福建省PVY变异株系的分布情况,生产上迫切需要实现PVY的快速检测并准确鉴定出PVY株系。【拟解决的关键问题】通过RT-PCR扩增、基因克隆获得福建省长乐市、福清市两个马铃薯产区PVY样品的P1、VPg和CP 3个基因序列,结合序列特征,采用多基因联合方法实现PVY株系的快速、准确鉴定,为该病毒的流行、变异趋势及有效防控打下基础。
1 材料与方法
试验于2015年在福建农林大学植物病毒研究所完成。
1.1 毒源
2015年利用随机采样法在福建省长乐市、福清市马铃薯种植区采集17份带有重型花叶、叶脉坏死和垂叶条斑坏死症状等典型PVY症状的马铃薯病叶,将新鲜病叶分别放置在不同密封袋中并标记采样时间、地点及编号,置于-80℃超低温冰箱保存待用。
1.2 试剂
DNA聚合酶(TransTaq DNA Polymerase HiFi)、DNA Marker和连接载体试剂盒(pEASY-T5 Zero Cloning Kit)均购自北京全式金(TransGen)公司;ELISA检测试剂盒购自美国Agdia公司;植物总RNA提取试剂盒(RNAsimple Total RNA Kit)和DNA回收试剂盒(Gel Purification Kit)均购自天根(TianGen)生化科技公司;反转录试剂盒(ReverTra Ace qPCR RT Kit)购自TaKaRa公司。
1.3 血清学检测
按照ELISA检测试剂盒说明书进行操作,先将待测样品加入预包被抗体的酶标板中,放置在4℃冰箱孵育过夜,接着用PBST洗涤,之后将酶标抗体加入并对抗体进行检测,在室温下孵育2 h,再用PBST洗涤,再将底物(pNPP)加入以显色,使用Thermo Multiskan MK3酶标仪读取405 nm处的OD值,通过比较405 nm处的OD值并判断阴性、阳性。试验检测了17份样品,并且每份样品做3个重复试验,同时设计了3组对照,分别为阴性对照、阳性对照及空白对照。
1.4 RNA提取及基因克隆
根据植物总RNA提取试剂盒(RNA Simple Total RNA)说明书提取17份疑似受PVY感染的马铃薯叶片的总RNA。参照反转录试剂盒(ReverTra Ace qPCR RT Kit)说明书,使用引物oligo(dT)18进行反转录试验扩增双链PVY全基因组的cDNA。由英潍捷基(上海)贸易有限公司合成3对简并引物,该引物序列分别为: P1-F(5′-CVATGGCAAYYTACAYGTCAAC-3′)和 P1-R(5′-AGGRTATCTCADYHGTGCCC-3′),KV-F(5′-TAATATTGGTGGAGAGAYTGCTTG-3′)和 KV-R(5′-GCTTCATGCTCYACYTCCTG-3′),CP-F(5′-GSAAAYGAYACAATYGATGC-3′)和 CP-R(5′-CACATGTTYTTVACTCCAAG-3′)[15]。利用合成的3对简并引物,分别对P1、VPg及CP基因进行PCR扩增,其预期的片段大小分别为915、737和803 bp。试验中PCR反应体系为25 μL,包括2.5 μL 10×TransTaq HiFi Buffer II,2 μL dNTPs(2.5 mmol·L-1),正向引物和反向引物(10 μmol·L-1)各1 μL,0.25 μL TransTaq HiFi Polymerase(5 U·μL-1),1 μL cDNA和17.25 μL ddH2O。PCR反应程序:首先在94℃下预变性处理4 min,随后在94℃下变性处理30 s,在53—55℃温度下退火30 s(P1、VPg和CP基因的退火温度分别为53、55和55℃),再在72℃下延伸若干分钟(延伸时间按照1 min·kb-1计算),其中变性、退火和延伸这3个过程各进行30个循环,最后在72℃下终延伸5 min。取5 μL的PCR产物,加入到1 μL的6倍上样缓冲液(6×DNA Loading Buffer),混匀后滴至1%琼脂糖凝胶的泳道中,采用110 V电压电泳30 min检测PCR产物。检测成功的PCR产物利用DNA回收试剂盒回收纯化,根据连接载体试剂盒(pEASY-T5 Zero Cloning Kit)的说明书,将回收产物与T5 Vector连接载体25℃连接15 min,连接后转化到DH5α感受态细胞中,最后通过菌落的重组子鉴定得到阳性克隆,并从阳性克隆中随机选择3—6个送至英潍捷基(上海)贸易有限公司测序。
1.5 序列分析及重组检测
利用DNAMAN 9(Lynnon BioSoft,Canada)软件对测序获得的序列进行拼接。通过NCBI上的BLASTn(http://blast.ncbi.nlm.nih.gov/)在线工具分析序列一致性。使用重组分析软件RDP4对可能的重组事件进行检测,并通过Simplot 3.5分析潜在重组相似性作图(Similarity plot)。以PVYN株系的Mont分离物(GenBank登录号:AY884983)和PVYO株系的Oz分离物(GenBank登录号:EF026074)为参考亲本序列[23]。
1.6 多基因联合系统发育分析
从GenBank中选择32个其他已知株系的PVY分离物作为参考(表1)。建树前,使用MEGA 5[24]中的MUSCLE算法[25]分别对建树的P1、VPg和CP基因序列进行多重序列比对后,应用SequenceMatrix[26]进行序列串联,获得的合并序列采用邻接法(neighborjoining,NJ)重建系统发育关系,最后应用自举法(Bootstrap)重复抽样1 000次以评估各分支节点的可靠性。

表1 本研究采用到的PVY参考分离物的名称、株系分类及GenBank登录号Table1 Name, accession number and classification of reference isolates downloaded from GenBank in this study
2 结果
2.1 ELISA检测
在检测的17份样品中,共有13份样品与PVY抗体呈阳性反应,其余4份样品均呈阴性反应,检出率为76.47%(表2),其中采自福建长乐的样本阳性检出率为83.33%,而采自福建福清的样本阳性检出率略低,为72.73%,两者相差10.60%。
2.2 目的基因的PCR扩增
利用简并引物P1-F/P1-R、KV-F/KV-R、CP-F/CPR扩增到的PCR产物与预期的目的片段大小相一致,分别约为915、737和803 bp。福建长乐和福清两个产区的13个分离物以及阳性对照均可扩增到目的片段,阴性(健康马铃薯叶片)和空白对照都未扩增到相应的片段。
RT-PCR扩增获得的基因片段裁去冗余序列和载体序列后,经DNAMAN、BLAST等分析,确定P1、VPg和CP基因编码区大小分别825 bp(GenBank登录号KX376914-KX376918、KX376921、KX376922、KX376925、KX376927-KX376931)、564 bp(GenBank登录号KX376932-KX376936、KX376939、KX376940、KX376943、KX376945、KX376946-KX376949)和801 bp(GenBank登录号KX376950-KX376954、KX376957、KX376958、KX376961、KX376963-KX376967)。
2.3 目的基因的序列特征
序列一致性分析显示,福建13个PVY分离物的P1基因与8个参考株系的核苷酸序列一致性为72%— 99%(表3),其中分离物CL01、CL02、CL05、FQ08、FQ13与Oz的序列一致性最低,为72%—73%,与Wilga5、34/1、SYR-II-2-8的序列一致性最高,为88%—99%。而VPg和CP基因与8个PVY参考株系核苷酸序列一致性都比较高,分别为85%—99%和88%—99%(表3)。其中,VPg基因中分离物FQ01与Mont的核苷酸序列一致性最低,为85%,与Oz、PB209、Wilga5和HN2的序列一致性最高,为99%;CP基因中分离物FQ08与PB209、Wliga5、SYR-II-2-8、Oz、HN2的序列一致性最低,为88%—89%,与Mont、PB312、34/1的序列一致性最高,为93%—95%。结合3个基因的序列一致性分析结果表明,CL01、CL02、CL05和CL13 4个分离物与PVYNTN-NW株系SYR-I型的核苷酸序列一致性最高,CL03、CL04、FQ02、FQ06、FQ09、FQ11和CL12 7个分离物与PVYNTN-NW株系SYR-II型的核苷酸序列一致性最高,而FQ01分离物与PVYN-Wi株系的核苷酸序列一致性最高,FQ08分离物与PVYE株系的核苷酸序列高度同源。
Simplot分析显示,在长乐、福清两个产区的PVY分离物中,除了CP基因未检测到重组信号外,P1和VPg基因中均检测到潜在的重组信号(图1为FQ09分离物的Simplot分析图),重组位点分别位于P1和VPg 基因区域内的第291和第128核苷酸位置,表明这些分离物均为重组型。
进一步统计分析结果显示,长乐产区的P1基因为N型(60%)和N×O重组型(40%),VPg基因均为N×O重组型,CP基因均为O型;福清产区的P1基因为N型(25%)和N×O重组型(75%),VPg基因为N×O重组型(87.5%)和O型(12.5%),CP基因除了一个分离物为N型外,其他均为O型(表4)。

表2 17份疑似PVY感染的马铃薯叶片的ELISA检测Table2 ELISA detection of the 17 potato leaves with PVY-alike symptoms sampled

表3 PVY福建分离物和GenBank下载参考分离物之间的P1(单元格中第一个数据)、VPg(单元格中第二个数据)和CP(单元格中第三个数据)基因序列一致性Table3 Nucleotide identity in the P1 (1st value in the cells), VPg (2nd value in the cells) and CP (3rd value in the cells) sequences between PVY isolates from Fujian and reference isolates downloaded from GenBank (%)
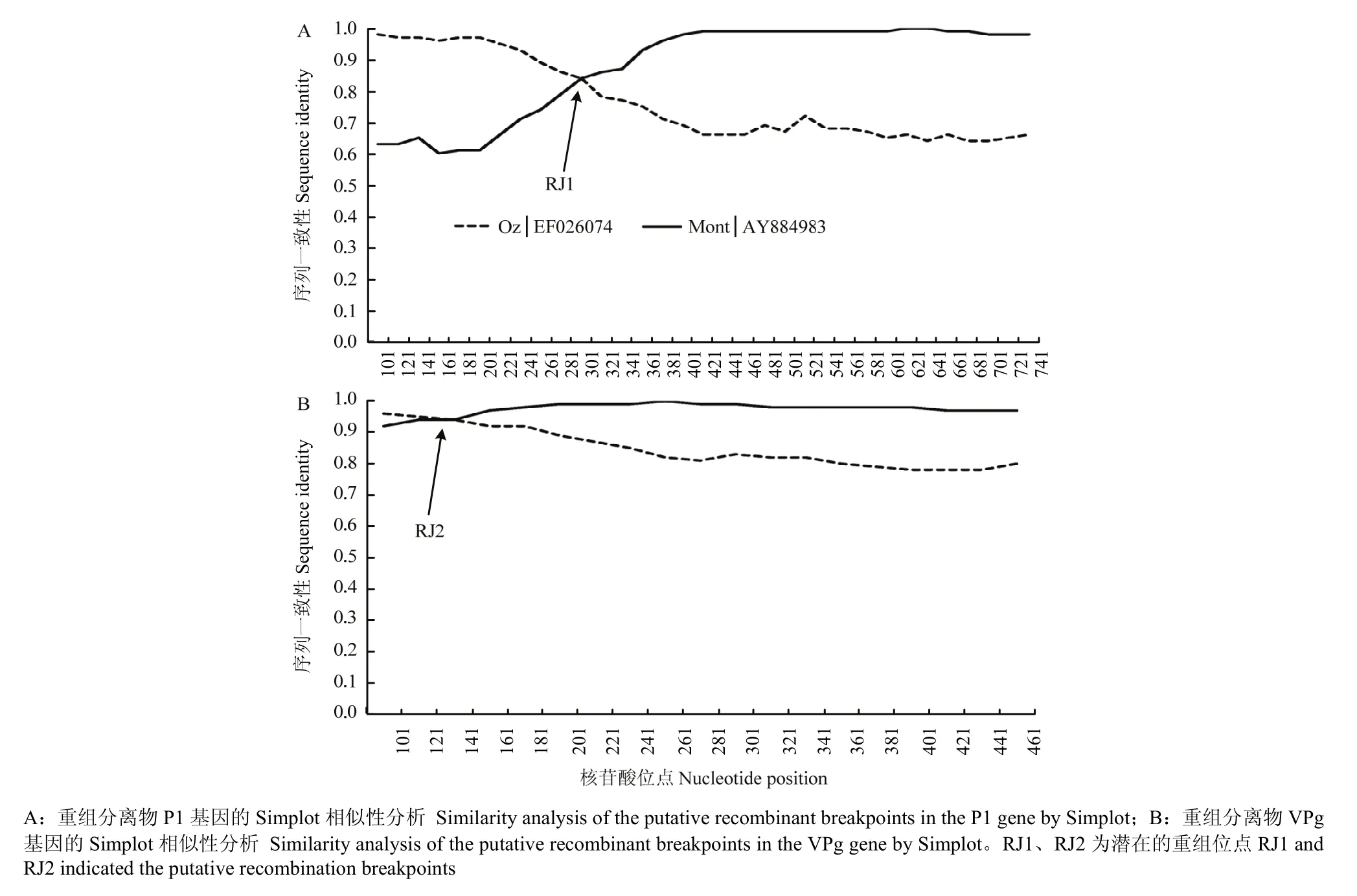
图1 PVY福建分离物重组位点检测Fig. 1 Detection of recombination breakpoints in the PVY isolates from Fujian Province

表4 福建长乐、福清产区PVY株系组成Table4 Strain composition of PVY isolates sampled from Changle and Fuqing of Fujian Province
2.4 多基因联合系统发育分析
使用 NJ 法构建的系统发育树如图2所示,所有的PVY分离物共形成了11个簇,其中来自同一株系的分离物以较高的置信度相聚在一起,表明PVY具有显著的株系特异性。本研究获得的4个分离物CL01、CL02、CL05和FQ13与PVYNTN-NW株系SYR-I型的3个分离物以较高的自举值(BP=97)聚为一簇,CL03、CL04、FQ02、FQ06、FQ09、FQ11和FQ12 7个分离物与PVYNTN-NW株系SYR-II型的3个分离物也以较高自举值(BP=97)聚为一簇,但FQ01分离物与PVYN-Wi株系的3个分离物以较高的自举值(BP=97)聚为一簇,FQ08分离物与PVYE株系的两个分离物以较高的自举值(BP=97)聚为一簇。系统发育分析表明,在进化关系上CL01等4个分离物与PVYNTN-NW株系SYR-I型最近,CL03等7个分离物与PVYNTN-NW株系SYR-II型最近,而FQ01分离物与 PVYN-Wi株系最近,FQ08分离物与PVYE株系最近。
3 讨论
PVY可以由40种多种蚜虫频繁传播,从而加快了流行速度[10]。农产品全球化销售及其本身快速变异的特点,也增加了PVY准确鉴定的难度。福建省是重要的南方马铃薯冬作区,鉴定并了解福建省PVY株系的分布情况和组成,对该省PVY防控和发展中国马铃薯产业至关重要。ELISA是PVY检测上最为常用的血清学方法之一,虽然可以快速、简便地检测大量的疑似样品,但灵敏度低于以核苷酸为基础的PCR方法,因而对于PVY含量低的材料容易漏检。例如,高芳銮等[4]分别用ELISA分析了采自重庆的28份感染PVY的马铃薯叶片,阳性检出率仅为43%(12/28),而改用RT-PCR方法时,阳性检出率则是100%。本试验通过ELISA检测了17份疑似PVY样品,其中4份样品呈阴性反应,并经CP基因的PCR扩增确定不含有PVY。17份样品中有13份呈阳性反应,检出率为76.47%,相比2011年福建长乐和福建福清的PVY检出率(75.00%)略高一些[4],说明PVY在福建省马铃薯主要种植生产区长乐和福清普遍发生,且发生频率稍有提高。究其原因,可能归因于福建省农产品贸易发达,马铃薯种薯和产品的调运,加快了PVY的传播。

图2 基于NJ法重建的PVY P1、VPg和CP联合基因系统发育树Fig. 2 Phylogenetic tree reconstructed from the concatenated sequences of P1, VPg and CP genes using neighbor-joining method
由于PVY株系分化现象明显,传统上普遍采用的以CP单一基因为基础的分子分类方法无法满足PVY株系的快速、准确鉴定需求。采用CP单基因系统发育分析,可以将PVYO外的其他株系很好地区分开来,但无法正确识别PVYN:O、PVYN-Wi和PVYNTN-NW3种株系,因为它们的CP基因都是O型。通过全基因组序列分析可以获得不同分离物的株系结构类型[27],但试验成本较高,且实用性较差[28]。而多基因联合分析弥补了这些不足,为准确、快速地鉴定PVY常见株系提供了可能。在PVY基因组中,P1、HC-pro、VPg和CP基因是重组热点的区域[23]。为实现PVY的快速检测和鉴定,高芳銮[22]以这4个基因为研究对象,采用不同基因组合分别进行系统发育分析,并通过系统发育与性状关联分析进一步验证各种组合中代表分离物与株系的关联系数及相关参数,结果表明联合P1、VPg和CP 3个基因即可将PVY的常见株系区分开。利用此联合多基因方法,笔者实验室鉴定到PVY一株新的重组分离物GF_YL21[22,29],表明该方法不仅可以实现对PVY常见株系的准确鉴定,还可以实现对新重组分离物的预判。本文对福建省马铃薯主栽区PVY分离物P1、VPg和CP基因进行RT-PCR扩增,并联合PVY基因组重组热点所在的3个基因进行系统发育分析,表明PVY在福建省长乐市、福清市两个马铃薯种植区普遍发生,重组株系已成为田间的主流株系,且PVYNTN-NW是优势株系。梁五生等[30]通过统计分析发现中国的PVY群体主要以PVYN株系为主,但高芳銮等[4]通过对中国14个省PVY分离物的检测鉴定,表明PVY在马铃薯主要种植区普遍发生的多为重组株系。导致两种不同结论的可能原因主要在于PVY基因组中P1、HC-pro、CP等基因区段重组频繁,仅通过一个或两个基因进行分析,无法获得PVY株系的准确鉴定。本研究的多基因联合分析结果显示,重组株系已经成为了田间的主流株系(100%),包括PVYNTN-NW株系SYR-I型、PVYNTN-NW株系SYR-II型、PVYN-Wi株系和PVYE,进一步支持高芳銮等的鉴定结果[4]。重组株系被认为可能比非重组株系具有更强的致病力[11],并已在世界各马铃薯种植区越来越流行,因此生产上需要密切关注这些重组株系的发展动态。
4 结论
通过ELISA方法对采自福建省马铃薯产区的17份疑似PVY样品进行检测,阳性检出率平均为76.47%,表明PVY在福建省马铃薯长乐市、福清市两地普遍发生;通过P1、VPg和CP 3个基因的序列特征及联合多基因系统发育分析,表明PVY重组株系已成为田间的主流株系,尤其是PVYNTN-NW株系。
[1] BIRCH P J, BRYAN G, FENTON B, GILROY E, HEIN I, JONES J,PRASHAR A, TAYLOR M, TORRANCE L, TOTH I. Crops that feed the world 8: Potato: are the trends of increased global production sustainable? Food Security, 2012, 4(4): 477-508.
[2] 胡新喜, 何长征, 熊兴耀, 刘明月, 宋勇, 聂先舟. 马铃薯Y病毒研究进展. 中国马铃薯, 2009, 23(5): 293-300.
HU X X, HE C Z, XIONG X Y, LIU M Y, SONG Y, NIE X Z. Progress on Potato virus Y. Chinese Potato Journal, 2009, 23(5): 293-300. (in Chinese)
[3] WANG B, MA Y, ZHANG Z, WU Z, WU Y, WANG Q, LI M. Potato viruses in China. Crop Protection, 2011, 30(9): 1117-1123.
[4] 高芳銮, 沈建国, 史凤阳, 方治国, 谢联辉, 詹家绥. 中国马铃薯Y病毒的检测及CP基因的分子变异. 中国农业科学, 2013, 46(15): 3125-3133.
GAO F L, SHEN J G, SHI F Y, FANG Z G, XIE L H, ZHAN J S. Detection and molecular variation of Potato virus Y CP gene in China. Scientia Agricultura Sinica, 2013, 46(15): 3125-3133. (in Chinese)
[5] WHITWORTH J L, NOLTE P, MCINTOSH C, DAVIDSON R. Effect of Potato virus Y on yield of three potato cultivars grown under different nitrogen levels. Plant Disease, 2006, 90(1): 73-76.
[6] AKANDA A K. Performance of seed potato produced from sprout cutting, stem cutting and conventional tuber against PVY and PLRV. Bangladesh Journal of Agricultural Research, 2010, 34(4): 609-622.
[7] KING A M, LEFKOWITZ E, ADAMS M J, CARSTENS E B. Virus Taxonomy, Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam: Elsevier Academic Press, 2011.
[8] CHUNG B Y, MILLER W A, ATKINS J F, FIRTH A E. An overlapping essential gene in the Potyviridae. Proceedings of the National Academy of Sciences of the United States of America, 2008,105(15): 5897-5902.
[9] ROHOŽKOVÁ J, NAVRÁTIL M. P1 peptidase-a mysterious protein of family Potyviridae. Journal of Biosciences, 2011, 36(1): 189-200.
[10] QUENOUILLE J, VASSILAKOS N, MOURY B. Potato virus Y: a major crop pathogen that has provided major insights into the evolution of viral pathogenicity. Molecular Plant Pathology, 2013, 14(5): 439-452.
[11] 史凤阳, 高芳銮, 沈建国, 常飞, 詹家绥. 马铃薯Y病毒福建分离物P1基因的分子变异和结构特征. 遗传, 2014, 36(7): 713-722.
SHI F Y, GAO F L, SHEN J G, CHANG F, ZHAN J S. Sequence variation of P1 gene in Potato virus Y isolated from Fujian Province. Hereditas, 2014, 36(7): 713-722. (in Chinese)
[12] LORENZEN J, NOLTE P, MARTIN D, PASCHE J S, GUDMESTAD N C. NE-11 represents a new strain variant class of Potato virus Y.Archives of Virology, 2008, 153(3): 517-525.
[13] GALVINO-COSTA S B F, FIGUEIRA A D R, CAMARGOS V V,GERALDINO P S, HU X J, NIKOLAEVA O V, KERLAN V,KARASEV A V. A novel type of Potato virus Y recombinant genome,determined for the genetic strain PVYE. Plant Pathology, 2012, 61(2): 388-398.
[14] ALI M C, MAOKA T, NATSUAKI T, NATSUAKI K. PVYNTN-NW, a novel recombinant strain of Potato virus Y predominating in potato fields in Syria. Plant Pathology, 2010, 59(1): 31-41.
[15] 高芳銮, 常飞, 沈建国, 谢联辉, 詹家绥. PVYNTN-NW榆林分离物的全基因组序列测定与分析. 中国农业科学, 2015, 48(2): 270-279.
GAO F L, CHANG F, SHEN J G, XIE L H, ZHAN J S. Complete genome analysis of a PVYNTN-NWrecombinant isolate from Yulin of China. Scientia Agricultura Sinica, 2015, 48(2): 270-279. (in Chinese)
[16] 贾广成, 周增强, 侯珲, 王丽, 朱建兰. 苹果轮纹病菌的多基因联合鉴定. 生物技术通报, 2013(7): 161-166.
JIA G C, ZHOU Z Q, HOU H, WANG L, ZHU J L. Botryosphaeria dothidea of multiple genes joint identification. Biotechnology Bulletin,2013(7): 161-166. (in Chinese)
[17] 刘威, 叶乃兴, 刘伟, 金珊, 连玲丽, 赖建东, 谢运海. 茶树炭疽菌的鉴定及致病力分析. 福建农林大学学报 (自然科学版), 2015,44(6): 581-586.
LIU W, YE N X, LIU W, JIN S, LIAN L L, LAI J D, XIE Y N. Identification and pathogenic analysis of the pathogen causing tea plant (Camellia sinensis) anthracnose. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2015, 44(6): 581-586. (in Chinese)
[18] 周明霞, 陈勇喆, 王辉, 陈智, 邓乐. 发光细菌PB-P3.9的鉴定与gyrB、luxA基因系统发育分析. 食品科学, 2011, 32(15): 220-224.
ZHOU M X, CHENG Y Z, WANG H, CHEN Z, DENG L. Identification and phylogenetic analysis based on gyrB and luxA of photobacterium PB-P3.9. Food Science, 2011, 32(15): 220-224. (in Chinese)
[19] 李河, 周国英, 徐建平, 朱丹雪. 一种油茶新炭疽病原的多基因系统发育分析鉴定. 植物保护学报, 2014, 41(5): 602-607.
LI H, ZHOU G Y, XU J P, ZHU D X. Pathogen identification of a new anthracnose of Camellia oleifera in China based on multiple-gene phylogeny. Acta Phytophylacica Sinica, 2014, 41(5): 602-607. (in Chinese)
[20] 杨庆东, 吴兴泉, 陈士华, 刘晓磊. PVY株系间的分子变异及分子鉴定方法. 中国马铃薯, 2011, 25(3): 166-169.
YANG Q D, WU X Q, CHEN S H, LIU X L. Molecular variation and identification of Potato virus Y strains. Chinese Potato Journal, 2011,25(3): 166-169. (in Chinese)
[21] 刘洪义, 梁五生, 刘忠梅, 张洪祥, 杨立群. 黑龙江省部分地区马铃薯Y病毒株系检测. 东北农业大学学报, 2014, 45(1): 47-52.
LIU H Y, LIANG W S, LIU Z M, ZHANG H X, YANG L Q. Identification of Potato virus Y strains in infected potato plants from some places of Heilongjiang Province. Journal of Northeast Agricultural University, 2014, 45(1): 47-52. (in Chinese)
[22] 高芳銮. 中国马铃薯Y病毒群体遗传结构及其分子进化机制[D].福州: 福建农林大学, 2014.
GAO F L. Population genetic structure of Potato virus Y and its molecular evolutionary mechanisms[D]. Fuzhou: Fujian Agriculture and Forestry University, 2014. (in Chinese)
[23] HU X, KARASEV A V, BROWN C J, LORENZEN J H. Sequence characteristics of Potato virus Y recombinants. The Journal of General Virology, 2009, 90(12): 3033-3041.
[24] TAMURA K, PETERSON D, PETERSON N, STECHER G, NEI M,KUMAR S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[25] EDGAR R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 2004, 32(5): 1792-1797.
[26] VAIDYA G, LOHMAN D J, MEIER R. SequenceMatrix: concatenation software for the fast assembly of multi-gene datasets with character set and codon information. Cladistics, 2011, 27(2): 171-180.
[27] KARASEV A V, GRAY S M. Continuous and emerging challenges of Potato virus Y in potato. Annual Review of Phytopathology, 2013, 51: 571-586.
[28] 韦茂春. 侵染烟草的四个马铃薯Y病毒分离物基因组序列比较分析[D]. 南宁: 广西大学, 2011.
WEI M C. Comparative analysis of genome sequences of four isolates of Potato virus Y infecting tobacco[D]. Nanning: Guangxi University,2011. (in Chinese)
[29] GAO F L, CHANG F, SHEN J G, SHI F Y, XIE L H, ZHAN J S. Complete genome analysis of a novel recombinant isolate of Potato virus Y from China. Archives of Virology, 2014, 159(12): 3439-3442.
[30] 梁五生, 温雪玮, 刘洪义, 刘忠梅. 马铃薯Y病毒株系分类及中国大田马铃薯感染的马铃薯Y病毒株系谱研究进展. 中国农学通报,2015, 31(21): 136-143.
LIANG W S, WEN X W, LIU H Y, LIU Z M. Progress of Potato virus Y strain classification and analysis of profile of Potato virus Y strains infecting potato in China. Chinese Agricultural Science Bulletin, 2015,31(21): 136-143. (in Chinese)
(责任编辑 岳梅)
Strain Composition of Potato virus Y in Fujian Province Detected with the Concatenated Sequence Approach
SHEN Lin-lin, ZOU Wen-chao, GAO Fang-luan, ZHAN Jia-sui
(Institute of Plant Virology, Fujian Agriculture and Forestry University/Key Laboratory of Plant Virology of Fujian Province,Fuzhou 350002)
【Objective】Potato virus Y (PVY) is one of the most destructive pathogens constraining sustainable development ofpotato industry. The objective of this study is to develop a fast, easy-to use and accurate approach to timely detect PVY strains and apply the approach to investigate the occurrence, distribution and composition of PVY strains in Fujian Province. 【Method】ELISA method was used to confirm the presence of virus in the PVY-alike leaf samples randomly collected from Changle and Fuqing cities in Fujian Province. Three pairs of degenerate primers designed from the conserved regions of P1, VPg and CP genes in the reference PVY sequences downloaded from GenBank were used to amplify the positive samples by ELISA test. Nucleotide identity and recombination events between isolates from Fujian and strains downloaded from GenBank were evaluated and phylogenetic tree was reconstructed using concatenated sequences of the three genes. 【Result】ELISA confirmed that 13 out of the 17 samples collected from the two potato producing areas were infected with PVY. RT-PCR amplifications of all 13 ELISA-positive samples generated the expected sizes of fragments corresponding to the three genes. Individual gene analyses showed that P1, VPg and CP sequences in Fujian isolates shared 72%-99%, 85%-99% and 88%-99% nucleotide identity with the
trains, respectively. Concatenated sequence analysis showed that FQ01 and FQ08 isolates shared the highest sequence identity with PVYN-Wiand PVYE, respectively;CL01, CL02, CL05 and CL13 isolates shared the highest sequence identity with PVYNTN-NW(SYR-I), whereas CL03, CL04, FQ02,FQ06, FQ09, FQ11 and CL12 shared the highest sequence identity with PVYNTN-NW(SYR-II). Recombination signals were identified in P1 and VPg genes but not in CP of PVY, which isolated from Changle and Fuqing cities in Fujian Province. Further analyses showed that in Changle City, P1 gene was composed of two lineages, with 60% being N lineage and 40% being N×O lineage. All VPg genes were N×O lineage and all CP genes were classified as O lineage. Similarly in Fuqing City, P1 gene was consisted of two lineages, with 25% being N lineage and 75% being N×O lineage; 87.5% VPg genes were N×O lineage and 12.5% was O lineage; and all except one (which was classified as N lineage) CP genes were O lineage. Phylogenetic tree revealed that isolates CL01, CL02,CL05 and CL13 were grouped with PVYNTN-NW(SYR-I) and isolates CL03, CL04, FQ02, FQ06, FQ09, FQ11 and CL12 were clustered with the PVYNTN-NW(SYR-II), whereas isolate FQ01 was grouped with PVYN-Wiand isolate FQ08 was clustered with the PVYE, respectively. These results indicated that evolutionarily, CL01, CL02, CL05 and CL13 are closer to PVYNTN-NW(SYR-I);CL03, CL04, FQ02, FQ06, FQ09, FQ11 and CL12 are closer to PVYNTN-NW(SYR-II), FQ01 is closer to PVYN-Wiand FQ08 is closer to PVYE, respectively. 【Conclusion】PVY occurs frequently in Changle and Fuqing cities in Fujian Province and recombinant strains,particularly PVYNTN-NWare dominant in the PVY isolates from the province.
Potato virus Y (PVY); ELISA; concatenated sequence; phylogeny; recombinant strain
2016-06-23;接受日期:2016-08-25
国家现代农业马铃薯产业技术体系(CARS-10)
联系方式:沈林林,E-mail:18649708815@163.com。邹文超,E-mail:tsou2015@163.com。沈林林和邹文超为同等贡献作者。通信作者高芳銮,Tel:0591-83793031;E-mail:raindy@fafu.edu.cn。通信作者詹家绥,Tel:0591-83856973;E-mail:jiasui.zhan@fafu.edu.cn

