原生质体融合提高产谷氨酰胺转氨酶菌株产量
侯孝仑,刘雅清,郭玮婷,高红亮,常忠义,步国建,鲁 伟,解秀娟,金明飞,*
(1.华东师范大学生命科学学院,上海 200241;2.泰兴市东圣食品科技有限公司,江苏 泰兴 225411)
原生质体融合提高产谷氨酰胺转氨酶菌株产量
侯孝仑1,刘雅清1,郭玮婷1,高红亮1,常忠义1,步国建2,鲁 伟2,解秀娟2,金明飞1,*
(1.华东师范大学生命科学学院,上海 200241;2.泰兴市东圣食品科技有限公司,江苏 泰兴 225411)
目的:研究并建立产谷氨酰胺转氨酶茂原链霉菌原生质体制备技术,通过原生质体融合技术筛选高产菌株。方法:以溶菌酶处理茂原链霉菌获得原生质体,采用原生质体融合技术,通过96 孔板高通量初筛、试管复筛、摇瓶验证选育高产菌株。结果:茂原链霉菌形成的原生质体再生出的菌落直径比较小,而菌丝生成的菌落直径比较大,两种形态的菌落96 孔板发酵后酶活力有显著性差异。不同的茂原链霉菌株制备原生质体,酶解程度、原生质体纯度、再生率有很大的差异,再生率最高可达1 804.25%,最低仅为12.76%。原生质融合后的高产菌株,选取96 孔板初筛酶活力前3%的菌株进行试管发酵,得到高产菌株比例为32.2%,酶活力比亲本高22.4%,经摇瓶验证产酶比对照提高16.28%。结论:利用基因组重排技术,可以初步对茂原链霉菌进行高产菌株选育。
茂原链霉菌;原生质体;再生;融合;基因组重排
谷氨酰胺转氨酶(transglutaminase,TGase)是一种巯醇酶,能够催化蛋白质与蛋白质、蛋白质与氨基酸之间发生酰胺基转移反应,从而使蛋白质发生交联。这种性质能够使其广泛地应用于食品行业中,在烘焙面制品[1-2]、酸奶[3-4]、鱼糜及其他肉制品[5-6]中改善食品中蛋白质的特性,提高保水性、强度、起泡性、乳化性,引入必需氨基酸等,有很大的应用价值,被誉为“21世纪超级黏合剂”,另外还在医药[7]、纺织品、保健护理等行业有很大的应用前景[8-10]。
目前商品化的TGase主要由微生物——茂原链霉菌[11]发酵获得。随着TGase商业化的不断加深及更加广泛的应用,对TGase产量的要求越来越高,大量学者通过培养基优化、发酵条件优化、菌种选育等方面来提高TGase的产量以满足商业化需求,降低生产成本。由于茂原链霉菌本身性质,其发酵条件难以控制,通过培养基优化和发酵条件的优化已经很难使TGase的产量有大幅度的提高,一般在1~3 U/mL左右[12-13]。目前主要通过选育优良的菌株来提高TGase的产量。在茂原链霉菌的菌种选育上,主要有自然选育或人工诱变筛选等方法,如张莹[14]、田沛霖[15]等已对茂原链霉菌菌株的选育做了大量的工作,但是随着菌种一定的驯化,会产生饱和效应,单位产量难以持续提高,一般维持在3~5 U/mL[15-16]。随着原生质体制备技术不断的成熟,有大量学者通过原生质体融合技术选育微生物菌种[17-18],2002年Zhang Yingxin等[19]将原生质体融合技术应用到弗氏链霉菌(Streptomyces fradiae)中,使其合成泰乐菌素的能力大大提高,是传统方法进行20~30 轮诱变才能得到的结果,并首次提到基因组重组技术。Patnaik[20]和Wang Yuhua[21]等分别报道了采用原生质体融合技术多轮融合筛选乳酸杆菌耐酸菌株,筛选到的新菌株大大提高了乳酸杆菌合成乳酸的能力。Yu Lei等[22]将鼠李糖乳酸杆菌经过两轮的原生质体融合,乳酸产量提高71.5%。多轮融合后玫瑰孢链霉菌产达托霉素能力达到野生菌株的6.5~7 倍[23]。纳他霉素产生菌经过4 轮的原生质体融合后纳他霉素产量比亲本菌株高97.1%[24]。绿色产色链霉菌通过5 轮的原生质体融合筛选后,高产菌株阿维霉素产量分别比两株出发菌株高4.85 倍和36.8 倍[25]。刺糖多胞菌和阿维链霉菌原生质体紫外处理后进行属间融合后,高产菌株多杀菌素产量比野生菌株提高447.22%[26]。多轮的原生质体融合即基因组重排技术在微生物育种中显示了极大的优势,但目前还未有在茂原链霉菌中的报道。基因组重排技术是从基因上改变菌株本身的特性,原生质体在聚乙二醇(polyethylene glycol,PEG)的作用下发生融合,基因发生交叉互换,菌株获得新的特性,在热稳定、产量、发酵时间等方面都会产生变化,因此在微生物菌种改造中有很大的优势,不仅能够满足食品安全等要求,又能提高所需产品的产量,备受广大研究者的青睐。基因组重排技术中,原生质体的制备条件、再生等起着关键性因素,不少研究者对微生物菌种的基因组重排技术做了大量研究,尤其是对原生质体制备及再生的影响因素,早在1981年Baltz等[27]在原生质体制备再生中就对菌丝体的培养温度、时间,及原生质体的培养温度,固体培养基的湿度等做了研究,发现了原生质体的制备及再生在各个方面受到影响。国内外对产TGase茂原链霉菌基因组重排技术的研究还比较少,本实验对茂原链霉菌的原生质体制备做了研究,观察原生质体的再生情况,并在基因组重排中作了初步的探索。
1 材料与方法
1.1 菌株、试剂与培养基
茂原链霉菌,由华东师范大学生命科学学院微生物发酵实验室保存。
溶菌酶、PEG 2000 美国Sigma公司;其他试剂为国产分析纯。
高氏一号培养基(g/L):甘油20、鱼粉蛋白胨25、酵母膏5、MgSO4•7H2O 2、K2HPO4•3H2O 2、调pH值至7.4,1×105Pa灭菌20 min。发酵培养基(g/L):甘油20、鱼粉蛋白胨25、酵母膏6、MgSO4•7H2O 2、K2HPO4•3H2O 2、调pH值至7.4,1×105Pa灭菌20 min。高渗培养基[28]:培养前预培养基中加入体积分数为20%的甘氨酸645 μL、5 mol/L的MgCl2•6H2O 50 μL。再生培养基[28](g/L):蔗糖103、葡萄糖10、K2SO40.25、酵母膏5、N-三(羟甲基)甲基-2-氨基乙磺酸(N-Tr is(hydroxymethyl) methyl-2-aminoethanesulfonic acid,TES)5.73、酶水解干酪素0.1、KH2PO40.05、L-脯氨酸(L-Pro) 3、MgCl2•6H2O 10.12、CaCl2•2H2O 2.22、琼脂粉12、微量元素溶液2 mL,115 ℃高压灭菌20 min。微量元素溶液[28](mg/L):ZnCl240、FeCl3•6H2O 200、CuCl2•2H2O 10、MnCl2•4H2O 10、Na2B4O7•10H2O 10、(NH4)6Mo7O24•4H2O 10。P Buffer:蔗糖103 g、K2SO40.25 g、MgCl2•6H2O 2.02 g、微量元素溶液2 mL、补足蒸馏水800 mL,115 ℃高压灭菌20 min,使用前加入0.5%(质量分数,下同)KH2PO410 mL、3.68% CaCl2•2H2O 100 mL、5.73% pH 7.2的TES缓冲液100 mL。
1.2 仪器与设备
DK-S22电热恒温水浴锅、GNP-9160隔水式恒温培养箱 上海精宏实验设 备有限公司;CDA-1650-2超净工作台 上海上净净化设备有限公司; DKY-Ⅱ恒温调速回转摇床 上海杜科自动化设备有限公司;BX-51TF相差显微镜 日本Olympus公司;SyergyHT多功能微孔板检测仪 基因有限公司;721可见分光光度计 上海元析仪器有限公司。
1.3 方法
1.3.1 孢子培养
将甘油保存的孢子悬液稀释涂布高氏一号培养基上,28 ℃条件下倒置培养6~8 d。
1.3.2 菌丝体预培养
以107个孢子的接种量,接种到高渗培养基中,30 ℃、200 r/min条件下培养36 h(培养前预培养基中加入体积分数20%的甘氨酸645 μL,5 mol/L的MgCl2•6H2O 50 μL)。
1.3.3 原生质体制备及再生
菌丝体培养36 h后,取菌悬液3 000 r/min离心10 min,然后弃去上清,用10.3%蔗糖溶液清洗2 次后(记作A),加入含6 mg/mL溶菌酶的P Buffer溶液,混合均匀,置于30 ℃条件下的水浴锅中孵育,每隔15 min缓慢上下颠倒5 次,用相差显微镜观察菌悬液,当出现大量原生质体,仅含少量菌丝体时,将菌液3 000 r/min离心7 min,然后弃去上清,用P Buffer重悬清洗2 次,此时仍然有部分未完全酶解的菌丝体(记作B),500 r/min离心15 min取上层溶液即得到纯度较高的原生质体(D),保存于-80 ℃低温冰箱。
原菌液(A)用10.3%的蔗糖溶液10 倍梯度稀释。原生质体菌丝混合液(B)及原生质体(D)用P Buffer 10 倍梯度稀释,并分别做水处理对照,分别记作C和D,取合适浓度的稀释液于R2YE再生培养基再生;28 ℃条件下倒置培养,8~10 d后进行菌落计数。
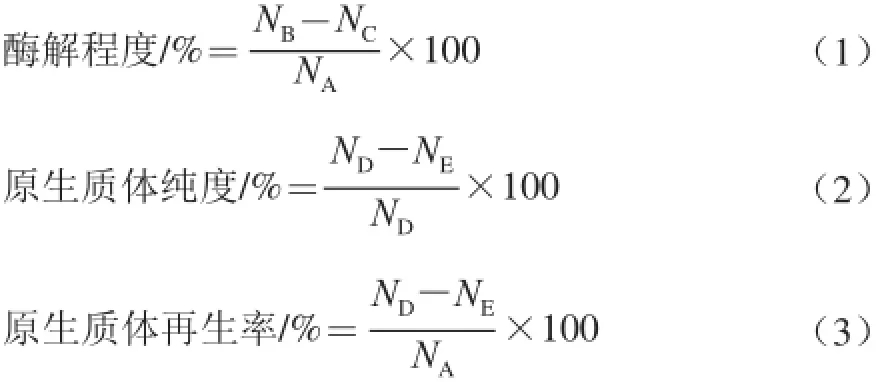
式中:NA、NB、NC、ND、NE分别为1.3.3节中菌液A、B、C、D、E再生后的菌落总数/(CFU/mL)。
1.3.4 原生质体融合
不同茂原链霉菌菌株制备成纯度较高的原生质体,然后取1 mL等量混合,3 000 r/min离心7 min后弃上清,用200 μL P Buffer重悬混合均匀,再加入800 μL含50% PEG2000的P Buffer高渗溶液,混合均匀后置于30 ℃恒温水浴锅中孵育10 min,立即用P Buffer 10 倍梯度稀释涂布到再生培养基平板上,并同时用水处理作对照,28 ℃条件下倒置培养8~10 d。
1.3.5 发酵培养
96 孔板发酵:用牙签刮取再生菌落,加入到96 孔板(每孔含150 μL发酵培养基)中,同时将每个菌落对应扩增,96 孔板200 r/min,30 ℃条件下培养4 d;试管发酵,扩增后的菌落产生孢子后,刮取一定量孢子加入到含有3 mL发酵培养基的试管(24 mm×110 mm)中,200 r/min、30 ℃条件下培养48 h;摇瓶发酵,取平板孢子总量的1/4接种到种子培养基中,30 ℃、200 r/min条件下培养24 h,以10%的接种量接入到发酵培养基中,200 r/min、30 ℃条件下培养。
1.3.6 酶活力测定
采用氧肟酸比色法[29]:一个酶活力单位定义为恒温37 ℃,孵育1 min后谷氨酰胺转氨酶催化底物苯甲基氧化碳酰-L-谷氨酰胺甘氨酸(benzyloxycarbonyl-L-glutaminyl glycine,CBZ-Gln-Gly)生成1 μmol L-谷氨酸-γ-单羟氧肟酸所需要的酶量(U)。
1.4 数据分析
采用GraphPad Prism 5、Origin 9.0、SPSS 20进行软件作图和数据分析。
2 结果与分析
2.1 原生质体的制备
在溶菌酶的作用下茂原链霉菌失去细胞壁形成大量的原生质体,但有部分难以破壁的菌丝体未形成原生质体,从而存在于菌悬液中。在低渗溶液中小分子极易透过细胞膜进入原生质体,导致原生质体胞内渗透压升高,致使原生质体破碎。相差显微镜观察培养36 h后的菌丝及原生质体形态,如图1a所示,茂原链霉菌菌丝成丝状均匀分布,相互交联;经过溶菌酶的处理后,菌丝体失去细胞壁变成大量的原生质体,图1b可以观察到产谷氨酰胺转氨酶茂原链霉菌的原生质体,成球状大小不均匀,与丝状的菌丝体有显著性差异。而经过水处理的原生质体由于胞内渗透压过高,致使细胞膜破碎,图1c中只能观察到细胞碎片和少量未被溶菌酶破壁的菌丝体,可见原生质体制备效果良好。

图1 茂原链霉菌丝和原生质体显微镜图(×1 000)Fig.1 Micrograph of Streptomyces mobaraensis and protoplasts (× 1000)
2.2 原生质体的再生
将获得的原生质体用P Buffer溶液稀释成10 倍和1 000 倍,并以水处理1 000 倍为对照组。由图2a可知,原生质体稀释10 倍后涂布再生,可观察到大量直径较大的菌落存在,菌落直径较小的数量远少于图2b中观察到的结果,推测是由于原生质体没有细胞壁,在再生过程中生长较慢,稀释倍数小的菌液中(图2a)含细胞壁的菌丝体数量较多,生长较快从而影响原生质体的再生,甚至抑制原生质体再生,致使直径较小的菌落数量快速下降。由图2b可知,原生质体稀释1 000 倍后可以观察到大量直径较小的菌落,少量直径较大的菌落。由图2c可知,原生质体用水稀释至1 000 倍后再生出的菌落的直径均比较大。可能是原生质体不含细胞壁,在低渗透势的溶液中,容易致使破碎,失去再生能力,经水处理后大量原生质体无法再生,而含有细胞壁的菌丝体不受渗透势的影响可正常再生。推测直径较小的菌落是由原生质体再生出来,直径较大的菌落是由未被酶解的菌丝体生成的。
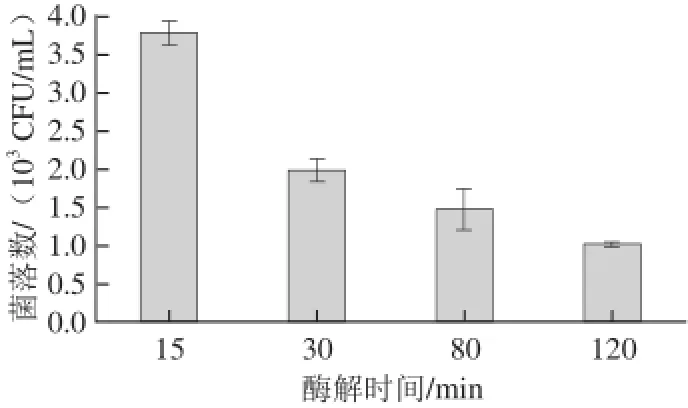
图3 酶解时间对直径较大菌落再生的影响Fig.3 Effect of enzymatic digestion time on regeneration of larger-diameter colony
以6 mg/mL的溶菌酶处理,以酶解时间为单一变量制备原生质体,考察再生后直径较大菌落的总菌落数。如图3所示,酶解时间在一定范围时,直径较大菌落的总菌落数不断下降,80 min以后再生出的直径较大菌落数量下降缓慢,趋于稳定,这表明酶解超过一定时间后,溶菌酶的处理不再使原生质体数量增加。同时用相差显微镜观察不同酶解时间的菌悬液,发现随着酶解时间延长,原生质体数量不断增加,菌丝体数量不断减少,当酶解90 min后,原生质体数量变化不明显。实验确定了茂原链霉菌原生质体制备的酶解时间在90~120 min,并选择酶解100 min制备原生质体进行融合。
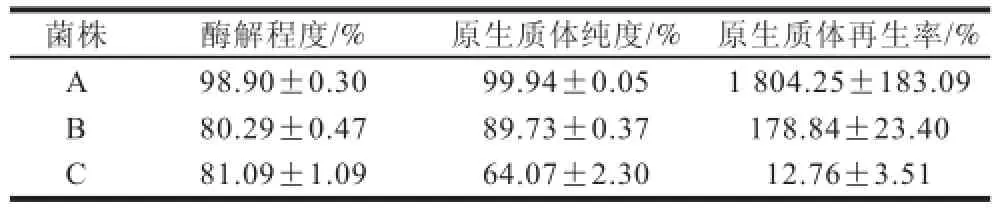
表1 不同来源茂原链霉菌原生质体制备及再生Table1 Protoplasts formation and regeneration of different mutant strains of Streptomyces mobaraensis
同一株茂原链霉菌紫外化学诱变后,从菌株库中选取酶活性有差异的3 株菌,以相同的条件制备原生质体。由表1可知,3 株不同的菌株之间有很大的差异。菌株A很容易酶解,而菌株B和C酶解程度较A低,推测诱变后不同的细胞壁成分有所差异,影响了溶菌酶对细胞壁的酶解过程。制备得到的原生质体纯度也是菌株A最高,菌株B次之,菌株C最差。得到的原生质体再生,其再生率也有所不同,其中以菌株A最好,高达1 804.25%,菌株B再生率达到178.84%,而菌株C再生率相当差,仅仅只有12.76%。推测是由于在预培养的过程中一些菌株的菌丝易形成孢子,溶菌酶处理后,每个孢子都能形成一个原生质体,便可观察到原生质体的再生率达到1 804.25%,同时也有一些菌株的细胞壁成分可能有所不同,致使溶菌酶难以破壁,难以制备成原生质体。不同来源菌株原生质体制备及再生的差异极大,这对基因组重排技术中原生质体的制备增加了难度。
2.3 不同直径大小的再生菌落产酶比较

图4 两种不同直径大小的菌落产酶比较Fig.4 Comparison of TGase activity between two different diameter colonies
由图4可知,直径较大的菌落平均酶活力为2.21 U/mL,直径较小菌落的平均酶活力为1.81 U/mL。运用对两组数据进行独立t检验,得到P=0.006<0.05,表明两组酶活力有差异显著。表明菌落直径大小对酶活力有影响,即茂原链霉菌经过溶菌酶处理后对其产酶有显著影响。
2.4 原生质体融合在茂原链霉菌育种的初探

图5 PEG 2000处理后的原生质体(×1 000)Fig.5 Protoplasts after PEG 2000 treatment (× 1000)
不同菌株的原生质体等量混合,用PEG 2000处理促进原生质体融合,在相差显微镜下观察,结果见图5。如图5a所示,原生质体在PEG 2000的作用下汇集在一起两两聚集,或者大量聚集在一起成团。图5b中可以观察到原生质体聚集在一起后,细胞膜发生融合,形成比正常原生质体大的细胞,原生质体发生融合后,发生基因组交叉互换,形成具有新特性的菌株,能够增加菌株的遗传多态性。
同一菌株进行紫外化学诱变后,从菌种库中选取4 株酶活力和菌落形态有差异的菌株(亲本1、亲本2、亲本3、亲本4)进行原生质体融合,再生后选取直径较小的再生菌落进行96 孔板发酵,30 ℃、200 r/min条件下培养 4 d后测酶活力,选取其中酶活力最高的3%进行试管复筛(图6),可以看到其中有32.2%的菌株的酶活力高于菌株亲本2,其中菌株58比对照中酶活力最高的亲本2提高了22.4%,比亲本3提高了249.48%。对高产菌株与亲菌株的酶活力进行t检验,P值为0.037<0.05,这说明高产菌株与亲本的酶活力有显著性差异。
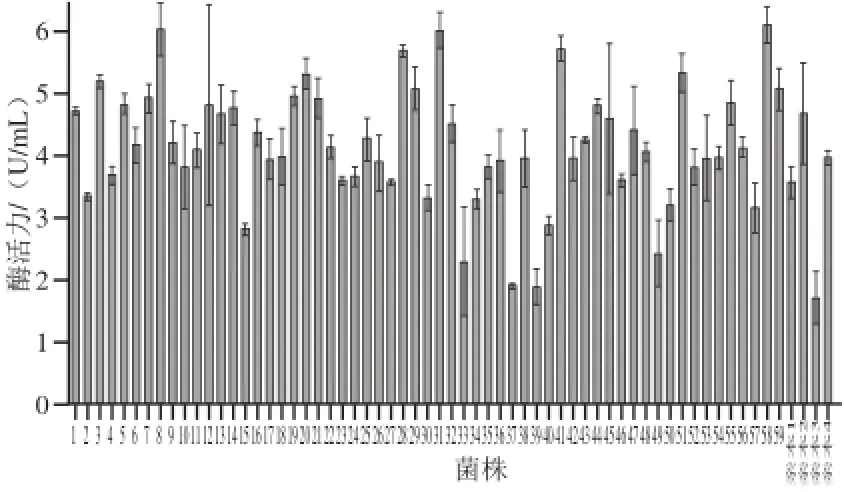
图6 96 孔板初筛高产酶活力菌株试管发酵Fig.6 TGase activities of high-yield strains from 96-well plate screening in tube fermenta tion
在试管发酵中通过接种环接种,接种量的控制精确度低于摇瓶发酵中接种量控制,试管发酵酶活力存在一定偏差,从而运用摇瓶发酵验证高产菌株。试管发酵中共含有19 株菌,酶活力高于产酶最高的亲本2,选取菌株58与亲本2进行摇瓶发酵验证,如图7所示,两株菌在36 h时酶活力达到最高,菌株58为6.1 U/mL,比对照亲本2提高了16.28%,表明基因组重排技术能够有效地提高茂原链霉菌的谷氨酰胺转氨酶产量,筛选更加优良的菌株,为工厂生产降低了成本。

图7 高产菌株和亲本菌株发酵曲线Fig.7 Fermentation curves of high-yield strain and parental strain
3 结 论
通过本研究,可以看到在产谷氨酰胺转氨酶茂原链霉菌的原生质体制备中,溶菌酶处理后,原生质体不含细胞壁,生长缓慢,再生出的菌落直径较小。而菌丝体再生后的菌落直径较大。两种不同直径大小的菌落进行96 孔板发酵培养,发现它们的平均酶活力不同,经t检验得到P=0.006<0.05,说明两种直径大小的菌落产酶差异显著。在基因组重排菌株选育中,可以通过直径大小的筛选,减小亲本对筛选的影响,提高高通量筛选效率。
在酶解时间实验中,确定茂原链霉菌溶菌酶处理时间为90~120 min,并选择100 min作为原生质体制备的酶解时间。以相同的制备条件处理不同的3 株菌,发现酶解程度均可达到80%以上,原生质体纯度差异明显,在60%~100%之间,不同菌株原生质体的再生率差异极大,最高可达1 804.25%,最低仅为12.76%。基因组重排的过程中,需要不同来源的茂原链霉菌作为亲本,增加遗传的多样性,由于原生质体制备条件及再生情况不同,基因组重排便增加了难度。
在原生质体融合中, 选取4 株诱变后的茂原链霉菌制备原生质体,进行谷氨酰胺转氨酶高产菌株的选育。96 孔板高通量筛选后,选择前3%的菌株进行试管发酵,在试管发酵中有32.2%的菌株高于对照组中产酶最高的亲本2,对获得高产菌株与亲本菌株进行t检验,具有十分显著的差异。选取菌株58和亲本2进行摇瓶发酵验证,发现这两株菌同在36 h有最高酶活力,高产菌株58比亲本2提高16.28%。表明了原生质体融合技术在产谷氨酰胺转氨酶茂原链霉菌菌种改造中具有明显优势,可以运用多轮原生质体融合即基因组重排技术来改造茂原链霉菌,筛选高产谷氨酰胺转氨酶的菌株。但不同的菌株原生质体制备及再生条件要求不同,在茂原链霉菌的基因组重排技术中仍有很多的问题需要解决。
[1] 李鑫, 赵燕, 李建科. 微生物谷氨酰胺转氨酶对小麦粉品质的影响[J].食品科学, 2013, 34(1): 135-139.
[2] ONYANGO C, MUTUNGI C, UNBEHEND G, et al. Rheological and baking characteristics of batter and bread prepared from pregelatinised cassava starch and sorghum and modifi ed using microbial transglutaminase[J]. Journal of Food Engineering, 2010, 97(4): 465-470. DOI:10.1016/j.jfoodeng.2009.11.002.
[3] JAROS D, JACOB M, OTTO C, et al. Excessive cross-linking of caseins by microbial transglutaminase and its impact on physical properties of acidifi ed milk gels[J]. International Dairy Journal, 2010, 20(5): 321-327. DOI:10.1016/j.idairyj.2009.11.021.
[4] GUYOT C, KULOZIK U. Effect of transglutaminase-treated milk powders on the properties of skim milk yoghurt[J]. International Dairy Journal, 2011, 21(9): 628-635. DOI:10.1016/j.idairyj.2010.10.010.
[5] BASARAN P, BASARAN-AKGUL N, RASCO B A. Dielectric properties of chicken and fish muscle treated with microbial transglutaminase[J]. Food Chemistry, 2010, 120(2): 361-370. DOI:10.1016/j. foodchem.2009.09.050.
[6] MORENO H M, CARBALLO J, BORDERIAS A J. Use of microbial transglutaminase and sodium alginate in the preparation of restructured fi sh models using cold gelation: effect of frozen storage[J]. Innova tive Food Science & Emerging Technologies, 2010, 11(2): 394-400. DOI:10.1016/j.ifset.2009.12.002.
[7] 谢霞, 田江克, 于腾飞, 等. 谷氨酰胺转氨酶交联的原位明胶止血剂体外凝血实验的研究[J]. 明胶科学与技术, 2012, 32(1): 25-27. DOI:10.3969/j.issn.1004-9657.2012.01.004.
[8] MOTOKI M, SEGURO K. Transglutaminase and its use for food processing[J]. Trends in Food Science & Technology, 1998, 9(5): 204-210. DOI:10.1016/S0924-2244(98)00038-7.
[9] KASHIWAGI T, YOKOYAMA K, ISHIKAWA K, et al. Crystal structure of microbial transglutaminase from Streptoverticillium mobaraense[J]. The Journal of Biological Chemistry, 2002, 277(46): 44252-44260. DOI:10.1074/jbc.m203933200.
[10] ZHU Y, RINZEMA A, TRAMPER J, et al. Microbial transglutaminase: a revie w of its production and application in food processing[J]. Applied Microbiology and Biotechnology, 1995, 44(3/4): 277-282. DOI:10.1007/BF00169916.
[11] MOTOKI M, KUMAZAWA Y. Recent research trends in transglutaminase technology for food processing[J]. Food Science and Technology Research, 2000, 6(3): 151-160. DOI:10.3136/fstr.6.151.
[12] 黄六容, 何冬兰, 刘梅芳. 产谷氨酰胺转胺酶菌株的筛选及其产酶条件研究[J]. 华中农业大学学报, 2005, 24(4): 369-372. DOI:10.3321/j.issn:1000-2421.2005.04.011.
[13] 燕国梁, 堵国成, 陈坚. 谷氨酰胺转氨酶发酵条件的优化研究[J]. 工业微生物, 2003, 33(1): 4-8. DOI:10.3969/ j.issn.1001-6678.2003.01.002.
[14] 张莹, 郭琛琛, 黑东燕, 等. 复合诱变法选育谷氨酰胺转氨酶高产菌株[J]. 食品科学, 2014, 35(11): 139-142. DOI:10.7506/spkx1002-6630-201411028.
[15] 田沛霖. 产谷氨酰胺转氨酶的茂原链霉菌的发酵优化及诱变育种[D].上海: 华东师范大学, 2013: 17-25.
[16] 柏映国, 燕国梁, 堵国成, 等. Streptomyces hygroscopicus产谷氨酰胺转氨酶摇瓶发酵条件的优化[J]. 食品与发酵工业, 2004, 30(2): 27-31. DOI:10.3321/j.issn:0253-990X.2004.02.007.
[17] 田丰伟, 程传荣, 袁维涵, 等. 原生质体诱变选育ε-聚赖氨酸高产菌株[J]. 微生物学通报, 2010, 37(10): 1457-1461.
[18] 王丽宁, 赵妍, 张宝粉, 等. 利用原生质体紫外诱变技术选育耐高温香菇菌株[J]. 微生物学通报, 2014, 41(7): 1350-1357. DOI:10.13344/ j.microbiol.china.140226.
[19] ZHANG Y X, PERRY K, VINCI V A, et al. Genome shuffl ing leads to rapid phenotypic improvement in bacteria[J]. Nature, 2002, 415: 644-646. DOI:10.1038/415644a.
[20] PATNAIK R, LOUIE S, GAVRILOVIC V, et al. Genome shuffl ing of Lactobacillus for improved acid tolerance[J]. Nature Biotechnology, 2002, 20(7): 7 07-712. DOI:10.1038/nbt0702-707.
[21] WANG Y H, LI Y, PEI X L, et al. Genome-shuffl ing improved acid tolerance and L-lactic acid volumetric productivity in Lactobacillus r hamnosus[J]. Journal of Biotechnology, 2007, 129(3): 510-515. DOI:10.1016/j.jbiotec.2007.01.011.
[22] YU L, PEI X L, LEI T, et al. Genome shuffling enhanced L-lactic acid production by improving glucose tolerance of Lactobacillus rhamnosus[J]. Journal of Biotechnology, 2008, 134(1): 154-159. DOI:10.1016/j.jbiotec.2008.01.008.
[23] YU G H, HU Y S, HUI M M, et al. Genome shuffl ing of Streptomyces roseosporus for improving daptomycin production[J]. App lied Biochemistry and Biotechnology, 2014, 172(5): 2661-2669. DOI:10.1007/s12010-013-0687-z.
[24] LUO J M, LI J S, LIU D, et al. Genome shuffling of Streptomyces gilvosporeus for improving natamycin production[J]. Journal of Agricultural and Food Chemistry, 2012, 60(23): 6026-6036. DOI:10.1021/jf300663w.
[25] LV X G, JIN Y Y, LI Y D, et al. Genome shuffl ing of Streptomyces viridochromogenes for improved production of avilamycin[J]. Applied Microbiology and Biotechnology, 2013, 97(2): 641-648. DOI:10.1007/ s00253-012-4322-7.
[26] WANG C, ZHANG X L, CHEN Z, et al. Strain construction for enhanced production of spinosad via intergeneric protoplast fusion[J]. Canadian Journal of Microbiology, 2009, 55(9): 1070-1075. DOI:10.1139/w09-064.
[27] BALTZ R H, MATSUSHIMA P. Protoplast fusion in Streptomyces: conditions for effi cient genetic recombination and cell regeneration[J]. Journal of General Microbiology, 1981, 127(1): 137-146. DOI:10.1099/00221287-127-1-137.
[28] KIESER T, BIBB M J, BUTTNER M J, et al. Practical streptomyces genetics[M]. England: John Innes Foundation Norwich, 2000: 33-67.
[29] GROSSOWICZ N, WAINFAN E, BOREK E, et al. The enzymatic formation of hydroxamic acids from glutamine and asparagine[J]. Journal of Biological Chemistry, 1950, 187(1): 111-125.
Strain Improvement by Protoplast Fusion for Enhanced Transglutaminase Production
HOU Xiaolun1, LIU Yaqing1, GUO Weiting1, GAO Hongliang1, CHANG Zhongyi1, BU Guojian2, LU Wei2, XIE Xiujuan2, JIN Mingfei1,*
(1. School of Life Sciences, East China Normal University, Shanghai 200241, China; 2. Taixing Dongsheng Food Co. Ltd., Taixing 225411, China)
Objective: To obtain a high-yield transglutaminase (TGase)-producing str ain through protoplast fusion between mutant strains of Streptomyces mobaraensis. Methods: Protoplasts were obtained by lysozyme digestion, and after protoplast fusion, the selected fusants were screened for TGase production by primary high-throughput screening in 96-well plates and fermentation in test tubes and shake flasks, respectively. Results: The coloni es regenerated from prot oplasts had a smaller diameter, and the diameter of colonies formed by mycelia was larger. The two different colonies had a significant difference in TGase activity during fermentation in 96-well microplates. Different mutant strains of Streptomyces mobaraensis were very different with respect to TGase activity, protoplast purity and protoplast regeneration rate. The maximum protoplast regeneration rate was 1 804.25%, and the minimum was only 12.76%. After genome shuffling, we selected the top 3% of f usant strains with high TGase activity through high-throughput screening in 96-well plates to carry out fermentation i n test tubes. Of these strains, 32.2% were high-yield strains having a 22.4% higher TGase activity than the parental strain. The production was in creased by 16.28% in shake flask fermentation. Conclusion: Genome shuffling can be used to improve Streptomyces mobaraensis for enhanced TGase production.
Streptomyces mobaraensis; protoplasts; regeneration; fusion; genome shuffling
10.7506/spkx1002-6630-201603027
Q939.97
A
1002-6630(2016)03-0145-06
侯孝仑, 刘雅清, 郭玮婷, 等. 原生质体融合提高产谷氨酰胺转氨酶菌株产量[J]. 食品科学, 2016, 37(3): 145-150. DOI:10.7506/spkx1002-6630-201603027. http://www.spkx.net.cn
HOU Xiaolun, LIU Yaqing, GUO Weiting, et al. Strain improvement by protoplast fusion for enhanced transglutaminase production[J]. Food Science, 2016, 37(3): 145-150. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201603027. http://www.spkx.net.cn
2015-03-19
中央高校基本科研业务费专项资金项目(78210203)
侯孝仑(1989—),男,硕士研究生,研究方向为应用微生物。E-mail:houxiaolun@126.com
*通信作者:金明飞(1979—),男,讲师,博士,研究方向为应用微生物。E-mail:mfjin@bio.ecnu.edu.cn

