SETDB1在乳腺癌中的表达及对癌细胞株增殖和迁移能力的影响
王小利,许德英,刘 刚,常永超
SETDB1在乳腺癌中的表达及对癌细胞株增殖和迁移能力的影响
王小利1,许德英1,刘 刚2,常永超1
目的 研究组蛋白甲基化酶SETDB1在乳腺癌中的表达情况及对其癌细胞株增殖和迁移能力的影响。方法
乳腺癌;SETDB1;基因沉默;细胞增殖;细胞迁移
世界卫生组织最新统计数据显示,乳腺癌在全世界女性中成最高发的癌症,而中国乳腺癌亦位于女性发病率第一位,故探讨乳腺癌的发生发展机制,寻找新的治疗靶点与药物,对其诊断和预后评估具有重大意义。SETDB1(SET domain bifurcated 1)又称ESET或KMT1E,是一种组蛋白赖氨酸甲基转移酶(histone lysine methyltransferases,HMT),属Suvar3-9家族。SETDB1在染色体结构中由高度保守的150个氨基酸序列结合而成[1],其编码的蛋白质位于人染色体1q21[2],可促使组蛋白H3-K9甲基化[3],这种甲基化作用可作为一种异染色质蛋白1(heterochromatin protein 1,HP1)甲基化组蛋白表观遗传转录抑制的标签[4]。已被证实SETDB1在肺癌[5]、黑色素瘤[6]、前列腺癌[7]、肝癌[8]等多种肿瘤中高表达,目前有报道[9]SETDB1在乳腺癌细胞株中亦高表达,但在乳腺癌组织及细胞功能学方面研究尚未见报道。该研究拟首先在乳腺癌组织及细胞株中检测其表达情况,再通过转染手段检测其被沉默后对癌细胞株增殖和迁移能力的影响。
1 材料与方法
1.1 临床标本及细胞系来源 收集河南科技大学第一附属医院乳腺科原发性乳腺癌及癌旁(距病灶≥2 cm以上)组织38例,所有样本经本院病理中心确诊,采集均经过患者及家属知情同意,所有患者术前未行放化学等治疗,无其他原发肿瘤;此38例患者年龄34~81(52.97±12.14)岁。T47D、ZR75-1、ZR75-30、BT549、MDA-MB-453、HBL100共6株细胞系均为复旦大学肿瘤医院乳腺癌研究所赠送(其中HBL-100为乳腺上皮细胞株,余为乳腺癌细胞株),293T细胞株为实验室自备。
1.2 主要试剂 2×SYBR Green qPCR Mix(瑞士Roche公司);SETDB1及GAPDH引物(上海生工生物公司);RPMI 1640、DMEM培养基及胎牛血清(FBS)(以色列BI公司);SETDB1(H300)抗体(美国Santa Cruz公司);Alexa Fluor 488荧光抗兔IgG Fab2(美国Cell signaling公司);流式细胞周期试剂盒(德国Miltenyi公司)。
1.3 主要仪器 实时荧光定量PCR仪(美国ABI-7500公司);凝胶成像系统(美国Bio-Rad-Universal HoodⅡ公司);全自动酶标仪(美国Thermo-354公司);荧光显微镜(日本Nikon-TI-DH公司);流式细胞仪(美国BD-FACS Calibur公司)。
1.4 细胞培养 常规培养人乳腺细胞系,ZR75-1、ZR75-30、HBL100用含10%FBS的RPMI 1640培养基,T47D、BT549、MDA-MB-453及293T用含10% FBS的DMEM培养基,置于37℃、5%CO2饱和湿度培养箱,取对数生长期细胞进行试验。
1.5 慢病毒包装稳定转染 SETDB1 4个shRNA干扰序列分别为GGGTATCTCTATGGAGGAACT、GCATGCGAATTCTGGGCAAGA、GCTCAAGAGTGGCCAGCTTAT、GCGGTTGACAGTGATGATATC,利用293T细胞对shRNA表达质粒及含GFP的对照组质粒进行慢病毒包装,将收获的24 h和48 h病毒加入刚贴壁的每孔约5×105个目的细胞的6孔板中,观察荧光,待出现荧光细胞数达50%以上时用嘌呤霉素(1 μg/ml)筛掉未感染细胞,以此克隆出沉默靶基因的细胞,提取总蛋白及RNA检测SETDB1表达,筛选出沉默效果最佳的两组克隆细胞,进行下一步实验。
1.6 RT-PCR实验 提取38例癌及癌旁组织和融合度至80%~90%的细胞株中总RNA,取1 μg RNA样品20 μl体系行逆转录反应,条件为:42℃、60 min,25℃、5 min,70℃、5 min,4℃、5 min;取1 μl cDNA产物进行RT-PCR反应,体系为20 μl,反应条件为:95℃、15 min 1个循环预变性,之后95℃、10 s,60℃、32 s,共40个循环。荧光定量PCR引物由Primer 5.0软件设计(以GAPDH为内参),序列如下:SETDB1上游引物:AAGACCAGAAGCTCCGTGAA,下游引物:CCTGGGAACTGCTCTTCTTG;GAPDH上游引物:CTCTCTGCTCCTCCTGTTCGAC,下游引物:TGAGCGATGTGGCTCGGCT。以HBL-100细胞株作对照,用2-ΔΔCt法计算SETDB1相对表达量,实验重复3次,选择表达量最高的细胞株用以沉默SETDB1基因,行后续实验。
1.7 IHC实验 取38例患者癌及癌旁石蜡组织切片,经烤片、脱蜡、水化、抗原修复,滴加过氧化物酶阻断剂,经血清封闭后加一抗SETDB1(1∶100稀释)过夜,翌日用PBS清洗过后加二抗结合及链霉卵白素工作液,然后DAB染色至细胞核着色时终止反应,再经苏木精复染,盐酸酒精分化,返蓝、脱水、透明、封片,由两位病理医师阅片。
1.8 Western blot实验 收获融合度80%~90%的细胞,加入250~500 μl蛋白裂解液,提取总蛋白定量并变性,经8%SDS-PAGE电泳、转膜、5%脱脂牛奶封闭,孵育SETDB1一抗4℃过夜,室温孵育二抗2 h,TBST洗膜后ECL显影,凝胶成像系统检测,实验重复3次,Image Lab软件分析目标条带的分子量和光密度值,用以检测SETDB1蛋白水平上的相对表达情况。
1.9 免疫荧光(IF)实验 计数5×103个细胞接种于载玻片培养瓶,待细胞平铺均匀时,固定、通透、封闭后孵育SETDB1一抗4℃过夜,PBS洗3次,加荧光二抗室温避光孵育2 h,DAPI染核,荧光观察,PBS及蒸馏水洗后封片,荧光拍照。
1.10 CCK-8检测细胞活性 制备单细胞悬液,调整细胞浓度为3×104个/ml,每孔加100 μl细胞悬液接种于96孔板,行常规培养,酶标仪测吸光度(optical density,OD)值,前4 h每孔加10 μl CCK-8溶液,在450 nm波长处测其OD值,共设置7个时间检测点,即第1、3、5、7、9、11、12天,每组每个检测点设6个复孔,绘制生长曲线。
1.11 流式细胞术检测细胞周期 每孔2×106个细胞接种于6孔板,待长至对数生长期后收获细胞,经固定、洗涤、计数,使每管细胞量约1×106个,加入400 μl溴化乙锭(PI,50 μg/ml)及100 μl RNase A(100 μg/ml),4℃避光孵育30 min,上机检测,每组实验重复3次。
1.12 软琼脂克隆形成率 1.2%Argrose与2×完全培养基(20%FBS+2×DMEM+2×抗生素)1∶1混合,加1.5 ml下层胶于6孔板中,室温静置待其凝固;用2 ×完全培养基配好单细胞悬液与0.7%Argrose 1∶1混合,取1 ml铺上层胶,每孔铺1×103个细胞,待上层琼脂凝固后,置于培养箱培养4~5周,间隔2 d补加200 μl完全培养基,每组细胞设置3个复孔。计数视野中>0.05 mm的克隆和所有克隆数,克隆形成率(%)=大于0.05 mm克隆数/接种细胞数×100%。
1.13 干细胞成球率 配制专用培养基,每500 ml DMEM基础培养基中加入10 ml B-27(50×)及终浓度为20 ng/ml的EGF,取处于对数生长期的克隆细胞组及对照组细胞制备单细胞悬液,接种于低吸附6孔板中,每孔约5×103个细胞,每2 d加入1 ml专用培养基,培养12 d后拍照,利用图像以球直径≥50 μm为标准计数成球数,每组细胞设置3个复孔,微球体形成率(%)=球体形成数/接种细胞数× 100%。
1.14 Transwell方法测定迁移能力 选择Corning FlouroBlok基底膜的12孔板Transwell小室,上室加50 μl无血清培养基置于37℃平衡1 h,取5×105个/ml无血清细胞悬液200 μl加至上室,600 μl 20%DMEM培养基加至下室,室温静置30 min。荧光显微镜下(×100)计数滤膜下表面穿膜细胞数,培养48 h后,随机计数5个视野,计算穿膜细胞均数。
1.15 IHC半定量分析 采用学术界公认的方法:①癌细胞阳性细胞数为0、≤25%、26%~50%、51%~75%、>75%,分别计为0、1、2、3和4分;②癌细胞染色强度棕褐色小块状、棕色粗颗粒状、浅棕色细颗粒状、未染色分别计为3、2、1和0分。两项的乘积>3分为免疫反应阳性。
1.16 统计学处理 采用Graph Pad Prism 5.0软件对数据进行分析,计量资料以±s表示,两组均数比较采用t检验,多组均数比较采用方差分析;计数资料采用χ2检验或Fisher's检验。
2 结果
2.1 SETDB1在乳腺癌旁及癌组织和细胞株中的表达情况 IHC结果显示,38例样本中有23例癌组织、13例癌旁组织SETDB1阳性表达,15例癌组织、25例癌旁组织阴性表达(Fisher's检验,P= 0.04),见图1。癌组织中的蛋白表达与患者的临床病理特征关系见表1。RT-PCR检测结果显示,38例乳腺癌与癌旁组织的相对表达量分别为(1.20± 0.08)、(0.96±0.05)(t=2.44,P<0.05),见图2。以HBL-100作对照,余5种乳腺癌细胞株BT549、ZR75-30、ZR75-1、MDA-MB-453和T47D中SETDB1蛋白水平相对表达量分别为(1.30±0.20)、(2.18±0.16)、(1.83±0.06)、(2.59±0.10)和(3.77±0.25),均高于HBL-100细胞株(F=541.20,P<0.001),其中T47D相对表达水平最高,见图3A、3B。RT-PCR结果显示,SETDB1的mRNA相对表达量分别是(1.21± 0.07)、(2.41±0.10)、(4.57±0.17)、(5.06±1.76)、(6.43±0.12),表达最高亦为T47D细胞株,差异具有统计学意义(F=643.30,P<0.05),见图3C。故在细胞功能学试验中选取T47D细胞株。

图1 SETDB1在癌组织、癌旁组织中表达情况 SP×400
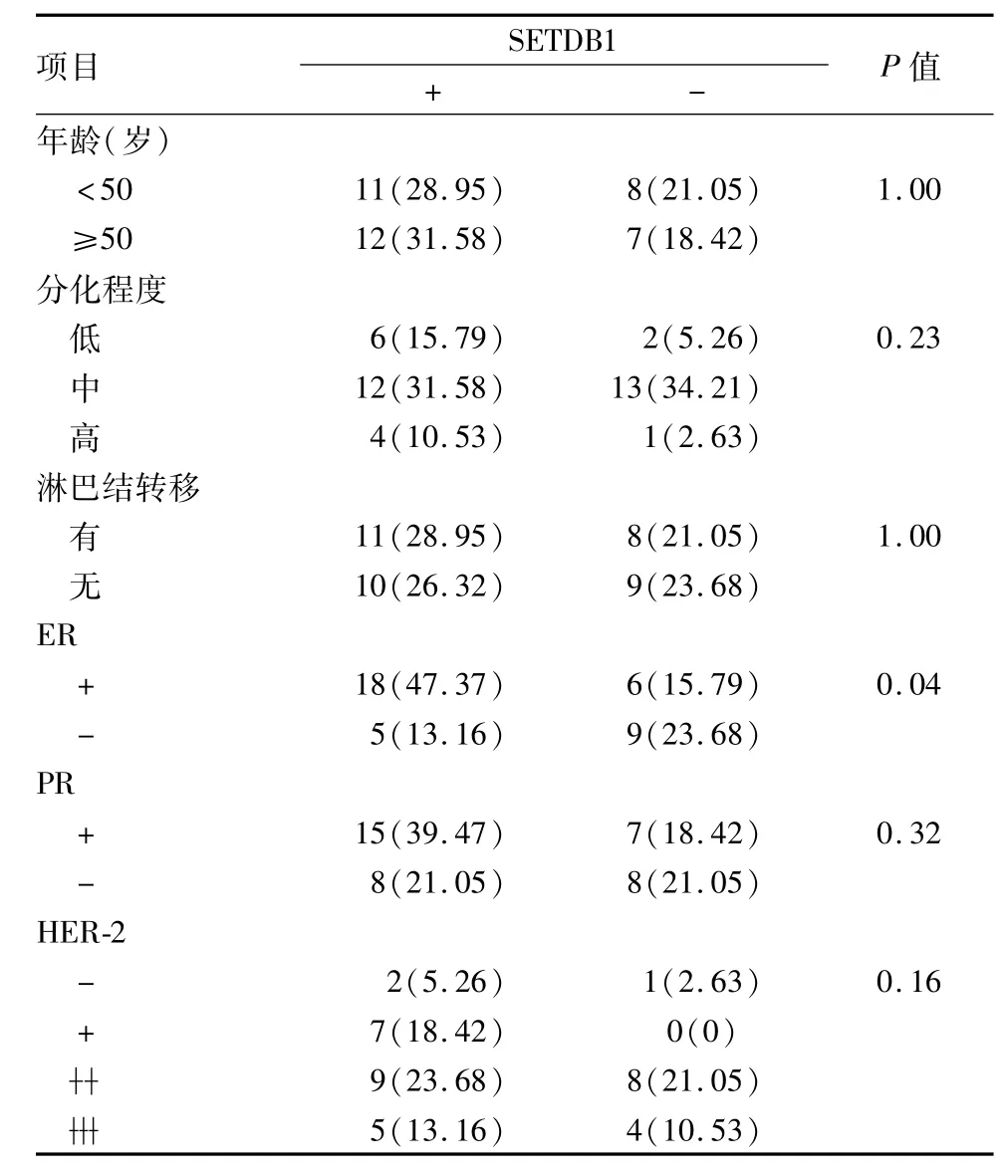
表1 SETDB1蛋白在乳腺癌组织中表达与患者临床病理学特征[n(%)]
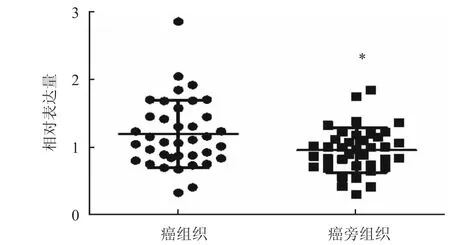
图2 SETDB1在38例配对组织中mRNA水平表达情况
2.2 在T47D细胞中沉默SETDB1基因后其表达情况 利用慢病毒转染RNAi技术筛选出沉默SETDB1基因的克隆细胞株,以GFP株为对照组,在蛋白水平,sh1、sh2、sh3、sh4的相对表达量依次是(0.48±0.03)、(0.36±0.03)、(0.54±0.05)、(0.22±0.03),相对表达水平亦降低(F=181.50,P<0.05),见图4A、4B;在mRNA水平,4个沉默克隆组sh1、sh2、sh3、sh4相对表达水平均明显降低,表达量依次为(0.28±0.01)、(0.39±0.05)、(0.39± 0.01)、(0.34±0.02),差异具有统计学意义(F= 56.70,P<0.05),见图4C;故综合选择克隆株sh2、sh4做下游细胞功能学实验。

图3 乳腺细胞株中SETDB1蛋白水平相对表达量
2.3 SETBD1在乳腺癌细胞中表达定位 T47D细胞IF实验可明显观察到SETDB1的表达在细胞核及细胞质中均有表达,但以细胞核表达(天青色)为主,见图5。
2.4 沉默SETBD1对乳腺癌细胞增殖能力的影响
利用CCK-8、软琼脂克隆形成及细胞周期实验检测沉默SETDB1对细胞增殖的影响。CCK-8实验结果显示在细胞对数生长期的增殖活性GFP株明显高于sh2株和sh4株,以GFP株作对照,在第5、7、
9、11天即细胞对数生长期时,sh2株和sh4株的增殖活力明显降低(P<0.05)。见图6。软琼脂克隆形成结果显示,GFP、sh2、sh4株形成克隆数目分别是(70.00±2.00)、(42.67±1.53)和(13.33± 1.53)(F=834.00,P<0.001),即沉默SETDB1可抑制癌细胞株的群体依赖性和恶性增殖,见图7。流式检测细胞周期显示sh2株出现了G0/G1期阻滞(t=14.39,P<0.01),而sh4株出现了G2/M期阻滞(t=6.95,P<0.01),见图8。
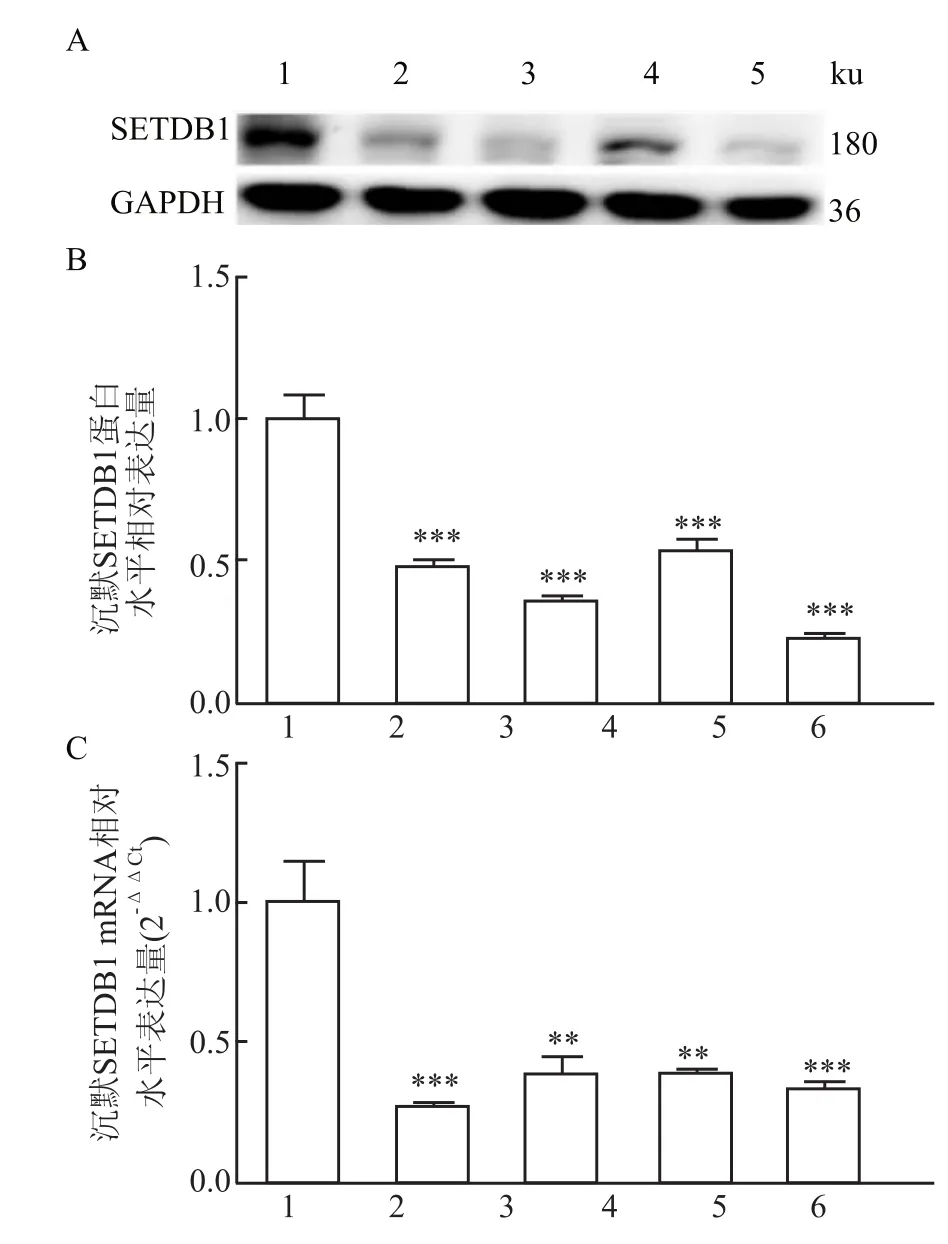
图4 沉默SETDB1基因后其表达情况
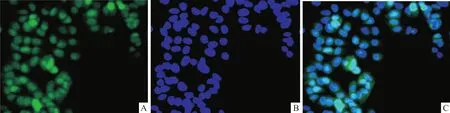
图5 SETDB1在T47D细胞中的免疫荧光表达定位情况 SP×400
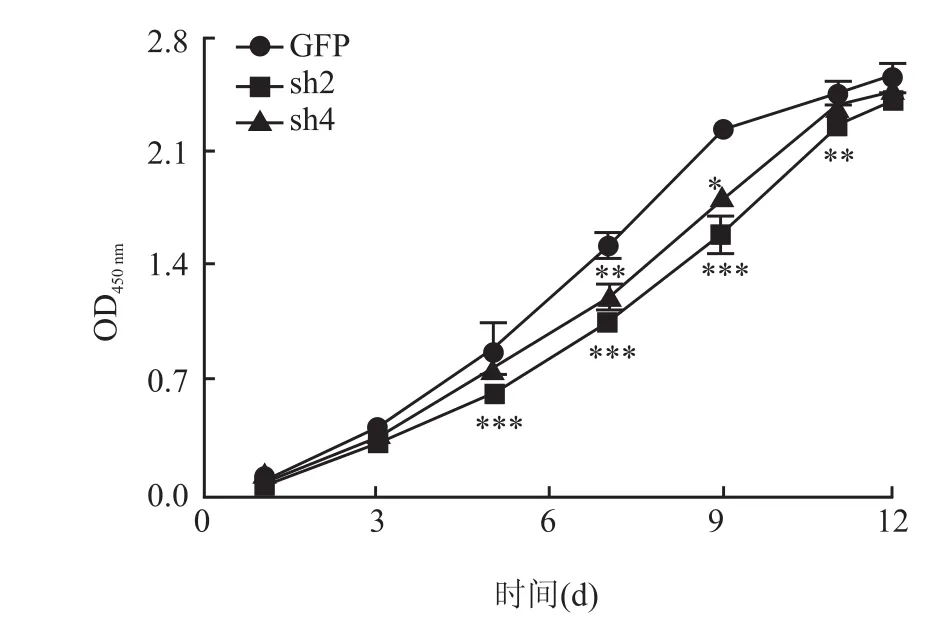
图6 CCK-8法检测克隆细胞株的增殖活力情况
2.5 沉默SETBD1对乳腺癌细胞干细胞形成能力的影响 利用干细胞培养基培养GFP、sh2和sh4克隆细胞株,3者的微球体形成率分别为(4.50± 0.07)%、(2.61±0.05)%和(1.66±0.07)%(F= 493.50,P<0.001),即沉默SETDB1后癌细胞株的干细胞活性和自我更新能力降低,见图9。
2.6 沉默SETBD1对乳腺癌细胞株迁移能力的影响 用荧光倒置显微镜可观察Corning FlouroBlok Transwell小室中穿膜的细胞数,GFP、sh2和sh4克隆细胞株在迁移48 h后,3者的穿膜个数分别是(54.25±1.71)、(16.50±1.29)、(9.00±0.82)个(F=1 344.00,P<0.001),见图10。
3 讨论
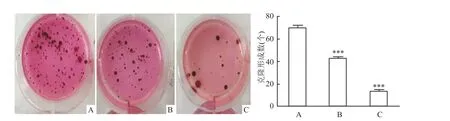
图7 软琼脂克隆形成实验检测克隆细胞株的增殖情况
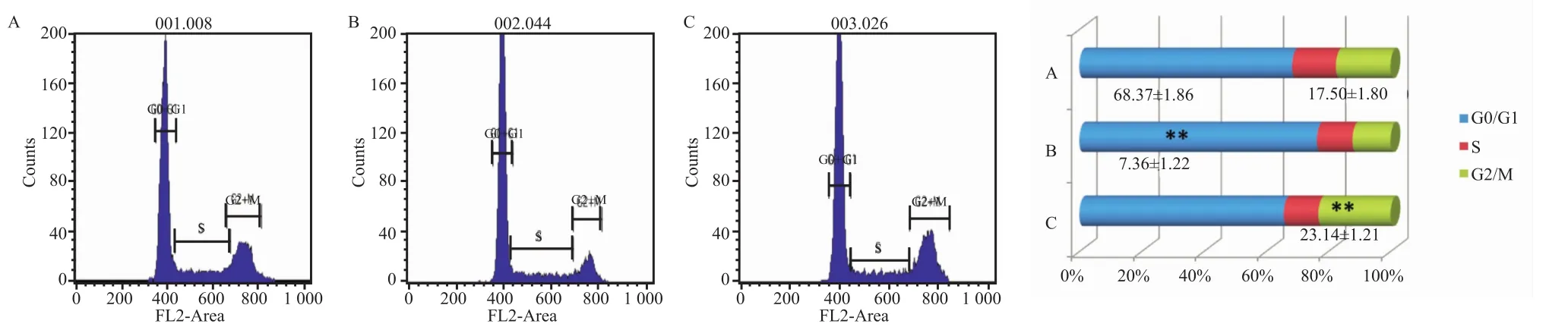
图8 流式细胞术检测克隆细胞株的细胞周期情况

图9 克隆细胞株干细胞形成能力情况 SP×200
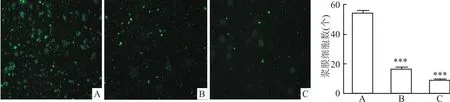
图10 克隆细胞株迁移48 h情况 SP×100
Ryu et al[10]首次报道了在人类“亨廷顿舞蹈症”(Huntington's disease,HD)患者和R6/2转基因HD小鼠模型中SETDB1的异常表达,发现SP1和SP3可作为SETDB1启动子转录激活神经元,而光神霉素和乌洛托品联合药物治疗可下调SETDB1表达;癌症基因组图谱(TCGA)数据库亦显示SETDB1是肿瘤高表达基因中排名最靠前基因之一[11]。本研究显示SETDB1在乳腺癌组织及细胞株中高表达,IF实验亦表明SETDB1细胞核、质均有表达,但以细胞核表达为主,临床病理相关性分析表明,SETDB1阳性表达与患者年龄、分化程度、淋巴结转移情况及病理指标PR、C-erbB-2间差异无统计学意义,但与ER却存在相关性。
在人类肝细胞癌(HCC)细胞株中沉默SETDB1能抑制其增殖、迁移和侵袭能力,临床病理相关性分析表明HCC的不同器官转移灶中SETDB1亦高表达且高表达的患者预后疗效差[8];在肺癌组织及细胞系中SETDB1表达水平增高,沉默该基因则可抑制癌细胞增殖并促进癌细胞凋亡,通过体内实验证实沉默SETDB1后小鼠的成瘤能力减弱[5];本研究亦从细胞功能学方面证实在乳腺细胞株中沉默SETDB1可使癌细胞的增殖、克隆形成、迁移能力显著降低,在细胞周期中沉默SETDB1后前列腺癌细胞株出现G0/G1期阻滞[7],而在本实验中sh2克隆株出现G0/G1期阻滞,sh4克隆株却出现G2/M期阻滞,该现象可能是因为不同的克隆细胞株所使用的shRNA干扰序列不同所导致的。通过实验显示沉默SETDB1后,细胞株形成干细胞的能力显著下降,自我更新能力降低,研究[12-13]显示,SETDB1可维持小鼠胚胎干细胞(embryonic stem cells,ESCs)的自我更新能力,下调SETDB1将会导致ESCs标记OCT4表达降低,而滋养外胚层细胞(trophectoderm,TE)标记如Cdx2和Tcfap2a表达升高,从而加速ESCs向TE分化;且有助于调控ESCs发育过程中的整体H3K9me3甲基化水平,亦参与调控沉默逆转录酶病毒(transcription of endogenous retroviruses, ERVs),特别是ERV-I和II类。SETDB1在人类干细胞形成过程中存在怎样的功能,仍需进一步探索。
本研究推测SETDB1在乳腺癌中扮演癌基因角色,其过表达可能促使乳腺癌的发生发展。研究[8]显示,SETDB1在肝细胞癌中常染色体1q21区域基因扩增,调控多个下游靶基因如IGFBP3、CXCL3和NOS3等,SP1转录因子在转录水平上可促进SETDB1活性,转录后水平上miR-29与SETDB1负相关;经致癌药物光神霉素治疗亦可有效抑制癌细胞中SETDB1的表达水平,基因芯片及qCHIP结果显示在肺癌中可能调控的下游靶基因是ADRB2、ANXA3、DNER、GPR64、NAV3、SGK1及TFAPIA等[5];染色质修饰抗癌药物DZNep(3-Deazaneplanocin A)在转录水平上也可以调控SETDB1启动子活性使其表达降低从而诱导肺癌细胞株凋亡[14];SETDB1调控的靶基因参与多种肿瘤相关的调控通路,包括钙黏蛋白介导的细胞黏附及Wnt信号通路[15]。所以SETDB1参与癌症的发生和进展过程,且具有细胞来源或肿瘤类型的特异性,SETDB1在乳腺癌中的下游调控靶基因以及参与的调控通路还未知,需要进一步探索。
近年来,肿瘤的靶向治疗成为人们研究的热点与难点,而本研究结果将对乳腺癌的靶向治疗提供新的分子生物学依据,且对下一步实验通过基因芯片技术探索其下游靶基因及寻找确切的调控通路奠定坚实基础。
[1] Nomura N,Nagase T,Miyajima N,et al.Prediction of the coding sequences of unidentified human genes.II.The coding sequences of 40 new genes(KIAA0041-KIAA0080)deduced by analysis of cDNA clones from human cell line KG-1[J].DNA Res,1994,1(5):223-9.
[2] Harte P J,Wu W,Carrasquillo M M,et al.Assignment of a novel bifurcated SET domain gene,SETDB1,to human chromosome band 1q21 by in situ hybridization and radiation hybrids[J].Cy-togenet Cell Genet,1999,84(1-2):83-6.
[3] Yang L,Xia L,Wu D Y,et al.Molecular cloning of ESET,a novel histone H3-specific methyltransferase that interacts with ERG transcription factor[J].Oncogene,2002,21(1):148-52.
[4] Wang H,An W,Cao R,et al.mAM facilitates conversion by ESET of dimethyl to trimethyl lysine 9 of histone H3 to cause transcriptional repression[J].Mol Cell,2003,12(2):475-87.
[5] Rodriguez-Paredes M,Martinez de Paz A,Simó-Riudalbas L,et al.Gene amplification of the histone methyltransferase SETDB1 contributes to human lung tumorigenesis[J].Oncogene,2014,33(21):2807-13.
[6] Ceol C J,Houvras Y,Jane-Valbuena J,et al.The histone methyltransferase SETDB1 is recurrently amplified in melanoma and accelerates its onset[J].Nature,2011,471(7339):513-7.
[7] Sun Y,Wei M,Ren S C,et al.Histone methyltransferase SETDB 1 is required for prostate cancer cell proliferation,migration and invasion[J].Asian J Androl,2014,16(2):319-24.
[8] Wong C M,Lai W,Law C T,et al.Up-regulation of histone methyltransferase SETDB1 by multiple mechanisms in hepatocellular carcinoma promotes cancer metastasis[J].Hepatology,2016,63(2):474-87.
[9] Liu L,Kimball S,Liu H,et al.Genetic alterations of histone lysine methyltransferases and their significance in breast cancer[J]. Oncotarget,2015,6(4):2466-82.
[10]Ryu H,Lee J,Hagerty S W,et al.ESET/SETDB1 gene expression and histone H3(K9)trimethylation in Huntington's disease[J].Proc Natl Acad Sci U S A,2006,103(50):19176-81.
[11]Chen Y,McGee J,Chen X,et al.Identification of druggable cancer driver genes amplified across TCGA datasets[J].PLoS One,2014,9(5):e98293.
[12]Cho S,Park J S,Kwon S,et al.Dynamics of Setdb1 expression in early mouse development[J].Gene Expr Patterns,2012,12(5-6):213-8.
[13]Yeap L S,Hayashi K,Surani M A.ERG-associated protein with SET domain(ESET)-Oct4 interaction regulates pluripotency and represses the trophectoderm lineage[J].Epigenetics Chromatin,2009,2(1):12.
[14]Lee J K,Kim K C.DZNep,inhibitor of S-adenosylhomocysteine hydrolase,down-regulatesexpressionofSETDB1H3K9me3 HMTase in human lung cancer cells[J].Biochem Biophys Res Commun,2013,438(4):647-52.
[15]Sun Q Y,Ding L W,Xiao J F,et al.SETDB1 accelerates tumourigenesis by regulating the WNT signalling pathway[J].J Pathol,2015,235(4):559-70.
SETDB1 expression in breast cancer and the effect on the proliferation and migration ability of cancer cells
Wang Xiaoli1,Xu Deying1,Liu Gang2,et al
(1Clinical Laboratory,2Cancer Epigenetics Laboratory,First Affiliated Hospital of Henan University of Science and Technology,Luoyang 471003)
Objective To study the histone methyltransferase SETDB1 expression in breast cancer and the proliferation and migration ability of breast cancer cells.Methods The SETDB1 expression on 38 cases of breast cancer tissues,cancer adjacent tissues and breast cancer cells were detected by RT-PCR,Western blot and IHC.The knockdown SETDB1 gene cell clones were screened out with stable transfection method,and the knockdown SETDB1 tumor cells proliferation ability was detected by CCK-8,clone forming and flow cytometry assay.The migration ability changes were detected by transwell assay.Results In 38 cases,23 cases of carcinoma tissues and 13 cases of tissue adjacent to carcinoma were positively expressed(P<0.05),and the mRNA relative expression levels of SETDB1 were(1.20±0.08),(0.96±0.05)(t=2.44,P<0.05).The formed clone numbers in GFP,sh2,sh4 were(70.00±2.00),(42.67±1.53),(13.33±1.53)(F=834.0,P<0.001).sh2 blocked cell cycle in G0/G1phase,while sh4 was arrested in G2/M phase.The numbers of migration respectively were(54.25± 1.71),(16.50±1.29),(9.00±0.82)(F=1 344.00,P<0.001).Conclusion SETDB1 is frequently overexpressed in breast cancer.Knockdown SETDB1 can inhibit the proliferation and migration capacity of cancer cell,which indicates that SETDB1 is a new target for diagnosis and treatment of breast cancer.
breast cancer;SETDB1;knockdown gene;cell proliferation;cell migration
R 73-37
A
1000-1492(2016)08-1081-07
时间:2016-6-22 14:44:58
http://www.cnki.net/kcms/detail/34.1065.R.20160622.1444.004.html
2016-04-19 接收
国家自然科学基金联合项目(编号:U1204821);国家自然科学基金面上项目(编号:81572794)
河南科技大学临床医学院、河南科技大学第一附属医院1检验科、2肿瘤表观遗传实验室,洛阳 471003
王小利,女,硕士研究生;
刘 刚,男,教授,责任作者,E-mail:liugang72@163.com;常永超,男,教授,硕士生导师,责任作者,E-mail:ychchang123@163.com
采用RT-PCR、Western blot和IHC方法检测38例乳腺癌与癌旁组织及癌细胞株中SETDB1的表达水平;采用稳定转染方法筛选出沉默SETDB1基因的克隆细胞株,采用CCK-8、软琼脂克隆形成及流式细胞术检测沉默SETDB1后对肿瘤细胞增殖能力的影响;采用Transwell方法检测沉默后对肿瘤细胞迁移能力的影响。结果 38例乳腺癌与癌旁组织中23例癌组织、13例癌旁组织阳性表达(P<0.05),SETDB1的mRNA水平相对表达量分别为(1.20±0.08)、(0.96± 0.05)(t=2.44,P<0.05);沉默T47D中SETDB1基因后,GFP、sh2、sh4株形成克隆个数分别是(70.00±2.00)、(42.67±1.53)和(13.33±1.53)个(F=834.00,P<0.001);细胞周期中sh2株出现了G0/G1期阻滞,而sh4株出现了G2/M期阻滞(P<0.01);48 h穿膜个数分别是(54.25±1.71)、(16.50±1.29)、(9.00±0.82)个(F=1 344.00,P<0.001)。结论 SETDB1在乳腺癌中高表达,沉默该基因可影响癌细胞株的增殖和迁移能力,有望作为乳腺癌诊断及治疗的新靶点。

