油茶柠檬酸合成酶(CS)基因的克隆和表达分析
叶思诚 姚小华 王开良 林 萍 龚洪恩 卓仁英
(中国林业科学研究院亚热带林业研究所,富阳 311400)
油茶柠檬酸合成酶(CS)基因的克隆和表达分析
叶思诚 姚小华*王开良 林 萍 龚洪恩 卓仁英
(中国林业科学研究院亚热带林业研究所,富阳 311400)
采用RT-PCR技术从油茶中分离出一个柠檬酸合成酶基因,该基因的cDNA全长1 416 bp,编码471个氨基酸,推导的蛋白分子量为52.74 kD,理论等电点(PI)为6.95。同源比对显示其与其他植物的CS蛋白序列高度同源,将该基因命名为CoCS(Gen Bank登录号:KU161147)。系统进化树分析表明油茶CoCS与杜鹃和葡萄的CS蛋白的亲缘关系较近。荧光定量PCR分析结果表明,油茶受到低磷胁迫后根系CoCS基因的表达受到低磷诱导,表达量呈现先升高后降低的趋势;不同油茶品种不同组织(根、茎、叶)中的CoCS基因在不用磷处理下的表达模式不同。
油茶;柠檬酸合成酶;低磷;基因克隆;表达分析
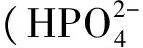
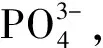
植物体内的柠檬酸合成是由柠檬酸合成酶(citrate synthase,CS,EC4.1.3.7)催化草酰乙酸和乙酰辅酶A合成的。柠檬酸合成酶是三羧酸循环和乙醛酸循环的关键酶,所参与的三羧酸循环和乙醛酸循环能够将体内三大物质氨基酸、糖和脂类的代谢联系起来。柠檬酸合成酶根据其在真核细胞中的定位不同,可分为线粒体CS(mCS)、乙醛酸循环体CS(gCS)、过氧化物酶体CS(pCS)[17]。柠檬酸合成酶与多种生理过程相关,如线粒体能量代谢、种子萌发、耐低磷和抗铝毒等[18~21]。De la Fuente等[22]将绿脓杆菌(Pseudomonasaeruginosa)的柠檬酸合成酶基因(CS)导入烟草(Nicotianatabacum)和木瓜(Chaenomelessinensis)中超表达,结果发现转基因植株根系柠檬酸合成能力比对照提高10倍以上,根系分泌的柠檬酸比对照高出4倍以上。Koyama等[23]研究发现,过表达CS基因的胡萝卜(Daucuscarota)细胞对磷的吸收显著提高。Koyama等[20]在拟南芥(Arabidopsisthaliana)中过表达CS基因,结果发现转基因植株根部分泌的柠檬酸比对照高出3倍,对磷的吸收能力也得到很大提高。López-bucio[24]等研究表明表达CS基因的烟草植株对磷的吸收能力大大增强。王祎[25]等研究表明,油菜过表达CS基因显著提高植株从土壤中获取磷的能力,转基因植株苗期地上部和籽粒中的磷累积量显著高于WT植株。
油茶(CamelliaoleiferaAbel.)是我国特有的木本食用油料树种[26]。油茶林地土壤大多属酸性红壤,土壤中有效磷含量很低,磷缺乏是油茶增产的主要限制因子[27]。低磷胁迫下,油茶根系和组织中分泌的有机酸(含柠檬酸)含量增多[28~29]。研究油茶根系分泌物对土壤和难溶磷化合物(AlPO4,Ca3(PO4)2FePO4·2H2O)中磷的活化作用,结果表明,油茶根系分泌物能够明显促进土壤磷的释放,不同难溶磷化合物在根系分泌物中的溶解性不同,溶解性AlPO4>Ca3(PO4)2>FePO4·2H2O;利用有机酸化学试剂验证其对土壤中P的能力,结果表明柠檬酸的活化能力最强[30]。目前,已知油茶在低磷胁迫下根系合成和分泌的柠檬酸含量升高,但是对催化合成柠檬酸的柠檬酸合成酶基因作用机制尚不清楚。因此,本研究以普通油茶磷高效基因‘长林166号’和磷低效基因型‘长林4号’两个无性系为试验材料,通过分离油茶柠檬酸合成酶基因CS全长cDNA和DNA序列并对其进行生物信息学分析,同时分析不同磷水平下不同无性系不同器官组织中CoCS基因在表达模式及柠檬酸合成酶活性的差异,为进一步探索油茶适应低磷和磷高效利用的分子机制以及磷高效油茶新品种的选育奠定理论基础。
1 材料与方法
1.1 材料
1.1.1 植物材料
试验材料为普通油茶(CamelliaoleiferaAbel.)‘长林4号’和‘长林166号’无性系一年生扦插苗,取自浙江省江山市林业局种苗站采穗圃。选择苗高均在10 cm左右和长势基本一致的无性系植株用于后续实验。
1.1.2 试剂
大肠杆菌(E.coli)DH5α和Topo10感受态细胞购自CWBIO公司(北京,中国)。PMD-18T克隆载体、荧光定量试剂盒SYBR PrimeScriptTMRT-PCR Kit购自TaKaRa公司(大连,中国)。植物RNA快速提取试剂盒(RN38 EASY spin Plus)购自Aidlab公司(北京,中国)。反转录试剂盒(SuperScripTMIII First-STRAND Synthesis System for RT-PCR)购自invitrogen公司(Carlbad,USA)。质粒DNA小量提取试剂盒(AP-MN-P-50)、琼脂糖凝胶DNA回收试剂盒购(AP-GX-50)自Axygen公司(杭州,中国)。超保真DNA聚合酶(PhantaTMEVO HS)、DL15000 DNA Marker购自Vazyme公司(南京,中国),核酸染料DNA GREEN购自TIANDZ(北京,中国)。引物合成和测序由上海生工和擎科生物公司完成。
1.2 方法1.2.1 试验苗木处理
沙培试验:选择‘长林4号’和‘长林166号’无性系一年生扦插苗作为沙培试验的材料。将油茶无性系扦插苗根系用清水冲洗干净,进行适当修剪后用0.15%的福尔马林溶液对其表面灭菌,用蒸馏水清洗后,移植到20 cm×30 cm塑料盆中,在塑料盆底垫块纱布,然后装1 kg 1~2 mm的石英砂(10%稀HCl浸泡一天后洗净)在盆底,上面装2 kg 0.5~1 mm的石英砂,每盆种植3株幼苗,放在温棚中进行培养。对油茶苗进行2个磷水平处理,分为缺磷LP(5 μmol·L-1KH2PO4)、常磷HP(1 mmol·L-1KH2PO4)2个处理水平,每处理6盆,每盆3株,采用完全随机区组设计。营养液配方参照林郑和[31]的方法并稍作修改。营养液中缺少的K+用等量的KCl替代。用1 mol·L-1HCl和1 mol·L-1NaOH把全营养液pH值调整到5.2左右。苗木于2014年4月种下,种下第1个月使用常磷(KH2PO4,1 mmol·L-1)完全营养液浇灌,每隔2天1次,每次200 mL。30天后开始进行低磷和常磷胁迫处理。苗木处理4个月后于2014年8月采集油茶苗的根、茎、叶组织,用锡箔纸包裹好样品立即放入液氮中,保存于-80℃冰箱中备用。
水培试验:选择磷高效基因型‘长林166号’无性系一年生扦插苗作为水培试验材料。将‘长林166号’油茶苗常磷水平沙培一个月,然后将长出新根的油茶苗转移常磷全营养液中进行水培培养。采用塑料筐作为水培的容器,带孔泡沫板作为固定板。将油茶苗固定在泡沫板上,根系浸入营养液中,同时利用氧气泵进行连续充气。每筐定植12棵,共水培4筐。先让油茶苗在常磷全营养液中培养一个星期,使其适应常磷水培环境。一个星期之后,将其中两盆用低磷LP(5 μmol·L-1KH2PO4)营养液进行低磷胁迫处理和另外两盆用常磷HP(1 mmol·L-1KH2PO4)营养液处理作为对照,每5天更换一次营养液。采集低磷胁迫0、12、24、48、72和96 h的根系样品。常磷和低磷水培一个月后,采集两个处理的根系样品。采集的根系样品用锡箔纸包裹立即后放入液氮中,保存于-80℃冰箱备用。
1.2.2 RNA的提取及逆转录
植物组织总RNA的提取按照植物RNA快速提取试剂盒(RN38 EASY spin Plus)说明书进行。用UV-Vis Spectrophotometer Q5000微量分光光度计测定RNA的浓度,并进行1%琼脂糖电泳分析其完整性。参照Invitrogen公司的反转录试剂盒(SuperScripTMIII First-STRAND Synthesis System for RT-PCR)逆转录合成第一链cDNA,合成后的cDNA稀释后用于基因PCR扩增和实时荧光定量PCR。
1.2.3 引物设计与合成
利用Pfam数据库中CS结构域(PF00285)的模型,通过Hmmer3.0本地软件对课题组前期的油茶转录组的数据[32进行比对搜索,筛选获得CS基因相关的Unigenes。利用DNAstar 7.0中Editseq程序和ORF-Finder寻找开放阅读框,并进行blast分析验证,选择含有CS基因完整开放阅读框(ORF)的Unigene作为油茶CS基因研究的参考序列。
利用Primer Premier 5和oligo7设计CS基因的全长引物,上下游引物分别为FP:5′-ATGGTGTTCTTCAGAAGCGTTTC-3′,RP:5′-TTAAGCTGATTTCTTGCAGTAATTTTC-3′,基因组全长扩增引物与cDNA全长引物扩增引物相同;利用primer3设计荧光定量PCR引物,上游为CS F1 5′-GGCTTCTTCCTCTTCACTCA-3′,下游为CS R1 5′-AACTTACACCGAATCCTCAC-3′;利用primer-blast验证引物的的特异性。引物由上海生工和擎科生物合成。
1.2.4油茶CS基因cDNA全长和基因组全长的克隆
以‘长林4号’植物的根系的cDNA为模板,进行PCR扩增cDNA全长。PCR反应体系(50 μL):5×EVO Buffer(with 10 mmol·L-1MgCl2)10 μL,dNTP Mix 1 μL,5×PCR Enhancer 10 μL,模板cDNA 4 μL,上下游引物各2 μL,ddH2O 20 μL。扩增条件:95℃ 3 min;95℃ 15 s,56℃ 15 s,72℃ 1 min,35个循环;72℃ 7 min;4℃保持。以长林4号植物的根系的DNA为模板,进行PCR扩增CS基因基因组全长。PCR反应体系和扩增条件与cDNA全长扩增基本相同。将PCR产物用琼脂糖凝胶DNA回收试剂盒回收,将cDNA全长和基因组DNA全长扩增的回收产物分别与pMD-18T和pTOPO-Blunt Simple克隆载体连接,并转化大肠杆菌(E.coli)DH5α,菌液PCR鉴定后将阳性结果送擎科生物公司测序。
1.2.5 油茶CS基因的生物信息学分析
测序获得的序列利用DNAstar 7.0软件分析基因的ORF,并推导出氨基酸序列;利用NCBI中BLAST程序对测序序列进行比对,搜索同源氨基酸序列;利用Clustal Omeg、DNAMAN7.0和MEGA6.0等软件对同源序列进行多序列比对、系统发育进化树构建;使用MEME数据库分析蛋白质的保守区;利用InterProscan、Motif Scan和PROSITE分析蛋白质的家族、结构域、模体和功能位点;利用ExPASy上的protparam和ProtScal分析蛋白的相对分子质量、等电点理化参数和亲疏水性;利用InterProscan和CCTOP在线工具对跨膜区和跨膜螺旋进行预测;采用SignalP 4.1对信号肽进行预测;利用Plant-PLoc、CELLO v.2.5、WoLF PSORT、targetP1.1 Server、SubLoc v1.0预测蛋白质的亚细胞定位;采用SOMPA预测蛋白质的二级结构;采用SWISS-MODEL同源建模的方法模拟CoCS蛋白的三维结构,使用UCSF Chimera软件对构建的模型进行观察和分析。
1.2.6 实时荧光定量PCR
以‘长林4号’根系cDNA为模板利用primer3设计的荧光定量引物进行PCR扩增,PCR产物经琼脂糖凝胶电泳检测。结果表明,两对引物均能扩增出特异的目的片段。以稀释15倍的各样品的cDNA样品为模板,以油茶Actin7α基因[33]作为本实验的内参基因,使用ABI7300实时荧光定量PCR仪进行RT-PCR实验。RT-PCR反应体积为20 μL,2×SYBR Premix Ex TaqTM(2×)10.0 μL,ROX Reference Dye(×50) 0.4 μL,正反向引物(10 μmol·L-1)各0.4 μL,cDNA 2.0 μL,ddH2O 6.8 μL,每个反应重复4次。RT-PCR反应程序:95℃ 30 s;95℃ 5 s,60℃ 31 s,40个循环;95℃ 15 s,60℃ 60 s,95℃ 15 s。qRT-PCR反应检测数据采用2-△△Ct方法分析。
1.2.7 数据处理与分析
利用Excel 2010和GraphPad Prism 5软件进行数据处理和图表制作;利用IBM SPSS Statistics 22软件进行统计分析,用单因素方差分析法(one-way ANOVA,Duncan)进行显著性检验。
2 结果与分析
2.1 油茶CS基因cDNA和DNA克隆
以油茶无性系‘长林4号’的根系总RNA(图1A)反转录的cDNA为模板,以CSFP和CSRP为上下游引物,PCR扩增得到一条1 500 bp左右的特异条带(图1B)。将PCR产物回收的接到pMD-18T载体,转化大肠杆菌感受态细胞DH5α,进行菌液PCR检测获得阳性克隆后送公司测序。测序结果表明,该PCR产物的片段长度为1 416 bp。测序结果序列与转录组序列一致。该基因全长1 416 bp,编码471个氨基酸。将氨基酸序列在NCBI中进行BlaxtP搜索同源序列结果显示全是CS基因,因此将基因命名为CoCS,登录号为KU161147。以油茶无性系4号的根系DNA为模板,以CSFP和CSRP为上下游引物进行PCR扩增,分离得到油茶CS基因的基因组序列(图1C),经比对与CS基因的cDNA序列一致,说明该基因DNA序列不含内含子。

图1 总RNA的电泳、油茶CS基因cDNA和DNA的克隆Fig.1 Electrophoresis of total RNA,Cloning of cDNA and DNA of CS in C.oleifera
2.2 CoCS蛋白的同源性和系统进化树分析
将分离得到的油茶CS基因的cDNA序列推导出氨基酸序列,通过NCBI中的BLASTp程序搜索其同源序列。利用Clustal Omeg和DNAMAN6软件对油茶CS基因与其他物种同源序列进行多序列比对和同源分析,结果显示(图2),油茶CS基因与其他植物的CS基因所编码的氨基酸序列相似度均在84.8%以上,其中与芝麻(Sesamumindicum)的相似度最高为91.1%,与葡萄(Vitisvinifera)、麻风树(Jatrophacarcas)和桑属(Morusnotabilis)相似度为分别为90.3%、90.3%和89.2%。
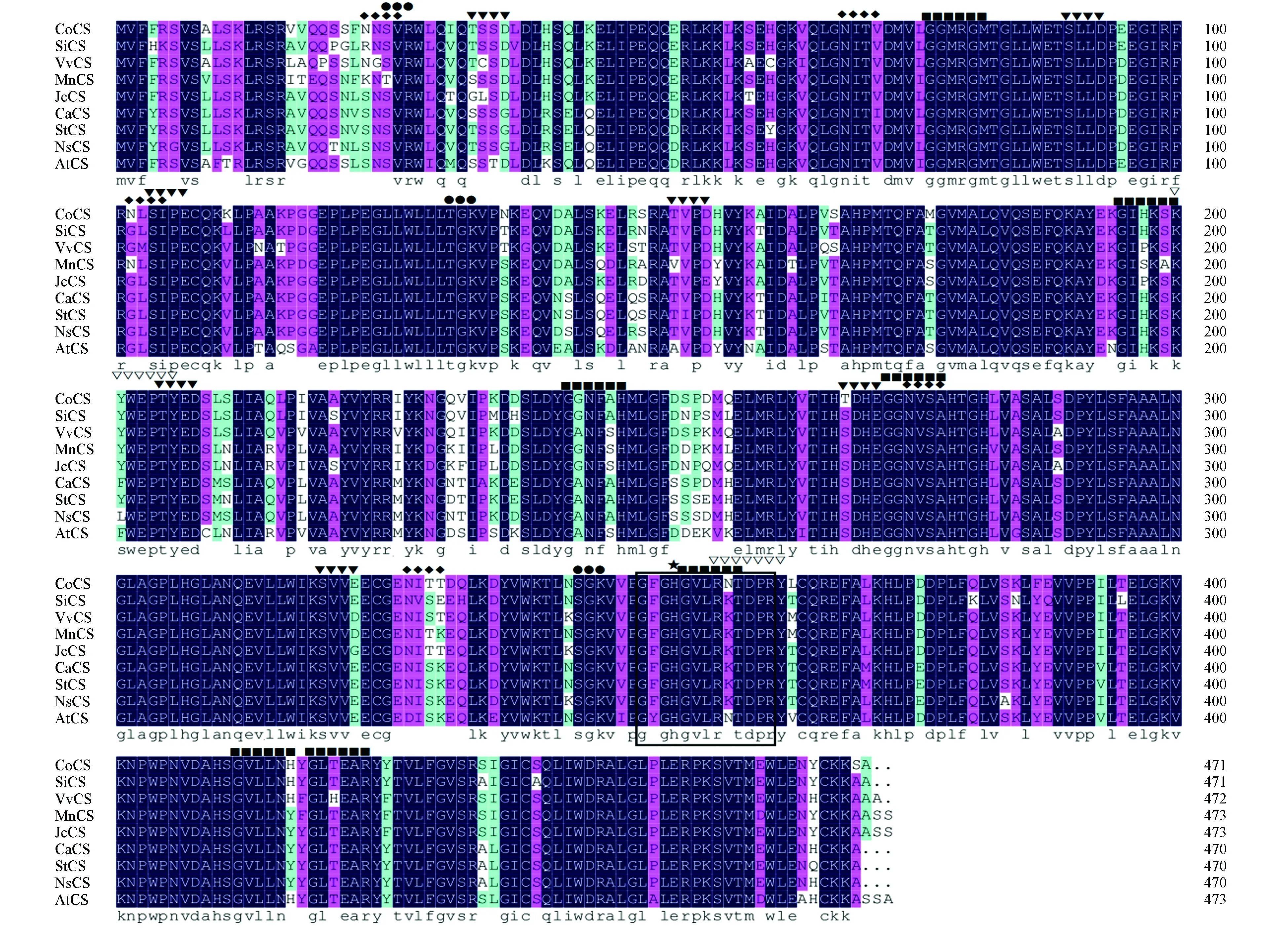
图2 CoCS与其他植物同源蛋白的氨基酸多序列比对 Co.油茶;Si.芝麻;Vv.葡萄;Mn.桑属;Jc.麻风树;Ca.甜椒;St.马铃薯;Ns.烟草;At.拟南芥 ◆N-糖基化位点;▼酪蛋白激酶Ⅱ的磷酸化位点;■肉豆蔻酰基化位点;●蛋白激酶C磷酸化位点;▽酪氨酸激酶磷酸化位点;□柠檬酸合酶标签;★柠檬酸合酶活性位点Fig.2 Multiple alignment result of CoCS and homologous proteins of other plants Co.Camellia oleifera;Si.Sesamum indicum;Vv.Vitis vinifera;Mn.Morus notabilis;Jc.Jatropha curcas;Ca.Capsicum annuum;St.Solanum tuberosum;Ns.Nicotiana sylvestris;At.Arabidopsis thaliana ◆N-glycosylation site; ▼Casein kinase II phosphorylation site; ■N-myristoylation site; ●Protein kinase C phosphorylation site; ▽Tyrosine kinase phosphorylation site; □Citrate synthase signature; ★Citrate synthase active site
为进一步分析油茶CS蛋白与其他物种CS蛋白之间的进化关系,利用Clustal Omeg对油茶CS蛋白与其他植物CS蛋白序列进行多序列比对,利用MEGA6.0中Neighbor-joining法构建了CS蛋白的系统进化树,选择小立碗藓为外群,作为进化树分支参考。结果显示(图3),被子植物中的双子叶植物和单子叶植物聚为一大类,目科属亲缘关系相近的植物归为一类,如锦葵目(Malvales)中的雷蒙德氏棉(Gossypiumraimondii)与可可(Theobromacacao)聚在一起,十字花科(Cruciferae)中的亚麻荠(Camelinasativa)、欧洲油菜(Brassicanapus)和拟南芥(Arabidopsisthaliana)聚在一起。油茶与杜鹃(Rhododendronmicranthum)、葡萄(Vitisvinifera)聚在一起,说明他们的亲缘关系最为接近,其次与中粒咖啡(Coffeacanephora)和芝麻(Sesamumindicum)的亲缘关系较近。CS蛋白在草本植物中的进化速度比木本中的进化速度快,其中十字花科的进化速度最快。
2.3 CoCS蛋白的生物信息学分析
采用InterProscan、Motif Scan和PROSITE在线工具预测油茶CS蛋白的家族、结构域、模体、功能位点等,结果表明,CoCS属于真核生物柠檬酸合酶家族成员(IPR010109),含有一个柠檬酸合酶(Citrate synthase)结构域(PF00285,78-455),含有一个柠檬酸合酶家族的标签基序(Citrate synthase signature)“GFGHGVLRNTDP”(PS00480),共同模式(Consensus pattern)为“G-[FYAV]-[GA]-H-x-[IV]-x(1,2)-[RKTQ]-x(2)-[DV]-[PS]-R”,组氨酸(H)是柠檬酸合酶活性位点残基。氨基酸序列中存在N-糖基化位点、肉豆蔻酰基化位点、酪蛋白激酶Ⅱ的磷酸化位点、蛋白激酶C磷酸化位点、酪氨酸激酶磷酸化位点(图2)。

图3 CoCS与其他物种的CS蛋白质的进化树分析Fig.3 Phylogenetic tree relationships among the CoCS and homologous protein in other plants
利用ProtParam对CoCS蛋白质进行理化性质分析,结果显示该蛋白的分子量为52.74 kD,理论等电点(PI)为6.95;带负电荷残基(Asp+Glu)数为51个,带正电荷残基(Arg+Lys)数为50个,分子式为C2379H3745N639O683S16;不稳定指数(II)为35.33,属于稳定蛋白;脂肪系数为95.80,总平均亲水性为-0.208,可以判断CoCS是疏水性蛋白。
利用在线软件CCTOP对CoCS蛋白的跨膜区与跨膜拓扑结构预测,结果表明CoCS非跨膜蛋白。利用SignalP 4.1 Server预测CoCS的信号肽,结果显示CoCS不存在信号肽;利用SubLoc v1.0、targetP1.1 Server、Plant-PLoc、CELLO v.2.5和WoLF PSORT预测CoCS的亚细胞定位,预测结果显示均定位在线粒体上。采用SOPMA对CoCS蛋白二级结构预测表明,α-螺旋是油茶CoCS蛋白的主要结构元件,α-螺旋(Alpha helix)占45.65%,β折叠(Extended strand)占12.10%、β转角(Beta turn)占9.98%、无规则卷曲(Random coil)占32.2%。
2.4 CoCS基因的表达分析
利用qRT-PCR技术分析了‘长林166号’油茶无性系根系受到低磷胁迫不同时间CoCS基因的表达情况,结果表明(图4A),低磷胁迫后,根系中CoCS基因的表达量均比胁迫前高,呈先增升高后降低的趋势,其中在胁迫24 h表达量达到最高,为对照的2.6倍,96 h时表达量为对照的1.4倍。分析不同磷水平处理水下水培30 d后‘长林166号’根系CoCS基因表达量的结果显示(图4B),低磷处理下根系CoCS基因的表达量高于对照,为对照的1.4倍。
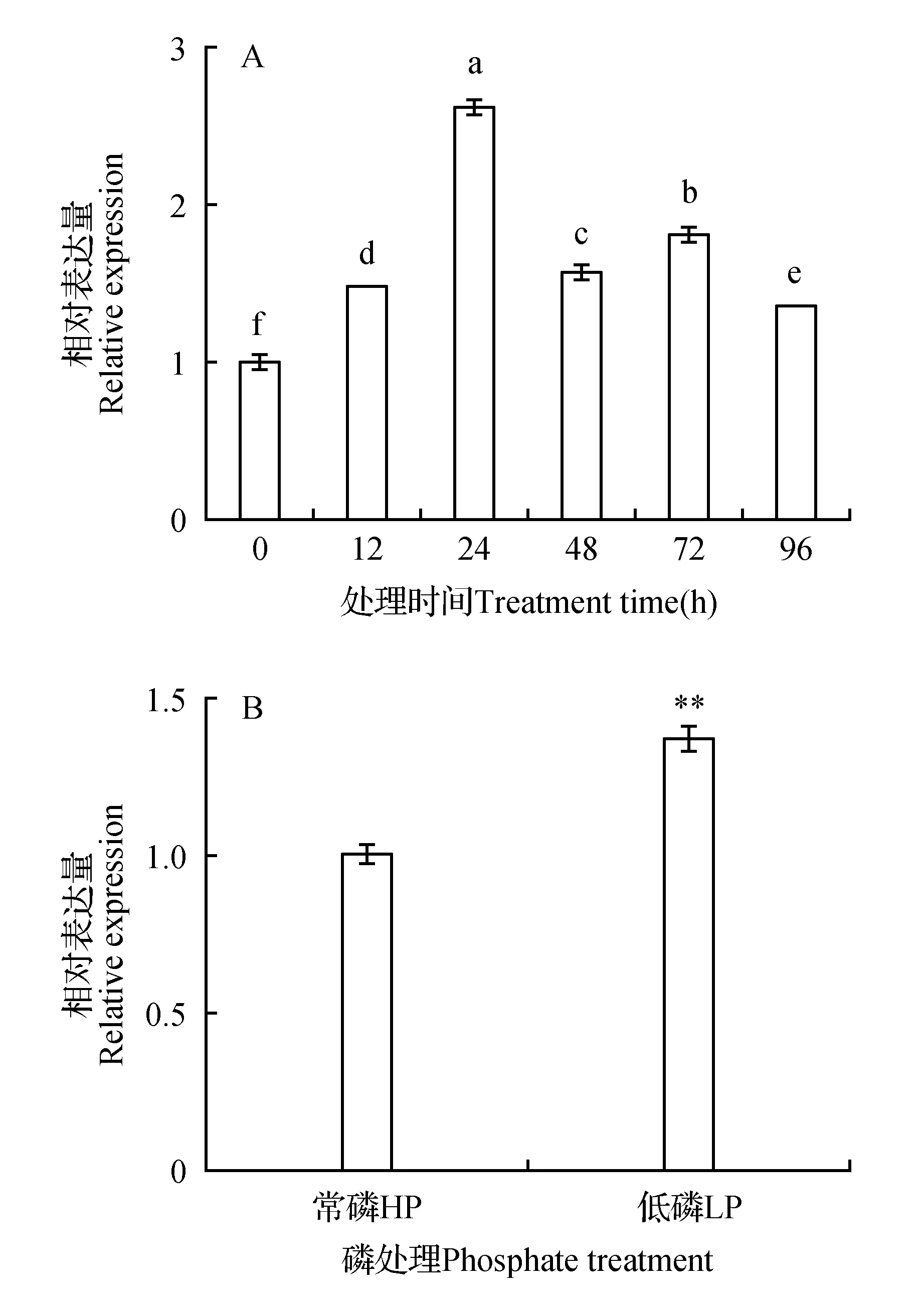
图4 不同低磷处理时间(A)和不同磷水平(B)根系CoCS基因的表达 不同字母表示差异显著(P<0.05);**表示差异极显著(P<0.01) 下同。Fig.4 Expresson of CoCS in root under different treatment time and different phosphate treatment levelDifferent letters indicated significant difference at 5% level; ** indicated significant difference at 1% level The same as below.
分析不同磷水平处理下‘长林4号’和‘长林166号’根、茎、叶中CoCS基因表达的结果表明(图5):在根部(图5A),低磷处理下‘长林4号’CoCS基因表达量和对照相当没有显著变化,而‘长林166号’表达量稍微升高,比对照提高了的15.84%;在低磷处理和对照处理下,‘长林166号’中该基因的表达量均比‘长林4号’高,其中对照处理下该基因的表达量是‘长林4号’的2.5倍,低磷处理下是‘长林4号’的3.2倍。在茎中(图5B),该基因的表达模式与根部相反,低磷处理下,‘长林4号’茎中表达量相对对照增加了1倍,而在‘长林166号’相对对照减少了40.29%;正常磷水平处理下‘长林166号’是‘长林4号’的1.5倍,而低磷处理‘长林4号’则是‘长林166号’的2.3倍。在叶片中(图5C),该基因的表达模式与茎部相似,低磷处理下‘长林4号’茎中表达量较对照升高了1.5倍,而‘长林4号’表达量较对照降低了27.27%。

图5 不同磷水平处理下根、茎、叶中CoCS基因的表达 A.根;B.茎;C.叶Fig.5 Expression of CoCS in root,stem and leaf under different phosphate treatment level A.Root; B.Stem; C.Leaf
3 讨论
柠檬酸是三羧酸循环(TCA)中重要的中间产物,柠檬酸合酶(citrate synthase,CS)具有催化草酰乙酸(OAA)和乙酰辅酶A(CoA)合成柠檬酸的作用。三羧酸循环在三大营养素糖类、脂类、氨基酸的代谢中起着枢纽作用,是它们的最终代谢通路,对生物体的各种氧化还原反应过程都起着至关重要的作用。柠檬酸合酶催化合成的柠檬酸通过根系分泌到土壤中可以能够活化土壤中的难容磷,提高植物的磷利用效率[9,21]。超表达柠檬酸合酶基因可以提高转基因植株提高磷的吸收和利用效率[20,23,25]。
本研究从油茶中克隆了CS基因的全长,并对其进行了生物信息学分析。分析结果显示,CoCS其它植物的CS蛋白序列高度相似,与杜鹃和葡萄的CS蛋白具有较近的进化关系;该基因的蛋白序列具有典型的CS蛋白的活性位点和结构域,与其他植物CS蛋白序列的特征一致[34~36]。因此说明CoCS基因属于高等植物CS基因家族成员。荧光定量PCR结果表明,低磷胁迫后,根系中的CoCS的表达受到低磷诱导,表达量呈现先升高后降低的趋势。正常和低磷处理一个月后,低磷处理下根系中CoCS表达量低磷较正常处理略有增强,结果与茶树CS基因在低磷下表达略有升高的结果一致[31]。这说明低磷可以地诱导柠檬酸合酶的增强表达。沙培4个月后,低磷处理下‘长林4号’根系CoCS基因表达量较对照没有显著变化,而‘长林166号’表达量略有提高,正常和低磷处理下,‘长林166号’根系中的CoCS基因表达量均比‘长林4号’高,这说明油茶根系中的CoCS表达量的高低与自身的基因型有关。叶和茎中CoCS的表达模式相同,与根不同。低磷处理下,在茎和叶中‘长林4号’升高,而‘长林166号’降低,这说明CoCS基因在不同组织中的表达模式不同;叶和茎中的表达模式相同,可能是茎的采样部位是茎尖幼嫩部位与茎尖分化出的嫩叶的部位距离相近导致的;‘长林166号’和‘长林4号’不同,可能是由于自身基因型差异有关。正常处理和低磷下在根和叶中,‘长林166号’的表达量均比‘长林4号’的高,但在茎中低磷下‘长林4号’的反比‘长林166号’的高,这可能是由于在低磷处理下,‘长林166号’根系中CoCS的表达量比‘长林4号’高,导致在茎中‘长林166号’更早适应低磷,CoCS表达量降低的速度比‘长林4号’快,从而导致‘长林4号’反比‘长林166号’的高。综上结果表明,低磷能诱导根系中CoCS基因轻微上调表达,但在不同无性系不同组织中的表达模式不同,这暗示CoCS基因的功能在不同油茶品种和不同组织中存在差异,下一步将通过转基因来验证CoCS基因的功能,为揭示油茶适应低磷的分子机制和选育磷高效油茶品种奠定分子生物学基础。
1.Zhang Z L,Liao H,Lucas W J.Molecular mechanisms underlying phosphate sensing,signaling,and adaptation in plants[J].Journal of Integrative Plant Biology,2014,56(3):192-220.
2.Lynch J P,Beebe S E.Adaptation of beans(PhaseolusvulgarisL.) to low Phosphorus availability[J].Hort Science,1995,30(6):1165-1171.
3.Shen J B,Yuan L X,Zhang J L,et al.Phosphorus dynamics:from soil to plant[J].Plant Physiology,2011,156(3):997-1005.
4.Franco-zorrilla J M,González E,Bustos R,et al.The transcriptional control of plant responses to phosphate limitation[J].Journal of Experimental Botany,2004,55(396):285-293.
5.马祥庆,梁霞.植物高效利用磷机制的研究进展[J].应用生态学报,2004,15(4):712-716.
Ma X Q,Liang X.Research advances in mechanism of high Phosphorus use efficiency of plants[J].Chinese Journal of Applied Ecology,2004,15(4):712-716.
6.Wu P,Shou H X,Xu G H,et al.Improvement of Phosphorus efficiency in rice on the basis of understanding phosphate signaling and homeostasis[J].Current Opinion in Plant Biology,2013,16(2):205-212.
7.Veneklaas E J,LAmbers H,Bragg J,et al.Opportunities for improving phosphorus-use efficiency in crop plants[J].New Phytologist,2012,195(2):306-320.
8.Pérez-torres C A,López-bucio J,Cruz-ramírez A,et al.Phosphate availability alters lateral root development in Arabidopsis by modulating auxin sensitivity via a mechanism involving the TIR1 auxin receptor[J].The Plant Cell,2008,20(12):3258-3272.
9.Plaxton W,Tran H T.Metabolic adaptations of Phosphate-Starved plants[J].Plant Physiology,2011,156(3):1006-1015.
10.Péret B,Clément M,Nussaume L,et al.Root developmental adaptation to phosphate starvation:better safe than sorry[J].Trends in Plant Science,2011,16(8):442-450.
11.Niu Y F,Chai R S,Jin G L,et al.Responses of root architecture development to lowPhosphorusavailability:a review[J].Annals of Botany,2013,112(2):391-408.
12.王学敏.低磷供应对拟南芥根系构型的影响[J].植物研究,2010,30(4):496-502.
Wang X M.Effect of Low-Phosphate supply on the root system architecture inArabidopsisthaliana[J].Bulletin of Botanical Research,2010,30(4):496-502.
13.Czarnecki O,Yang J,Weston D J,et al.A dual role of strigolactones in phosphate acquisition and utilization in plants[J].International Journal of Molecular Sciences,2013,14(4):7681-7701.
14.Neumann G,Römheld V.Root excretion of carboxylic acids and protons in phosphorus-deficient plants[J].Plant and Soil,1999,211(1):121-130.
15.王树起,韩晓增,乔云发,等.缺磷胁迫条件下大豆根系有机酸的分泌特性[J].大豆科学,2009,28(3):409-414.
Wang S Q,Han X Z,Qiao Y F,et al.Characteristics of organic acids exudated from soybean(GlycinemaxL.) Roots under P deficiency stress[J].Soybean Science,2009,28(3):409-414.
16.陈凯,马敬,曹一平,等.磷亏缺下不同植物根系有机酸的分泌[J].中国农业大学学报,1999,4(3):58-62.
Chen K,Ma J,Cao Y P,et al.Exudation of organic acids by the roots of different plant species under phosphorus deficiency[J].Journal of China Agricultural University,1999,4(3):58-62.
17.Schnarrenberger C,Martin W.Evolution of the enzymes of the citric acid cycle and the glyoxylate cycle of higher plants:a case study of endosymbiotic gene transfer[J].European Journal of Biochemistry,2002,269(3):868-883.
18.Pracharoenwattana I,Cornah J E,Smith S M.Arabidopsisperoxisomal citrate synthase is required for fatty acid respiration and seed germination[J].The Plant Cell,2005,17(7):2037-2048.
19.Larsen P B,Degenhardt J,Tai C Y,et al.Aluminum-resistantArabidopsismutants that exhibit altered patterns of Aluminum accumulation and organic acid release from Roots[J].Plant Physiology,1998,117(1):9-17.
20.Koyama H,Kawamura A,Kihara T,et al.Over expression of mitochondrial citrate synthase inArabidopsisthalianaimproved growth on a phosphorus-limited soil[J].Plant & Cell Physiology,2000,41(9):1030-1037.
21.Khorassani R,Hettwer U,Ratzinger A,et al.Citramalic acid and salicylic acid in sugar beet root exudates solubilize soil Phosphorus[J].BMC Plant Biology,2011,11:121.
22.de la Fuente J M,Ramírez-rodríguez V,Cabrera-ponce J L,et al.Aluminum tolerance in transgenic plants by alteration of citrate synthesis[J].Science,1997,276(5318):1566-1568.
23.Koyama H,Takita E,Kawamura A,et al.Over expression of mitochondrial citrate synthase gene improves the growth of carrot cells in Al-phosphate medium[J].Plant & Cell Physiology,1999,40(5):482-488.
24.López-bucio J,de la Vega O M,Guevara-garcía A,et al.Enhanced Phosphorus uptake in tobacco transgenic plants that overproduce citrate[J].Nature Biotechnology,2000,18(4):450-453.
25.Wang Y,Xu H,Kou J J,et al.Dual effects of transgenicBrassicanapusoverexpressing CS gene on tolerances to Aluminum toxicity and Phosphorus deficiency[J].Plant and Soil,2013,362(1-2):231-246.
26.庄瑞林.中国油茶[M].2版.北京:中国林业出版社,2008:3-5.
Zhuang R L.Oil-tea camellia in China[M].2nd ed.Beijing:China Forestry Publishing House,2008:3-5.
27.何方,何柏.油茶栽培分布与立地分类的研究[J].林业科学,2002,38(5):64-72.
He F,He B.Cultural distribution and site classification forCamelliaoleifera[J].Scientia Silvae Sinicae,2002,38(5):64-72.
28.叶思诚,谭晓风,袁军,等.油茶根系及分泌物中有机酸的HPLC法测定[J].南京林业大学学报:自然科学版,2013,37(6):59-63.
Ye S C,Tan X F,Yuan J,et al.Determination of organic acids in oil tea root tissue and root exudation with HPLC[J].Journal of Nanjing Forestry University:Natural Sciences Edition,2013,37(6):59-63.
29.叶思诚.油茶适应低磷胁迫的根系生理响应[D].长沙:中南林业科技大学,2013.
Ye S C.The research on the physiological adapting responses of the root ofCamelliaoleiferato Phosphorus deficiency[D].Changsha:Central South University of Forestry and Technology,2013.
30.Yuan J,Tan X F,Ye S C,et al.The organic acids in root exudates of oiltea and its role in mobilization of sparingly soluble phosphate in red soil[J].Journal of Chemical and Pharmaceutical Research,2013,5(11):572-577.
31.林郑和,陈常颂,邬龄盛.低磷胁迫下茶树根系CS基因的克隆及表达分析[J].茶叶科学,2010,30(5):362-366.
Lin Z H,Chen C S,Wu L S.Cloning and expression analysis on citrate synthase gene of tea root under low phosphorous stress[J].Journal of Tea Science,2010,30(5):362-366.
32.胡玉玲,姚小华,任华东,等.油茶花发育转录组测序及相关基因表达分析[J].林业科学,2014,50(9):36-43.
Hu Y L,Yao X H,Ren H D,et al.Sequencing of transcriptome relevant to flowering and analysis of Floral-Related genes expression inCamelliaoleifera[J].Scientia Silvae Sinicae,2014,50(9):36-43.
33.Zhou C F,Lin P,Yao X H,et al.Selection of reference genes for quantitative real-time PCR in six oil-tea camellia based on RNA[J].Molecular Biology,2013,47(6):836-851.
34.任磊,王雁,周琳,等.牡丹PsCS基因的克隆及序列分析[J].生物技术通报,2011,27(9):81-85.
Ren L,Wang Y,Zhou L,et al.Cloning and sequence analysis ofPsCSgene in tree peony[J].Biotechnology Bulletin,2011,27(9):81-85.
35.王滕旭,李正国,杨迎伍,等.甜橙柠檬酸合酶基因的克隆及其表达分析[J].中国农学通报,2010,26(10):65-69.
Wang T X,Li Z G,Yang Y W,et al.Cloning and expression analysis of citrate synthase gene in orange[J].Chinese Agricultural Science Bulletin,2010,26(10):65-69.
36.杨林通,林郑和,陈立松.酸柚根系CS和PEPC基因的克隆及序列分析[J].福建农林大学学报:自然科学版,2012,41(3):253-258.
Yang L T,Lin Z H,Chen L S.Cloning and sequence analysis of citrate synthase and phosphoenolpyruvate carboxylase in the root of ‘Sour pummelo’(Citrusgrandis)[J].Journal of Fujian Agriculture and Forestry University:Natural Science Edition,2012,41(3):253-258.
This work was supported by Zhejiang Agriculture Major Project ‘Breeding and demonstration of the new cultivars ofCamelliaoleiferawith high yield and good quality’(2012C12908)
introduction:YE Si-Cheng(1985—),male,Dr.,main research interest is non-timber tree breeding and cultivation.
date:2016-03-16
CloningandExpressionAnalysisofCitrateSynthase(CS)GeneinCamelliaoleifera
YE Si-Cheng YAO Xiao-Hua*WANG Kai-Liang LIN Ping GONG Hong-En ZHUO Ren-Ying
(Research Institute of Subtropical Forestry,Chinese Academy of Forestry,Fuyang 311400)
A citrate synthase(CS) gene were isolated fromCamelliaoleiferaby RT-PCR. The full-length cDNA of theCSis 1 416 bp in size, encodeding a deduced polypetide of 471 amino acids with estimated molecular weight of 52.74 kD and theoretical isoelectric point of 6.95. Homologous alignment showed that the deduced protein had high identities with the CS proteins of other plants, therefore the gene was named asCoCS(Genbank No.KU161147). By phylogenetic tree analysis, CoCS had close genetic relationships withRhododendronmicranthumandVitisvinifera. By qRT-PCR, the expression ofCoCSin root was induced by phosphate deficiency and increased at first and decreased subsequently. The expression patterns ofCoCSwere different among different tissues and different cultivars.
Camelliaoleifera;citrate synthase;phosphate deficient;gene cloning;expression analysis
浙江省重大农业专项“油茶高产优质新品种选育及示范”(2012C12908)
叶思诚(1985—),男,博士研究生,主要从事经济林栽培育种研究。
* 通信作者:E-mail:yaoxh168@163.com
2016-03-16
* Corresponding author:E-mail:yaoxh168@163.com
S794.4
A
10.7525/j.issn.1673-5102.2016.04.011

