白桦BpCesA基因的生物信息学及表达分析
申婷婷 姜 静 刘桂丰 许思佳 李慧玉* 袁红梅,2
(1.东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040; 2.黑龙江省农业科学院经济作物研究所,哈尔滨 150086)
白桦BpCesA基因的生物信息学及表达分析
申婷婷1姜 静1刘桂丰1许思佳1李慧玉1*袁红梅1,2
(1.东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040;2.黑龙江省农业科学院经济作物研究所,哈尔滨 150086)
从白桦45个转录组数据中分析获得6条白桦纤维素合成酶基因,其中2条为该家族的新基因,通过生物信息学及实时定量PCR技术探讨白桦BpCesA在不同材性白桦中的表达量情况。结果表明,6条白桦BpCesA基因的ORF长度在2 958~3 312 bp,编码的氨基酸数目在985~1 103 aa,相对分子量在110.40~124.29 kD,理论等电点为5.90~7.43;6条BpCesA均含有D,D,D,QXXRW保守结构域和8个跨膜螺旋,其中2个位于N端,6个位于C端。对来自6个双子叶植物,4个单子叶植物和1个裸子植物的52条CesA进行了聚类分析发现:白桦BpCesA与同为木本的杨树PtrCesA具有较高的同源性,其中BpCesA2与PtrCesA3同源性达到92%、另外BpCesA5与PtrCesA6、BpCesA1与PtrCesA8也具有较高的同源性,分别为89%和82%。BpCesA1、BpCesA2、BpCesA3、BpCesA5基因随纤维素含量的增加,表达量也呈现增加趋势,推测这些基因在白桦的纤维素合成中起到促进的作用。本研究为揭示纤维素合成酶基因的功能分析提供了基础。
白桦;纤维素;木质素;生物信息学;表达量
白桦(BetulaplatyphyllaSuk.)属桦木科(Betulaceae)桦木属(Betula),广泛分布于我国东北、华北、西北、西南等14个省区。白桦形态优美,树皮光滑洁白,有较高的观赏价值,适合作为城市绿化树种[1]。20世纪90年代初白桦被列为国家科技攻关研究对象之一,目前白桦作为国家科技支撑计划研究的树种,我国已在其种源分析[2]、木材形成[3]、花发育调控[4]以及扦插繁殖等方面进行了大量的研究工作,而日益成熟的生物技术也为这些研究提供了全新的思路。白桦的生长速度较快,抗逆性和适应性较强,纤维长宽比为56.7,适合造纸使用[5]。在纸浆造纸中,木质素被降解掉之后,留下纤维素,纤维素的长度决定了纸张的硬度和弹性,纤维素越长,纸张越硬,弹性也越好。对白桦来说,了解哪些基因与木材形成相关具有重大的意义和作用。
纤维素是由β-1,4糖苷键连接形成的不分支多糖,其重复结构单位是纤维二糖,但是其合成机制却非常复杂,涉及到多种酶。Pear等采用cDNA随机测序和序列分析法,首次从棉花中克隆了编码纤维素合成酶催化亚基的β-1,4糖苷转移酶基因(CesA)。此后,在水稻、拟南芥、玉米、杨树等更多的植物中分离克隆出CesA基因[6~7]。CesAs形成一个庞大的基因家族,家族内的每个酶彼此相关,协同作用。同时,利用突变体研究已经了解到CesA在纤维素合成中的作用及表达调控[8]。此外,在植物中还存在大量的纤维素合成酶相似蛋白,其功能还不清楚。
在前期研究中,以控制授粉获得的49个白桦家系建立的6年生子代为材料,对其材性进行了遗传变异分析,并对这49个家系的纸浆材材性进行评估。本研究从材性评价高、中、低的6个家系:0628、0655、0616、0617、0613、0630中各选取一个单株进行无性繁殖,分别提取木质部和韧皮部的总RNA,参考已知的杨树的研究结果,选择了6条与白桦纤维素合成相关的白桦基因,通过实时定量PCR分析这些基因在白桦木材形成中的作用,筛选直接参与木材形成的基因,为林木遗传改良提供优良的理论基础。
1 材料与方法
1.1 实验材料
用于材性分析的白桦来自东北林业大学帽儿山实验林场6年生的白桦控制授粉子代,根据前期材性评估结果,分别从材性高、中、低的6个家系(0628、0655、0616、0617、0613、0630)中选择一个单株(材性高:5、6;材性中:3、4;材性低:1、2),取同一方向的一级侧枝进行材性测定。
实时定量PCR分析的试验材料为从上述6个单株上取的芽外植体消毒后,进行无性扩繁,生根苗在培养钵中生长一年后,分别取其木质部和韧皮部,经过液氮速冻后,放入-80℃冰箱中备用。
1.2 材性测定方法
纤维长度测定方法采用Taylor精确测定法[9~10]。用于测定相应含量的试样先研磨后过筛按照国家标准GB/T 26771-93处理。木质素和纤维素含量按照ANKOM公司的A2000i型全自动纤维分析仪进行的测定,即滤袋法进行测定。
1.3 统计分析方法
数据分析采用SPSS 20.0软件进行方差和多重比较分析。
1.4白桦纤维素合成酶基因的获得及生物信息学分析
利用本团队已有的45个白桦的转录组数据,通过NCBI进行比对搜索,筛选获得6条纤维素合成相关基因:BpCesA1、BpCesA2、BpCesA3、BpCesA4、BpCesA5、BpCesA6。利用NCBI在线寻找程序(ORF founder)确定该基因的开放读码框;对蛋白质的分子量和理论等电点进行了在线计算(http://web.expasy.org/protparam/);用BioEdit中的ClustalW进行氨基酸的多序列比对;选取白桦、拟南芥和杨树的CesA基因进行系统发育分析;在NCBI和TAIR数据库搜索已知的CesA蛋白质序列,使用MEGA 5.1绘制系统发育进化树,Bootstrap分析进行1 000个重复,以默认参数构建邻接树(Neighbor-joining tree,NJ-tree)[11],借助MEGA软件完成作图[12]。
1.5 实时定量荧光PCR分析
用植物总RNA提取试剂盒(离心柱型)提取总RNA,琼脂糖凝胶电泳检测RNA完整性。并使用TOYOBO的ReverTra Ace® qPCR RT Master Mix with gDNA Removerr试剂盒进行cDNA合成。
根据6条BpCesA基因序列,利用primer5.0设计定量PCR引物,由博仕生物公司合成,引物序列见表1。18SrRNA为本研究中选择的内参基因。
将合成的第一链cDNA加去离子水稀释10倍,作为模版,进行实时定量PCR扩增,反应体系为:SYBR® Premix ExTaqTMⅡ(2×)6 μL,引物各0.48 μL(10 μmol·L-1),水3.6 μL,模板2 μL,Rox DyeⅡ 0.24 μL,反应参数为:95℃预变性30 s,95℃变性5 s,60℃退火延伸34 s,循环40次,绘制熔解曲线,温度由95℃ 15 s,60℃ 1 min,至95℃ 15 s止,所有试样均进行3次技术重复,反应结束后,获取数据并通过-△△Ct法进行基因表达分析。
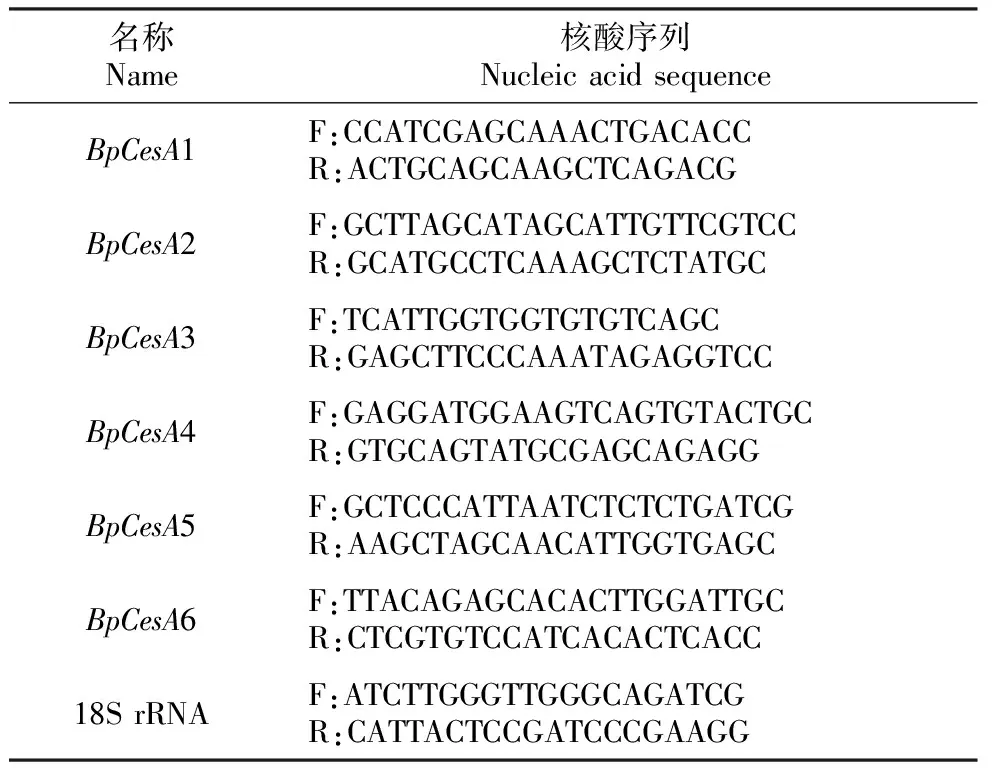
表1 实时定量PCR引物
2 结果与分析
2.1 优良单株的材性分析
对6个白桦单株的纤维素的4个相关性状进行了方差分析,由表2可以看出,6个单株纤维素和综纤维素含量方面的差异都达到了极显著水平,半纤维素差异显著,纤维长宽比差异不显著,进一步进行了多重比较(Duncan)(表3)。
表2白桦株系间生长性状方差分析
Table2VarianceanalysisofeachgrowthtraitofB.platyphyllastrains
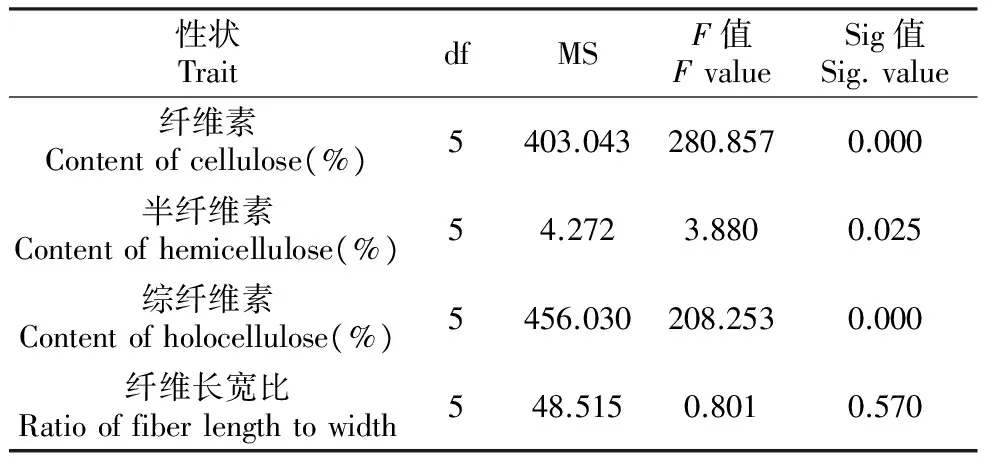
性状TraitdfMSF值FvalueSig值Sig.value纤维素Contentofcellulose(%)5403.043280.8570.000半纤维素Contentofhemicellulose(%)54.2723.8800.025综纤维素Contentofholocellulose(%)5456.030208.2530.000纤维长宽比Ratiooffiberlengthtowidth548.5150.8010.570
注:Sig.中0.000代表P>F0.01,差异极显著。
Note:Sig.0.000 representativesP>F0.01,the difference was significant.
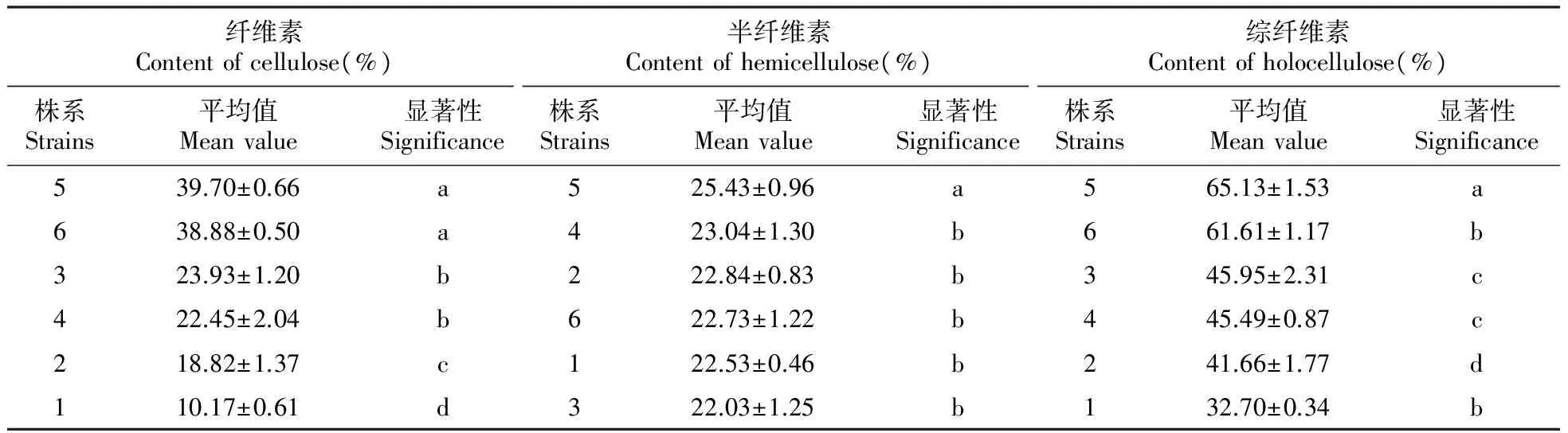
表3 白桦各株系纤维素相关性状的多重比较结果
由表3可以看出,来自白桦材性优良家系的单株5、6与分别来自材性中及差家系的单株3、4、2、1的纤维素及综纤维素含量差异显著,单株5、6的纤维素及综纤维素含量显著高于其他4个株系,分别比含量最低的1株系提高290%、282%和99%、88%。在半纤维素含量方面,单株5与其他5个单株的半纤维素含量差异显著,较单株3的半纤维素含量提高了15%。
2.2 白桦纤维素合成酶基因的生物信息学分析2.2.1 白桦BpCesA基因的特性分析
利用本团队已有45个转录组数据,获得白桦CesA家族共6条全长基因,其中BpCesA1、BpCesA2、BpCesA3、BpCesA4与NCBI已提交的4条BpCesA基因相同,BpCesA5、BpCesA6为白桦纤维素合成酶基因家族的新基因。我们对白桦6个BpCesA基因的特性进行分析发现:这6个基因ORF长度范围在2 958~3 312 bp,编码的氨基酸数目在985~1 103 aa,相对分子质量在110.40~124.29 kD,理论等电点的范围在5.90~7.43。
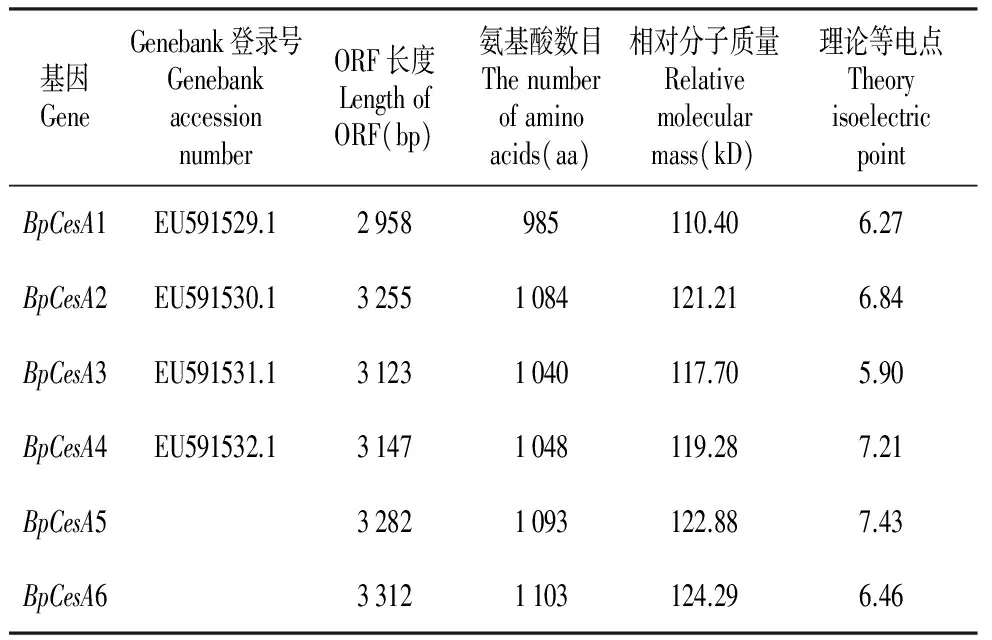
表4 白桦BpCesA基因特性分析

图1 白桦纤维素合成相关基因的氨基酸序列比对 蓝色:D,D,D,QXXRW序列的结构域;绿色:RING-finger domain;红色:Zinc-finger motifsFig.1 Amino acid sequence alignment of B.platyphylla cellulose synthesis related genes Blue:Domain of D,D,D,QXXRW sequence; Green:RING-finger domain; Red:Zinc-finger motifs
2.2.2 白桦BpCesA基因的多序列比对
我们对这6条白桦BpCesA基因的氨基酸序列进行分析,发现白桦BpCesA均含有纤维素合成酶保守的结构域:3个保守的天冬氨酸残基和QXXRW motif,构成含有D,D,D,QXXRW序列的保守结构域(图1),研究发现该结构域在细胞质膜表面形成糖基转移酶活性位点。通过软件预测发现6个白桦BpCesA均含有8个跨膜螺旋,其中2个位于N端,6个位于C端。有研究表明,8个跨膜螺旋在D,D,D,QXXRW序列的结构域处形成通道,将葡聚糖链分泌出去[13]。除此之外,在蛋白的N端还含有1个Zinc-finger motifs和RING-finger domain。Zinc-finger motifs包含4个保守的半胱氨酸残基(X3CX2-4CX12-15CX2C),而RING-finger domain含有一系列的保守的半胱氨酸残基及组氨酸残基(CX2CX9-39CX1-3HX2-3C/HX2CX4-48CX2C)(图1)。它们分别具有DNA-binding特性和将2个锌原子结合到N端的“cross-brace”上的作用[14]。
2.2.3 白桦纤维素合成相关基因的聚类分析
我们对来自6个双子叶植物,4个单子叶植物和1个裸子植物的52条CesA进行了聚类分析。有研究报道高等植物的纤维素合成酶基因共分为6类,这些基因参与植物的初生或次生细胞壁形成[15],我们的研究结果与之一致,从图2中可以看出:BpCesA1与PtrCesA1,BpCesA4与PtrCesA4、AtCesA4具有较高的同源性,同属于一个亚类;BpCesA2与PtrCesA5、AtCesA3属于一个亚类;BpCesA3与PtrCesA2、AtCesA7属于一个亚类;BpCesA5与PtrCesA6属于一个亚类;整体上看,白桦CesA与同为木本植物的杨树CesA具有较高的同源性,其中BpCesA2与PtrCesA3同源性达到91%,另外BpCesA5与PtrCesA6、BpCesA1与PtrCesA8也具有较高的同源性,分别为89%和81%。

图2 白桦BpCesA与其他植物CesA序列的系统发育树 Bp.白桦;At.拟南芥;Gh.棉花;Hv.大麦;Os.水稻;Pr.松树;Ptr.杨树;St.马铃薯;Ta.小麦;Ze.百日草;Zm.玉米Fig.2 Phylogenetic tree of B.platyphylla BpCesA with some other plants CesA sequence Bp.Betula platyphylla Suk; At.Arabidopsis thaliana; Gh.Gossipium hirsutum; Hv.Hordeum vulgare; Os.Oryza sativa; Pr.Pinus radiata; Ptr.Populus tremuloides; St.Solanum tuberosum; Ta.Triticum aestivum; Ze.Zinnia elegans; Zm.Zea mays
2.3 实时定量qRT-PCR分析
我们以材性不同的白桦为试材,通过实时定量PCR技术,研究这些纤维素及木质素合成相关基因在白桦木材形成中的作用。
由图3可以看出,在白桦的木质部中,基因的表达量随白桦材性的提高并未看到规律的变化。在白桦的韧皮部中,除单株5外,BpCesA1基因的表达量与白桦纤维素含量呈正相关,该基因在单株6中表达量最高。BpCesA2、BpCesA3及BpCesA5基因的表达趋势与BpCesA1相似,除个别单株外,均呈现随白桦纤维素含量提高,表达量上升的趋势,3个基因在单株5中的表达量最高,分别比表达量最低的株系提高了。BpCesA4基因在单株1、2、3、4中的表达量呈现与纤维素含量负相关的趋势,但在单株6表达量最高。BpCesA6基因的单株在表达量上呈现不规则变化,纤维素含量中等的单株4表达量最高。综合材性及基因表达量分析的结果可以看出,随着白桦纤维素及综纤维素含量的提高,6个白桦纤维素合成相关基因的表达量发生变化,尤其BpCesA1、BpCesA2、BpCesA3及BpCesA5随纤维素含量的增加,表达量也呈现增加的趋势,说明这些基因在白桦的纤维素合成中起到促进作用。

图3 纤维素合成相关基因的表达 A. BpCesA1;B. BpCesA2;C. BpCesA3;D. BpCesA4;E. BpCesA5;F. BpCesA6Fig.3 RNA expression level of cellulose synthesis related genes A. BpCesA1; B. BpCesA2; C. BpCesA3; D. BpCesA4; E. BpCesA5; F. BpCesA6
3 讨论
纤维素合成酶为多基因家族,现在己知植物的纤维素合成酶基因家族包括很多成员,拟南芥基因组中含有10个CesA基因,杨树中至少含有18个,玉米[16]中含有13个基因,水稻[17~18]至少11个基因[19],我们从白桦中克隆出6个BpCesA基因。所有的白桦纤维素合成酶基因均含有保守的3个天冬氨酸残基和8个跨膜螺旋。根据拓扑结构模型,3个天冬氨酸残基位点接近,并且与QXXRW motif在质膜表面形成活性位点。其中D1和D2残基协助UDP结合,而D3为糖苷链延伸的催化位点[20]。也有研究将纤维素合成酶的保守结构域分为A和B两个区,其中2个天冬氨酸旋残基位于A区,2个跨膜螺旋位于A区前面;而B区含有另外一个天冬氨酸残基和6个跨膜螺旋[21]。在白桦纤维素合成酶N端的2个跨膜螺旋前面含有23氨基酸残基(X3CX2CX12CX2C),该区域为DNA-binding位点。
不同纤维素合成酶参与不同细胞细胞壁的纤维素合成[22~26]。研究表明AtCesA1、AtCesA3、AtCesA6及其同源基因参与初生细胞壁中纤维素合成[27];AtCesA4、AtCesA7、AtCesA8及其同源基因参与次生细胞壁中纤维素合成。由此可以推测,BpCesA2和BpCesA6可能参与初生细胞壁中纤维素合成;而BpCesA1、BpCesA3和BpCesA4可能在次生细胞壁中纤维素合成中起作用。研究发现纤维素合成酶基因具有组织特异性。AtCesA2、AtCesA5和AtCesA9在种子上表皮细胞的细胞壁中表达;水稻OsCesA5/OsCesA6与OsCesA3形成复合体,在胚芽和胚根中表达[28];AtCesA1、AtCesA3、与AtCesA6在子叶的下胚轴中表达。AtCesA4/AtCesA7和AtCesA8相互作用形成复合体,在木质部细胞中表达[29]。对2到12个月大的辐射松植株的茎进行原位杂交发现PrCesA10在次生木质部中表达,参与次生细胞壁的形成。与BpCesA3高度同源的PtrCesA1和PtrCesA2参与次生细胞壁的合成,且PtrCesA1在木质部特异表达并参与应拉胁迫应答[30]。在本研究中,我们选取了材性不同的单株无性系,研究BpCesA基因在木质部和韧皮部中的表达情况,可以看到在白桦韧皮部中,BpCesA3基因随着纤维素含量的增加,其表达量也是逐渐增加。BpCesA1、BpCesA2、BpCesA4及BpCesA5基因在纤维素最高的植株中表达量也是最高,尤其是BpCesA1。说明这些基因可能参与纤维素合成。其深层的分子调控机制及基因间的协同作用,尚有待于进一步研究。
1.郁书君,汪天,金宗郁,等.白桦容器栽培试验(Ⅰ)—最适基质配方的筛选[J].北京林业大学学报,2001,23(1):24-28.
Yu S J,Wang T,Kim J W,et al.Container culture experiments ofBetulaplatyphylla(Ⅰ)Screening of the optimum formula for substrate[J].Journal of Beijing Forestry University,2001,23(1):24-28.
2.Wei Z G,Zhang K X,Yang C P,et al.Genetic linkage maps ofBetulaplatyphyllaSuk based on ISSR and AFLP markers[J].Plant Molecular Biology Reporter,2010,28:169.
3.宋福南,邢磊,陈肃,等.白桦苯丙氨酸解氨酶(PAL)基因的分离及其表达[J].东北林业大学学报,2009,37(12):14-17,47.
Song F N,Xing L,Chen S,et al.Expression and functional analysis of correlative enzyme genes of lignin phenylpropanoid biosynthetic pathway in Betula PLATPHYLLA[J].Journal of Northeast Forestry University,2009,37 (12):14-17,47.
4.刘雪梅,杨传平,耿峰.白桦胚珠发育及胚、胚乳发育关系的研究[J].植物研究,2005,25(3):322-326.
Liu X M,Yang C P,Geng F.Development of ovule and relation between embryo and endosperm developments ofBetulaplatyphylla[J].Plant research,2005,25(3):322-326.
5.陈鹏飞.白桦纤维素合成酶基因克隆与表达特征分析[D].哈尔滨:东北林业大学,2008.
Chen P F.Cloning and expression characteristics of birch cellulose synthase gene analysis[D].Harbin:Northeast Forestry University,2008.
6.Arioli T,Peng L C,Betzner A S,et al.Molecular analysis of cellulose biosynthesis inArabidopsis[J].Science,1998,279(5351):717-720.
7.Gardiner J C,Taylor N G,Turner S R.Control of cellulose synthase complex localization in developing xylem[J].The Plant Cell,2003,15(8):1740-1748.
8.关录凡.白桦纤维素合成酶基因BpCesA4的遗传转化[D].哈尔滨:东北林业大学,2009.
Guan L F.Genetic transformation of birch cellulose synthase geneBpCesA4[D].Harbin:Northeast Forestry University,2009.
9.Taylor F W.Fiber length measurements-an accurate inexpensive-technique[J].Tapp,1975,12(58):126-127.
10.李斌,顾万春,夏良放,等.鹅掌楸种源材性遗传变异与选择[J].林业科学,2001,37(2):42-50.
Li B,Gu W C,Xia L F,et al.Study on genetic variation and selection of main wood characteristics among provenances of liriodendron[J].Forestry Science,2001,37(2):42-50.
11.Strimmer K,von Haeseler A.Quartet puzzling:a quartet maximum-likelihood method for reconstructing tree topologies[J].Molecular Biology and Evolution,1996,13(7):964-969.
12.Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA) software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
13.Richmond T A,Somerville C R.The cellulose synthase superfamily[J].Plant Physiology,2000,124(2):495-498.
14.Yang X,Sun C,Hu Y,et al.Molecular cloning and characterization of a gene encoding RING zinc finger ankyrin protein from drought-tolerantArtemisiadesertorum[J].Journal of Biosciences,2008,33(1):103-112.
15.Ranik M,Myburg A A.Six new cellulose synthase genes from Eucalyptus are associated with primary and secondary cell wall biosynthesis[J].Tree Physiology,2006,26(5):545-556.
16.Appenzeller L,Doblin M,Barreiro R,et al.Cellulose synthesis in maize:isolation and expression analysis of the cellulose synthase(CesA) gene family[J].Cellulose,2004,11(3-4):287-299.
17.Wang L,Guo K,Li Y,et al.Expression profiling and integrative analysis of theCesA/CSLsuperfamily in rice[J].BMC Plant Biology,2010,10(1):282-297.
18.Yin Y,Huang J,Xu Y.The cellulose synthase superfamily in fully sequenced plants and algae[J].BMC Plant Biology,2009,9:99.
19.Richmond T.Higher plant cellulose synthases[J].Genome Biology,2000,1(4):reviews3001.1.
20.Saxena I M,Brown RM JR,Fevre M,et al.Multidomain architecture of beta-glycosyl transferases:implications for mechanism of action[J].Journal of Bacteriology,1995,177(6):1419-1424.
21.Festucci-Buselli R A,Otoni W C,Joshi C O.Structure,organization,and functions of cellulose synthase complexes in higher plants[J].Brazilian Journal of Plant Physiology,2007,19(1):1-13.
22.Roberts A W,Bushoven J T.The cellulose synthase(CESA) gene superfamily of the mossPhyscomitrellapatens[J].Plant Molecular Biology,2007,63(2):207-219.
23.Hamann T,Osborne E,Youngs H L,et al.Global expression analysis ofCESAandCSLgenes inArabidopsis[J].Cellulose,2004,11(3-4):279-286.
24.Roberts A W,Roberts E.Cellulose synthase(CesA) genes in algae and seedless plants[J].Cellulose,2004,11(3-4):419-435.
25.Mackinnon I M,turcová A,Sugimoto-Shirasu S K,et al.Cell-wall structure and anisotropy in procuste,a cellulose synthase mutant ofArabidopsisthaliana[J].Planta,2006,224(2):438-448.
26.Haigler C H,Ivanova-Datcheva M,Hogan P S,et al.Carbon partitioning to cellulose synthesis[J].Plant Molecular Biology,2001,47(1-2):29-51.
27.Burn J E,Hocart C H,Birch R J,et al.Functional analysis of the cellulose synthase genesCesA1,CesA2 andCesA3 inArabidopsis[J].Plant Physiology,2002,129(2):797-807.
28.Wang L,Guo K,Li Y,et al.Expression profiling and integrative analysis of theCESA/CSLsuperfamily in rice[J].BMC Plant Biology,2010,10:282.
29.Krauskopf E,Harris P J,Putterill J.The cellulose synthase genePrCESA10 is involved in cellulose biosynthesis in developing tracheids of the gymnospermPinusradiata[J].Gene,2005,350(2):107-116.
30.Samuga A,Joshi C P.A new cellulose synthase gene(PtrCesA2) from aspen xylem is orthologous toArabidopsisAtCesA7(irx3) gene associated with secondary cell wall synthesis[J].Gene,2002,296(1-2):37-44.
National Science and Technology Planning Project of “12th Five-Year”(2012BAD21B02); State Key Laboratory of Tree Genetics and Breeding Northeast Forestry University(201203)
introduction:SHEN Ting-Ting(1990—),female,master,tree genetics and breeding research.
date:2016-09-09
BioinformaticsandExpressionAnalysisofBpCesAGenesofBetulaplatyphylla
SHEN Ting-Ting1JIANG Jing1LIU Gui-Feng1XU Si-Jia1LI Hui-Yu1*YUAN Hong-Mei1,2
(1.State Key Laboratory of Tree Genetics and Breeding,Northeast Forestry University,Harbin 150040;2.Industrial Crops Institute of Heilongjiang Academy of Agricultural Sciences,Harbin 150086)
Six cellulose synthase genes ofBetulaplatyphyllafrom 45 transcriptome data sets were obtained, in which two genes were new genes of cellulose synthase gene family. The sequence characteristic and expression levels in Birch xylem ofBpCesAwere analyzed using bioinformatics and quantitative real-time PCR. The ORF length of 6BpCesAgenes varied from 2 958-3 312 bp; Number of amino acids encoded varied from 985-1 103 aa, and relative molecular weight varied from 110.40-124.29 kD; Theoretical isoelectric point was from 5.90-7.43. SixBpCesAgenes all contained D, D, D, QXXRW conservative structure domain and eight transmembrane helices, in which two were located at the end of the N, and the others were at the end of the C. 52CesAsfrom six dicotyledonous plants, four monocotyledons, and one gymnosperms were analyzed by cluster analysis, andBpCesAshad higher homology withPtrCesA. The homology betweenBpCesA2 andPtrCesA3 reached 92%.BpCesA5 andPtrCesA6,BpCesA1 andPtrCesA8 also had high homology of 89% and 82%, respectively. The RNA level ofBpCesA1,BpCesA2,BpCesA3 andBpCesA5 was increased with the increase of cellulose content. This results suggested that these genes play a positive role in cellulose synthesis in birch. This study provides a basis for the functional analysis of cellulose synthase gene.
Betulaplatyphylla;cellulose;lignin;bioinformatics;expression quantity
“十二五”农村领域国家科技计划课题(2012BAD21B02);东北林业大学林木遗传育种国家重点实验室开放基金项目(201203)
申婷婷(1990—),女,硕士研究生,主要从事林木遗传育种研究。
* 通信作者:E-mail:lihuiyu0519@aliyun.com
2016-09-09
* Corresponding author:E-mail:lihuiyu0519@aliyun.com
S792.153
A
10.7525/j.issn.1673-5102.2016.06.015

