2个慈竹NAC转录因子的克隆与组织表达分析
李会萍 黎帮勇 胡尚连 曹 颖
(1.西南科技大学植物细胞工程实验室,绵阳 621010; 2.四川省生物质资源利用与改性工程技术研究中心, 绵阳 621010)
2个慈竹NAC转录因子的克隆与组织表达分析
李会萍 黎帮勇 胡尚连*曹 颖
(1.西南科技大学植物细胞工程实验室,绵阳 621010;2.四川省生物质资源利用与改性工程技术研究中心, 绵阳 621010)
以慈竹转录组数据为基础,结合毛竹基因组数据,采用同源克隆技术从慈竹中克隆到两个NAC基因BeNAC1(GenBank注册号:KU550706)和BeNAC2(GenBank注册号:KU821586),分别编码313和389个氨基酸残基。蛋白功能域预测和保守结构域多重对比分析表明,BeNAC1和BeNAC2基因均含有NAM保守域,其模拟的蛋白质三维结构与已知晶体结构的水稻模型蛋白高度相似。亚细胞定位预测分析显示,BeNAC1主要集中在细胞质中;BeNAC2则主要集中在细胞核中。表达分析表明,BeNAC1和BeNAC2具有相似的组织特异性表达,在茎中表达量均明显高于笋和叶片,且BeNAC1在除笋之外各组织中的表达量均显著高于BeNAC2。
慈竹;NAC转录因子;克隆;生物信息学分析;组织表达模式
慈竹(Bambusaemeiensis)是西南地区尤其四川省的主要竹种之一,其纤维具有一定的机械强度,故具备生产较高强度纸张的条件[1]。由于开花周期难以预测,且开花竹生长即衰败[2],使其传统遗传改良方法受到极大的限制。以往对慈竹在生长状况[3]、生理生化[4]、遗传多样性[5]、功能基因克隆[6]等方面的研究,为其品种改良提供了一定的基础,但有关NAC转录因子方面的研究鲜见报道。
NAC转录因子是植物特有的一类转录因子,它的N端为高度保守的结构域,C端为高度变异的转录激活区[7~8]。自从Souer等从矮牵牛(Petuniahybrida)中克隆到第一个NAC基因NAM(no apical meristem)以来[9],NAC在植物生长发育和逆境胁迫的响应中的重要作用不断被揭示[10]。如拟南芥(Arabidopsisthaliana)NAC1转录因子受生长素诱导,同时介导生长素信号来促进侧根的发育[11]。水稻NAC29/31影响次生壁的发育,能够激活MYB61,进而激活次生壁纤维素合成基因(CESA)表达,调控通路NAC29/31-MYB61-CESA影响水稻次生壁纤维素合成[10];而水稻OsNAC5和OsNAC6则参与调控逆境应答,其表达受到旱、冷、高盐等非生物胁迫和ABA的诱导[12];但对慈竹NAC转录因子的相关研究却鲜见报道。
随着高通量测序技术的发展,RNA-Seq测序技术已经成为了转录组分析的标准技术,利用该技术得到慈竹转录组数据库[13],本研究基于课题组慈竹转录组数据库,克隆得到两个NAC基因,并对其进行生物信息学分析以及组织特异性表达分析,为慈竹品质改良提供一定基础。
1 材料与方法
1.1 植物材料
以慈竹的笋(100 cm)、未展开叶、展开叶、茎为试验材料,于液氮中保存,置于-80℃超低温冰箱中备用(以上植物材料皆取自西南科技大学生命科学与工程学院资源圃)。
1.2 主要试剂和菌种
Plant RNA Kit试剂盒、DNA胶回收试剂盒购自Omega Bio-tec公司;LA-Taq聚合酶、pMDTM19-T载体、PrimeScript®RT reagent Kit(Perfect DNA)反转录试剂盒、EcoRⅠ、HindⅢ等购自宝生物工程(大连)TaKaRa公司;质粒小提试剂盒、X-Gal等购自北京天根公司;Green View染料购自BioBRK公司;大肠杆菌DH5α感受态由本试验室制备。
1.3 试验方法1.3.1 慈竹总RNA的提取和cDNA的合成
参照Plant RNA Kit试剂盒说明书方法提取慈竹笋(高100 cm)、未展开叶、展开叶、茎等组织总RNA。以Oligo dT为引物,按照PrimeScript®RT reagent Kit prefect real Time反转录试剂盒说明书的方法将总RNA反转录成cDNA。
1.3.2 慈竹BeNAC1和BeNAC2的克隆
以慈竹转录组数据为基础,结合转录因子数据库(plantTFDB)中的毛竹NAC基因序列,采用Primer Premier 5.0软件设计2对引物(BeNAC1-F和BeNAC1-R;BeNAC2-F和BeNAC2-R,表1),以慈竹笋的cDNA为模版分别进行扩增。PCR的反应条件为:95℃预变性3 min,95℃变性30 s,55℃退火30 s,72℃延伸分为1 min(BeNAC1)和1∶20 min(BeNAC2),30个循环,72℃延伸10 min,4℃保持。
表1慈竹BeNAC1和BeNAC2基因克隆和Real-timePCR分析所用引物
Table1PrimersusedinBeNAC1andBeNAC2genescloningandreal-timePCRanalysisinB.emeiensis
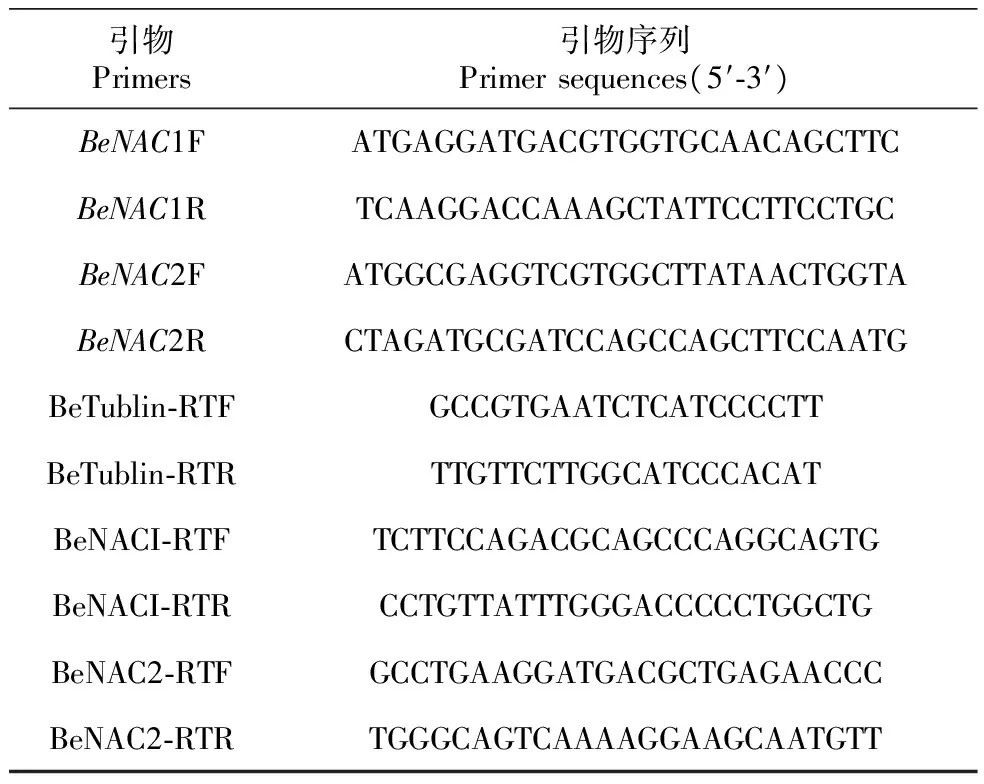
引物Primers引物序列Primersequences(5′⁃3′)BeNAC1FATGAGGATGACGTGGTGCAACAGCTTCBeNAC1RTCAAGGACCAAAGCTATTCCTTCCTGCBeNAC2FATGGCGAGGTCGTGGCTTATAACTGGTABeNAC2RCTAGATGCGATCCAGCCAGCTTCCAATGBeTublin⁃RTFGCCGTGAATCTCATCCCCTTBeTublin⁃RTRTTGTTCTTGGCATCCCACATBeNACI⁃RTFTCTTCCAGACGCAGCCCAGGCAGTGBeNACI⁃RTRCCTGTTATTTGGGACCCCCTGGCTGBeNAC2⁃RTFGCCTGAAGGATGACGCTGAGAACCCBeNAC2⁃RTRTGGGCAGTCAAAAGGAAGCAATGTT
将回收纯化的的PCR产物插入到pMDTM19-T载体(大连TaKaRa公司)克隆载体,在上海英潍捷基公司测序。
1.4慈竹BeNAC1和BeNAC2基因的生物信息学分析
用NCBI在线工具Conserved对两个NAC编码的氨基酸序列的结构域进行预测分析。从NCBI数据库中获得其它物种的NAC功能性氨基酸和核苷酸序列,DAMBE检测饱和度[14~15],用ModelGenerator选择最适建树模型[16],整理后导入Mega7.0软件,用最大似然法(Maximum Likehood,ML)构建系统进化树(Bootstrap参数为1000 replicates)。用ClustalX1.8.3软件对不同植物的NAC转录因子的NAM结构域进行多重序列比对分析。MEME、SOPMA、Phyre2.0等在线工具分别对NAC蛋白序列进行模体、二级结构和三级结构分析。用Yloc[17~18]进行亚细胞定位预测分析(选择模型:Plants YLoc-HighRes)。
1.5 慈竹BeNAC1和BeNAC2的组织表达分析
根据测序的慈竹BeNAC1和BeNAC2基因的全长序列,设计real-time PCR引物(BeNAC1-RTF和BeNAC1-RTR;BeNAC2-RTF和BeNAC2-RTR,表1)。并以慈竹Tublin基因作为内参,引物为BeTublin-RTF和BeTublin-RTR。按照Real Master Mix(SYBR Green)试剂盒中20 μL体系的说明加样。在iQ5 Multicolor Real-Time PCR自动扩增仪上进行real-time PCR反应。每个样品重复3次,最后采用2-ΔΔCt法[19]进行结果分析。
2 结果与分析
2.1 慈竹BeNAC1和BeNAC2基因的克隆及鉴定
以慈竹笋的cDNA为模版进行克隆,获得两条约1 000 bp的片段。测序结果显示,获得的BeNAC1和BeNAC2基因的开放阅读框长度分别为939和1 167 bp,分别编码313和389个氨基酸(图1)。用NCBI在线工具Conserved[20]分别对其进行保守结构域预测,BeNAC1和BeNAC2转录因子分别在67-208和57-201氨基酸位点含有NAM保守结构域。NAM保守结构域是NAC家族转录因子的特征结构域,进一步确认BeNAC1和BeNAC2转录因子属于NAC家族。将所得的新序列分别命名为BeNAC1和BeNAC2,GenBank注册号分别为KU550706和KU821586。

图1 慈竹BeNAC1(A)和BeNAC2(B)基因的核苷酸序列及其编码的氨基酸序列 下划线部分为NAM结构域;“*”代表终止密码子Fig.1 The nucleotide and encoded amino acid sequences of BeNAC1(A)and BeNAC2(B) The underlined part is the NAM domain; “*” represents the stop codon
2.2 慈竹BeNAC1和BeNAC2基因进化树分析
分别以BeNAC1和BeNAC2的氨基酸序列为探针,通过Blast P程序从NCBI在线数据库中检索到与慈竹BeNAC1和BeNAC2同源性较高其他植物NAC序列。拟南芥ANAC036(NP_565404)、ANAC001(EFH68348)、CUC1(NP_188135)、SND2(AT4G28500.1)、SND3(AT1G28470.1)、VND6(NP_201044)、VND7(NP_177338)、ANAC008(At1g25580);水稻ONANC003(AK061716);矮牵牛NAM(CAA63102)和烟草TERN(BAA78417)。利用MEGA7.0软件构建系统发育进化树(建树模型JTT+I+F+G)。如图2所示,BeNAC1与ONAC003、SND2和SND3聚在一个分支上;BeNAC2与ANAC008聚在一个分支上。这两个分支皆属于第II组中ONAC003亚家族[21],可以推测BeNAC1和BeNAC2分别与ONAC003/SND2/SND3和ANAC008在进化上的亲缘关系上具有一定的相似性。用ModelGenerator进行氨基酸替代率分析,每个氨基酸的频率分别为5.46%(A),4.88%(R),4.75%(N),5.64%(D),1.91%(C),4.19%(Q),7.14%(E),7.14%(G),3.77%(H),4.08%(I),6.83%(L),6.66%(K),2.22%(M),3.68%(F),6.06%(P),9.08%(S),5.84%(T),1.86%(W),3.28%(Y)和5.53%(V)。根据分析的数据显示,氨基酸的替换率在0.00~2.19。
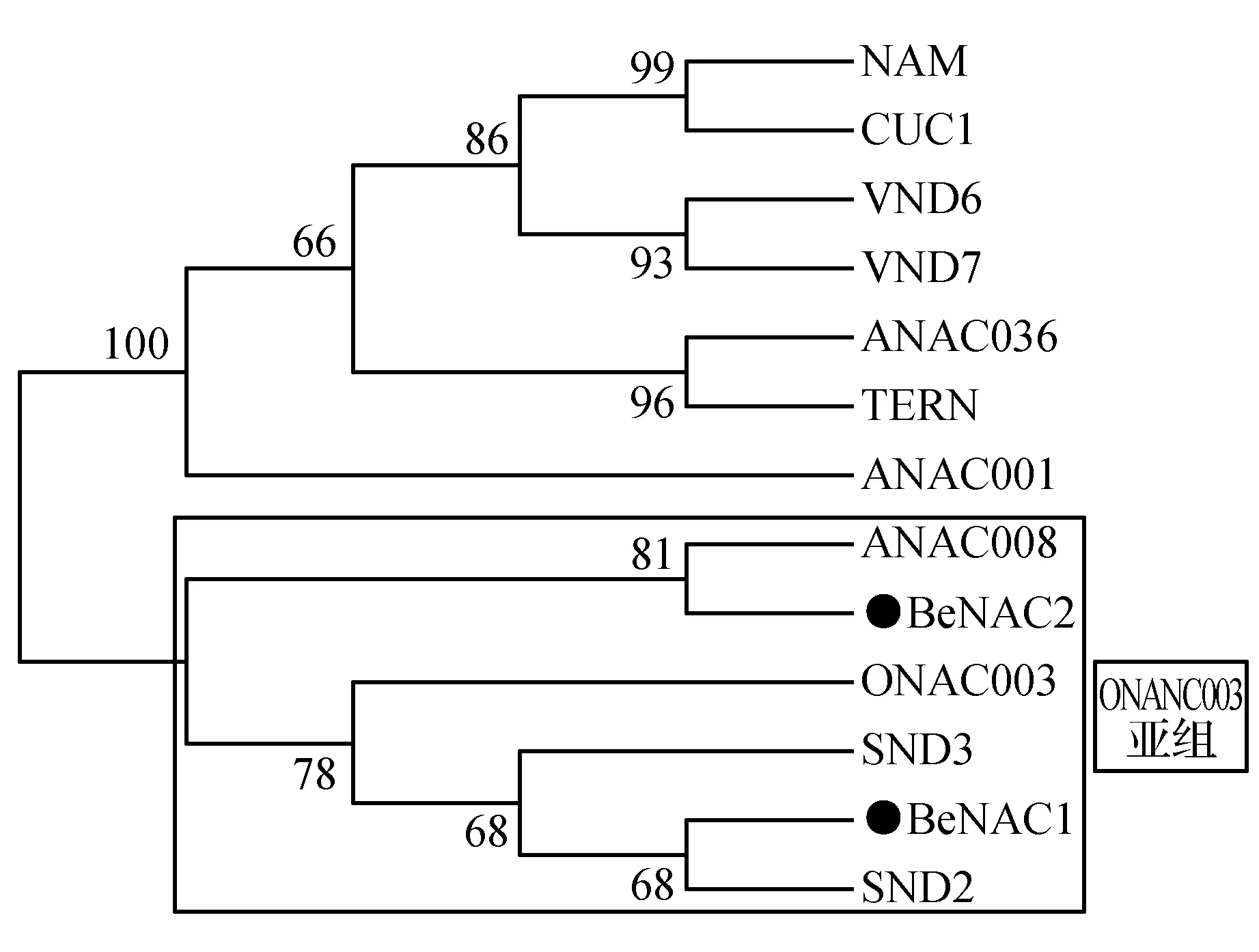
图2 BeNAC1、BeNAC2和其他植物NAC的系统进化树Fig.2 The phylogenetic tree of BeNAC1 and BeNAC2 protein
用Clustal W程序将BeNAC1和BeNAC2与拟南芥ANAC036(NP_565404)、ANAC001(EFH68348)、CUC1(NP_188135);水稻ONANC003(AK061716)、OsNAC5(BAA89799)和矮牵牛NAM(CAA63102)的NAC家族成员进行保守结构域多重比对分析。结果表明,BeNAC1和BeNAC2与ONAC003转录因子N端保守域的氨基酸相似性最高(图3),这与上述进化树分析的结果保持一致。且整个NAM结构域均含有相同的、保守性高的氨基酸。
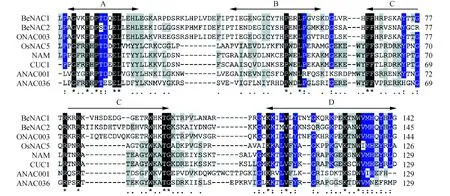
图3 BeNAC1和BeNAC2的NAM保守结构域多重比对分析 “*”表示保守氨基酸;“:”表示保守替换;“.”表示非保守替换;“-”表示缺失;阴影的黑色、蓝色、灰色分别表示保守性为100%、≥75%、≥50%的氨基酸;A~D为NAC转录因子N端保守区的保守亚域Fig.3 Multiple alignment analysis of NAM conservative structure domain in BeNAC1 and BeNAC2 “*” represents a conserved amino acid; “:” represents a conservative replacement; “.” represents non conservative replacement; “-” means missing; The shadow of the black, blue, grey represent conservative amino acid was 100%, ≥75%, ≥50%; A-D is a conserved subunit of the NAC transcription factor N terminal conserved region
2.3慈竹BeNAC1和BeNAC2蛋白的保守基序分析
利用MEME[22]在线工具对慈竹、水稻、拟南芥的NAC转录因子编码的蛋白序列模体进行识别分析(图4)。通常情况下,系统进化树分析中聚在一个亚组的蛋白质会有相似的motif组成,可以看出,BeNAC1、BeNAC2、ONAC003、SND2、SND和ANAC008均具有5个Motif。Motif 4、1、3、2分别对应NAM多重比对分析A、B、C和D四个亚域,其中Motif 2基序末端序列中包含了NAM多重比对分析中未列出的E亚族序列,可以看出其氨基酸序列保守性不高。
蛋白质二级结构预测表明,这两种蛋白都是以无规则卷曲为主,其中BeNAC1和BeNAC2中分别含有26.84%和32.47%的α-螺旋,10.03%和10.05%的β-转角,18.88%和15.72%的延伸链与44.25%和41.75%的无规则卷曲。以c3ulxA为模型[23],用Phyre2.0[24]对BeNAC1和BeNAC2进行三级结构进行预测。结果显示,这两个蛋白与c3ulxA的三级构型高度相似(c3ulxA:已经被X-Ray解析出晶体模型;PDB ID:3ULX)。图5可以看出BeNAC1和BeNAC2的三级结构主要以β-折叠为主,有少部分的α-螺旋。Yloc亚细胞定位预测结果表明,BeNAC1集中于细胞质中的概率是75.5%(可信度为0.69);BeNAC2则集中于细胞核中的概率为88.7%(可信度为0.93)。
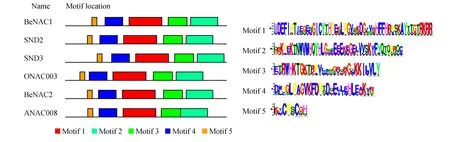
图4 BeNAC1和BeNAC2蛋白保守基序分析Fig.4 The conserved motif analysis of BeNAC1 and BeNAC2 protein
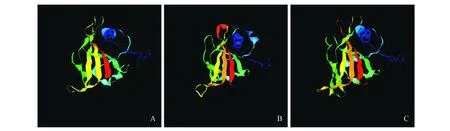
图5 NAC蛋白质三级结构预测 A.模型c3ulxA的三级结构;B. BeNAC1的三级结构;C. BeNAC2的三级结构Fig.5 NAC protein tertiary structure prediction A. The tertiary structure of model c3ulxA; B. The tertiary structure of BeNAC1; C. The tertiary structure of BeNAC2
2.4慈竹BeNAC1和BeNAC2的组织表达模式分析
分别取慈竹的笋(100 cm)、未展开叶、展开叶和茎,通过荧光定量检测BeNAC1和BeNAC2基因在不同组织中的表达情况。BeNAC1在慈竹中的表达量为茎>展开叶>笋>未展开叶;BeNAC2中为茎>笋>展开叶>未展开叶(图6)。可以看出两个基因在茎中的表达量皆大于在其它组织中的表达量。特别是BeNAC1在茎中表达量几乎是笋中表达量的12倍,未展叶表达量最低;BeNAC2在茎中表达量为笋中表达量的2倍多,在展开叶和未展叶中表达量最少。
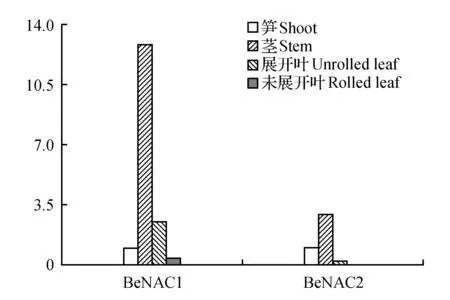
图6 同一慈竹NAC基因在不同组织中的表达分析Fig.6 The same sinocalamus affinis NAC gene expression analysis in different organizations
3 讨论
近年来植物细胞次生壁形成的调控取得了显著的进展,而NAC作为关键调节因子,在其中起着举足轻重的作用。目前,对次生生长相关NAC的研究大都集中在水稻、拟南芥等模式植物中[10]。慈竹是造纸工业的优质原料,提高其纤维素含量有着重要的意义,但目前对慈竹次生生长相关的NAC研究鲜见报道。
本研究克隆了2个慈竹NAC转录因子BeNAC1和BeNAC2。序列分析表明,这两个基因都含有NAM特征结构域。根据保守氨基酸的分布,该NAM结构域可以分为A、B、C、D等四个亚域[21],其中A、C和D亚域高度保守,B和E两个亚域的氨基酸富于变化[9](B和E子结构域在Ⅱ组中不保守,而在Ⅰ组的NAP,AtNAC3,ATAF和OsNAC3亚组中保守)[25],因为E亚域氨基酸在此处的的保守性极低[21],变化很大。此处未将E亚域序列包含在保守结构域内。由于N端存在保守的碱性氨基酸,这些氨基酸使NAC蛋白相应的表面上富含正电荷,很有可能直接参与了与DNA的结合[26]。
研究已经表明,ONAC003(SNAC3)能增强水稻对高温、干旱和渗透胁迫的耐受性。SNAC3介导活性氧(ROS)代谢,正向调控ROS清除基因的表达,故而增强了水稻对胁迫的耐受能力[27]。SND2/3除了能够调节纤维素和半纤维素的生物合成外,还参与木质素聚合和信号传导[28]。ANAC008能够编码γ反应抑制因子(SOG1),可能对DNA的损伤有多重响应。通过进化树比对分析发现,这些功能性NAC与BeNAC1和BeNAC2进化关系比较近,同属于ONAC003亚族。所以推测他们在功能上也具有一定的相似性,BeNAC1转录因子除了能参与植物的非生物胁迫下的防御应答,还可能参与纤维细胞的发育调控;BeNAC2除了有上述功能外,还可能参与SOG1的合成调控。另外,表达分析表明BeNAC1和BeNAC2基因在茎中的表达量都远高于其它组织。与其他组织相比,茎部具有较高的纤维素含量,这些证据暗示,这两个转录因子可能参与慈竹茎部的纤维素合成调控过程,与系统发育树的聚类结果一致。但以上推测均需要进一步的深入研究与验证。
1.周宗昌.全慈竹浆生产纸袋纸研究[J].西南造纸,1989(2):30-31.
Zhou C Z.Study on the production of paper bags fromBambusaemeiensispulp[J].Southwest Pulpand Paper,1989(2):30-31.
2.谢寅峰,张春霞,丁雨龙.竹子开花人工诱导与逆转研究现状及前景[J].浙江农林学院学报,2000,17(4):436-440.
Xie Y F,Zhang C X,Ding Y L.Research status and Prospect of artificial induction and reversion of bamboo flowering[J].Journal of Zhejiang A &F University,2000,17(4):436-440.
3.刘庆,何海,沈昭萍.成都地区慈竹生长状况及其与环境因子关系的初步分析[J].四川环境,2001,20(4):43-46.
Liu Q,He H,Shen S P.Study on the Growth Status and Correlative Environment Factors of Bamboo,Bambusaemeiensisfrom Chengdu Region[J].Sichuan Environment,2001,20(4):43-46.
4.胡尚连,蒋瑶,陈其兵,等.四川2种丛生竹理化特性及纤维形态研究[J].植物研究,2010,30(6):708-712.
Hu S L,Jiang Y,Chen Q B,et al.Physical and Chemical Properties of 2 Species in Bamboos from the Different Regions in Sichuan Province[J].Bulletin of Botanical Research,2010,30(6):708-712.
5.陈其兵,蒋瑶,卢学琴,等.四川不同地区慈竹的遗传多样性研究[J].西北农林科技大学学报:自然科学版,2009,37(6):187-193.
Chen Q B,Jiang Y,Lu X Q,et al.Assessment of genetic diversity in Neosinocalamus affinis from different regions of Sichuan province[J] Journal of Northwest A&F University:Natural Science Edition,2009,37(6):187-193.
6.胡尚连,曹颖,黄胜雄,等.慈竹4CL基因的克隆及其生物信息学分析[J].西北农林科技大学学报:自然科学版,2009,37(8):204-210.
Hu S L,Cao Y,Huang S X,et al.Cloning and bioinformation analysis of 4CL gene inNeosinocalamusaffinis[J].ournal of Northwest A&F University:Natural Science Edition,2009,37(8):204-210.
7.Olsen A N,Ernst H A,Leggio L L,et al.NAC transcription factors:structurally distinct,functionally diverse[J].Trends in Plant Science,2005,10(2):79-87.
8.Riechmann J L,Heard J,Martin G,et al.Arabidopsistranscription factors:genome-wide comparative analysis among eukaryotes[J].Science,2000,290(5499):2105-2110.
9.Souer E,van Houwelingen A,Kloos D,et al.The no apical meristem gene ofPetuniais required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell,1996,85(2):159-170.
10.Huang D,Wang S,Zhang B,et al.A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice[J].Plant Cell,2015,27(6):1681-1696.
11.Xie Q,Frugis G,Colgan D,et al.Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J].Genes & Development,2000,14(23):3024-3036.
12.Takasaki H,Maruyama K,Kidokoro S,et al.The abiotic stress-responsive NAC-type transcription factor OsNAC5 regulates stress-inducible genes and stress tolerance in rice[J].Molecular Genetics and Genomics,2010,284(3):173-183.
13.杨传凤,曹颖,胡尚连,等.基于慈竹转录组MYB基因的克隆及胁迫诱导表达[J].森林与环境学报,2015,35(1):60-66.
Yang C F,Cao Y,Hu S L,et al.Cloning of MYB gene based onBambusaemeiensistranscriptome and their induced expression under stresses[J].Journal of Forest and Environment,2015,35(1):60-66.
14.Xia X H,Xie Z,Salemi M,et al.An index of substitution saturation and its application[J].Molecular Phylogenetics and Evolution,2003,26(1):1-7.
15.Xia X,Lemey P.Assessing substitution saturation with DAMBE[M].//Lemey P,Salemi M,Vandamme A M.The phylogenetic handbook.Cambridge:Cambridge University Press,2009:611-626.
16.Keane T M,Creevey C J,Pentony M M,et al.Assessment of methods for amino acid matrix selection and their use on empirical data shows that ad hoc assumptions for choice of matrix are not justified[J].BMC Evolutionary Biology,2006,6:29.
17.Briesemeister S,Rahnenführer J,Kohlbacher O.Going from where to why-interpretable prediction of protein subcellular localization[J].Bioinformatics,2010,26(9):1232-1238.
18.Briesemeister S,Rahnenführer J,Kohlbacher O.YLoc-an interpretable web server for predicting subcellular localization[J].Nucleic Acids Research,2010,38(S2):W497-W502.
19.Pfaffl M W.A new mathematical model for relative quantification in real-time RT-PCR[J].Nucleic Acids Research,2001,29(9):e45.
20.Marchler-Bauer A,Derbyshire M K,Gonzales N R,et al.CDD:NCBI’s conserved domain database[J].Nucleic Acids Research,2015,43(D1):D222-D226.
21.Ooka H,Satoh K,Doi K,et al.Comprehensive analysis of NAC family genes inOryzasativaandArabidopsisthaliana[J].DNA Research,2009,34(6):239-247.
22.钟慧.两个油菜NAC基因的鉴定及其表达和功能的初步研究[D].武汉:华中师范大学,2011.
Zhong H,Idengtification of twoNACgenes and their expression and function inBrassicanapus[D].Wuhan: Huazhong Normal Universiry,2011.
23.Bailey T L,Elkan C.Fitting a mixture model by expectation maximization to discover motifs in biopolymers[C].//Proceedings of the international conference on intelligent systems for molecular biology.Stanford,CA,1994.2:28-36
24.Chen Q F,Wang Q,Xiong L Z,et al.A structural view of the conserved domain of rice stress-responsive NAC1[J].Protein & Cell,2011,2(1):55-63.
25.Kelley L A,Mezulis S,Yates C M,et al.The Phyre2 web portal for protein modeling,prediction and analysis[J].Nature Protocols,2015,10(6):845-858.
26.张进艳.玉米NAC转录因子家族特性分析及与水分胁迫相关性[D].兰州:甘肃农业大学,2014.
Zhang J Y.Characteristic analysis of NAC transcription factor family and correlation with water stress inZeamaysL.[D].Lanzhou:Gansu Agricultural University,2014.
27.Fang Y J,Liao K F,Du H,et al.A stress-responsive NAC transcription factor SNAC3 confers heat and drought tolerance through modulation of reactive oxygen species in rice[J].Journal of Experimental Botany,2015,66(21):6803-6817.
28.Hussey S G,Mizrachi E,Spokevicius A V,et al.SND2,a NAC transcription factor gene,regulates genes involved in secondary cell wall development inArabidopsisfibres and increases fibre cell area inEucalyptus[J].Bmc Plant Biology,2011,11:173.
National Natural Science Foundation of China(Nos.31400257,31400333); Breeding Program Fund Project by the 13th Five-Year Plan of Sichuan Province(Nos:16ZS212301); Fund of Engineering Research Center for Biomass Resource Utilizaiton and Modification of Sichuan Province(Nos.12zxsk07,13zxsk01); Graduate innovation fund of Southwest University of Science and Technology(Nos.16ycx069)
introduction:LI Hui-Ping(1991—),female,master,research mainly focuses on plant genetics and variety improvement.
date:2016-08-30
CloningandExpressionAnalysisofTwoNACTranscriptionFactorsinBambusaemeiensis
LI Hui-Ping LI Bang-Yong HU Shang-Lian*CAO Ying
(1.Lab of Plant Cell Engineering Southwest University of Science and Technology,Mianyang 621010;2.Engineering Research Center for Biomass Resource Utilization and Modification of Sichuan Province,Mianyang 621010)
Based on the transcriptome database ofBambusaemeiensisand genome information ofPhyllostachysheterocycla, the two NAC genes were cloned by homology cloning technology from the shoots ofB.emeiensis, named asBeNAC1(GenBank: KU550706) andBeNAC2(GenBank: KU821586), encoding 313 and 389 amino acids, respectively. By protein function prediction and conserved domain multiple alignment, both ofBeNAC1 andBeNAC2 contained NAM conserved region. By protein structure prediction, the putatived three-dimensional structure ofBeNAC1 andBeNAC2 protein was similar to a rice protein with known crystal structure. By subcellular localization prediction analysis, BeNAC1 was mainly concentrated in the cytoplasm, and BeNAC2 was mainly concentrated in the nucleus. Tissue expression pattern analysis showed thatBeNAC1 andBeNAC2 displayed similar tissue-specific, both having the highest expression on stems, whereas in addition to the bamboo shoots, the expression ofBeNAC1 in each tissue tested was higher than that ofBeNAC2.
Bambusaemeiensis;NAC transcription factors;clone;bioinformatics analysis;tissue expression pattern
国家自然科学基金(31400257,31400333);四川省“十三五”重点公关资助项目(16ZS212301);四川省生物质资源利用与改性工程技术研究中心基金资助(12zxsk07,13zxsk01);西南科技大学研究生创新基金资助项目(16ycx069)
李会萍(1991— ),女,硕士研究生,从事植物遗传与品种改良研究。
* 通信作者:E-mail:hushanglian@126.com
2016-08-30
* Corresponding author:E-mail:hushanglian@126.com
S795.5
A
10.7525/j.issn.1673-5102.2016.06.013

