羌活(Notopterygium incisum)nrDNAITS和cpDNA rpl20-rps12序列分子进化特点的分析
杨路存 刘何春 周学丽 徐文华 周国英*
(1.中国科学院西北高原生物研究所,810008; 2.中国科学院藏药研究重点实验室,西宁 810008; 3.中国科学院大学,北京 100049; 4.青海省铁卜加草原改良试验站,西宁 810008)
* 通信作者:E-mail:zhougy@nwipb.cas.cn
羌活(Notopterygiumincisum)nrDNAITS和cpDNArpl20-rps12序列分子进化特点的分析
杨路存1,2刘何春1,3周学丽4徐文华1,2周国英1,2*
(1.中国科学院西北高原生物研究所,810008;2.中国科学院藏药研究重点实验室,西宁 810008;3.中国科学院大学,北京 100049;4.青海省铁卜加草原改良试验站,西宁 810008)
应用PCR产物直接测序法分析了羌活居群间nrDNA(核糖体DNA)ITS序列和cpDNA(叶绿体DNA)rpl20-rps12的碱基差异,从而初步研究两套植物基因组的变异速率。采用改良的CTAB法从硅胶干燥的羌活叶片中提取总DNA,并对nrDNA ITS和cpDNA rpl20-rps12区域进行扩增、纯化、测序。nrDNAITS序列共有635 bp,有变异位点17处,变异位点百分率为2.68%,(G+C)含量为57.83%。cpDNA rpl20-rps12序列共有767 bp,有变异位点35处,变异位点百分率4.56%,(G+C)含量为33.06%。比较发现,羌活nrDNAITS区域较cpDNA rpl20-rps12序列保守,变异速率较慢。通过对ITS和rpl20-rps12序列单倍型(haplotype)进行分析发现,两者得出的结论一致,即现有分布范围经历了居群近期范围扩张。因此,羌活nrDNA ITS序列适合该种的谱系地理学研究。
羌活;ITS序列;rpl20-rps12序列
羌活(Notopterygiumincisum)隶属于伞形科(Umbelliferae)羌活属(Notopterygium),是中国的特有种[1~2]。主要分布在青海、甘肃、四川[3]。生于海拔2 500~3 500 m的高山灌木林、亚高山灌丛、草丛及高山林缘地上。羌活与同属的宽叶羌活(N.forbesii)同为药材羌活的基源植物,以根茎及根入药[4],是中、藏、羌医药体系中常用药材,主要含挥发油和香豆素类成分。具有散寒、祛风、除湿、止痛功效,主治风寒感冒头痛、风湿痹痛等症,现代药理研究表明对心脑血管疾病也有确切疗效[5]。目前,用羌活的中(藏)成药有200余种,用药需求量非常大,长期仅靠野生采集满足国内外市场需求,以致资源已濒临灭绝,1987年即被国务院《中国野生药材资源保护管理条例》列为Ⅲ级保护物种,2005年又载入《中国物种红色名录》[6]。目前,羌活植物化学[7~8]、药理[9]、地理分布[3,10]、分类学[1~2]、环境土壤学[11]和驯化栽培[12~13]等方面的研究比较多,对其nrDNA ITS序列和cpDNA rpl20-rps12序列的研究尚属首次。
随着分子生物学技术的快速发展,用于分子系统学和谱系地理学研究的分子标记从RFLP、RAPD、SSR和ISSR过渡到核糖体DNA(nrDNA)的18S基因及ITS等非编码区,以及叶绿体DNA的编码基因及非编码区序列和部分线粒体DNA(mtDNA)的基因片段。目前应用最广泛的核基因组是nrDNAITS序列,它是核糖体DNA中介于18S和5.8S之间(ITS1)以及5.8S和26S之间(ITS2)的非编码转录间隔区,具有较高的重组率,成为植物谱系地理学(Phylogeography)研究和探讨科内属间及属下种间关系中最有效的手段之一[14~15]。叶绿体DNA(cpDNA)多数具有单亲遗传的特性,在遗传过程中不易发生重组,并且没有基因的重叠、缺失或假基因的现象,拥有独立的进化路线,不依赖其他数据,可以通过自身数据构建分子系统来推测物种的进化历史[16~17]。而rpl20-rps12属于叶绿体非编码区域,有较高的突变频率,且这些突变不受自然选择等其他因素的影响,属于中性突变,可以保留经长期的变迁的植物的遗传结构(如过去的迁移路线、建群的原动力等),经过长年累月的变化可以提供丰富的信息,是植物系统地理学研究的首选分子标记[18]。本文分析了羌活12个居群,116个个体的nrDNAITS和cpDNA rpl20-rps12序列区域,研究该种nrDNAITS和cpDNA rpl20-rps12序列的分子特点和变异情况,并从这两个序列初步确定该种形成现有分布范围的成因。
1 材料与方法
1.1 材料
用于本实验的12个居群为2014年6~9月份分别采自青海、四川和甘肃3省区,基本包括了我国羌活主要产区。采样时每个居群选取成年植株6~15株,根据随机取样原则,取样的羌活株距在50 m以上。野外采集新鲜、完整的植物幼嫩叶片,置于盛有硅胶的塑胶袋中干燥保存。采样编号及地点见表1。
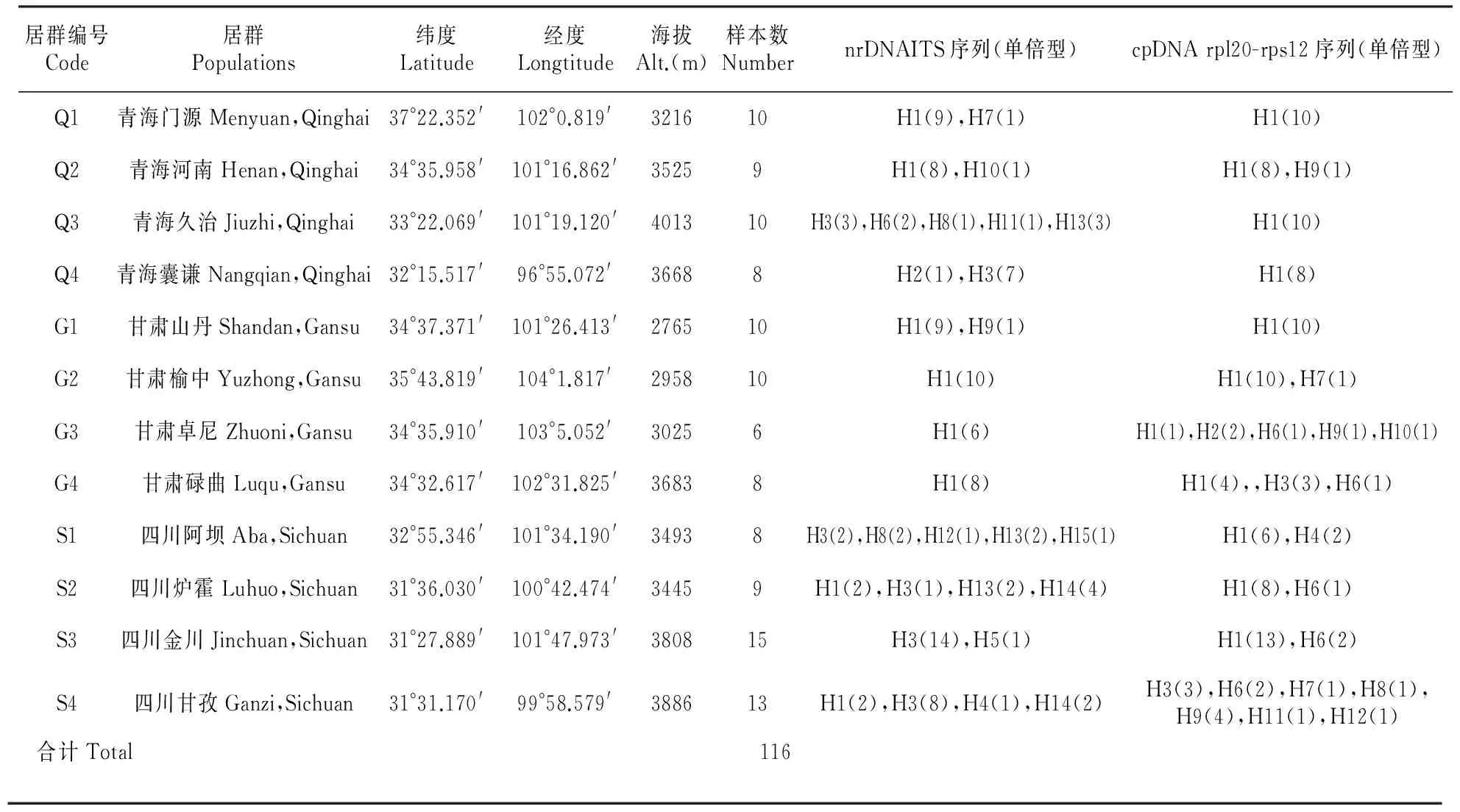
表1 12个羌活居群的采样表
1.2 实验方法
1.2.1 基因组DNA的提取与检测
依据改良的CTAB法从硅胶干燥的叶片中提取总DNA。通过测定紫外光吸收值来确定DNA浓度和纯度。利用0.8%琼脂糖凝胶电泳检查DNA完整性。
1.2.2 PCR扩增与DNA测序
采用通用引物“ITS1”(5′-AGAAGTCGTAACAAGGTTTCCGTAGG-3′)和“ITS4”(5′-TCCTCCGCTTATTGATATGC-3′);“rpl20”(5′-TTTGTTCTACGTCTCCGAGC-3′)和“rps12”(5′-GTCGAGGAACATGTACTAGG-3′)分别扩增nrDNA ITS序列和cpDNA rpl20-rps12序列。扩增反应在Eppendorf Mastercycler Gradient PCR仪上进行,反应体系为25 μL,内含25 mmol·L-1MgCl22 μL、2.5 mmol·L-1dNTPs 2 μL、10 μmol·L-1引物各0.6 μL、25 ng模板、10×buffer 2.5 μL和1U Taq DNA聚合酶(TaKaRa)。nrDNA ITS扩增程序为:95℃预变性3 min;35个循环:95℃变性0.5 min,51℃退火0.5 min,72℃延伸0.5 min;最后72℃延伸10 min。cpDNA rpl20-rps12扩增程序为:95℃预变性3 min;35个循环:95℃变性0.5 min,52℃退火0.5 min,72℃延伸0.5 min;最后72℃延伸10 min。
将PCR扩增产物经电泳检测纯化后由上海美吉生物医药科技有限公司测序。
1.3 数据处理
序列对位排列用ClustalX软件自动完成后[19],并手工适当校正。用DnaSP软件统计变异位点[20],单倍型(haplotype)类型和变异位点百分率,以及核苷酸多样性,并将nrDNAITS、和cpDNA rpl20-rps12序列进行比较。Tajima’s D和Fu’s Fs两种无限突变位点模型的中性检验方法及歧点分布(Mismatch distribution)分析也在DnaSP程序中完成,来检验羌活居群间是否经历了近期居群范围扩张。用MEGA4.1软件进行核苷酸组成分析,采用Kitmura-2-Parameter distance双参数模型(Kimura,1980)计算遗传距离,并用邻接(Neighbour-Joining,NJ)法构建系统树,通过1 000次重复获得的自展检验(bootstrap)数值标记在分支上[21]。
2 结果与分析
2.1nrDNAIT和cpDNArpl20-rps12目的片段的扩增
PCR扩增体系对不同居群羌活nrDNAITS和cpDNA rpl20-rps12片段进行扩增,得到单一片段条带,电泳显示条带非常清晰,没有拖带和非特异条带(图1)。
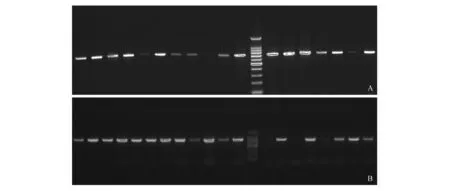
图1 nrDNAITS区域(A)和cpDNA rpl20-rps12区域(B)PCR扩增电泳结果Fig.1 The results of PCR amplification on ITS(A) and cpDNA rpl20-rps12(B) region
2.2羌活各居群间nrDNAITS和cpDNArpl20-rps12序列及分析
对羌活116个个体nrDNA ITS区域进行测序,并用ClustalX进行对位排列。经对位排列,获得我国主要羌活产地12个居群的ITS序列,碱基长度均为635 bp。合并相同的序列共得到15个单倍型(haplotype,H),其中H1是分布最广的单倍型(分布于Q1,Q2,G1,G2,G3,G4,S2,S4居群中),其次是H3(分布于Q3,Q4,S1,S2,S3,S4居群中),H13和H14分别为Q9,S3,S5居群和S5,S8居群所共享,H8为Q9,S3居群所共享,其它特有的单倍型(H2,H4,H5,H7,H8,H9,H10,H11,H12,H15)分布到各自相应的居群中。羌活的cpDNA rpl20-rps12序列,碱基长度767 bp。合并相同的序列共得到12个单倍型,其中单倍型H1是分布最广的单倍型,它分布于全部12个居群中,其次是单倍型H6和H9,分别被5个和4个居群所共享,其它特有的单倍型(H2,H3,H4,H5,H7,H8,H10,H11,H12)分布到各自相应的居群中。
羌活nrDNA ITS序列的(G+C)含量为57.83%,变异位点百分率为2.68%。在635 bp的碱基序列中,有17处变异位点,其中1处为插入/缺失(位于26 bp位点处,表2),16处为碱基置换。羌活nrDNA ITS序列(G+C)含量(57.83%)明显高于rpl20-rps12序列的(G+C)含量(33.06%)。cpDNA rpl20-rps12序列变异位点百分率为4.56%,而nrDNA ITS序列变异位点百分率是2.68%,cpDNA rpl20-rps12序列变异位点百分率远大于nrDNA ITS序列的变异位点百分率(表3)。
cpDNA rpl20-rps12的核苷酸多样性也远大于nrDNA ITS序列的核苷酸多样性。
表2羌活ITS片段15种单倍型进行序列比对的变异位点
Table2Variablesitesofthealignedsequencesamong15haplotypesoftheITSfragmentofN.incisum

单倍型HaplotypeVariablesites126112555555566663660900225669112289723467873889H1GGGGGTAAAGGTACGCGH2····A············H3······G··A·······H4······G··········H5······G···T·····TH6····A·G··········H7····A·GG·········H8··AA·············H9A·A··············H10······GG·········H11······G·T··CTTTTTH12·—A··············H13··A··············H14··A··A···········H15······G··········
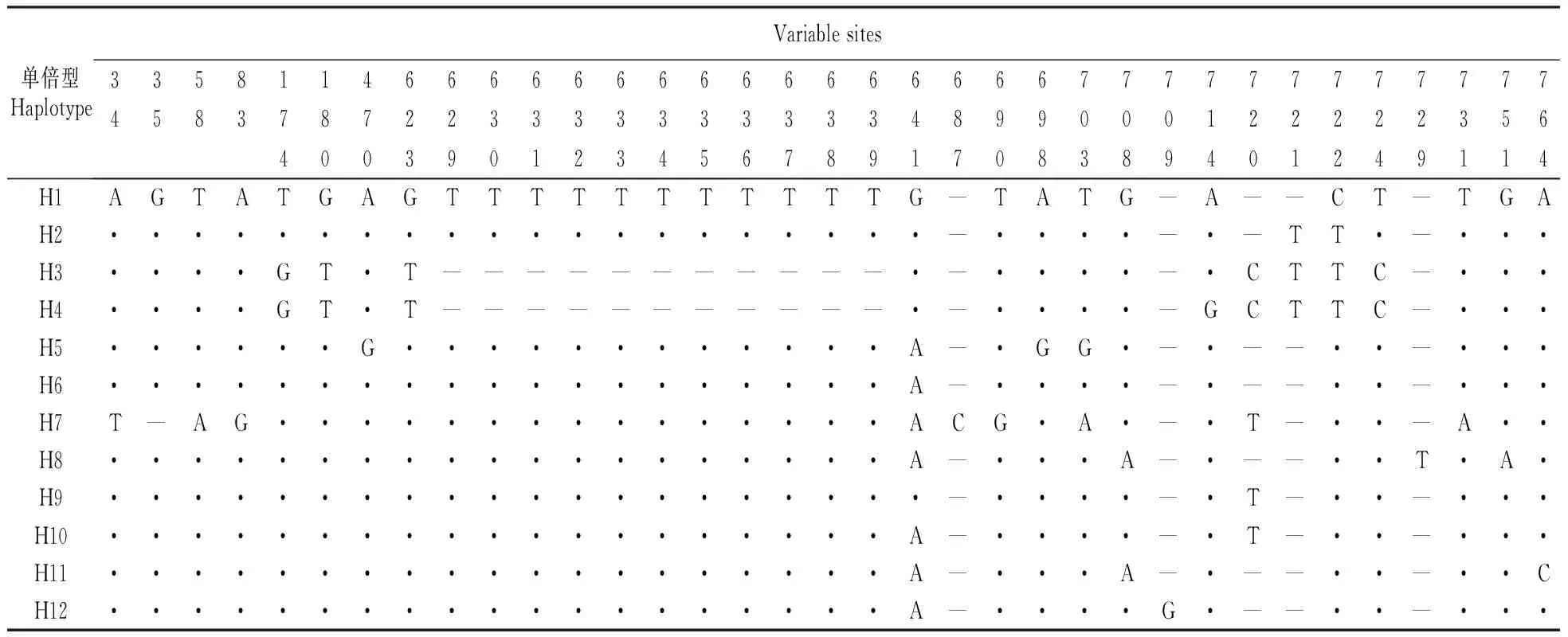
表3 羌活cpDNA rpl20-rps12片段12种单倍型进行序列比对的变异位点
运用MEGA软件对羌活nrDNAITS序列和cpDNA rpl20-rps12所得单倍型进行系统发育树重建。由图2可知,由nrDNAITS序列构建的NJ树分为四支,H2、H3、H4、H5、H15聚为一支,H1、H6、H7、H8、H11、H12、H13、H14、H15聚为一支,H10为一支,H9为另一支。由图3可知,cpDNA rpl20-rps12序列构建的NJ树分为两支,H1、H2、H3、H4、H5、H9聚为一支,H6、H7、H8、H10、H11、H12聚为一支。
采用nrDNA ITS和cpDNA rpl20-rps12序列对羌活的野生种群进行种群历史变动分析,釆用单倍型错配分布、无限突变位点模型的中性检验值Tajima’s D和Fu’s Fs等方法同时调查羌活的种群历史变动错配分布。分析结果显示,羌活种群单倍型和碱基差异呈明显的单峰分布(图4),较为保守的Tajima’s D(nrDNA ITS:-1.447 04,P<0.05;cpDNA rpl20-rps12:-2.102 63,P<0.05)显著偏离中性平衡,Fu and Li’s D*(nrDNA ITS:-4.260 68,P<0.02;cpDNA rpl20-rps12:-3.518 20,P<0.02)和 Fu and Li’s F*(nrDNA ITS:-3.84182,P<0.02;cpDNA rpl20-rps12:-3.567 84,P<0.02)均为显著的负值,支持羌活种群经历了种群选择或扩张事件。

图2 基于ITS序列的15种单倍型的NJ树Fig.2 NJ tree based on the 15 haplotypes of ITS sequences
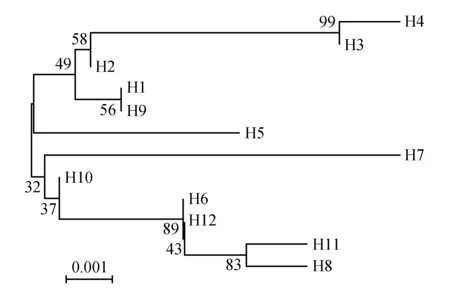
图3 基于cpDNA rpl20-rps12序列的12种单倍型的NJ树Fig.3 NJ tree based on the 12 haplotypes of cpDNA rpl20-rps12 sequences
Table4ComparisonsbetweennrDNAITSandcpDNArpl20-rps12sequencesforN.incisum
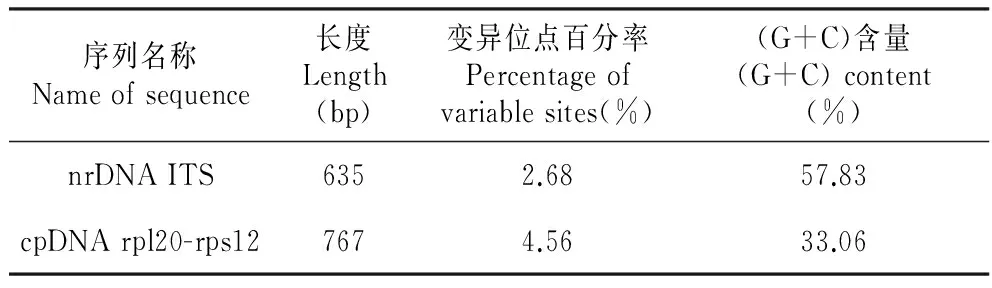
序列名称Nameofsequence长度Length(bp)变异位点百分率Percentageofvariablesites(%)(G+C)含量(G+C)content(%)nrDNAITS6352.6857.83cpDNArpl20-rps127674.5633.06
3 讨论
一般而言,在大多数的被子植物中由于叶绿体DNA是母性遗传、非重组的并且由于进化速度比较慢[22],因此常被用来研究群体遗传学和系统地理学中的遗传瓶颈和奠基者效应,并且用于测量遗传漂变[16]。而核DNA(nDNA)的进化较快,可用于解决属下不同种和种内的系统发育和分类问题,并且可解决由于叶绿体变异的水平太低而不能阐明居群的关系的问题[23]。因此,在植物谱系地理学的研究中采用叶绿体和核基因组联合分析来彻底的阐明调查物种的遗传结构。
本研究中,应用PCR产物直接测序法分析了羌活居群间nrDNA(核糖体DNA)ITS序列和cpDNA(叶绿体DNA) rpl20-rps12的碱基差异,初步确定了两套植物基因组的变异速率,即羌活nrDNA ITS序列较cpDNA rpl20-rps12 序列保守,变异速率较慢,这与上面的研究结果不一致,但与段义忠等[24]对窄叶鲜卑花的研究结果一致。
nrDNA ITS序列共发现15个单倍型,其中H1是分布最广的单倍型,分布于12个居群中的8个居群中,且青藏高原东南部边缘的居群遗传多样性较高。这与叶绿体rpl20-rps12序列单倍型所得出的结果较相近。nrDNA ITS和cpDNA rpl20-rps12序列所得到的两种中性检验值均为负值(nrDNA ITS:-1.447 04,P<0.05;cpDNA rpl20-rps12:-2.102 63,P<0.05),羌活nrDNA ITS和cpDNA rpl20-rps12基因区间序列数据的歧点分布(mismatch distribution)分析结果表明,观测值与期望值之间比较,得到了一个明显的单峰分布曲线图,表明羌活居群可能经历过居群近期范围扩张。虽然nrDNA ITS序列变异位点较少,但其反应的居群间的进化关系与叶绿体基因组一致,nrDNA ITS序列可以推测出羌活居群扩张的大致时间,因此nrDNAITS序列适合羌活以居群进化历史为主要目的的谱系地理学研究。
1.溥发鼎,王萍莉,郑中华,等.重订羌活属的分类[J].植物分类学报,2000,38(5):430-436.
2.王幼平,溥发鼎,王萍莉,等.中国特有属——羌活属的系统分类研究[J].云南植物研究,1996,18(4):424.
3.周毅,蒋舜媛,马小军,等.羌活资源危机和保护[J].中草药,2003,34(10):12.
4.中华人民共和国国家药典委员会.中华人民共和国药典一部[M].北京:化学工业出版社,2010:170-171.
5.徐惠波,孙晓波.羌活挥发油的药理作用研究[J].中草药,1991,22(1):28-30.
6.汪松,解焱.中国物种红色名录:第1卷[M].北京:高等教育出版社,2004:8507.
7.刘卫根,王亮生,徐文华,等.不同商品等级羌活中有机酸和香豆素类化合物的测定[J].中成药,2012,34(011):2181-2186.
8.刘卫根,王亮生,周国英,等.羌活不同部位有机酸和香豆素类化合物含量的比较研究[J].药物分析杂志,2012,32(011):1950-1956.
9.张明发,沈雅琴.羌活药理学研究[J].药学服务,2008(5):28-30.
10.马玉林,李建民,马莉,等.药用植物羌活的研究进展[J].安徽农业科学,2010,38(24):13092-13093.
11.蒋舜媛,孙辉,黄雪菊,等.羌活和宽叶羌活的环境土壤学研究[J].中草药,2005,36(6):917-921.
12.董生健.野生羌活驯化栽培技术[J].中药材,2010(1):38-39.
13.方子森,陈小莉,张恩和.野生羌活的生态环境与驯化栽培[J].中草药,2004,35(9):1071-1073.
14.赵一之,成文连,尹俊,等.用rDNA的ITS序列探讨绵刺属的系统位置[J].植物研究,2003,23(4):402-406.
15.Baldwin B G,Sanderson M J,Porter J M,et al.The Its Region of Nuclear Ribosomal DNA-a Valuable Source of Evidence on Angiosperm Phylogeny[J].Annals of the Missouri Botanical Garden,1995,82(2):247-277.
16.Petit R J,Pineau E,Demesure B,et al.Chloroplast DNA footprints of postglacial recolonization by oaks[J].Proceedings of the National Academy of Sciences of the United States of America,2003,94(18):9996-10001.
17.Fujii N,Ueda K.,Watano Y,et al.Intraspecific sequence variation of chloroplast DNA inPedicularischamissonisSteven(Scrophulariaceae) and geographic structuring of the Japanese “Alpine” plants[J].Journal of Plant Research,1997,110(1098):195-207.
18.Gao Q B,Zhang D J,Duan Y Z,et al.Intraspecific divergences ofRhodiolaalsia(Crassulaceae) based on plastid DNA and internal transcribed spacer fragments[J].Botanical Journal of the Linnean Society,2009,168(2):204-215.
19.Thompson J D,Gibson T J,Plewniak F,et al.The Clustal-X windows interface:flexible strategies for multiple sequence alignment aided by quality analyses tools[J].Nucleic Acids Research,1997,25 :4876-4882.
20.Rozas J,Sanchez-DelBarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
21.Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA) software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
22.Reboud X,Zeyl C.Organelle Inheritance in Plants[J].Heredity,1994,72:132-140.
23.Wolfe K,Li W,Sharp P.Rates of nucleotide substitution vary greatly among plant mitochondrial,chloroplast,and nuclear DNAs[J].Proceedings of the National Academy of Sciences,1987,84:9054-9058.
24.段义忠,张得钧,高庆波,等.窄叶鲜卑花(Sibiraeaangustata)nrDNAITS和cpDNAtrnL-F序列分子进化特点的分析[J].植物研究,2010,30(2):146-151.
CharacteristicofMolecularEvolutionofNotopterygiumincisumBasedonnrDNAITSandcpDNArpl20-rps12SequenceAnalysis
YANG Lu-Cun1,2LIU He-Chun1,3ZHOU Xue-Li4XU Wen-Hua1,2ZHOU Guo-Ying1,2*
(1.Northwest Institute of Plateau Biology,Chinese Academy of Sciences,Xining 810008;2.Key Laboratory of Tibetan Medicine Research,Chinese Academy of Sciences,Xining 810008;3.University of Chinese Academy of Sciences,Beijing 100049;4.Tiebujia Grassland Improvement Experiment Station,Xining 810008)
We analyzed the differences between the nrDNAITS and cpDNA rpl20-rps12 sequences inNotopterygiumincisumby PCR direct sequencing. Total DNA was extracted from silica-dried leaves ofN.incisumusing modified CTAB method. With the extracted DNA as template, nrDNA ITS and cpDNA rpl20-rps12 regions were amplified, then purified and sequenced. The length of nrDNA ITS sequence ofN.incisumwas 635 bp, of which 17 were variable sites with a percentage of 2.68%, the(G+C) content was 57.83%. The length of cpDNA rpl20-rps12 sequence ofN.incisumwas 767 bp, of which 35 was variable site with a percentage of 4.56%, the(G+C) content was 33.06%. The nrDNAITS region ofN.incisumwas more conserved and evolved more slowly than the cpDNA rpl20-rps12sequence. The present distribution range ofN.incisumexperienced range expansion by the haplotype analysis of this species, which consisted with the conclusion resulting from cpDNA genome. Therefore, the nrDNA ITS sequence of rpl20-rps12 was fit to the phylogeographic study of this species.
Notopterygiumincisum;ITSsequence;rpl20-rps12 sequence
青海省青年基金项目(珍稀濒危药用植物羌活的濒危机制研究[2013-Z-942Q])
杨路存(1981—),女,博士,助理研究员,主要从事药用植物分子生态学研究工作。
2015-06-30
Q949.763.3
A
10.7525/j.issn.1673-5102.2016.02.019

