基于核基因序列JRD5680的核桃群体遗传多样性和遗传结构研究
张 甜 王玛丽 赵 鹏
(西北大学生命科学学院,西安 710069)
* 通信作者:E-mail:pengzhao@nwu.edu.cn
基于核基因序列JRD5680的核桃群体遗传多样性和遗传结构研究
张 甜 王玛丽 赵 鹏*
(西北大学生命科学学院,西安 710069)
以40个核桃(Juglansregia)地理群体及5个外类群植物为材料,利用苯丙氨酸解氨酶基因片段JRD5680序列对265个个体研究了我国不同地区核桃的群体遗传多样性、群体遗传结构以及单倍型地理分布。结果表明:核基因JRD5680序列长度为809 bp,其中G+C碱基含量为46.2%,74个简约性信息位点。共30个单倍型,单倍型多样性变异小,表现出较低的遗传多样性(Hd=0.370,π=0.005 3)。核桃群体间有明显的谱系地理学结构(NST>GST),呈现地域性分布格局的特点。空间遗传结构分析表明,地理隔离距离与遗传距离之间也显示两者之间相关性显著(r=0.263 2;P=0.032 4*),不同地理分布核桃群体间存在着明显地理隔离效应。综合失配分析及中性检验的结果,可推断出近期历史上不同地区核桃没有发生群体扩张事件。遗传结构显示,核桃群体可分为两组(西南地区与其他地区)。AMOVA分析结果表明,核桃群体遗传变异主要存于群体之间(56.54%),不同种之间的遗传分化水平较高(FST=0.885)。单倍型系统发育树中核桃与外类群物种均能形成单系分支,说明JRD5680序列在胡桃科中的具有较高的分子鉴别率。
核桃;苯丙氨酸解氨酶基因;遗传多样性;遗传结构
植物群体遗传变异是进化的基础,这些变化的速率与现有的遗传多样性的数量成比例[1~2]。遗传多样性水平可以反映植物自身的生物学特征及适应自然环境变化和人为干扰的能力[3]。遗传结构是物种在空间上不同群体间和同一群体内不同个体间的遗传变异[4],群体遗传结构差异也是衡量植物遗传多样性的一种重要体现[5]。了解群体遗传多样性与遗传结构,可以揭示物种或群体的进化历史,为其进化潜力的研究提供重要资料,也为物种保护提供基本的遗传信息。
核桃(JuglansregiaL.)也称胡桃,具有很高的经济、食用和药用价值[6]。核桃在我国的分布范围十分广泛,由于地理因素、气候条件等不尽相同,导致不同地理群体的核桃在形态特征、生理及遗传特性等方面存在一定的差异,具有丰富的遗传多样性。同时,由于核桃经长期的自然和人为选择,导致核桃的遗传背景非常复杂[7]。近二十年来,国内外学者采用不同的分子标记技术,对核桃的不同种质资源进行了研究。主要包括:限制性片段长度多态性(RFLP)[8]、随机扩增多态DNA(RAPD)[9]、简单序列重复长度多态性(SSR)[10]和扩增片段长度多态性(AFLP)[11]等。但鉴于这些遗传标记本身存在的局限性,群体样本数较少的限制。对中国整个地理分布范围的核桃遗传多样性和遗传结构仍需进一步认识和研究。植物细胞中存在3套基因组,其中叶绿体基因(cpDNA)和线粒体基因(mtDNA)都是单亲遗传[12],反映的遗传信息有限,不适宜作为核桃种内遗传多样性研究。而核基因DNA序列因为双亲遗传标记,包含着更为丰富的遗传信息,在植物遗传多样性研究方面已有报道[13~14],但在胡桃属植物的种质资源研究中还未见报道。
本研究拟通过核桃转录组数据中筛选出苯丙氨酸解氨酶基因片段JRD5680序列对核桃40个地理群体遗传多样性和遗传结构进行了分析,以2个核桃楸(J.mandshurica)、1个野核桃(J.cathayensis)、1个山核桃(Caryacathayensis)及1个黄杞(Engelhardtiaroxburghiana)地理群体植物为外类群,探讨在核基因的水平评估全国不同地理群体核桃的遗传多样性、群体遗传结构及历史动态,以期为核桃种质资源的开发和利用提供有价值的参考。
1 材料与方法
1.1 材料采集
实验所用材料采自中国40个地理分布核桃群体、2个核桃楸群体、1个野核桃群体、1个山核桃群体及1个黄杞群体。各群体的采集地理位置、群体代码、海拔、经纬度等详细信息(表1)。采集过程中,每个自然群体选择9~20个植株进行采样,采样间隔距离为50~100 m。在每棵核桃树上采集健康幼嫩的树叶数片,将其迅速放入盛有变色硅胶的封口塑料袋内,带回实验室保存备用。
1.2 实验方法
1.2.1 DNA的提取与检测
基因组DNA的提取与分离方法是参照Doyle and Doyle提出的CTAB方法[14],并在此基础上进行了改良,方法详见Zhao and Woeste和赵鹏等[9,15]。利用分光光度计和琼脂糖凝胶电泳检测基因组DNA的浓度和纯度。
1.2.2核基因JRD5680序列的引物设计、PCR扩增、检测与测序
从核桃的叶片、芽、雌花和雄花不同组织提取RNA后获得核桃的转录组数据,对转录组中拼接得到的基因进行功能注释,从中我们筛选了一些功能基因。根据对一个注释结果为抗逆性基因JRD5680序列(2 369 bp)在美国国家生物技术信息中心(National center biolotechnology information,NCBI,http://www.ncbi.nlm.nih.gov/)数据库上在线BLAST后,发现该基因序列编码苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL),分别与白桦(Betulaplatyphylla)的1 308 bp(GenBank No.AKN79308.1)、蓖麻(Ricinuscommunis)的1 271 bp(GenBank No.AGY49231.1)、木薯(Manihotesculenta)的1 269 bp(GenBank No.AAK60275.1)等序列相似(鉴定度分别为87%,85%和85%),说明核桃JRD5680可能与其非生物逆境抗性密切相关[16~17]。用Primer3分别对全长为2 369 bp的JRD5680序列设计了两对引物,其中一对扩增效果好,数据分析采用了扩增效果好的序列片段。JRD5680序列的PCR扩增引物序列是通过上海生物工程公司(Sangon Biotech Co.,Ltd.,Shanghai)合成(F:TGGACATGGCTAGTGACTGG;R:TCCCTCTTGCCTACATTGC)2X Taq PCR Mix(0.1 U Taq polymerase,500 μmol·L-1dNTPs,20 mmol·L-1Tris-HCl,pH 8.3,3 mmol·L-1MgCl2,100 mmol·L-1KCl)购自润德生物技术有限公司(RUNDE,西安),牛血清蛋白(Bovine Serum Albumin,BSA)购自ProMEGA公司(Madison,Wisconsin,USA)。根据Zhao and Woeste[10]的PCR反应基础做了改进,即在25 μL的PCR扩增反应体系中,含50 ng·μL-1模版DNA 2 μL,2X Taq PCR Mix 12.5 μL,引物正反向都为1.0 μL(10 μmol·L-1),牛血清蛋白(BSA,0.5 mg·mL-1)2.5 μL,H2O 6 μL。PCR扩增反应程序:95℃预变性3 min;93℃变性15 s,60℃退火1 min,72℃延伸1.5 min,35个循环;72℃延伸10 min后置10℃保存。扩增产物通过1%的琼脂糖凝胶电泳进行检测后,将扩增产物送到上海生工生物工程有限公司进行测序。
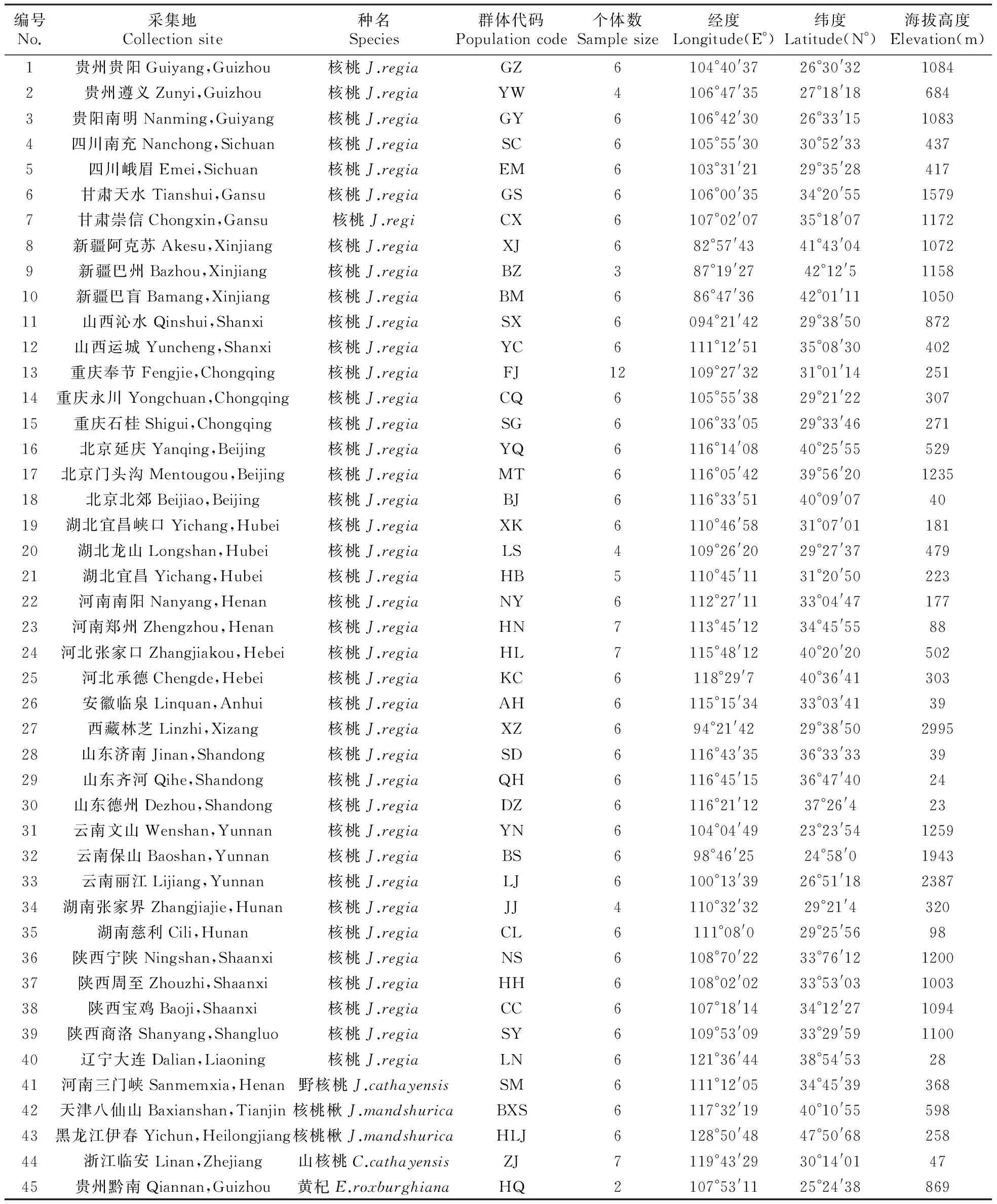
表1 全国各地45个群体植物材料地理与样品信息
1.3 数据分析
对核桃265个个体的PCR扩增产物测序后,用软件BioEditv.7.0.9对测序结果进行排列、比对和校正[18]。使用Dnasp 5.0软件[19]分析核苷酸多态性(π)、单倍型个数(H)、单倍型多样性(Hd)等遗传多样性指标,将全部数据分为两组进行分析:第一组,核桃群体材料,第二组全部样本植物材料(核桃群体,以及外类群:包括胡桃属野核桃40个群体,核桃楸2个群体,山核桃属山核桃1个群体,黄杞属黄杞1个群体);对不同地理群体之间的基因流(Nm)进行估算,并对群体进行Tajima’s、Fu的中性检验,以确定目标DNA序列在进化过程中是否遵循中性学说[20~21];并利用该软件对所有群体的动态扩张进行失配分布分析(Mismatch Distribution Analysis)[22]。利用软件NETWORK 4.2.0.1基于最大简约性原则的中间连接网法分析(Median-joining networks),构建单倍型间的网络关系图[23]。利用软件ARELEQUIN 3.11中的AMOVA(Analysis of Molecular Variance)分析方法[24]分别检测群体间和群体内的遗传变异。运用软件STRUCTURE2.0[25]进行贝叶斯聚类分析,其中采用马尔可夫链蒙特卡罗(MCMC)迭代(1 000 000),重复运行10次,K值设置为2-7。利用在线软件STRUCTURE HAVESTER计算最优K值,并利用软件DISTRUCT进行遗传组分聚类图绘制。运用运用软件MEGA6.0溯祖理论推测单倍型间的亲缘关系,构建JRD5680序列单倍型的邻接(Neighor-joining,NJ)系统发育树[26]。应用IBD(Isolation by Distance)软件(http://ibdws.sdsu.edu/~ibdws/)中IBDWS方法[27]对群体遗传距离与地理距离间的相关性及遗传距离与海拔之间的相关性进行Mantel检验分析。
2 结果与分析
2.1 核酸多样性分析
对所有样品测序的265条序列用BioEdit5.0和MEGA6.0软件进行排列、对比、校正,JRD5680序列片段长度为809 bp。全部序列GC核苷酸含量占总核苷酸的46.2%。根据Dnasp 5.0的分析结果表明,共有75个变异位点,包括1个单碱基变异位点,74个简约信息位点,共30个单倍型,不存在插入缺失。全部样本个体的单倍型多样性(Hd)较高为0.482,不同地理分布的40个核桃群体的单倍型多样性(Hd)较低为0.370(表2)。两组的核苷酸多态性结果与单倍型多样性的结果一致(核桃π=0.005 27、全部个体π=0.008 99)(表2)。用Dnasp5.0对群体植物材料的基因流分析发现,不同地理核桃群体间的基因流较大(Nm=0.34),全部个体的基因流小(Nm=0.10)(表2)。遗传分化系数FST,也表现出明显的差异,其全部个体的FST值最大为0.885,其核桃群体材料遗传分化系数最小,其FST值为0.435。

表2 核基因序列JRD5680的遗传多样性分析
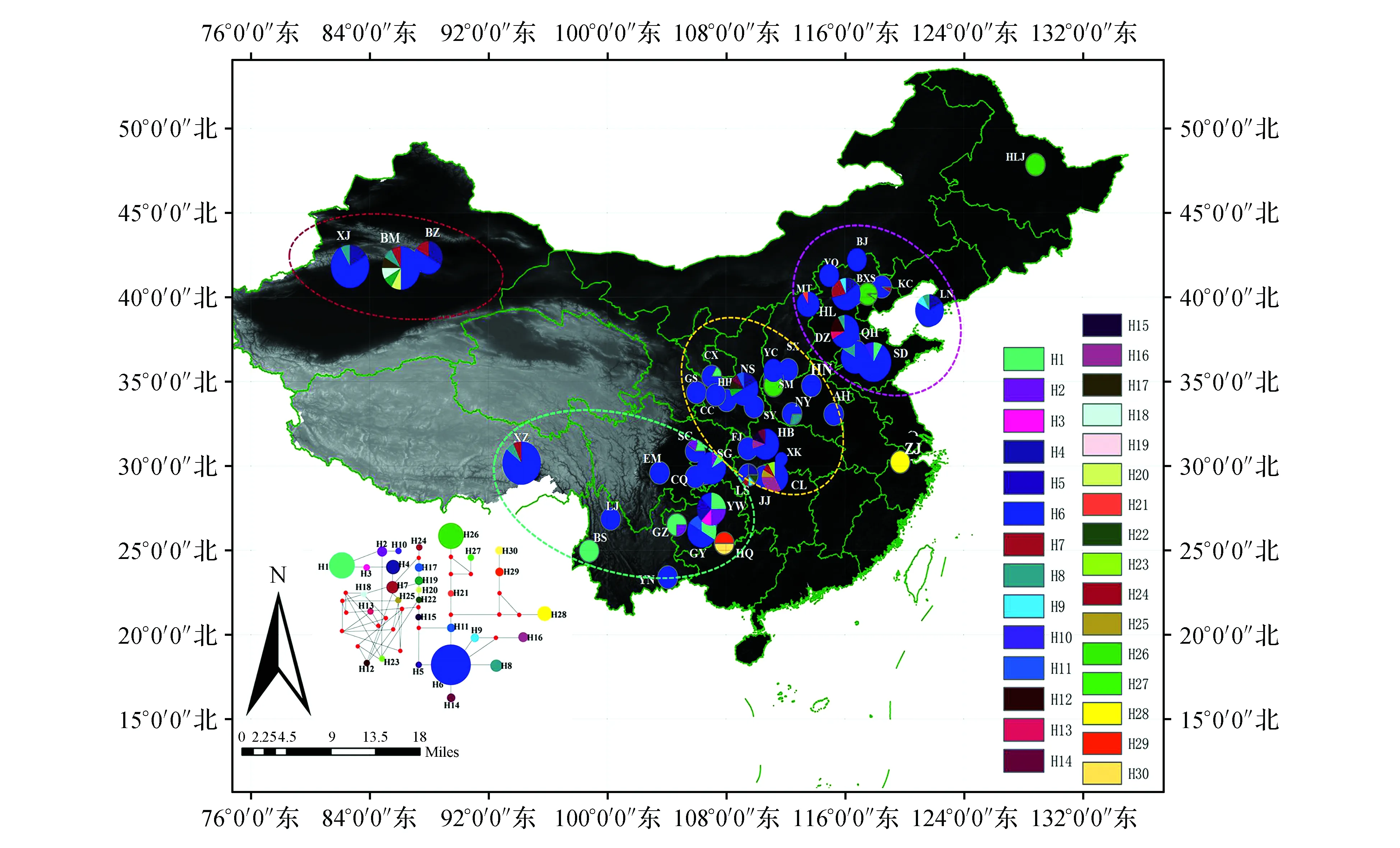
图1 核桃群体JRD5680序列单倍型地理分布及单倍型Network网络拓扑结构图 饼图代表各单倍型在该群体中的分布比例,标注框内不同颜色代表不同的JRD5680序列单倍型,群体代码同表1,单倍型网络拓扑结构图位于左下角。Fig.1 Geographical distribution of JRD5680 haplotypes and network of haplotype for 45 populations of J.regia Different patterns were assigned for each haplotype to the legend at the right side of the figure. Circumference size indicates that the number of each population. Population symbols are identified in Table1. The network of haplotype based on JRD5680 data were located in the lower left corner.
这些结果都表明不同种之间遗传差异比较大。在群体水平上,45个居群中单倍型多态性最高的两个群体分别为贵州遵义(YW)(Hd=0.929,π=0.008 88)与新疆省巴盲(BM)(Hd=0.773,π=0.009 79)。
2.2 基因谱系
2.2.1 单倍型及地理分布与网络拓扑结构
从单倍型地理分布图可以看出,所有样品具有的30个单倍型中有25个单倍型存在核桃群体中(Genbank No.KT820730~KT820759),其余5个单倍型存在于外类群中(图1)。核桃群体中频率最高的单倍型为H6,除了贵州贵阳(GZ)与云南保山(BS)群体拥有独特单倍型H1外,38个(95%)群体具有单倍型H6。单倍型H1主要分布于西南地区的5个群体中:四川南充(SC)、重庆石桂(SG)、贵州遵义(YW)、贵州贵阳(GZ)、贵阳南明(GY)、云南保山(BS)。此外,拥有单倍型最多的群体分别为新疆巴盲(BM,7个单倍型:H6、H7、H8、H17、H18、H19、H20)、陕西宁陕(NS,6个单倍型)、贵州遵义(YW,6个单倍型:H1-H6)、湖北龙山(LS,6个单倍型:)与湖南慈利(CL,6个单倍型)。比较遗传分化系数GST与NST发现,NST值都明显高于GST值,表明各群体间有明显的谱系地理学结构(表2)。
单倍型网络拓扑结构图表明,核桃共有25个单倍型,其中单倍型H6为古老单倍型,其他单倍型都是有单倍型H6经过一步或多步突变形成的,具有特有单倍型(图1)。外类群中野核桃与核桃楸具有相同的单倍型H26;山核桃具有特有单倍型H28;黄杞具有独特单倍型H29和单倍型H30。外类群的5种单倍型都是与核桃主流单倍型经过2-5步突变而形成的,不同种之间不存在共有单倍型,且单倍型的频率都比较低。核桃群体中单倍型H1位于网络图最外端,与其他群体分布的单倍型形成复杂的拓扑结构关系(图1)。
2.2.2 单倍型系统发育树构建
以40个核桃(J.regia)、2个核桃楸、1个野核桃、1个山核桃及1个黄杞地理群体植物为材料,运用软件Mega6.0构建了核基因JRD5680序列最大邻接树(NJ)。结果表明:山核桃(单倍型H28)与黄杞(H29、H30)作为外类群分布位于系统发育树的基部,各自为单系分支,自展支持率为99%。所有胡桃属植物自成一支,自展支持率为53%。NJ树的结果与单倍型网络结构一致。野核桃(H26)与核桃楸的独特单倍型H27分成两小支。
2.3 群体遗传结构
2.3.1 分子方差分析(AMOVA)
全国不同地理群体分子方差分析(AMOVA)见表3,群体间的遗传变异为43.46%,群体内的遗传变异为56.54%,即全国不同地区核桃群体的遗传变异主要存在于群体内。将外类群加入分子方差分析(AMOVA)分析时,结果表明,组间变异达到79.57%,群体间变异为8.90%,群体内的变异为11.53%。说明利用JRD5680序列分组后的遗传变异主要存在于不同物种之间。
2.3.2 遗传结构分析
运用Structure软件对45个群体的遗传结构进行分析,K的取值范围为2~7,运用Structure Harvester软件确定最佳分组时发现,K的最佳分组为4,但K=3与K=4的结果相差不大。当K=4时,发现外类群的遗传结构明显与核桃不同,山核桃(ZJ)与黄杞(HQ)分为一组,野核桃(SM)与核桃楸(BXS与HLJ)的遗传组分相似分为一组,核桃群体分为两组,这与单倍型的分布一致(图1)。遗传结构分析表明不同物种之间的遗传差异比较大。40个核桃群体间遗传差异不大,但贵州贵阳(GZ)、贵州遵义(YW)与云南保山(BS)三个群体明显能与其他核桃群体分开,出现了较为明显的遗传分化(图2)。
表3基于核基因序列JRD5680分子方差分析
Table3Analysisofmolecularvariance(AMOVA)forthepopulationsbasedonnuclearDNAJRD5680sequences
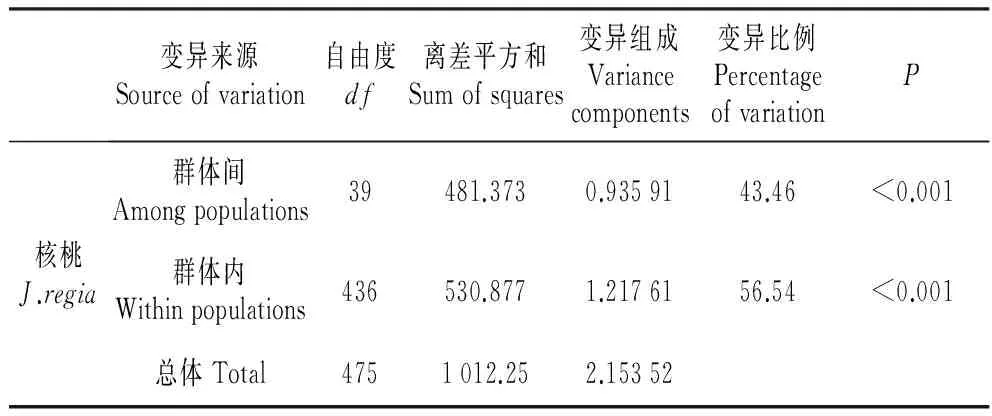
变异来源Sourceofvariation自由度df离差平方和Sumofsquares变异组成Variancecomponents变异比例PercentageofvariationP核桃J.regia群体间Amongpopulations39481.3730.9359143.46<0.001群体内Withinpopulations436530.8771.2176156.54<0.001总体Total4751012.252.15352
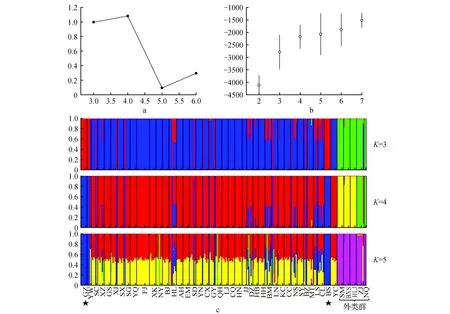
图2 基于抗旱基因核基因JRD5680序列的贝叶斯模型的群体遗传结构聚类分析a.超过10个重复估算出的数据的概率(△K)对K聚类分组的数据;(b)△K值的平均对数似然概率推断不同群体遗传结构(K)的范围从2~7;c.估计的遗传聚类(K=3,K=4,K=5),得到的遗传结构为256个个体,不同颜色代表不同遗传组分,数字表示不同颜色遗传成分所占比例,垂直线表示不同群体的分界标识线。Fig.2 Results of the Bayesian model-based clustering structure analysis of 256 individuals of common walnut using drought relative nuclear marker JRD5680a. The probability of the data(△K) against the number of K clusters,calculated over 10 replicates; b. △K values from the mean log-likelihood probabilities from structure runs where inferred clusters(K) ranged from 2-7; c. Estimated genetic clustering(K=2,K=3,and K=4) obtained with the structure program for 256 individuals,colors represent the cluster are separating according to the population,and the black vertical line in the bar chart is the population identifier.
2.3.3 中性检验与失配分布
核桃群体与外类群两组情况分别进行中性检验[23~24]结果表明:所有群体的Tajima’s D为不显著负值(Tajima’sD=-1.101 9,P>0.10),Fu and Li’s D值与Fu and Li’s F均为正值(Fu and Li’sD=2.3420,P>0.10;Fu and Li’sF=0.759 0,P>0.10)。核桃40个群体中性检验结果表明:Tajima’s D为不显著正值(Tajima’sD=0.270 8,P>0.10),Fu and Li’s D值为不显著负值(Fu and Li’sD=-0.066 7,P>0.10),Fu and Li’sF为不显著正值(Fu and Li’sF=0.093 2,P>0.10),表明全国不同地区的群体均未经过扩张事件。失配分布结果显示:观测值的曲线呈现为双峰现象,也表明不同地理分布的核桃群体近期历史上没有经历扩张事件。
2.3.4 IBD分析
对中国核桃群体进行遗传距离与地理距离(IBD)相关性分析,结果显示两者之间具有显著地相关性(r=0.263 2;P=0.032 4*),而对群体进行遗传距离与海拔距离(IBD)相关性分析,结果显示不显著相关性(r=0.012 4;P=0.668 9),上述结果说明,中国不同地理核桃群体间的遗传变异可能是由地理距离造成的。
3 讨论
3.1 群体遗传多样性
遗传多样性主要包括同一群体不同个体之间、种内不同群体之间遗传变异的总和[1,28]。遗传多样性处于生物多样性的核心地位,遗传多样性的高低,与植物与环境变化的适应力相关。一些相关的参数如:单倍型多样性、核苷酸多样性都用来反应遗传多样性,这些参数值越大,说明遗传多样性越高,基因的丰富度也就越高[1,29]。该研究采用一对可能具有抗逆功能的核基因JRD5680序列片段对核桃群体的单倍型多样性为0.482,比叶绿体、线粒体、核糖体内转录间隔区(ITS)的单倍型多样性高。如秦岭地区核桃的ITS序列分的单倍型多样性为0.274,核桃楸叶绿体单倍型多样性也很低,野核桃的叶绿体单倍型多样性为0.796,美国白核桃的叶绿体单倍型多态性为0~0.317,表明核基因更适于做群体遗传多样性分析。核基因存在明显的基因复制现象,非模式植物获得核基因比较困难。但随着高通量测序技术的发展,转录组数据为非模式生物核基因的获得提供可能。本文中JRD5680是从核桃转录组测序数据中得到的,PCR扩增产物通过琼脂糖凝胶电泳检测结果来看,所获得条带与目的条带大小一致,同时,Sanger测序得到的JRD5680序列在NCBI数据库中进行比对,发现与白桦(Betulaplatyphylla)等物种的基因编码序列(Coding sequence,cds)区域相似度很高,不存在四碱基以上的较大的间隔,说明我们所得到的序列不含内含子,为苯丙氨酸解氨酶编码区序列。同时,相对内含子来说,外显子更难发生突变,而核桃群体的JRD5680序列发生突变与其受到自然和人工选择压力有关。
从单倍型地理分布图中可以明显看出,核桃群体遗传多样性中心主要分布于秦岭巴山地区(NS、HB与CL)、西南地区(YW)、太行山北部地区(DZ、HL)及新疆地区(BM),且不同地区之间存在明显地单倍型差异,仅共享主流单倍型H6(图1)。这说明不同地区的地形、气候因子复杂多变,尤其温度与水份因子变化尤其剧烈,这些因素都有可能造成核桃群体适应不同的环境演化出许多特有单倍型。同时,核桃栽培历史已有6 800多年[1,30],在长期人工驯化过程中,一些核桃品种产生了优良的抗逆基因[31],如抗旱[32]与耐盐[32~33]等,由于商业活动及人为对核桃的传播,导致栽培核桃群体与野生核桃群体之间长期的基因交流,这些也是中国主要核桃产区的群体遗传多样性较高的原因。中国核桃的主要栽培中心为新疆、陕西、河北和云南等省和地区[34],本研究中遗传多样性较高的核桃群体(新疆巴盲、陕西宁陕、湖北宜昌、湖南慈利、贵州遵义、河北张家口及山东德州)也主要集中于上述四个地区(图1)。这一结果与奚声坷1987年将我国核桃的栽培实生群划分为新疆、华北山地、秦巴山地、西藏高地四个地理生态型一致[35]。Structure软件分析的遗传结构结果发现贵州贵阳(GZ)、贵州遵义(YW)与云南保山(BS)三个群体与其他群体出现较明显遗传分化,这可能是由于该三个地区拥有自己独特的单倍型H1的比例较高,从而导致遗传结构分为西南地区和其他地区两大组。另外,西南地区核桃群体遗传多样性较高且原始单倍型H6分布频率高,说明该地区也可能为核桃群体的遗传多样性起源中心之一。
3.2 群体遗传结构
遗传结构指遗传多样性大小和在群体中的分布形式,通过物种群体间和群体内的遗传分化来实现[4]。遗传分化可以通过基因流的大小来体现,基因流越大,群体间的遗传分化越小;相反,基因流越小群体间的遗传分化越大。除此之外,温度、海拔、气候、湿度等外部的因素也可能对遗传结构造成影响[4]。由于核基因JRD5680序列的NST值都明显高于GST值,表明各群体间有明显的谱系地理学结构。同时,核桃种群IBD(Isolation by Distance)空间遗传结构分析表明,地理隔离距离与遗传距离之间也显示两者之间相关性显著(r=0.263 2;P=0.032 4*),说明该研究中不同地

图3 基于核基因JRD5680序列的30个单倍型氨基酸序列变异位点Fig.3 The amino acid sequence variation sites based on nuclear gene JRD5680 sequences of 30 haplotypes
理分布核桃群体间存在着明显地理隔离效应。利用软件ArcGIS进行遗传分析结果也显示,核桃群体在JRD5680位点下的生物地理分布格局呈现地域性分布特点(图1)。而遗传距离与海拔梯度之间不存在显著相关性(R=0.012 4;P=0.668 9),这说明核桃群体间不存在海拔梯度的遗传变异。AMOVA分析结果表明不同物种之间的遗传变异比较大(79.57%),核桃群体间与群体内遗传变异大小相当(表3)。Hamrick[36]等研究发现广布种、寿命较长的多年生木本、异交风媒物种的群体间分化程度均小于其他物种。核桃为广布种,多年生木本,风媒传粉,花粉传播的距离较远,导致群体之间分化程度较小,这符合HamricK的结论。核桃群体遗传组分可分为两组,即西南地区与其他地区核桃群体分为开(图2),其中,云南宝山(BS)群体仅有单倍型H1,这说明,核桃可能在西南地区长期适应形成自己特有单倍型H1,这也可能与该地区适应环境有关。
3.3 核DNA基因片段JRD5680核桃进化历史分析
核基因片段JRD5680序列单倍型网络拓扑结构(Network)与邻接树(NJ)均表明了核桃与其他几个外类群植物(核桃楸、野核桃、山核桃、黄杞)并无共享的单倍型,表明不同种之间的遗传差异比较大,该核基因序列可用于胡桃科不同属间系统分类与物种分化方面的研究。同时,胡桃属植物中核桃与另外两个外类群核桃楸和野核桃也可以明显分开,没有共享单倍型的现象,核桃楸和野核桃共享单倍型H28,说明核桃楸与野核桃亲缘关系比较近。这个结果支持核桃(J.regia)属于核桃组(Sect.Juglans),核桃楸、野核桃归属于核桃楸组(Sect.Cardiocaryon)的结论[35~37]。
单倍型H6为主流单倍型广泛分布在核桃中,其他单倍型与单倍型H6之间存在较为复杂的网络进化关系。造成这种现象的原因,一方面可能与核桃的动态历史有关。本研究中,中性检验、失配分布分析结果都表明核桃在演化的过程中没有明显的扩张现象,可能在各地理区域内由于当地适应(local adaption)、自然选择及突变形成多个频率较低的单倍型。另一方面,由于该研究采用核基因属于双亲遗传,位点间存在重组和杂合性,可能导致网状进化的存在(图1)。植物中nrDNA的转录间隔区(ITS)由于进化速率较快,提供了较多的遗传进化信息,研究学者利用该序列进行了群体进化方面的研究[38~39]。JRD5680这个基因植物形成次级代谢中起到重要作用,对植物生长发育、抗病、抗旱、抗寒等方面具有重要意义和价值[16~17],而这些适应性会对物种的遗传多样性造成影响,导致群体的遗传分化。利用NCBI数据对该基因进行氨基酸序列分析,发现核桃及外类群中的不同单倍型序列也引起了氨基酸的变异(图3)。因此,这种功能上的差异可能与物种之间的分化及其环境适应有着密切相关。在物种分化过程中,由于选择或者功能不同,可能导致相应的种间基因流和遗传分化不同。这些结果对中国不同地理群体核桃的遗传多样性及种质资源保护和利用提供一定的理论依据。
1.Beer R,Kaiser F,Schmidt K,et al.Vegetation history of the walnut forests in Kyrgyzstan(Central Asia):natural or anthropogenic origin?[J].Quaternary Sci Rev,2008,27(5):621-632.
2.Hoban S M,Borkowski D S,Brosi S L,et al.Range‐wide distribution of genetic diversity in the North American tree Juglans cinerea:a product of range shifts,not ecological marginality or recent population decline[J].Mol Ecol,2010,19(22):4876-4891.
3.Ishihama F,Ueno S,Tsumura Y,et al.Gene flow and inbreeding depression inferred from fine-scale genetic structure in an endangered heterostylous perennial,Primulasieboldi[J].Molecular Ecology,2005,14(4):983-990.
4.Epperson B K.Spatial distributions of genotypes under isolation by distance[J].Genetics,1995,140(4):1431-1440.
5.Escudero A,Iriondo J M,Torres M E.Spatial analysis of genetic diversity as a tool for plant conservation[J].Biol Conserv,2003,113(3):351-365.
6.Vinson J A,Cai Y.Nuts,especially walnuts,have both antioxidant quantity and efficacy and exhibit significant potential health benefits[J].Food Funct,2012,3(2):134-140.
7.肖志娟,翟梅枝,王振元,等.微卫星DNA在分析核桃遗传多样性上的应用 [J].中南林业科技大学学报,2014,34(2):55-61.
8.Fjellstrom R G,Parfitt D E.Phylogenetic analysis and evolution of the genusJuglans(Juglandaceae) as determined from nuclear genome RFLPs[J].Plant Syst Evol,1995,197(1-4):19-32.
9.Zhao P,Woeste K E.DNA markers identify hybrids between butternut(JuglanscinereaL.) and Japanese walnut(JuglansailantifoliaCarr.)[J].Tree Genet Genomes,2011,7(3):511-533.
10.Dangl G S,Woeste K,Aradhya M K,et al.Characterization of 14 microsatellite markers for genetic analysis and cultivar identification of walnut[J].J Am Soc Hortic Sci,2005,130(3):348-354.
11.王红霞,张志华,赵书岗,等.核桃种质资源遗传多样性研究中的AFLP技术优化及引物筛选[J].华北农学报,2008,23(1):50-54.
12.丁铭,曾丽萍,马红,等.初探低拷贝核基因在低等分类阶元系统发育重建中的适用性—以十字花科为例[J].植物分类与资源学报,2012,34(3):211-221.
13.吴春燕,高立志.六个核基因片段在中国颗粒野生稻中的单核苷酸多态性及其在群体遗传学研究中的应用[J].植物分类与资源学报,2013,35(5):537-546.
14.Doyle J J.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochem bull,1987,19(1):11-15.
15.赵鹏,Keith Woeste,程飞,等.美国黑核桃SSR反应体系优化[J].植物研究,2012,32(2):213-221.
16.Pina A,Errea P.Differential induction of phenylalanine ammonia-lyase gene expression in response to in vitro callus unions ofPrunusspp.[J].J Plant Physiol,2008,165:705-714.
17.Olsen K M,Lea U S,Slimestad R,et al.Differential expression of fourArabidopsisPAL genes;PAL1 and PAL2 have functional specialization in abiotic environmental-triggered flavonoid synthesis[J].J Plant Physiol,2008,165:1491-1499.
18.Hall T A.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[C].//Nucleic acids symposium series,1999,41:95-98.
19.Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
20.Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
21.Fu Y X,Li W H.Statistical tests of neutrality of mutations[J].Genetics,1993,133(3):693-709.
22.Rogers A R,Harpending H.Population growth makes waves in the distribution of pairwise genetic differences[J].Mol Biol Evol,1992,9(3):552-569.
23.Bandelt H J,Forster P,Röhl A.Median-joining networks for inferring intraspecific phylogenies[J].Mol Biol Evol,1999,16(1):37-48.
24.Pritchard J K,Stephens M,Donnelly P.Inference of population structure using multilocus genotype data[J].Genetics,2000,155(2):945-959.
25.Excoffier L,Laval G,Schneider S.Arlequin(version 3.0):an integrated software package for population genetics data analysis[J].Evol Bioinform,2005,1:47.
26.Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.
27.Jensen J L,Bohonak A J,Kelley S T.Isolation by distance,web service[J].BMC Genet,2005,6(1):13.
28.李俊清.植物遗传多样性及保护研究进展[J].植物研究,1998,18(2):226-242.
29.王静,张小平,李文良,等.濒危植物连香树居群的遗传多样性和遗传分化研究[J].植物研究,2010(2):208-214.
30.Joly C,Visset L.Evolution of vegetation landscapes since the Late Mesolithic on the French West Atlantic coast[J].Rev Palaeobot Palyno,2009,154(1):124-179.
31.刘方春,邢尚军,马海林,等.干旱生境中接种根际促生细菌对核桃根际土壤生物学特征的影响[J].应用生态学报,2014,25(5):1475-1482.
32.Akca Y,Samsunlu E.The effect of salt stress on growth,chlorophyll content,proline and nutrient accumulation,and K/Na ratio in walnut[J].Pak J Bot,2012,44(5):1513-1520.
33.冯连芬,吕芳德,张亚萍,等.我国核桃育种及其栽培技术研究进展[J].经济林研究,2006,24(2):69-73.
34.Hamrick J L,Godt M J W.Conservation genetics of endemic plant species[M].Conservation genetics.Springer US,1996:281-304.
35.奚声珂.我国胡桃属(JuglansL.) 种质资源与核桃(JuglansregiaL.) 育种[J].林业科学,1987,23(3):342-349.
36.Aradhya M K,Potter D,Gao F,et al.Molecular phylogeny ofJuglans(Juglandaceae):a biogeographic perspective[J].Tree Genet Genomes,2007,3(4):363-378.
37.赵鹏,周惠娟,刘占林,等.胡桃属植物分子系统发育和生物地理研究进展[J].林业科学,2014,50(11):147-157.
38.赵大鹏,王康满,侯元同.基于叶绿体trnL-F,rbcL序列和核糖体ITS序列探讨廖属(廖科)头状廖组的系统发育[J].植物研究,2012,32(1):77-83.
39.胡昳恒,党萌,张甜,等.秦岭地区核桃自然群体和栽培群体的遗传多样性及其演化关系—基于nrDNA ITS序列分析[J].林业科学,2014,50(12):47-55.
SequenceAnalysisofNuclearDNAJRD5680forDeterminingGeneticDiversityandGeneticStructureAnalysisofCommonWalnut(JuglansregiaL.)
ZHANG Tian WANG Ma-Li ZHAO Peng*
(College of Life Sciences,Northwest University,Xi’an 710069)
We studied the genetic diversity and population genetic structure of 265 individuals from different regions of China by using the roles of phenylalanine ammonia-lyase(PAL) nuclear geneJRD5680 DNA sequence of 40 common walnut(Juglansregia) and 5 out group plant species. The length ofJRD5680 sequence was 809 bp, the content of G+C was 46.2% with 74 information sites. A total of 30 haplotypes, haplotype diversity variation showed low genetic diversity(Hd=0.370,π=0.005 3). There is a clear geographical structure(NST>GST), which is a regional distribution pattern. The spatial genetic structure analysis showed that the correlation between geographical isolation and genetic distance was significant(r=0.263 2;P=0.032 4*), and there was obvious geographical isolation effect among different geographic populations. The results of comprehensive mismatch analysis and neutral test can be deduced from the recent history of the different regions of the population expansion event. From the genetic structure, the walnut population can be divided into two groups(southwest and other regions). By AMOVA, the genetic variation among the populations was mainly stored in the populations(56.54%), and the genetic differentiation among the populations was higher(FST=0.885). The total of 5 species of Juglandaceae could be a single branch, and theJRD5680 sequence might be with high molecular identification radio in the members of the Juglandaceae.
Juglansregia;phenylalanine ammonia-lyase(PAL) gene;genetic diversity;genetic structure
国家自然科学基金项目(31200500;41471038);陕西省教育厅重点实验室科研计划(13JS094)
张甜(1989—),女,硕士研究生,主要是从事植物分子生态学研究。
2015-10-27
S664.1
A
10.7525/j.issn.1673-5102.2016.02.012

