异肽键稳定的MERS-CoV融合蛋白S2亚基N端重复序列三股α螺旋的构建
郑 汐, 梁国栋, 王 潮, 刘克良
(军事医学科学院毒物药物研究所, 抗毒药物与毒理学国家重点实验室, 北京 100850)
异肽键稳定的MERS-CoV融合蛋白S2亚基N端重复序列三股α螺旋的构建
郑汐, 梁国栋, 王潮, 刘克良
(军事医学科学院毒物药物研究所, 抗毒药物与毒理学国家重点实验室, 北京 100850)
通过在天然N肽的氨基端引入可以诱导螺旋三聚体形成的人工多肽序列, 并通过酰基转移反应在上述嵌合肽所形成的三股α螺旋间引入异肽键, 构建了中东呼吸综合征病毒(MERS-CoV)的N-trimer模型, 为MERS-CoV融合抑制剂的设计奠定了基础.
中东呼吸综合征病毒; 融合蛋白; 多肽; 异肽键
2012年, 一名60岁的男性沙特籍患者死于多器官衰竭[1], Zaki等[2~4]在该患者体内分离出一种新型的人兽共患冠状病毒, 并将其命名为中东呼吸综合征病毒(MERS-CoV). MERS-CoV先后在中东及南韩地区爆发了大规模流行, 在全球范围内共造成1600余例人类感染, 导致590人死亡[5,6]. 与2003年亚洲地区流行的严重急性呼吸窘迫综合征冠状病毒(SARS-CoV)相比[7], MERS-CoV的致死率高达36%. 目前, 临床的治疗手段是利巴韦林与干扰素联合治疗, 尚无特异性治疗方法[2,8~10].
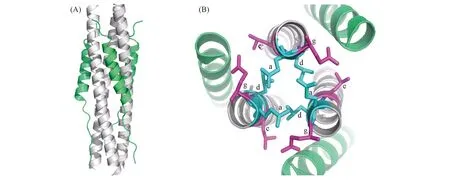
Scheme 1 Structure of 6-helix bundle(6-HB)(A) Side view of MERS-CoV S2 subunit 6-HB structure(PDB:4mod), peripheral helix present the C-terminal heptad repeat(CHR) in MERS-CoV, while the central helix trimer are N-terminal heptad repeat(NHR); (B) top view of 6-HB show the amino acid in a, d site(cyan) and e, g site.
MERS-CoV所属的冠状病毒科具有包膜结构[11]. 融合是包膜病毒入侵宿主细胞时的重要环节. 通过抑制融合, 可以将包膜病毒阻止在宿主细胞膜外, 最终被身体的免疫防线清除[12]. 晶体学研究结果表明, MERS-CoV包膜上具有融合功能的棘突糖蛋白(Spike protein)为Ⅰ类融合蛋白[12,13]. 病毒发生膜融合时, S蛋白的S1亚基会首先识别细胞表面的特异性受体CD26(或称DPP4)并与之结合, 然后在体内蛋白酶TMPRSS2的作用下暴露出S2亚基的膜融合肽FP片断, 并插入宿主细胞膜中[12~16]. 失去支撑的S2亚基沿着势能降低的方向发生折叠变构, 其中N端七重复序列(NHR)以α螺旋的形式形成同源三聚体并提供3个疏水沟槽; C端七重复序列(CHR)以α螺旋的形式结合至疏水沟槽内, 形成如Scheme 1所示的6-HB结构. 6-HB结构拉近了宿主细胞膜和病毒包膜之间的距离, 从而引发融合过程[17~20]. 6-HB结构是MERS-CoV入侵宿主细胞过程中的关键结构, 其中NHR三股螺旋位于其结构中心. 从MERS-CoV内源性6-HB晶体结构可知, NHR与CHR互为配基; 在药物研发领域, NHR与CHR互为靶标. 因此, 体外重现N肽三螺旋结构, 一方面可以作为开发C肽类MERS-CoV融合抑制剂的分子靶标, 另一方面N肽三螺旋本身又可以作为N肽类融合抑制剂用于阻止MERS-CoV感染宿主细胞. HIV-1与MERS-CoV同属I类包膜病毒, 具有相似的融合机制, 在融合过程中均经历由NHR-CHR相互作用形成的6-HB结构[21~23]. 因此, MERS-CoV肽类融合抑制剂的研究可以参考HIV-1融合抑制剂. Jiang等[24~26]报道了衍生于HIV-1 CHR的第一个HIV-1融合抑制剂多肽(C肽) T20, T20的上市开辟了利用C肽类融合抑制剂阻止Ⅰ类包膜病毒与宿主细胞融合的新领域. Jiang等[13]发现衍生于MERS-CoV融合蛋白S2亚基CHR区的36肽HR2P对MERS-CoV的半数抑制浓度达到1 μmol/L. 与此相反, 衍生于MERS-CoV融合蛋白NHR的多肽片段(N肽)HR1P(S蛋白第998~1039位)虽与HR2P互为靶点, 但因其长度缩短后结构不稳定, 从而其在离体状态下难以重现螺旋三聚体核心结构, 进而难以发挥生物学活性[27~32]. 因此, 如何按照已阐明的晶体结构和作用机制在体外重现MERS-CoV的NHR三螺旋结构, 成为开发N肽类MERS-CoV融合抑制剂的难点. 目前, 尚未见有关稳定MERS-CoV N肽方法的报道.
本文从MERS-CoV S蛋白S2亚基的NHR区域分段选取含有21个残基的N肽片段作为模板, 借鉴HIV-1 N肽的研究思路[33~35], 通过在N肽的氨基端引入人工设计的具有形成三股α螺旋能力的辅助序列, 改善N肽的水溶性, 并使N肽能自发组装成三聚体[36~38], 通过硫酯转移反应在特定氨基酸残基侧链间形成异肽键[39~41][Scheme 2(A)], 进一步稳定三聚体, 构建了MERS-CoV S2亚基N肽三聚体(N-trimer)模型. N-trimer模型的构建方法可以为MERS-CoV融合蛋白NHR三聚体的构建提供思路, 并为其它螺旋三聚体模型的构建提供参考.
Scheme 2 N-helix trimer stabled by isopeptide(A) Isopeptide bond was formed between lysine in e site and glutamic acid in g′ site of adjacent peptide chain; (B) schematic representation of isopeptide bond formation via an interhelical acyl transfer reaction.
1 实验部分
1.1试剂与仪器
Rink酰胺树脂(载量0.46 mmol/g)购自天津南开和成科技有限公司;N-芴甲氧羰基-L-丙氨酸(Fmoc-Ala-OH)、N-芴甲氧羰基-N′-三苯甲基-L-天冬酰胺[Fmoc-Asn(Trt)-OH]、N-芴甲氧羰基-N′-三苯甲基-L-谷氨酰胺[Fmoc-L-Gln(Trt)-OH]、N-芴甲氧羰基-O-叔丁基-L-谷氨酸[Fmoc-Glu(OtBu)-OH]、N-芴甲氧羰基-甘氨酸(Fmoc-Gly-OH)、N-芴甲氧羰基-L-异亮氨酸(Fmoc-Ile-OH)、N-芴甲氧羰基-L-亮氨酸(Fmoc-Leu-OH)、N-α-芴甲氧羰基-N′-叔丁氧羰基-L-赖氨酸[Fmoc-Lys(Boc)-OH]、N-芴甲氧羰基-L-甲硫氨酸(Fmoc-Met-OH)、N-芴甲氧羰基-L-苯丙氨酸(Fmoc-Phe-OH)、N-芴甲氧羰基-O-叔丁基-L-丝氨酸[Fmoc-Ser(tBu)-OH]、N-芴甲氧羰基-O-叔丁基-L-苏氨酸[Fmoc-Thr(tBu)-OH]、N-芴甲氧羰基-L-缬氨酸(Fmoc-Val-OH)、N-芴甲氧羰基-O-烯丙基-L-谷氨酸[Fmoc-Glu(OAll)-OH]、 苯并三氮唑-N,N,N′,N′-四甲基脲六氟磷酸盐(HBTU)和1-羟基苯并三唑(HOBt)购自上海吉尔生化有限公司; 双甲酮(Dimedone)、 四三苯基膦零价钯盐[Pd(PPh3)4]、 二乙基二硫代氨基甲酸钠盐(或称铜试剂)、 苄硫醇(HSBn)和N,N-二异丙基乙基胺(DIEA)购自J & K百灵威试剂公司;N-甲基吡咯烷酮(NMP)购自Acros试剂公司; 乙二硫醇(EDT)和间甲酚(m-Cresol)购自Alfa Aesar试剂公司; 三氟乙酸(TFA)和N,N-二甲基甲酰胺(DMF, 必要时CaH2浸泡过夜后减压蒸馏)购自北京博迈杰科技有限公司; 二氯甲烷(DCM)用无水K2CO3干燥处理, 避光静置24 h后使用; 哌啶与NaOH回流后重蒸或直接使用; 所用试剂均为分析纯.
CEM LibertyTM微波多肽合成仪(美国CEM公司); Shimadzu 10A型高效液相色谱仪(HPLC, 日本岛津株式会社); Venusil ASB C8液相色谱柱(4.6 mm×250 mm, 5 μm,天津博纳艾杰尔公司); Free-Zone 18 L型冷冻干燥机(美国Labconco公司); 9.4 T混合型串联傅里叶质谱仪(MS, 德国Bruker公司); REFLEX Ⅲ型MALDI-TOF质谱仪(MS, 德国Bruker公司); Waters Delta Prep-4000型高压制备液相色谱仪(HPLC, 美国Waters公司); X-Bridge C8高压制备液相色谱柱(19.5 mm×250 mm, 美国Waters公司); Milli-Q纯水机(美国MilliPore公司).
1.2实验过程
1.2.1多肽序列的固相合成使用CEM微波多肽合成仪, Rink酰胺树脂, 0.25 mmol规模. 采用标准的氨基芴甲氧羰基保护(Fmoc)固相方法合成多肽树脂载体, 以10 mL DMF和10 mL DCM作为溶胀溶剂, 以10 mL哌啶(体积分数20%)的DMF溶液作为脱保护试剂, 以HBTU/HOBt/DIEA作为缩合剂, 以0.5 mL乙酸酐与20 mL DCM溶液(含0.5 mL DIEA)作为封端试剂, 经[树脂溶胀-脱Fmoc保护-氨基酸缩合-]循环, 期间用DMF洗涤树脂残留溶液, 从C端依次向N端合成氨基酸, 必要时缩合2次. 最末端氨基酸脱除Fmoc后乙酰化封端保护, 得到侧链正交保护的多肽树脂载体.
采用侧链由烯丙氧羰基保护的L-型谷氨酸参与多肽序列的合成, 如Scheme 2(B)所示. 将Rink酰胺树脂同上述封端后, 用5 mmol零价钯Pd(PPh3)4和5 mmol双甲酮在避光条件下正交脱除谷氨酸的保护基团作为反应位点. 暴露的—COOH与5 mmol苄硫醇在EDC/HOBt催化下缩合形成硫脂键. 以B裂解液[V(三氟乙酸)∶V(间甲酚)∶V(苯甲醚)∶V(乙二硫醇)∶V(水)=16∶1∶1∶1∶1]与树脂以10 mL/g的比例裂解冻干得粗肽. 利用高压制备液相色谱纯化后冻干得到纯肽, 纯度大于95%(HPLC). 用ESI-Q-FT-MS或MALDI-TOF-MS鉴定多肽. 将冻干粉精密称量至每份约1 mg并于4 ℃保存[13].
1.2.2用异肽键交联成三聚体将上述硫酯化多肽在纯化、 浓缩、 冻干后溶解于pH=7.4的PBS缓冲液中(含15%体积分数乙腈), 浓度为1 mg/mL, 置于37 ℃培养箱中避光孵育. 在该条件下, IZ片段与N肽片段自发组装为三聚体的驱动力使多肽单体相互靠近, 使硫酯化修饰的Glu活泼酯与对应位点的Lys侧链发生酰基转移反应, 用分析型RP-HPLC监测形成异肽键的反应动力学RC-HPLC条件:A相V(TFA)∶V(H2O)=1∶1000, B相V(TFA)∶V(H2O)∶V(MeCN)= 1∶300∶700, 梯度0 min B相(体积分数10%), 5 min B相(体积分数50%), 20 min B相(体积分数100%); 流速1 mL/min.
2 结果与讨论
RP-HPLC动力学检测结果如图1(A)~(C)所示, 硫酯化修饰的单体由于非极性基团的引入, 原料峰保留时间最长, 均大于17.5 min. 在PBS缓冲液提供的弱碱离子环境下, 多肽发生自组装, 目标反应位点在空间距离和方向的限制下相互靠近, 发生缓慢的酰基转移反应形成异肽键. 由于形成的三聚体脱去了硫酯非极性基团, 极性增大; 形成三聚体时, 共价交联将多肽a,d位形成的疏水面包裹在分子内侧, 降低了与反相柱填料的作用概率; 形成三聚体时, 分子量增大, 使柱填料保留能力减弱, 故产物色谱峰出现在保留时间为11~12 min的位置. 由图1(D)~(I)可见, 经ESI-Q-FT-MS验证, 与多肽单体相比, 该产物为N-trimer共价交联三聚体. 结果表明, 经过修饰的N肽在生理条件下具有自组装成三螺旋的倾向, 并以这一驱动力为空间结构基础, 发生定点酰基转移反应, 生成了异肽键稳定的N-trimer.
Fig.1 RP-HPLC traces(A—C) and mass spectrometry(D—I) of isopeptide bond forming reactionRP-HPLC traces for the acyl transfer reaction of (IZN21L)3(A), (IZN21M)3(B) and (IZN21R)3(C) from t=2.5 to 150 h. Absorbance was monitored at 210 nm. (A—C):a. The reaction material; b. the reaction product; c. by-products. Mass spectrometry of monomer peptide, the reaction materal of (IZN21L)3(D), (IZN21M)3(E), (IZN21R)3(F) and reaction products, the trimeric peptide (IZN21L)3(G), (IZN21M)3(H) and (IZN21R)3(I).
螺旋间的酰基转移反应是一个定点特异的交联反应. Lai等[32,33,41]利用该反应在螺旋间的e/g′位定点特异性地形成异肽键用于稳定超螺旋结构. 本文序列中的Glu-11和Lys-16分别处于e/g′位(见表1), 因此可以确定Glu-11可以与相邻螺旋的Lys-16发生特异性反应形成异肽键交联的N-trimer. Lai等报道了杂质峰中的中间产物随着反应时间延长会逐渐减少, 本实验中也观察到相似的现象. 但由于MERS-CoV N肽高级结构的稳定性比HIV-1更差, 因此交联反应更困难, 不仅所需时间更长, 而且副产物更多. 本文交联反应的副产物主要是多肽在生理条件下的降解碎片; 从化学反应特性的角度推测, 其它副反应如硫酯基团的水解和非特异氨基位点反应也不可避免. 用人工序列修饰MERS-CoV融合蛋白跨膜亚基S2的NHR衍生肽, 使之不易聚集并具有自组装倾向, 再通过硫酯活化的酰基转移反应, 将螺旋间的一对谷氨酸-赖氨酸侧链以异肽键的方式连接在一起, 构建了由共价键稳定的N-trimer结构. 在进一步结构研究的指导下, 该模型可为中东呼吸综合征冠状病毒融合蛋白跨膜亚基体外N端三螺旋的构建提供参考.

Table 1 Designed peptide sequences derived from HR1P
*Lower-case letters(a—g) above indicate the amino acids sites in heptad repeat. Italic letters represent auxiliary sequence. IZm:IKKE IEAIKKE QEAIKKK IEAI. Isopeptide bonds are formed between Glu-11 and Lys-16 (underlined).
[1]Zaki A. M., Van B. S., Bestebroer T. M., Osterhaus A. D., Fouchier R. A.,N.Engl.J.Med., 2012, 367(19), 1814—1820
[2]Price S. M., Miazgowicz K. L., Munster V. J.,Path.Dis., 2014, 71(2), 121—136
[3]Bermingham A., Chand M. A., Brown C. S., Aarons E., Tong C., Langrish C., Hoschler K., Brown K., Galiano M., Myers R., Pebody R. G., Green H. K., Boddington N. L., Gopal R., Price N., Newsholme W., Drosten C., Fouchier R. A., Zambon M.,Euro.Surveill., 2012, 17(40), 20290
[4]Abdel-Moneim A. S.,Arch.Virol., 2014, 159(7), 1575—1584
[5]Memish Z. A., Zumla A. I., Assiri A.,N.Engl.J.Med., 2013, 369(9), 884—886
[6]World Health Organization(WHO),OutbreaksandEmergencies-MiddleEastRespiratorySyndromeCoronavirus(MERS-CoV). http://www.who.int/emergencies/mers-cov/en
[7]Drosten C., Günther S., Preiser W., van der Werf S., Brodt H. R., Becker S., Rabenau H., Panning M., Kolesnikova L., Fouchier R. A. M., Berger A., Burguiere A. M., Cinatl J., Eickmann M., Escriou N., Grywna K., Kramme S., Manuguerra J. C., Muller S., Rickerts V., Sturmer M., Vieth S., Klenk H. D., Osterhaus A., Schmitz H., Doerr H. W.,N.Engl.J.Med., 2003, 348(20), 1967—1976
[8]Majumder M. S., Kluberg S. A., Mekaru S. R., Brownstein J. S.,Emerg.Infect.Dis., 2015, 21(11), 2088—2090
[9]Chan J. F., Chan K. H., Kao R. Y., To K. K., Zheng B. J., Li C. P. Y., Li P. T. W., Dai J., Mok F. K. Y., Chen H., Hayden F. G., Yuen K.,J.Infection, 2013, 67(6), 606—616
[10]Kilianski A., Baker S. C.,Antivir.Res., 2014, 101(1), 105—112
[11]Chen Y., Rajashankar K. R., Yang Y., Agnihothram S. S., Liu C., Lin Y. L., Baric R. S., Li F.,J.Virol., 2013, 87(19), 10777—10783
[12]Gao J., Lu G. W., Qi J. X., Li Y., Wu Y., Deng Y., Geng H. Y., Li H. B., Wang Q. H., Xiao H. X., Tan W. J., Yan J. H., Gao G. F.,J.Virol., 2013, 87(24), 13134—13140
[13]Lu L., Liu Q., Zhu Y., Chan K. H., Qin L., Li Y., Wang Q., Chan J. F. W., Du L. Y., Yu F., Ma C. Q., Ye S., Yuen K. Y., Zhang R. G., Jiang, S. B.,Nat.Commun., 2014, 5(5), 3067—3078
[14]Chan J. F. W., Chan K. H., Choi G. K. Y., To K. K. W., Tse H., Cai J. P., Yeung M. L., Cheng V. C., Chen H., Che X. Y., Lau S. K., Woo P. C., Yuen K. Y.,J.Infect.Dis., 2013, 207(11), 1743—1752
[15]Lu G. W., Hu Y. W., Wang Q. H., Qi J. X., Gao F., Li Y., Zhang Y. F., Zhang W., Yuan Y., Bao J. K., Zhang B. C., Shi Y., Yan J. H., Gao G. F.,Nature, 2013, 500(7461), 227—231
[16]Gierer S., Bertram S., Kaup F., Wrensch F., Heurich A., Krämer-Kühl A., Welsch K., Winkler M., Meyer B., Drosten C., Dittmer U., von Hahn T., Simmons G., Hofmann H., Poehlmann S.,J.Virol., 2013, 87(10), 5502—5511
[17]Masters P. S.,Adv.VirusRes., 2006, 66, 193—292
[18]Lu M., Blacklow S. C., Kim P. S.,Nat.Struct.Biol., 1995, 2(12), 1075—1082
[19]Ma G., Feng Y., Gao F., Wang J., Liu C., Li Y.,Biochem.Bioph.Res.Commun., 2005, 337(4), 1301—1307
[20]Liu S., Zhao Q., Jiang S.,Peptides, 2003, 24(9), 1303—1313
[21]Apellániz B., Huarte N., Largo E., Nieva J. L.,Chem.Phys.Lipids, 2014, 181, 40—55
[22]Tripet B., Howard M. W., Jobling M., Holmes R. K., Holmes K. V., Hodges R. S.,J.Biol.Chem., 2004, 279(20), 20836—20849
[23]Eckert D. M., Kim P. S.,Annu.Rev.Biochem., 2001, 70(1), 777—810
[24]Korazim O., Sackett K., Shai Y.,J.Mol.Biol., 2006, 364(5), 1103—1117
[25]Jiang S., Lin K., Strick N., Neurath A. R.,Nature, 1993, 365, 113
[26]Kilby J. M., Hopkins S., Venetta T. M., DiMassimo B., Cloud G. A., Lee J. Y., Alldredge L., Hunter E., Lambert D., Bolognesi D., Mathews T., Johnson M. R., Nowak M. A., Shaw G. M., Saag M. S.,Nat.Med., 1998, 4(11), 1302—1307
[27]Muppidi A., Zhang H., Curreli F., Li N., Debnath A. K., Lin Q.,Bioorg.Med.Chem.Lett., 2014, 24(7), 1748—1751
[28]Bianchi E., Finotto M., Ingallinella P., Hrin R., Carella A. V., Hou X. S., Schleif W. A., Miller M. D., Geleziunas R., Pessi A.,PNAS, 2005, 102(36), 12903—12908
[29]Eckert D. M., Kim P. S.,PNAS, 2001, 98(20), 11187—11192
[30]Chen X., Lu L., Qi Z., Lu H., Wang J., Yu X., Chen Y. H., Jiang S.,J.Biol.Chem., 2010, 285(33), 25506—25515
[31]Kang H. J., Baker E. N.,TrendsBiochem.Sci., 2011, 36(4), 229—237
[32]Wang C., Lai W. Q., Yu F., Zhang T., Lu L., Jiang X. F. , Zhang Z., Xu X., Bai Y., Jiang S., Liu K. L.,Chem.Sci., 2015, 6(11), 6505—6509
[33]Liang G. D., Wang C., Shi W. G., Wang K., Jiang X. F., Xu X. Y., Liu K. L.,Chem.J.ChineseUniversities, 2014, 35(10), 2100—2103(梁国栋, 王潮, 史卫国, 王昆, 姜喜凤, 许笑宇, 刘克良. 高等学校化学学报, 2014, 35(10), 2100—2103)
[34]Li X., Lai W. Q., Jiang X. F., Wang C., Liu K. L.,Chem.J.ChineseUniversities, 2016, 37(5), 881—885(李雪, 来文庆, 姜喜凤, 王潮, 刘克良. 高等学校化学学报, 2016, 37(5), 881—885)
[35]Zhang X. Y., Deng. D. J., Tan J. J., He Y., Li C. H., Wang C. X.,Chem.Res.ChineseUniversities, 2014, 30(2), 297—305
[36]Suzuki K., Hiroaki H., Kohda D., Tanaka T.,ProteinEng., 1998, 11(11), 1051—1055
[37]Huyghues-Despointes B. M., Klingler T. M., Baldwin R. L.,Biochemistry, 1995, 34(41), 13267—13271
[38]Clinton T. R., Weinstock M. T., Jacobsen M. T., Szabo-Fresnais N., Pandya M. J., Whitby F. G., Herbert A. S., Prugar L. I., McKinnon R., Hill C. P., Welch B. D., Dye J. M., Eckert D. M., Kay M. S.,ProteinSci., 2015, 24(4), 446—463
[39]Bai Y., Xue H., Wang K., Cai L., Qiu J., Bi S., Lai L., Cheng M., Liu S., Liu K.,AminoAcids, 2013, 44, 701—713
[40]BaiY., Ling Y., Shi W. G., Cai L. F., Jia Q., Jiang S. B., Liu K. L.,Chem.Biochem., 2011, 12(17), 2647—2658
[41]Lai W. Q., Wang C., Yu F., Lu L., Wang Q., Jiang X., Xu X., Zhang T., Wu S., Zheng X., Zhang Z., Dong F., Jiang S., Liu K.,Chem.Sci., 2016, 7, 2145—2150
(Ed.:P, H, W, K)
† Supported by the National Natural Science Foundation of China(Nos.81373266, 81573266).
Construction of Isopeptide Bridge-tethered NHR-trimeric Coiled-coil in MERS-CoV Membrane Fusion Protein†
ZHENG Xi, LIANG Guodong, WANG Chao*, LIU Keliang*
(Beijing Institute of Pharmacology and Toxicology, State Key Laboratory of Toxicology andMedicalCountermeasures,Beijing100850,China)
In Middle East respiratory syndrome coronavirus(MERS-CoV) infection, the formation of a coiled-coil six-helical bundle(6-HB) between three C-terminal heptad repeats(CHRs) and an N-terminal heptad repeat(NHR) trimer of the MERS-CoV spike protein S2 subunit provided the energy necessary for virus-cell membrane fusion. Mimicry of the NHR-helical trimers in the MERS-CoV membrane fusion protein for the discovery of antiviral therapeutics hampered because of the strong aggregation properties of synthetic NHR-based peptides taken out of their parent protein structure. Herein, the article presents an efficient strategy to recapi-tulate MERS-CoV NHRα-helical trimersviacombining the concepts of grafting the NHR-derived peptides onto an exogenous trimerization motif with stabilization of the trimeric oligomers through isopeptide bonds. The covalent bridge was introduced by an acyl transfer reaction between lysine and glutamic acid in specific amino acid sites with thio-easter modified. The designed isopeptide bridge-stabilized chimeric trimers has strong potential for the development potent MERS-CoV fusion inhibitors.
Middle East respiratory syndrome coronavirus; Fusion protein; Peptide; Isopeptide bond
10.7503/cjcu20160237
2016-04-13. 网络出版日期:2016-08-26.
国家自然科学基金(批准号:81373266, 81573266)资助.
O629.72
A
联系人简介:刘克良, 男, 博士, 研究员, 博士生导师, 主要从事多肽药物、 核酸化学及药用高分子材料研究.E-mail:keliangliu55@126.com
王潮, 男, 博士, 助理研究员, 主要从事多肽药物研究. E-mail:chaow301@gmail.com

