人工合成小麦染色体区段在小麦优良新品系中的分布
王宏霞,陈文杰,曹 东,甯顺腙,刘宝龙,张 波,张连全,刘登才,,张怀刚
(1.中国科学院西北高原生物研究所高原生物适应与进化重点实验室,青海西宁 810008;2.四川农业大学小麦研究所,四川成都 611130; 3.中国科学院大学,北京 100049)
人工合成小麦染色体区段在小麦优良新品系中的分布
王宏霞1,3,陈文杰1,曹 东1,3,甯顺腙2,刘宝龙1,张 波1,张连全2,刘登才1,2,张怀刚1
(1.中国科学院西北高原生物研究所高原生物适应与进化重点实验室,青海西宁 810008;2.四川农业大学小麦研究所,四川成都 611130; 3.中国科学院大学,北京 100049)
从人工合成六倍体小麦SHW-L1改良后代中选育的5个春小麦新品系,在青海表现出比对照品种高原448更优的农艺性状和产量潜力,推测源于外源物种的野生不良性状被淘汰,保留在新品系中的外源染色体区段可能对遗传改良有贡献。为了了解源自人工合成小麦SHW-L1的外源染色体区段在这5个改良新品系中的分布,利用11 660个具有染色体位置信息的多态性DArTseq标记对这5个改良品系进行了外源染色体区段分析。结果表明,共检测到78个外源染色体区段,其中,65个为源于四倍体小麦的A和B基因组,13个为来自于节节麦的D基因组。24个源于四倍体小麦的外源染色体区段分布于3个以上的品系中,这些区段主要来自于A基因组,其中2A有8个,7A有4个,1A有3个,6A有3个。本研究材料来自于混合选择,不同品系共有的外源染色体区段可能含有对当前育种有价值的重要基因位点或基因簇,这样的区段将是下一步关注的重点。
染色体区段;人工合成六倍体小麦;DArTseq标记
普通小麦(TriticumaestivumL.,2n=6x=42,AABBDD)是由四倍体小麦(TriticumturgidumL.,AABB)与节节麦(AegilopstauschiiCoss.,DD)远缘杂交,再经染色体自然加倍而来[1-4]。它是世界上超过40个国家和35%的人口的主粮,为人类提供的能量和蛋白质营养超过总量的20%。预计到2050年,小麦年均产量需增加2%,才能满足需要,这将是一个巨大挑战[5]。增加单产是提高小麦总产的环境友好型方法[6]。拓宽小麦育种改良的遗传基础是进一步增加小麦单产潜力的重要基础工作。
普通小麦的基因组供体物种(四倍体小麦和节节麦)为普通小麦育种改良提供了大量的遗传变异[7-9]。人工模拟普通小麦的起源过程,将四倍体小麦与节节麦人工杂交,然后经染色体加倍产生的人工合成六倍体小麦(Synthetic hexaploid wheat,SHW)[10],可同时将四倍体小麦和节节麦的外源遗传多样性导入六倍体小麦。SHW与普通小麦的A、B、D染色体组同源,其优良目的基因可通过染色体同源配对重组转移至普通小麦。以SHW为“桥梁”,与普通小麦优良品种(系)杂交,并进一步用优良品种(系)回交,在改良SHW携带的不利性状的同时,可将位于SHW不同染色体位置的优良基因导入到普通小麦中。这种“SHW桥梁转移法”已被国际上广泛使用[11-14]。澳大利亚“Synthetic Evaluation Project”、英国“Wheat LOLA Project”、法国“BREEDWHEAT”、墨西哥“SeeD”等研究计划,均涉及SHW的遗传改良应用[15]。利用SHW已培育了一批小麦新品种并应用于小麦生产[14-15],例如巴基斯坦的小麦新品种Lalma和KT-2010、墨西哥的Maravilla及西班牙的Carmona。在国内,利用源于CIMMYT的“硬粒小麦-节节麦人工合成小麦”,选育出了川麦38、川麦42、川麦43、川麦47等小麦新品种[16-20];利用中国圆锥小麦地方品种AS2255与中东节节麦AS60创制的人工合成小麦SHW-L1选育出了优良新品种“蜀麦969”[21]。
SHW与现有推广小麦品种的遗传差异大[7-8,13-15],因此SHW的染色体区段导入推广小麦品种后易于被检测和跟踪。但是,囿于常规分子标记的检测效率,目前对源于SHW的染色体区段在改良小麦品种(系)中的分布信息还知之甚少。近年发展起来的高通量分子标记为检测源于SHW的染色体区段提供了新的手段。基于多样性序列芯片技术(Diversity Arrays Technology,DArT)[22]和下一代测序平台的高通量DArTseq(http://www.diversityarrays.com/dart-application-dartseq)提供了一种高效的多样性分型手段。本研究拟利用该技术,对人工合成六倍体小麦SHW-L1经遗传改良后得到的5个在前期田间观察中表现较好的春小麦新品系进行分析,以期了解源自人工合成小麦SHW-L1的外源染色体区段在这5个改良新品系中的分布,为下一步外源染色体区段的育种组装和优良基因发掘准备条件。
1 材料与方法
1.1供试材料
供试材料为青海小麦主栽品种高原448(对照)及前期田间观察综合农艺性状优良的人工合成小麦改良新品系B2025、B2026、B2027、B2028和B2029。以上5个新品系来自同一杂交组合SHW-L1/SY95-71//700-011689/3/MY68942,F2、F3、F4代采用单株混合选择,在F5代选择出5个综合性状较好的单株,分别自交选择至F8代。SHW-L1为人工合成小麦,来自于中国四倍体小麦(Triticumturgidumssp.turgidum)地方品种AS2255与中东节节麦(Aegilopstauschii)AS60杂种的染色体加倍[21];SY95-71、700-011689和MY68942为普通六倍体小麦(TriticumaestivumL.,2n=6x=42,AABBDD)。
1.2试验方法
1.2.1农艺性状调查
5个小麦新品系及高原448连续两年(2013年和2014年)种植于青海省西宁市城北区吧浪村。每个材料种2行,行长2.0 m,行距0.2 m,株距0.1 m,地膜覆盖。设3次重复。于成熟期调查株高、主穗长、主穗小穗数、穗粒数和千粒重等农艺性状。
1.2.2产量比较
5个小麦新品系及高原448连续两年(2013年和2014年)种植于青海省西宁市湟中县坝沟门村,进行产量比较试验。每个材料种10行,行长2.0 m,行距0.2 m,每行播种300粒(与青海生产上的用种量接近),田间管理一致。设3次重复。收获晒干后测定产量。同时,新品系B2026参加了2013年青海水地春小麦区域试验,区域试验编号为11-5966,区域试验资料来源于青海省种子管理站。
1.2.3DArTseq分析及源于SHW-L1的染色体区段鉴定
苗期剪取叶片,采用天根生化科技(北京)有限公司生产的植物基因组DNA提取试剂盒提取植物基因组DNA。获得的DNA样品送澳大利亚公司Diversity Arrays Technology Pty Ltd (DArT P/L)(http://www.diversityarrays.com)进行DArTseq分型。DArTseq获得的silicoDArTs标记为显性标记,分型的结果为1(有)、0(无)和-(不确定)。将获得的silicoDArTs标记按照“亲本-后代”的顺序整理后,根据公司提供的染色体上的标记位置信息,排列标记位置。在系谱的基础上,通过新品系与亲本间的比较确定每一个标记的来源,将新品系中来自同一亲本(例如SHW-L1)的多个连续标记看作是一个染色体区段。使用Perl语言编写的一个免费的数据可视化软件circos(http://circos.ca/circos)构建标记区段的分布图。
1.2.4数据处理
农艺性状及产量数据用SPSS软件进行统计分析。
2 结果与分析
2.1农艺性状分析
由表1可知,5个改良品系的株高(65.45~90.83 cm)均显著低于对照高原448(100.32 cm);B2028和B2029的主茎穗长与对照高原448无显著差异,其余3个品系均显著高于对照;B2029的主茎穗穗粒数和有效小穗数均与对照高原448无显著差异,其余4个品系均显著高于对照;B2025和B2028的千粒重与对照无显著差异,其余3个品系均显著高于对照。说明这5个改良品系为农艺性状优良的小麦品系。
2.2产量比较
由表1可知,B2026(8 187.19 kg·hm-2)和B2029(8 117.29 kg·hm-2)的产量显著高于对照高原448(6 782.05 kg·hm-2),其余3个品系与对照无显著性差异。说明这5个改良品系的产量均达到了一个比较高的水平。
由于条锈病新小种的出现,这些新品系从2013年开始在青海一些地区高感条锈病。但是,B2026在2013年青海省水地春小麦区域试验中仍表现高产,7个试点平均产量为7 122.00 kg·hm-2,居第一位,比对照品种高原448平均增产7.85%(表2)。
2.3DArTseq标记检测结果
为明确这些优良新品系导入的人工合成小麦染色体区段,本研究进行了DArTseq分析。基于公司提供的分型结果,共获得39 210个覆盖小麦全基因组的多态silicoDArTs标记,其中,11 660个具有染色体位置信息,覆盖基因组A、B和D染色体的总长度为5 961.15 cM,平均1 cM含1.96个标记,即相邻标记间的平均距离为0.51 cM(表3)。从染色体组来看,B染色体组上的平均标记密度最高,D染色体组最低。A染色体组中,2A标记密度最高,1A最低;B染色体组中,2B标记密度最高,4B最低;D染色体组中,2D标记密度最高,4D最低。
2.4源于人工合成小麦SHW-L1的染色体区段鉴定结果
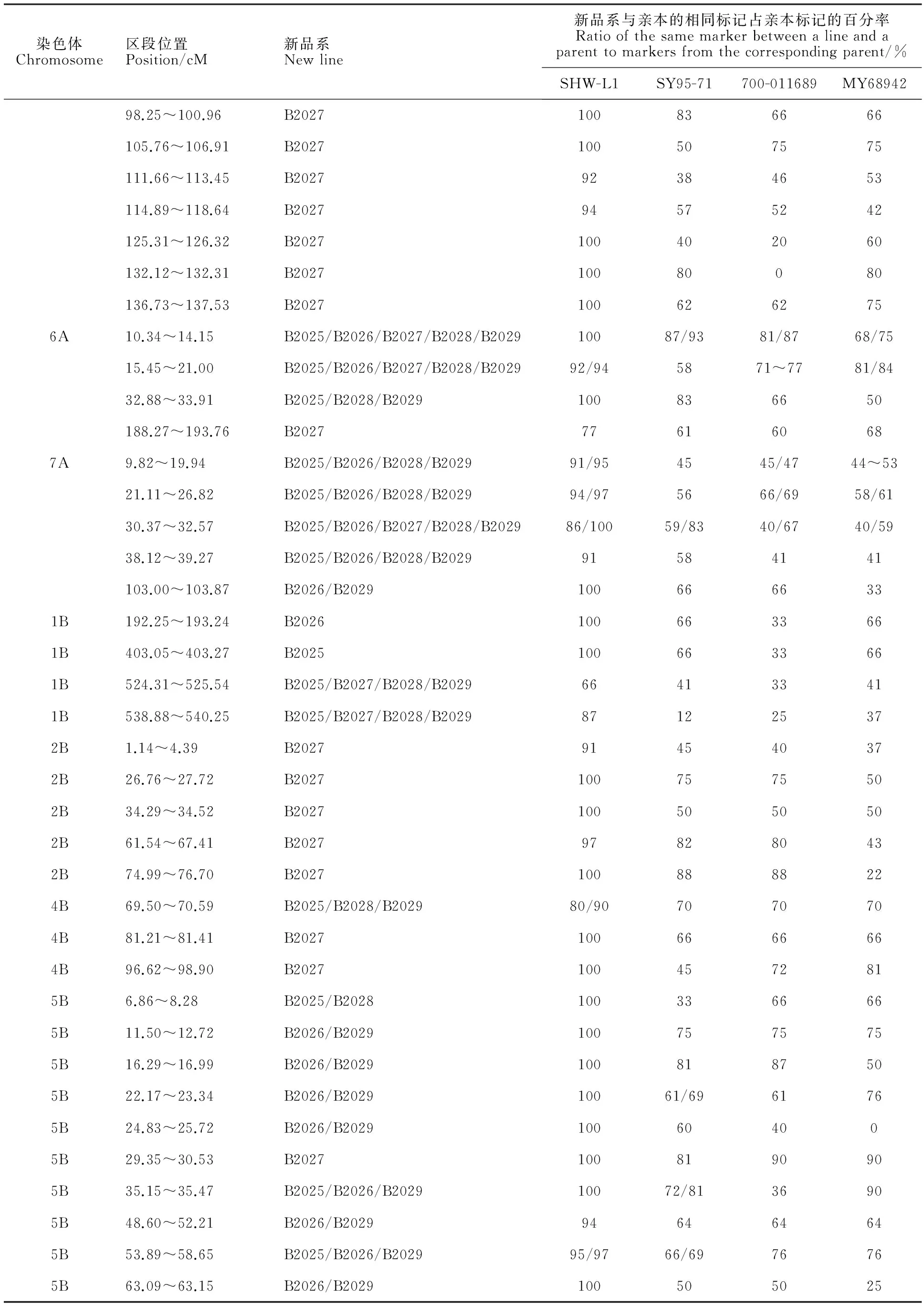
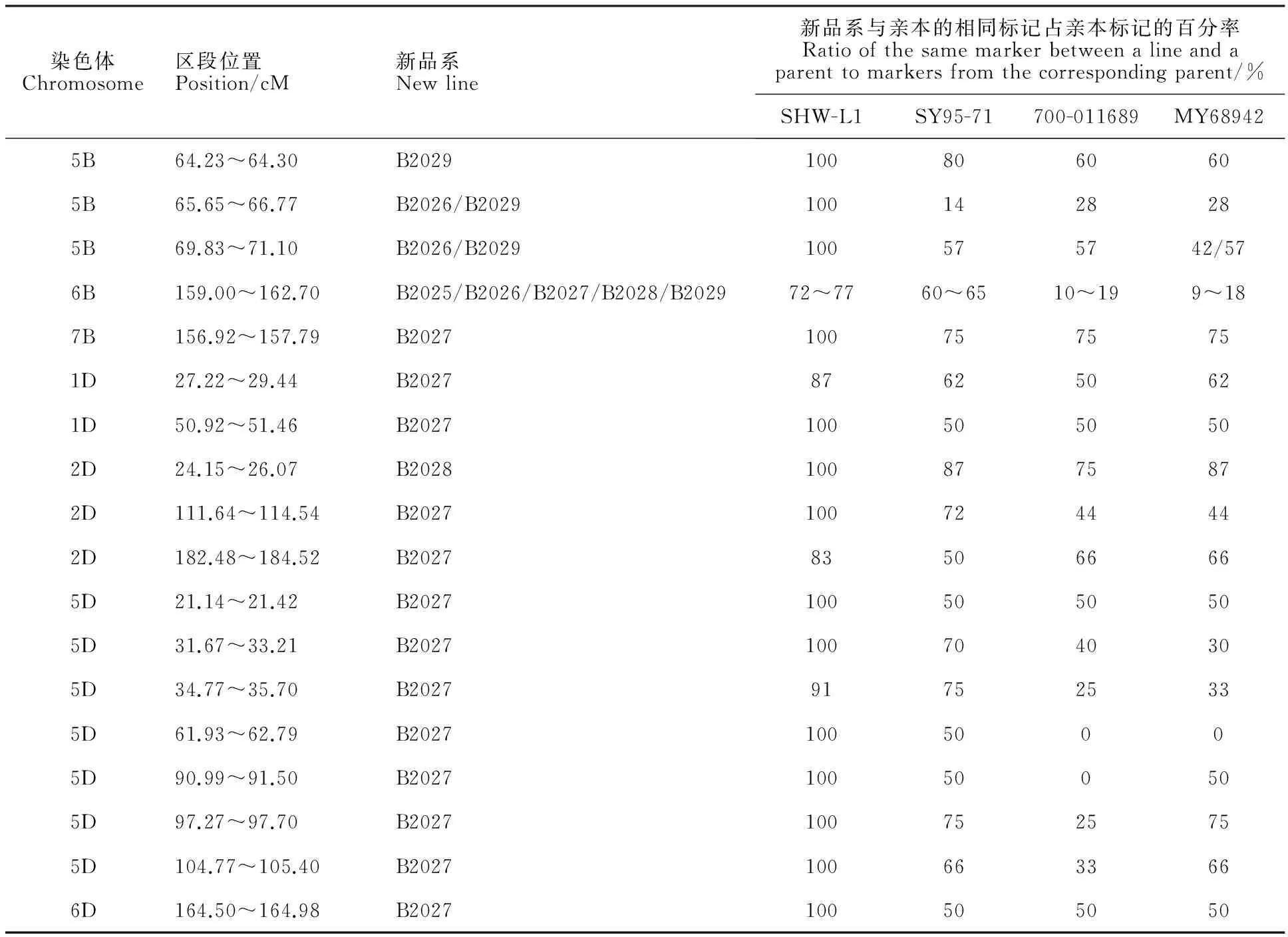
根据染色体上连续标记的分布情况,划分染色体区段。5个新品系总共包含755个区段,分布在不同染色体上(图略)。其中,78个区段源于SHW-L1,分布于除3B、3D、4D和7D以外的所有染色体上(表4),A染色体上最多,达38个;B染色体上27个;D染色体上最少,为13个。
新品系B2027、B2029、B2026、B2025和B2028含有的SHW-L1染色体区段数依次为46、34、33、26和24个。品系B2027的SHW-L1染色体区段分布于除2A、3A、3B、3D、4D和7D以外的15条染色体上,B2029分布于1A、2A、6A、7A、1B、4B、5B和6B等8条染色体上,B2026分布于1A、2A、3A、4A、6A、7A、1B、5B和6B等9条染色体上,B2025分布于1A、2A、6A、7A、1B、4B、5B和6B等8条染色体上,B2028分布于1A、
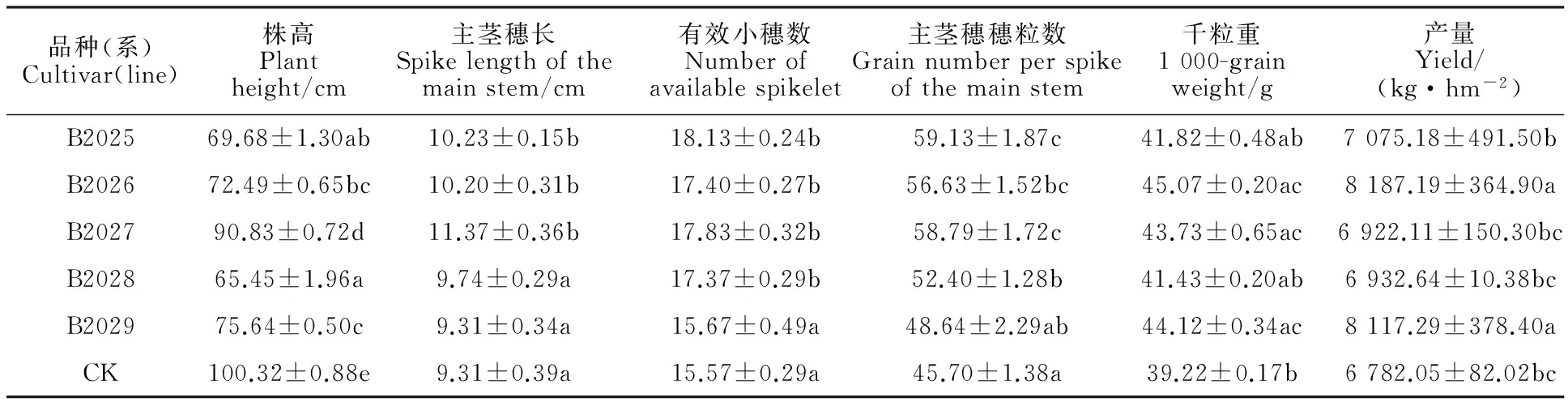
表1 人工合成小麦新品系和高原448(CK)的农艺性状及产量Table 1 Agronomic traits and yield of SHW lines and Gaoyuan 448(CK)
数据后不同字母表示品种(系)间差异在0.05水平上显著。
Different letters following values mean significant difference among different varieties(lines) at 0.05 level.
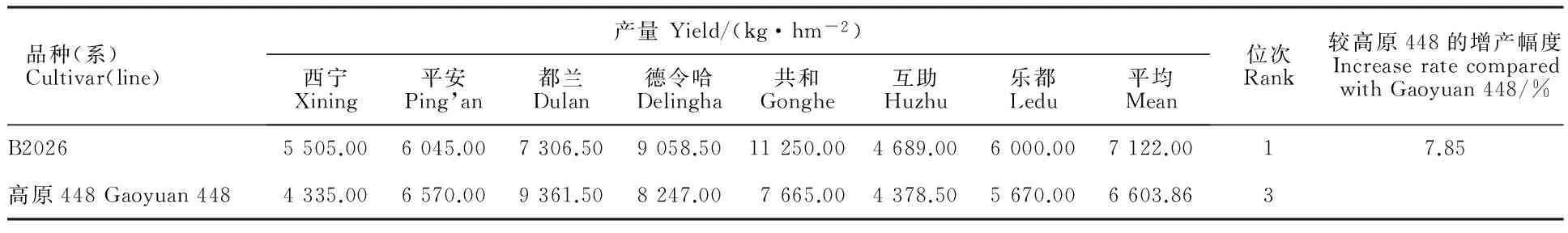
表2 2013年青海水地春小麦区试中B2026的产量表现Table 2 Yield of line B2026 in Qinghai provincial region trials for spring wheat in irrigated cropland
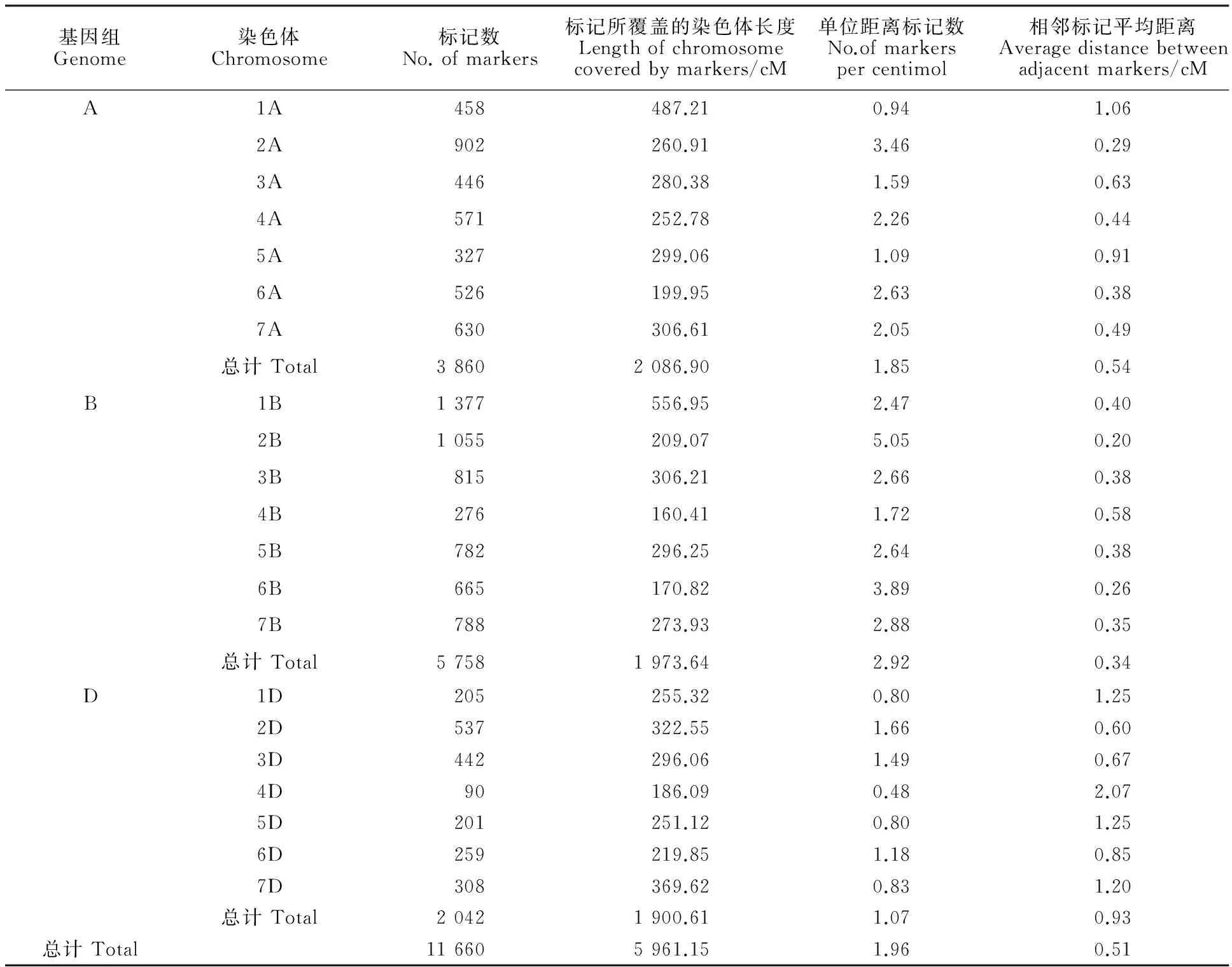
表3 DArT分子标记在小麦21条染色体上的分布Table 3 Distribution of DArT markers on 21 chromosomes of wheat
2A、6A、7A、1B、4B、5B、6B和2D等9条染色体上。导入的13个D基因组区段(12个导入到B2027,1个导入到B2028)中,7个来自5D染色体。
进一步分析不同品系共有的SHW-L1染色体区段发现,5个品系均含有源于SHW-L1的5个染色体区段:449.60~450.05 cM(1A)、10.34~14.15 cM(6A)、15.45~21.00 cM(6A)、30.37~32.57 cM(7A)和159.00~162.70 cM(6B);有15个区段同时存在于4个品系中,包括472.40~475.73 cM(1A)、480.14~482.56 cM(1A)、524.31~525.54 cM(1B)、538.88~540.25 cM(1B)、199.07~200.68 cM(2A)、210.59~213.15 cM(2A)、214.32~215.49 cM(2A)、220.95~221.10 cM(2A)、222.12~227.05 cM(2A)、230.40~234.76 cM(2A)、236.29~240.08 cM(2A)、243.29~244.61 cM(2A)、9.82~19.94 cM(7A)、21.11~26.82 cM(7A)和38.12~39.27 cM(7A);有4个外源染色体区段同时存在于3个品系中,包括69.50~70.59 cM(4B)、35.15~35.47 cM(5B)、53.89~58.65 cM(5B)和32.88~33.91 cM(6A)。从上述分布可以看出,这24个分布于3个以上品系的染色体区段全部来自A、B基因组,即源于人工合成小麦的四倍体小麦亲本,而没有片段源于节节麦D基因组。同时发现,这些区段主要来自于A基因组,其中2A有8个、7A有4个、1A有3个、6A有3个。
进一步分析源于SHW-L1染色体区段的临近区段,发现5个品系在6A染色体上含有两个相邻外源区段,包含在10.34~21.00 cM区间(图2、表3);品系B2025、B2026、B2028和B2029在2A染色体上的大约45 cM(199.07~244.61 cM)的区间内,含有8个相邻区段,约占2A染色体总长的1/6;类似地,在7A染色体上的一个大约30 cM(9.82~39.27 cM)的区间内包含4个相邻区段。
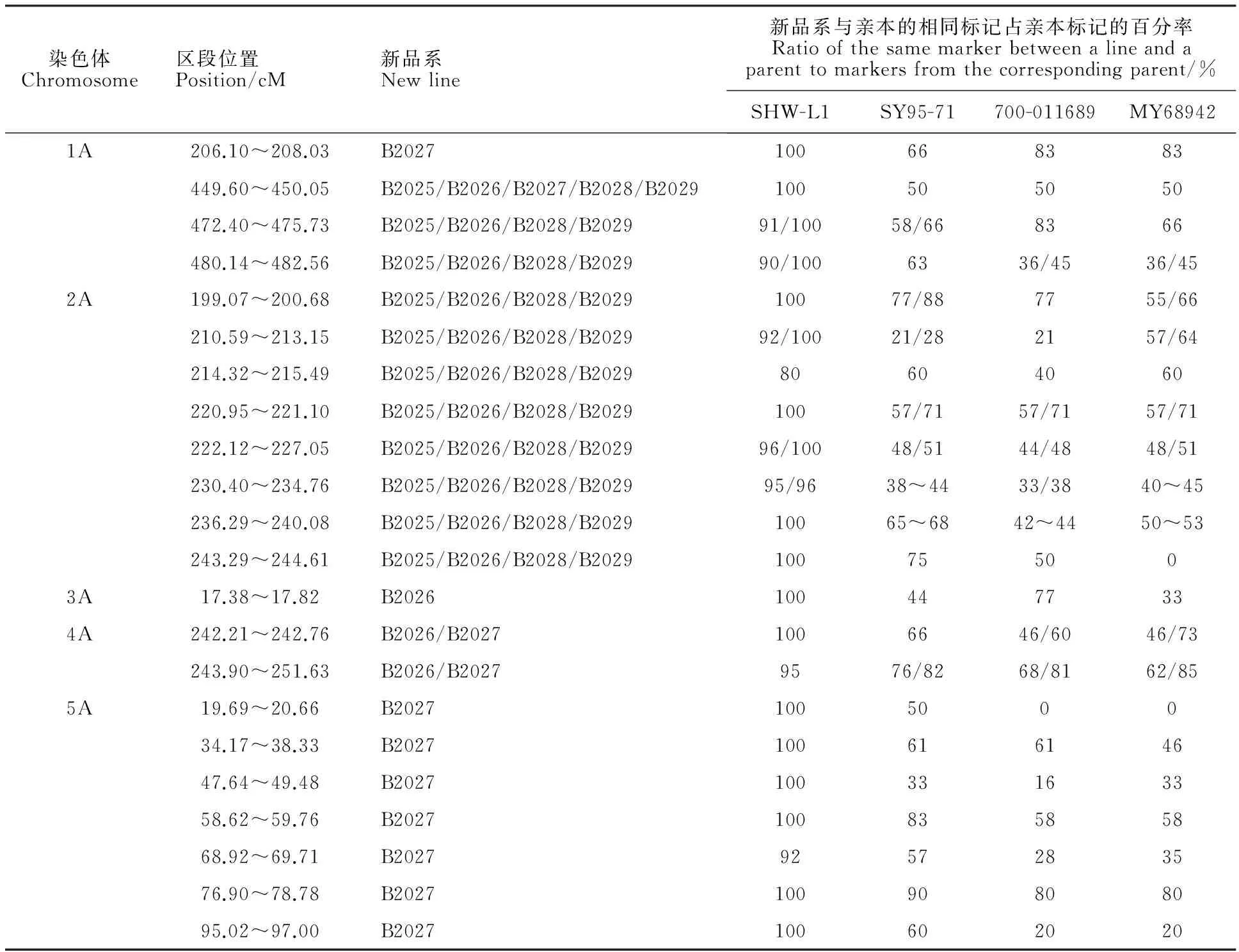
表4 源于合成小麦SHW-L1的染色体区段在小麦新品系中的分布Table 4 Chromosomal regions transferred from synthetic SHW-L1 into elite wheat lines
(续表4Continued table 4)
染色体Chromosome区段位置Position/cM新品系Newline新品系与亲本的相同标记占亲本标记的百分率Ratioofthesamemarkerbetweenalineandaparenttomarkersfromthecorrespondingparent/%SHW-L1SY95-71700-011689MY6894298.25~100.96B2027100836666105.76~106.91B2027100507575111.66~113.45B202792384653114.89~118.64B202794575242125.31~126.32B2027100402060132.12~132.31B202710080080136.73~137.53B20271006262756A10.34~14.15B2025/B2026/B2027/B2028/B202910087/9381/8768/7515.45~21.00B2025/B2026/B2027/B2028/B202992/945871~7781/8432.88~33.91B2025/B2028/B2029100836650188.27~193.76B2027776160687A9.82~19.94B2025/B2026/B2028/B202991/954545/4744~5321.11~26.82B2025/B2026/B2028/B202994/975666/6958/6130.37~32.57B2025/B2026/B2027/B2028/B202986/10059/8340/6740/5938.12~39.27B2025/B2026/B2028/B202991584141103.00~103.87B2026/B20291006666331B192.25~193.24B20261006633661B403.05~403.27B20251006633661B524.31~525.54B2025/B2027/B2028/B2029664133411B538.88~540.25B2025/B2027/B2028/B2029871225372B1.14~4.39B2027914540372B26.76~27.72B20271007575502B34.29~34.52B20271005050502B61.54~67.41B2027978280432B74.99~76.70B20271008888224B69.50~70.59B2025/B2028/B202980/907070704B81.21~81.41B20271006666664B96.62~98.90B20271004572815B6.86~8.28B2025/B20281003366665B11.50~12.72B2026/B20291007575755B16.29~16.99B2026/B20291008187505B22.17~23.34B2026/B202910061/6961765B24.83~25.72B2026/B2029100604005B29.35~30.53B20271008190905B35.15~35.47B2025/B2026/B202910072/8136905B48.60~52.21B2026/B2029946464645B53.89~58.65B2025/B2026/B202995/9766/6976765B63.09~63.15B2026/B2029100505025
(续表4Continued table 4)
染色体Chromosome区段位置Position/cM新品系Newline新品系与亲本的相同标记占亲本标记的百分率Ratioofthesamemarkerbetweenalineandaparenttomarkersfromthecorrespondingparent/%SHW-L1SY95-71700-011689MY689425B64.23~64.30B20291008060605B65.65~66.77B2026/B20291001428285B69.83~71.10B2026/B2029100575742/576B159.00~162.70B2025/B2026/B2027/B2028/B202972~7760~6510~199~187B156.92~157.79B20271007575751D27.22~29.44B2027876250621D50.92~51.46B20271005050502D24.15~26.07B20281008775872D111.64~114.54B20271007244442D182.48~184.52B2027835066665D21.14~21.42B20271005050505D31.67~33.21B20271007040305D34.77~35.70B2027917525335D61.93~62.79B202710050005D90.99~91.50B2027100500505D97.27~97.70B20271007525755D104.77~105.40B20271006633666D164.50~164.98B2027100505050
3 讨 论
普通小麦的A、B基因组供体(四倍体小麦)和D基因组供体(节节麦)为普通小麦育种提供了丰富的遗传基础[7-8,13-15]。以人工合成小麦为“桥梁”,可以通过同源重组方式,同时将四倍体小麦和节节麦的外源遗传物质导入到现有小麦遗传背景中。人工合成小麦在冬麦区的遗传改良实践已证明其在小麦育种中的重要利用价值[14-20]。
本研究中,利用人工合成小麦SHW-L1选育的5个小麦新品系在青海春麦区丰产性较好,表明人工合成小麦在春小麦产量育种中也有应用潜力。SHW-L1来自于具有多小穗的四倍体小麦AS2255与节节麦AS60的杂交。该杂交组合在不采用胚培的情况下,能够产生有生活力的杂种[23],且获得的单倍体杂种能够通过未减数配子的结合,实现染色体自动加倍[4,24]。节节麦AS60具有高分子量谷蛋白新亚基组合1Dx3.1t和1Dy11*t[25],它具有的光周期基因 Ppd-Dt1可能不存在于现有普通小麦群体中[26-27]。利用SHW-L1构建的遗传群体,发掘出了一些育种上有潜在利用价值的QTL位点[28- 34]。但是,该遗传群体存在许多野生不良性状,株系间的生育期、株高、分蘖等性状差异大,这可能影响QTL的鉴定结果,所发掘出的QTL在当前的小麦育种中的实际应用价值还有待于进一步的评价。
人工合成小麦SHW-L1植株高大,分蘖多,穗长较长,种子较大。但也存在着穗层极不整齐、生育期长、颖壳坚硬难以脱粒等不良野生性状。本研究涉及的5个SHW-L1改良新品系是SHW-L1与多个普通小麦优良品系杂交、回交后,再经多代系统选育而来。它们在青海表现出较优的农艺性状和较高的产量潜力。推测源于四倍体小麦和节节麦亲本的野生不良性状被选择淘汰,保留在新品系中的外源染色体区段可能对育种选择是有利的。因此,从中鉴定出导入的四倍体小麦和节节麦外源染色体区段,可为下一步的外源优良基因发掘及育种改良提供基础信息和材料。
本研究从人工合成改良品系中检测到78个外源染色体区段。其中,24个来源于四倍体小麦AABB基因组的外源染色体区段分布于3个以上的品系当中。位于1A、6A、7A、6B染色体上的5个区段存在于所分析的5个品系中,15个区段(1A/2个,1B/2个,2A/8个,7A/3个)分布于4个品系中,4个区段(4B、5B和6A)分布于3个品系中。因为本研究所分析的5个新品系来自于5个不同的F5单株后代,其F2~F4采用单株混合选择,所以推测这些分布于多个品系的外源染色体区段可能具有育种选择优势。这些共同存在于新品系中的外源染色体区段,可能具有重要的育种基因或基因簇,因此应是关注的重点。下一步,将深入评估这些外源染色体区段的遗传效应和育种价值,并从中发掘出优良新基因。同时,可利用它们进行外源染色体区段聚合育种,以提高普通小麦的遗传多样性。
在现有小麦A、B和D三个基因组中,小麦的D染色体组的遗传多样性最低。其中一个重要原因在于,普通小麦起源后,四倍体小麦与普通小麦容易相互杂交,它们存在遗传物质交流[35]。但是,节节麦通常很难与普通小麦天然杂交,节节麦与小麦的D基因组间缺乏遗传交流。本研究检测到D基因组1D、2D、5D、6D染色体上的节节麦外源区段13个,其中12个来自于小麦新品系B2027。该品系可作为丰富小麦D基因组遗传多样性的育种亲本材料予以重点关注。
[1]KIHARA H.Discovery of the DD-analyser,one of the ancestors ofTriticumvulgare[J].AgricutureandHorticulture,1944,19:889-890.
[2]MCFADDEN E S,SEARS E R.The artificial synthesis ofTriticumspleta[J].RecordsGeneticsSocietyofAmerica,1946,13:26-27.
[3]MATSUOKA Y.Evolution of polyploidTriticumwheats under cultivation:The role of domestication,natural hybridization and allopolyploid speciation in their diversification [J].PlantCellPhysiology,2011,52:750-764.
[4]HAO M,LUO J,ZENG D,etal. QTug.sau-3B is a major quantitative trait locus for wheat hexaploidization [J].G3:Genes/Genomes/Genetics,2014,4:1943-1953.
[5]HAWKESFORD M J,ARAUS J L,PARK R,etal.Prospects of doubling global wheat yields [J].FoodandEnergySecurity,2013,2(1):34-48.
[6]TILMAN D,BALZER C,HILL J,etal.Global food demand and the sustainable intensification of agriculture [J].ProceedingsoftheNationalAcademyofSciences,2011,108:20260-20264.
[7]OBONNAYA F C,HALLORAN G M,LAGUDAH E S.D Genome of Wheat-60 Years on from Kihara,Sears and McFadden [M].Yokohama:Kihara Memorial Foundation for the Advancement of Life Sciences,2005:205-220.
[8]REIF J C,ZHANG P,DREISIGACKER S,etal.Wheat genetic diversity trends during domestication and breeding [J].TheoreticalandAppliedGenetics,2005,110:859-864.
[9]JIA J,ZHAO S,KONG X,etal.Aegilopstauschiidraft genome sequence reveals a gene repertoire for wheat adaptation [J].Nature,2013,496:91-95.
[10]MUJEEB-KAZI A,ROSAS V,ROLDAN S.Conservation of the genetic variation ofTriticumtauschii(Coss.) Schmalh.(Aegilopssquarrosaauct.Non L.)in synthetic hexaploidwheats (T.turgidumL.×T.tauschii; 2n=6x=42,AABBDD) and its potential utilization for wheat improvement [J].GeneticResourcesandCropEvolution,1996,43:129-134.
[11]RASHID M R,MUHAMMAD B,SHOAIB U R,etal.Sythetics wheat;A new hope for the hungry world[J].AsianJournalofAgricultureandBiology,2013,1(2):91-94.
[12]HOISINGTON D,KHAIRALLAH M,REEVES T,etal.Plant genetic resources:What can they contribute toward increased crop productivity?[J].ProceedingsoftheNationaloftheUntitedstatesofAmerica,1999,96:5937-5943.
[13]WARBURTON M L,CROSSA J,FRANCO J,etal.Bringing wild relatives back into the family:Recovering genetic diversity in CIMMYT improved wheat germplasm[J].Euphytica,2006,149:289-301.
[14]VAN GINKEL M,OGBONNAYA F.Novel genetic diversity from synthetic wheats in breeding cultivars for changing production conditions [J].FieldCropsResearch,2007,104:86-94.
[16]邹裕春,杨武云,朱华忠,等.CIMMYT种质及育种技术在四川小麦品质改良中的利用[J].西南农业学报,2007,2(20):183-190.
ZOU Y C,YANG W Y,ZHU H Z,etal.Utilization of CIMMYT germplasm and breeding technologies in wheat improvement in Sichuan [J].SouthwestChinaJournalofAgriculturalSciences,2007,2(20):183-190.
[17]YANG W,LIU D,LI J,etal.Synthetic hexaploid wheat and its utilization for wheat genetic improvement in China[J].JournalofGeneticsandGenomics,2009,36:539-546.
[18]LI J,WAN H,YANG W.Synthetic hexaploid wheat enhances variation and adaptive evolution of bread wheat in breeding processes [J].JournalofSystematicsandEvolution,2014,52:735-742.
[19]LI J,WEI H T,HU X R,etal.Identification of a high-yield introgression locus in Chuanmai 42 inherited from synthetic hexaploid wheat [J].ActaAgronomicaSinica,2011,37:255-262.
[20]汤永禄,李朝苏,吴晓丽,等.人工合成小麦衍生品种的物质积累、冠层结构及群体光合特性[J].中国农业科学,2014,47(5):844-855.
TANG Y L,LI C S,WU X L,etal.Accumulation of dry matter,canopy structure and photosynthesis of synthetic hexaploid wheat-derived high yielding varieties grown in Sichuan basin,China [J].ScientiaAgriulturaSinica,2014,47(5):844-855.
[21]ZHANG L,LIU D,YAN Z,etal.Rapid changes of microsatellite flanking sequence in the allopolyploidization of new synthesized hexaploid wheat [J].ScienceinChinaSeriesC:LifeSciences,2004,47:553-561.
[22]JACCOUD D,PENG K,FEINSTEIN D,etal.Diversity arrays:A solid state technology for sequence information independent genotyping [J].NucleicAcidsReearchs,2001,29:e25.
[23]LIU D C,LAN X J,YANG Z J,etal.A uniqueAegilopstauschiigenotype needless to embryo rescue in cross with wheat [J].ActaBotanicaSinica,2012,44:508-613.
[24]ZHANG L Q,YEN Y,ZHENG Y L,etal.Meiotic restriction in emmer wheat is controlled by one or more nuclear genes that continue to function in derived lines [J].SexualPlantReproduction,2007,20:159-166.
[25]CHEN W,FAN X,ZHANG B,etal.Novel and ancient HMW glutenin genes fromAegilopstauschiiand their phylogenetic positions [J].GeneticResourcesandCropEvoutionl,2012,59:1649-1657.
[26]HUANG L,WANG Q,ZHANG L,etal.Haplotype variations of gene Ppd-D1 inAegilopstauschiiand their implications on wheat origin [J].GeneticResourcesandCropEvolution,2012,59:1027-1032.
[27]XIANG Z,ZHANG L,NING S,etal.Evaluation ofAegilopstauschiifor heading date and its gene location in a re-synthesized hexaploid wheat [J].AgriculturalSciencesinChina,2009,8:1-7.
[28]ZHANG L,LUO J,HAO M,etal.Genetic map ofTriticumturgidumbased on a hexaploid wheat population without genetic recombination for D genome [J].BMCGenetics,2012,13:69.
[29]YANG J,LIU Y,PU Z,etal.Molecular characterization of high pI α-amylase and its expression QTL analysis in synthetic wheat RILs [J].MolecularBreeding,2014,34:1075-1085.
[30]PU Z N,YU M,HE Q Y,etal.Quantitative trait loci associated with micronutrient concentrations in two recombinant inbred wheat lines [J].JournalofIntegrativeAgriculture,2014,13:2322-2329.
[31]YU M,MAO S L,CHEN G Y,etal.QTLs for waterlogging tolerance at germination and seedling stages in population of recombinant inbred lines derived from a cross between synthetic and cultivated wheat genotypes [J].JournalofIntegrativeAgriculture,2014,13:31-39.
[32]YU M,CHEN G Y,PU Z E,etal.Quantitative trait locus mapping for growth duration and its timing components in wheat [J].MolecularBreeding,2015,35:44.
[33]YU M,MAO S L,CHEN G Y,etal.QTLs for uppermost internode and spike length in two wheat RIL populations and their affect upon plant height at an individual QTL level [J].Euphytica,2014,200:95-108.
[34]YU M,CHEN G Y,ZHANG L Q,etal.QTL mapping for important agronomic traits in synthetic hexaploid wheat derived fromAegiliopstauschiissp.tauschii[J].JournalofIntegrativeAgriculture,2014,13:1835-1844.
[35]DVORAK J,AKHUNOV E D,AKHUNOV A R,etal.Molecular characterization of a diagnostic DNA marker for domesticated tetraploid wheat provides evidence for gene flow from wild tetraploid wheat to hexaploid wheat [J].MolecularBiologyandEvolution,2006,23:1386-1396.
Chromosomal Segments in Elite Wheat Lines Derived from Synthetic Hexaploid Wheat
WANG Hongxia1,3,CHEN Wenjie1,CAO Dong1,3,NING Shunzong2,LIU Baolong1,ZHANG Bo1,ZHANG Lianquan2,LIU Dengcai1,2,ZHANG Huaigang1
(1.Key Laboratory of Adaptation and Evolution of Plateau Biota,Northwest Institute of Plateau Biology,Chinese Academy of Sciences,Xining,Qinghai 810001,China; 2.Triticeae Research Institute,Sichuan Agricultural University,Chengdu,Sichuan 611130,China; 3.University of Chinese Academy of Sciences,Beijing 100049,China)
The genome donor species of common wheat,TriticumturgidumandAegilopstauschii,provide a lot of genetic variations for common wheat improvement. Genetic diversity of the two alien species can be introgressed into common wheat via synthetic hexaploid wheat as a bridge. Five spring wheat lines derived from synthetic hexaploid wheat SHW-L1 had better agronomic characters than Gaoyuan 448 and exhibited a high yield potential in Qinghai province. We suggested that the wild and disadvantage traits of alien species were eliminated during the breeding selection process,but the desirable alien chromosomal segments were kept. To find out the distribution of alien chromosomal segments in the five lines,we analyzed 11 660 polymorphic DArTseq markers with known chromosomal locations. The results showed that 78 alien chromosomal segments were found (65 donated from A and B genomes ofT.turgidumand 13 from D genome ofAe.tauschii). Out of them,24 segments existed in at least three wheat lines,most of which were from A genome,including eight from 2A,four from 7A,three from 1A,and three from 6A. Since these lines were selected by mixture selection,these common alien segments shared by different lines probably harbor key genes or gene clusters favor to breeding selection. Therefore,they are the main points in the future study.
Chromosomal segment; Synthetic hexaploid wheat; DArTseq marker
2016-04-10
2016-06-14
中国科学院战略性A类先导科技专项子课题(XDA08030106);青海省自然科学基金项目(2013-Z-942Q)
E-mail:zj511102365@163.com(王宏霞);cwj60905@163.com(陈文杰,与第一作者同等贡献)
刘登才(E-mail:dcliu7@yahoo.com);张怀刚(E-mail:hgzhang@nwipb.cas.cn)
S512.1;S330
A
1009-1041(2016)09-1130-09
网络出版时间:2016-08-31
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20160831.1649.004.html

