基于遗传算法的随机森林模型在特征基因筛选中的应用*
赵发林 张 涛 李 康
基于遗传算法的随机森林模型在特征基因筛选中的应用*
赵发林1张涛2李康3△
【提要】目的探索基于遗传算法的随机森林模型在特征基因筛选中的效果和特点。方法通过本文构建的基于遗传算法的随机森林模型(GARF)对真实基因数据和模拟数据进行特征基因筛选,以筛选后基因进行判别分析,计算ROC曲线下面积AUC值,同时观察GARF方法对模拟实验中预设的差异基因排序结果。结果对真实基因数据和模拟数据的分析结果均显示,采用GARF方法筛选得到的特征基因建立判别模型能获得更好的分类效果,在模拟实验中与随机森林相比能将预设的差异基因排在更靠前的位置。结论GARF方法能够有效地用于基因芯片数据特征基因筛选,在FDR控制上具备潜力,具有研究价值。
随机森林遗传算法特征基因筛选
特征基因筛选是基因组学研究的主要目的之一,不仅要求能够通过筛选到的特征变量对样本进行有效分类,而且要保证筛选得到的特征变量集有较小的假发现率(false discovery rate,FDR),否则将极大增加生物学验证的工作量,浪费大量资源,甚至无法实现生物学验证。
近年来,随机森林(random forests,RF)被广泛应用于高维数据分析并取得了良好的效果。RF可以在对样本分类的同时给出变量重要性评分(variable importance measures,VIMs)作为特征筛选的依据。但当变量个数非常多时,其中包含的大量对分类无作用的“噪声”变量对分类效果仍会有较大影响,使VIMs不稳定,真正对分类有作用的变量很可能在筛选得到的变量子集中排序靠后,甚至无法被选入[1]。遗传算法(genetic algorithm,GA)是按照随机搜索策略进行特征筛选的,可以由不同的染色体提供多样化的特征筛选结果,采用适当的GA与RF相结合将有可能降低“噪声”对筛选结果的影响,同时降低FDR水平。
本研究期望给出一种基于GA的RF搜索策略GARF,用于高维数据的特征筛选。GARF在遗传过程中加入基于VIMs的启发式变量搜索方法,能够降低噪声变量对分类的影响,并采用基于Permutation的方法确定最终入选模型的变量筛选界值,既能够避免人为确定筛选界值的主观性,又能够解决单纯采用多变量启发式搜索策略存在的变量竞争问题。
原理与方法
1.随机森林的基本原理
随机森林由Leo Breiman(2001)提出,它通过自助法(bootstrap)重采样技术,从原始训练样本集N中有放回地重复随机抽取b个样本生成新的训练自助样本集合,然后根据自助样本集生成b个分类树组成随机森林,新数据的分类结果按分类树投票多少形成的分数而定。随机森林的主要特点是在处理高维数据时不会产生过拟合现象,在分类的同时能够给出变量的重要性评分,依据该评分,可以筛选出对分类起重要作用的变量[2-4]。
2.遗传算法的基本原理
遗传算法由Michigan大学的J.Holland于1975年提出,是一种借鉴生物界自然选择和生物体遗传机制的随机搜索算法,其基本原理是进化机制和自然选择法则[5-6]。遗传算法的特点是采用简单编码技术表示复杂结构,并通对编码的遗传操作——复制(reproduction)、交叉(crossover)和变异(mutation)产生备择解集,通过优胜劣汰的选择机制进行导向性搜索。进化算法不需要了解问题的全部特征,就可以通过体现进化机制的进化过程完成问题求解。
3.GARF算法的基本原理
GARF采用RF模型对变量在样本分类中的作用进行评价,以permutation方法确定特征筛选界值作为最终确定特征变量的依据。为减少噪声变量对RF变量评价结果的干扰,每个RF模型仅包含由GA算法选取的部分变量,并且在遗传过程中加入了变量筛选步骤以进一步降低噪声变量影响。GA算法的启发特性使对分类作用较强的变量有更多的机会被评价,从而增强RF评价结果的稳定性;同时,GA算法的“变异”过程使搜索结果向一定方向收敛的同时具备较强的多样性,使分类作用较弱的变量也可以获得一定的被评价机会。GA算法的上述两点特性既保证了对变量评价的深度,也兼顾了评价的广度。遗传过程内的变量筛选中,采用permutation方法获得组间无差异变量重要性评分的经验分布,根据该经验分布自适应确定变量筛选界值。
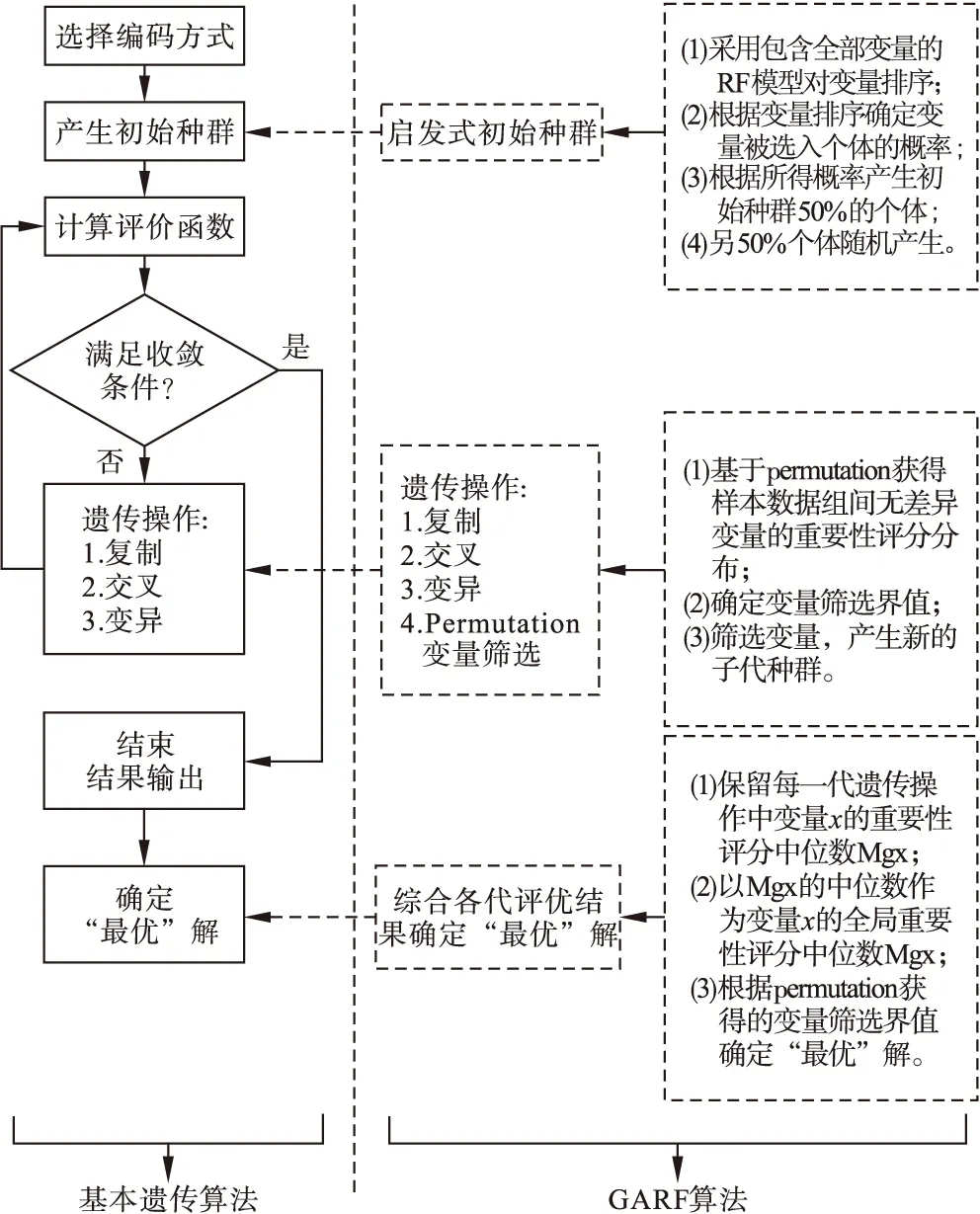
图1 GARF算法过程流程图
4.GARF算法实现
设数据分为A和B两类,样本量为N,变量个数为m,遗传算法每一代种群包含M条染色体,采用二进制编码方式,遗传过程中保留最优染色体的概率设为rE,变异率设为rM,变异中基因由0突变为1的概率为r0to1,算法的收敛条件为传代数达到G代,则GARF算法步骤如下:
(1)采用二进制编码方式,每一代种群均为由0,1构成的M行k列的矩阵。0表示对应位置的基因不表达(变量未被选入模型),1表示对应位置的基因表达(变量被选入模型)。
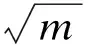
(3)以每条染色体中包含的变量建立随机森林模型,获取变量重要性评分作为自适应降噪的依据。
(4)自适应降噪:①计算每条染色体中包含的基因个数;②根据染色体中基因个数自适应选择相应的变量重要性评分筛选界值,剔除变量重要性评分小于该界值的变量;③形成降噪后的新种群。
(5)对降噪后的染色体进行评价:①用新种群中的染色体建立随机森林模型,获取森林对袋外数据分类的投票结果;②基于随机森林对袋外数据的投票计算模型判别结果的ROC曲线下面积AUC。AUC值在0.5~1.0之间,以1-AUC值作为对染色体的评价函数。
(6)获取降噪后随机森林模型对变量的评价结果,作为最终识别特征变量的依据。对变量在整个种群中获得的变量重要性评分求中位数,作为变量在这一代获得的评价结果。如某变量共获得r次评价,则对这r个变量重要性评分求中位数,如果该变量在这一代未获得评价则此处记为缺失。
(7)根据评价函数值由小到大的顺序对染色体进行排序,按设定的比例将排序靠前的部分染色体直接复制到子代种群中,不进行任何交叉和变异。子代中的其余染色体经由交叉和变异产生。
(8)根据染色体评价函数排序确定父代中的每条染色体被选中参与交叉操作的概率,保证评价函数较优的染色体有更高的概率被选中与其他染色体进行交叉。
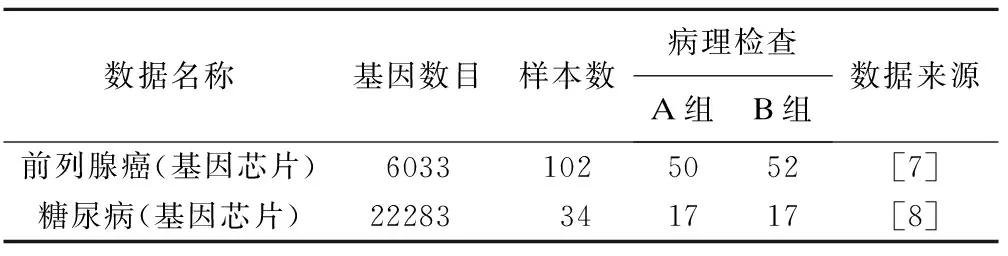
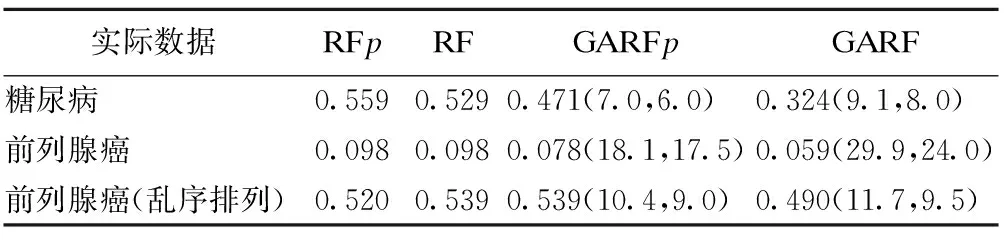
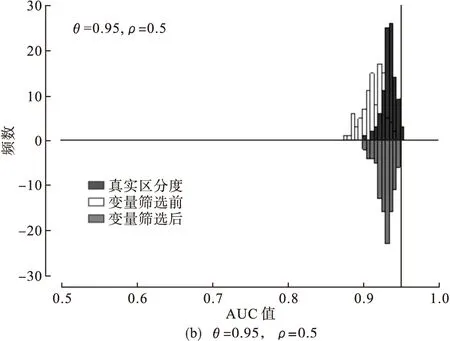
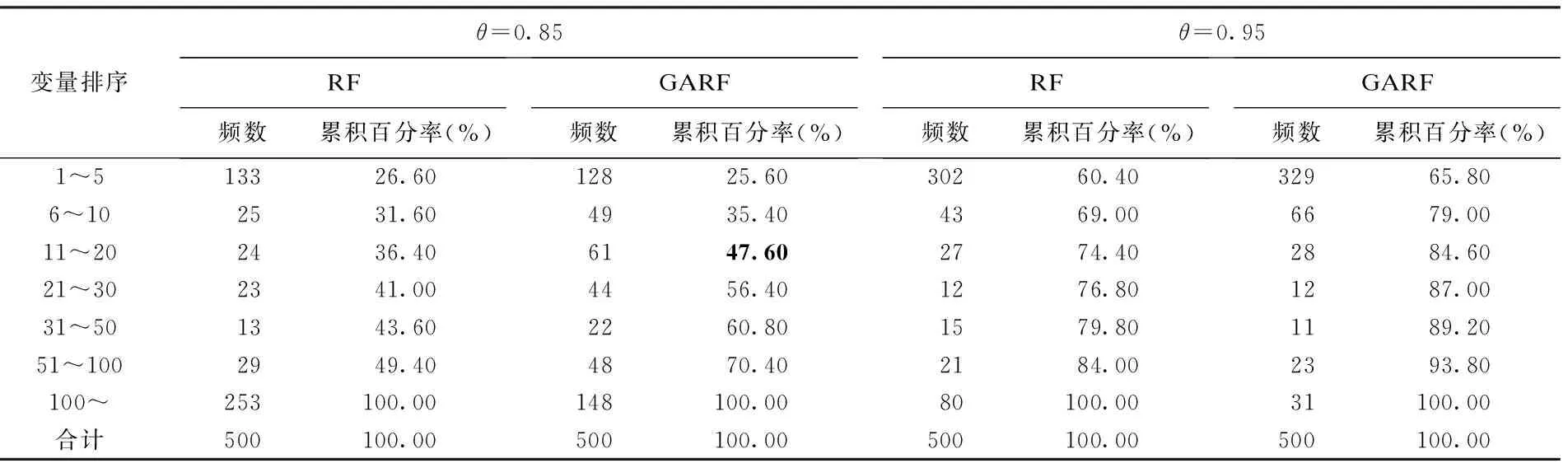
(9)交叉:由父代中按一定概率选取两条染色体,记为C1和C2,并从1-m的整数中随机抽取一个整数mC作为交叉点,当mC=1时以C1作为新产生的染色体,当mC=m时以C2作为新产生的染色体,当1 (10)变异:对由交叉产生的新染色体进行变异操作,如果染色体中的基因取值为0,则从0~1的均匀分布中随机产生一个数mM,与预先设定的突变率rM比较,如果mM (11)将步骤(7)与步骤(8)~步骤(10)产生的染色体合并,产生子代种群。 (12)重复上述步骤(3)~步骤(11)的操作,循环至第G代停止。 (13)将每一代中步骤(6)获得的变量评价结果取中位数,作为GARF特征筛选方法对变量的最终评价,记为VIgene。 (14)确定最终筛选界值,识别特征变量:①计算最后一代种群中每条染色体中包含的基因个数;②取其平均值记为Mgene,作为permutation抽样参数;③从数据集中随机抽取Mgene个变量,将分类标签随机打乱,建立随机森林模型,记录变量重要性评分,重复进行2000/Mgene次,共获得2000个变量重要性评分;④以上述2000个变量重要性评出的百分位数P95或P99作为GARF算法特征筛选界值,如VIgene大于该界值则将该变量识别为特征变量。 (15)结束。 1.数据来源 实例分析中,采用对未知分类样本的判别效果作为特征筛选结果的评价指标。本研究首先用GARF方法对真实基因芯片数据进行特征筛选,采用特征筛选结果建立随机森林分类模型,并与直接使用随机森林模型的结果进行对比。研究中分析的基因芯片数据有前列腺癌基因芯片数据和糖尿病基因芯片数据,数据由公开数据库获得。对数据的判别效果采用10-fold交叉验证评价,记录错分率。前列腺癌分为有病(A组)和无病(B组)两类,糖尿病分为空腹血糖正常组(A组)和糖尿病组(B组)。比较中采用的高维组学实验数据的基本信息如表1。 表1 基因芯片数据的样本分布情况 2.分析结果 由表2可见,经过GARF特征筛选,RF模型的判别能力增强。考虑到实际中经常采用单变量方法进行变量预筛选,我们同时采用了基于t检验的预筛选方法。首先根据t检验统计量绝对值大小排序,取绝对值较大的前2000个变量,然后分别采用随机森林、GARF法进行分析。结果显示,采用全部变量进行GARF特征筛选的效果要优于预筛选之后的2000个变量的分析结果。为考察各种方法是否会产生过拟合现象,我们还将前列腺癌数据的分类标签打乱,产生新的实际上不包含分类信息的数据集,采用上述方法对该数据集进行分析。分析结果显示,各种方法分类结果均很差,没有出现过拟合现象。 表2 随机森林及GARF对六个数据集分析的结果10-fold交叉验证错分率) *:RF为随机森林模型,后缀“p”表示采用基于t检验统计量对数据进行预筛选后再采用相应模型分析;括号内为交叉验证过程中分类模型包含的变量个数的均数和中位数。随机森林模型采用全部变量建模,基于预筛选的随机森林模型采用预筛选得到的2000个变量建模。 1.模拟实验条件设置 (1)模拟数据1:该部分模拟数据用于考察GARF特征筛选后的判别效果。设定训练样本为N=60,其中A类样本例数nA=30,B类样本例数nB=30;测试样本1000例,两类各500例。样本中含有5个有差异的变量,两类间的真实区分度用ROC曲线下面积AUC衡量,分别设AUC=0.85、AUC=0.95;ρ表示变量间的相关系数,研究中设定ρ=0.5;差异变量和无差异变量均服从正态分布;无差异变量的个数为2000,服从标准正态分布。模拟实验数据重复产生,形成100个随机样本。 (2)模拟数据2:该部分模拟数据用于考察GARF法对预设的差异变量的识别能力。设定每一个模拟数据样本为N=60,其中两分类的样本例数分别为nA=30和nB=30,差异变量和无差异变量均服从正态分布。两类间的总真实区分度用ROC曲线下面积θ衡量,分别设θ=0.85、θ=0.95;样本中含有5个有差异的变量,分别用X1,X2,X3,X4,X5表示,其中X1和X2两个变量的相关系数ρ=0.9,其余3个变量的相关系数ρ=0。为简单起见,各变量的方差和均数设为相同,方差σ2=1,均数μi(i=1,2,3,4,5)则根据θ值用编制的程序求出。在此基础上,加入2000个无差异变量。无差异变量来自实际基因芯片数据,即由前列腺癌基因芯片数据的两个分类中随机抽取30例样本,同时随机抽取2000个变量,打乱样本的分类标签。模拟实验数据重复产生,形成100个随机样本。 2.模拟实验结果 (1)GARF特征筛选后的判别效果 由表3可见,进行特征筛选后,随机森林模型只需用少量的变量就能达到很好的判别效果;尽管设定的类间区分度不同,但分类模型对数据判别分类呈现的趋势一致;设定的类间区分度值越高,则特征筛选后获得的“最优”分类模型的判别效果越接近实际区分度。 *:判别模型AUC估计的中位数;**斜线前为设定的差异变量被识别为特征变量的个数的中位数,斜线后为被识别为特征变量的变量个数的中位数。 图2给出了变量筛选前后ROC曲线下面积AUC估计值的频数分布情况。为便于分析和比较,同时给出了在不包含无差异变量干扰的情况下,随机森林算法AUC估计值的频数分布情况。结果显示,GARF特征筛选后,ROC曲线下面积均有明显的改善;当设定的AUC值较大时,变量筛选后的AUC频数分布与理想情况下(仅包含差异变量)的AUC频数分布几乎重合,得到了非常理想的结果。使用GARF算法进行变量筛选,有利于简化判别模型和增强预测效果。 图2 变量筛选前后及仅含差异变量的AUC频数变化情况 (2)GARF差异变量的识别结果 对变量的排序结果见表4,与RF模型相比GARF能将预先设定的差异变量排在更靠前的位置。即便在组间差异较小的情况下(θ=0.85),预设的差异变量被排在前20位的累积频率也接近50%。 表4 差异变量在RF和GARF分析结果中的排序分布情况 1.尽管随机森林具有较强的抗噪声能力,但基因芯片数据中的高噪声仍对其判别结果产生较大影响,通过GARF方法进行特征筛选后,根据筛选结果建立的判别模型判别能力有较大提升。 2.对基于前列腺癌基因芯片数据产生的分类标签乱序数据的分析结果表明,RF模型本身有良好的防止过拟合能力,同属于基于RF模型迭代算法的GARF法也没有出现过拟合。 3.GARF特征筛选方法无需进行预筛选,采用全部变量进行特征筛选的效果更理想。预筛选的主要作用是减少变量个数,通常预筛选采用单变量分析方法(如t检验、SAM法等),筛选结果不能体现变量间的相互作用。 4.本研究中没有直接考察GARF特征筛选方法的FDR,但通过比较GARF与随机森林对预设差异变量的排序结果可见,GARF能够将预设的差异变量排在更靠前的位置,即如果采用相同的筛选界值,GARF特征筛选结果更有可能获得较小的FDR。FDR的控制在生物学研究中十分重要,如果研究的主要目的是生物标志物提取,使用GARF算法是更有效的。 [1]Amaratunga D,Cabrera J,Lee YS.Enriched random forests.Bioinformatics,2008,24(18):2010-2014. [2]Cutler A,Stevens JR.Random forests for microarrays.Methods Enzymol,2006,411:422-32. [3]武晓岩,李康.基因表达数据判别分析的随机森林方法.中国卫生统计,2006,6:491-494. [4]李贞子,张涛,武晓岩,等.随机森林回归分析及在代谢调控关系研究中的应用.中国卫生统计,2012,29(2):158-160,163. [5]王小平.遗传算法——理论、应用与软件实现.西安:西安交通大学出版社,2002:1-4. [6]Yang J,Honavar V.Feature Subset Selection Using a Genetic Algorithm.IEEE Intelligent Systems,1998,13(2):44-49. [7]Diaz-Uriarte R,de Andres SA.Gene selection and classification of microarray data using random forest.BMC Bioinformatics,2006,7:3. [8]Mootha VK,Lindgren CM,Eriksson KF,et al.PGC-1alpha-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes.Nat Genet,2003,34(3):267-73. (责任编辑:郭海强) An Optimized Random Forest Based on Genetic Algorithm and its Application to Feature Selection for Gene Data Zhao Falin,Zhang Tao,Li Kang (Harbin Medical University(150081),Harbin) ObjectiveTo explore the effects and properties of an optimized random forest based on genetic algorithm(GARF)on feature selection.MethodsWe used GARF to select significant genes both on simulated data and real gene data then built discriminative models with significant genes.The area under the ROC curve was calculated to evaluate the discrimination performance.The ranks of significant genes assigned in simulated data were considered,too.ResultsThe discrimination performance based on the significant genes selected by GARF is better than on original data and selected by random forest.ConclusionThe proposed GARF method is suitable to analyze high dimensional data for feature selection and shows proper potency for controlling FDR. Random forest;Genetic algorithm;Feature selection 浙江省自然科学基金项目(LQ12H26002),杭州师范大学科研启动基金项目(2011QDL12) 李康,E-mail:likang@ems.hrbmu.edu.cn 1.杭州师范大学医学院健康管理系(310036) 2.山东大学公共卫生学院生物统计学系 3.哈尔滨医科大学卫生统计学教研室实例应用
模拟实验

讨 论

