基于CRISPR对大肠埃希菌O157∶H7的检测
梁文娟,张荣光,段广才,王颖芳,洪丽娟,
张 冰1,王鹏飞1,郗园林1,杨海燕1
(1. 郑州大学公共卫生学院流行病与卫生统计学系,河南郑州 450001;2. 新乡医学院分子诊断与医学检验技术河南省协同创新中心,河南新乡 453003;3. 河南科技大学预防医学教研室,河南洛阳 471023)
◇技术方法研究◇
基于CRISPR对大肠埃希菌O157∶H7的检测
梁文娟1,2,张荣光1,段广才1,2,王颖芳3,洪丽娟1,
张冰1,王鹏飞1,郗园林1,杨海燕1
(1. 郑州大学公共卫生学院流行病与卫生统计学系,河南郑州450001;2. 新乡医学院分子诊断与医学检验技术河南省协同创新中心,河南新乡453003;3. 河南科技大学预防医学教研室,河南洛阳471023)
目的基于成簇的规律间隔短回文重复序列(CRISPR),建立对大肠埃希菌O157∶H7的新型检测方法。方法应用PCR扩增实验室保存的443株肠道细菌(310株非O157∶H7大肠埃希菌、35株大肠埃希菌O157∶H7、89株志贺菌和9株沙门菌)的CRISPR1和CRISPR2,并将PCR产物测序;提取CRISPRdatabase数据库中(标准法)100株肠道细菌(47株非O157∶H7大肠埃希菌、5株大肠埃希菌O157∶H7、9株志贺菌和39株沙门菌)的CRISPR1和CRISPR2序列。使用CRISPRFinder在线软件分析PCR产物测序序列和CRISPRdatabase数据库的CRISPR序列。ClustalX软件进行间隔序列比对。比较标准法和PCR扩增CRISPR两种方法检测大肠埃希菌O157∶H7的一致性。结果共分析543株肠道细菌,其中75.6%的非O157∶H7大肠埃希菌、75.5%志贺菌、91.7%沙门菌和95%O157∶H7大肠埃希菌含有CRISPR1,其间隔序列数目为3~26、2~9、2~32、3。57.1%的非O157∶H7大肠埃希菌、77.6%志贺菌、85.4%沙门菌和100%O157∶H7大肠埃希菌含有CRISPR2,其间隔序列数目为1~20、1~6、2~27、1或4个。95%的O157∶H7大肠埃希菌的CRISPR1和90%CRISPR2分别含有3条独特间隔序列(S1-1,S1-2,S1-3)和1条独特间隔序列(S2-1)。间隔序列比对结果显示,S1-1+S1-2+S1-3和S2-1检测O157∶H7大肠埃希菌的特异性分别是100%和99.6%。标准法检测和PCR扩增CRISPR1和CRISPR2检测大肠埃希菌O157∶H7的一致性分别达99.6%和99.3%。基于CRISPR检测大肠埃希菌O157∶H7在模拟样品中的应用,结果显示在原样品大肠埃希菌O157∶H7浓度约2.3CFU/mL时,经12h增菌后即能检测出来。大肠埃希菌O157∶H7聚类分析显示,40株O157∶H7大肠埃希菌可分为3类。结论基于CRISPR的大肠埃希菌O157∶H7的检测方法,可以作为监测大肠埃希菌O157∶H7感染和高毒株大肠埃希菌O157∶H7有价值的流行病学工具。
大肠埃希菌O157∶H7;成簇的规律间隔短回文重复序列(CRISPR);聚合酶链反应;检测
大肠埃希菌O157∶H7是大肠埃希菌中一个重要的致病血清型。O157∶H7菌株感染不仅可引起出血性结肠炎、阑尾炎、食管狭窄和结肠穿孔等严重胃肠道并发症[1],而且在儿童和老年人群中还可引起溶血性尿路综合征(hemolyticuremicsyndrome,HUS)和血栓性血小板紫癜(thromboticthrombocytopenicpurpura,TTP)等并发症[2],严重时可致肾衰竭而死亡。目前大肠埃希菌O157∶H7检测方法主要有标准法检测、免疫学检测和分子生物学检测。分子生物学方法主要以PCR检测方法为主,具有简便、快速、敏感的特点;通常以特异性的编码基因O157脂多糖抗原基因(rfbE)、鞭毛抗原H7基因(flic)、志贺样毒素基因(stx)、溶血素基因(hly)、粘附素基因(eaeA)等作为靶基因[3];PCR扩增鉴定时,需要同时扩增rfbE和flic,而毒力因子基因stx、hly、eaeA只能作为辅助鉴定因子。
成簇的规律间隔短回文重复序列(ClusteredRegularlyInterspacedShortPalindromicRepeats,CRISPR)主要由重复序列(repeat)和间隔序列(spacer)等组成;其与CRISPR相关基因(CRISPR-associatedgenes,Cas)共同构成CRISPR/Cas系统[4]。这一系统是近年来发现的细菌处理噬菌体[5]、质粒[6]等外源遗传物质的原核生物免疫系统,广泛分布于细菌和古细菌基因组中。一个细菌中可以有多个CRISPR位点,在每个CRISPR位点中重复序列几乎完全一致;而spacer高度可变。目前,普遍认为spacer来源于外源噬菌体和质粒等可移动遗传因子[7],是细菌进行免疫识别的核心元件;cas可表达具有多种酶功能的Cas蛋白,从而发挥CRISPR/Cas系统的作用[8]。本课题组前期对CRISPR/Cas在志贺菌中的分布特征[9]及其与毒力[10]、耐药关系[11]等进行了研究,结果显示CRISPR中的spacer对于不同菌株具有一定的识别意义。
本研究通过对实验室保存和CRISPRdatabase数据库的大肠埃希菌(包括40株大肠埃希菌O157∶H7)、志贺菌和沙门菌的CRISPR位点进行检测与分析,探讨大肠埃希菌O157∶H7的CRISPR分布特征并建立一种基于CRISPR的大肠埃希菌O157∶H7细菌的检测方法,评价其特异性和灵敏性,并比较标准法和PCR扩增CRISPR检测大肠埃希菌O157∶H7的一致性。
1 材料与方法
1.1主要仪器和试剂酵母浸粉、胰蛋白胨(英国Oxoid公司);琼脂粉(Sigma公司);琼脂糖(BIOWEST公司进口分装);大肠埃希菌O157胶体金检测试剂条盒(郑州万泰);大肠埃希菌O157∶H7诊断血清(宁波天润);API20E生化鉴定板条(法国梅里埃);DNA试剂提取盒(上海莱枫生物科技有限公司)。PTC-100型基因体外扩增仪(MJRESEARCH公司);DYY-8C型稳压稳流定时电泳仪(北京六一仪器厂);Genesnap图像扫描仪(美国Syngene公司)。各种固体培养基及液体培养基在本实验室按配方配制。
1.2菌株共分析肠道细菌543株,其中本实验室细菌443株和CRISPRdatabase数据库(http://crispr.u-psud.fr/crispr/)细菌100株。菌株型分别为:O157∶H7大肠埃希菌40株(实验室35株,数据库5株),非O157∶H7大肠埃希菌357株(实验室310株,数据库47株),志贺菌98株(实验室89株,数据库9株),沙门菌48株(实验室9株,数据库39株)。所有实验室菌株均经过API20E生化鉴定条检定。
1.3实验室菌株CRISPR1和CRISPR2的检测及测序DNA模板制备时,大肠埃希菌采用煮沸法提取DNA,志贺菌和沙门菌根据试剂盒提取基因组DNA。
PCR扩增检测O157∶H7大肠埃希菌、非O157∶H7大肠埃希菌[12]、志贺菌[9]和沙门菌[9]的CRISPR1和CRISPR2的引物序列、扩增条件、体系均参考相应文献。所有引物(表1)由北京赛百盛基因技术有限公司合成。将CRISPR1和CRISPR2的PCR扩增产物经20g/L琼脂糖凝胶电泳后,由上海生工生物有限公司测序。
表1CRISPR引物序列
Tab.1TheprimersofCRISPRforPCR

CRISPR引物引物序列(5'~3')产物长度(bp)大肠埃希菌CRISPR1FGTTATGCGGATAATGCTACCRCGTAYYCCGGTRGATTTGGA497~1829大肠埃希菌和志贺菌CRISPR2FAAATCGTATGAAGTGATGCATRGTCGATGCAAACACATAAATA579~2318(大肠) 1357(志贺)志贺菌CRISPR1FAGCGACTAACTGGAATCTTGRCAATCTGGCTACTGGAAGTG711~1193沙门菌CRISPR1FGTRGTRCGGATAATGCTGCCRCGTATTCCGGTAGATBTDGATGG628~1261沙门菌CRISPR2FGAGCAATACYYTRATCGTTAACGCCRGTTGCDATAKGTYGRTRGRATGTRG764~1277
R=G或A;Y=T或C;K=G或T;D=G或A或T;B=G或T或C。
1.4同源比对实验室菌株测序的CRISPR序列和提取CRISPRdatabase数据库的菌株的CRISPR序列均通过CRISPRfinder在线软件(http://crispr.u-psud.fr/Server/)进行分析并获取重复序列和spacer信息,所有菌株的spacer比对使用ClustalX软件,CRISPRTarget在线工具(http://bioanalysis.otago.ac.nz/CRISPRTarget/crispr_analysis.html)查找大肠埃希菌O157∶H7的spacer的同源质粒或噬菌体。
1.5应用CRISPR检测大肠埃希菌O157∶H7方法对模拟样品的分析将大肠埃希菌O157∶H7接种于新鲜无菌的营养肉汤培养基中,37 ℃过夜(12h)培养。通过菌落计数方法推算其菌液原液的细菌数2.3×109CFU/mL。全脂奶粉10g溶于90mL生理盐水,灭菌,然后分别加入230CFU/mL和23CFU/mL的大肠埃希菌O157∶H7菌液1mL,混匀作为样品原液,即大肠埃希菌O157:H7的菌液浓度分别为23、2.3CFU/mL,放置2h。吸取5mL混合液加入45mL的营养肉汤放置37 ℃温箱进行培养,设置两组平行对照,一组阴性对照,然后于8、12、16、20h各取10mL混合液,分离培养细菌以提取DNA进行CRISPR1检测,方法同1.3项。
1.6聚类分析使用SPSS21.0对40株(实验室35株,数据库5株)大肠埃希菌O157∶H7的CRISPR1和CRISPR2的检测结果进行样本聚类分析。
2 结 果
2.1543株肠道细菌的CRISPR和spacer信息PCR扩增产物的测序序列和提取CRISPRdatabase数据库的菌株的CRISPR序列均使用CRISPRFinder软件进行分析,结果显示75.6%(270/357)的非O157∶H7大肠埃希菌、75.5%(74/98)志贺菌、91.7%(44/48)沙门菌、95%O157∶H7(38/40)大肠埃希菌含有CRISPR1,其spacer数目为3~26,2~9,2~32,3;57.1%(204/357)的非O157∶H7大肠埃希菌、77.6%(76/98)的志贺菌、85.4%(41/48)的沙门菌、100%(40/40)的O157∶H7大肠埃希菌含有CRISPR2,其spacer数目为1~20,1~6,2~27,1或4个(表2)。
2.2大肠埃希菌O157∶H7的CRISPR1和CRISPR2的电泳结果实验室保存O157∶H7大肠埃希菌的CRISPR1和CRISPR2位点的PCR扩增产物的电泳结果符合其长度(图1)。
2.3大肠埃希菌O157∶H7的spacer信息及比对结果通过CRISPRFinder软件分析40株大肠埃希菌O157∶H7的CRISPR,结果显示95%的(38/40)大肠埃希菌O157∶H7的CRISPR1可以检测出完全一致的4条重复序列(CGGTTTATCCCCGCTGGCGCGGGGAACAC)和3条独特spacer(分别命名为S1-1,S1-2,S1-3),90%(36/40)的大肠埃希菌O157∶H7的CRISPR2可以检测出完全一致的2条重复序列(GGTTTATCCCCACTGGCGCGGGGAAC-TC)和1条独特spacer(命名为S2-1)。其中实验室保存的4株大肠埃希菌O157∶H7CRISPR2分别可以检测到5条重复序列和4条spacer(未列出)。通过CRISPRTarget在线工具进行spacer的同源比对,结果显示没有发现与之匹配的同源质粒或噬菌体(表3)。
表2大肠埃希菌、志贺菌和沙门菌的CRISPR和spacer信息
Tab.2CRISPRandspacersofEscherichia coli,ShigellaandSalmonella

菌株组别CRISPR1菌株数[n(%)]Spacer数目CRISPR2菌株数[n(%)]Spacer数目非O157∶H7大肠埃希菌实验室(n=310)238(76.8)3~26172(55.5)1~15数据库(n=47)32(68.1)3~2532(68.1)1~20合计(n=357)270(75.6)3~26204(57.1)1~20志贺菌实验室(n=89)72(80.9)2~975(84.3)1~6数据库(n=9)2(22.2)2~31(11.1)1合计(n=98)74(75.5)2~976(77.6)1~6沙门菌实验室(n=9)9(100.0)7~159(100.0)10~16数据库(n=39)35(89.7)2~3232(82.1)2~27合计(n=48)44(91.7)2~3241(85.4)2~27O157∶H7大肠埃希菌实验室(n=35)33(94.3)335(100.0)1或4数据库(n=5)5(100.0)35(100.0)1合计(n=40)38(95.0)340(100.0)1或4

图1大肠埃希菌O157∶H7(mel-e200001/hn)的CRISPR1和CRISPR2
Fig.1TheCRISPR1andCRISPR2intheEscherichia coliO157∶H7 (mel-e200001/hn)
M:DL2000marker; 1:CRISPR1; 2:CRISPR2。
表3大肠埃希菌O157∶H7菌株的独特spacer信息
Tab.3ThespacersinEscherichia coliO157∶H7

名称Spacer长度(bp)同源质粒或噬菌体S1-1AGCGGCACGCTGGATTGAACAAATCCCTGGGC32无S1-2AAACCGAAACACACGATCAATCCGAATATGAG32无S1-3TTTGGTGACAGTTTTTGTCACTGTTTTGGTGA32无S2-1CTCGACTTTAATTTTTTCGCTGCCCGCTTTTGC33无
2.4CRISPR检测大肠埃希菌O157∶H7方法的特异性经过ClustalX比对和分析CRISPRdatabase数据库中47株非O157∶H7大肠埃希菌、9株志贺菌、39株沙门菌和实验室310株非O157∶H7大肠埃希菌、89株志贺菌、9株沙门菌的spacer,结果发现,spacerS1-1+S1-2+S1-3和S2-1对检测大肠埃希菌O157∶H7的特异性分别是100%和99.6%(CRISPRdatabase数据库中有2株大肠埃希菌O55:H7含有S1-1和S2-1)。
2.5标准法和CRISPR检测大肠埃希菌O157∶H7的一致性比较共选用543份肠道菌株,同时进行标准法和PCR扩增CRISPR1(S1-1+S1-2+S1-3)、CRISPR2(S2-1)的检测,结果显示CRISPR1和CRISPR2与标准法一致性分别达到99.6%(541/543)和99.3%(539/543)(表4)。
表4两种检测大肠埃希菌O157∶H7方法的比较
Tab.4TheconsistencyofthetwomethodsfordetectingE.coliO157∶H7
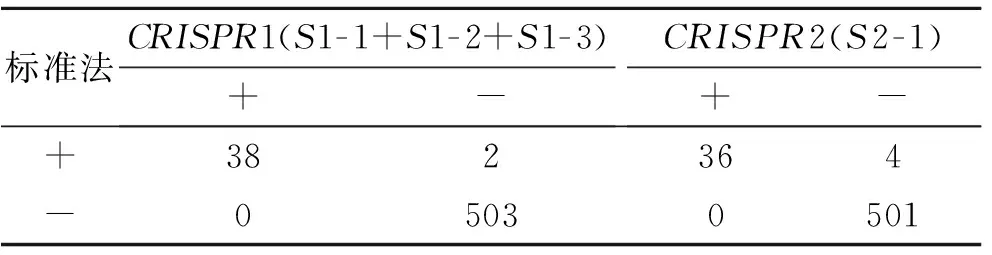
标准法CRISPR1(S1-1+S1-2+S1-3)+-CRISPR2(S2-1)+-+382364-05030501
2.6基于CRISPR检测大肠埃希菌O157∶H7对模拟样品的分析结果菌液浓度分别为23、2.3CFU/mL的样品原液经培养12h后PCR均能扩增出预期的目的片段(图2)。
2.7大肠埃希菌O157∶H7的CRISPR1和CRISPR2样品聚类分析40株O157∶H7大肠埃希菌的聚类分析结果显示可分为3类(图3)。
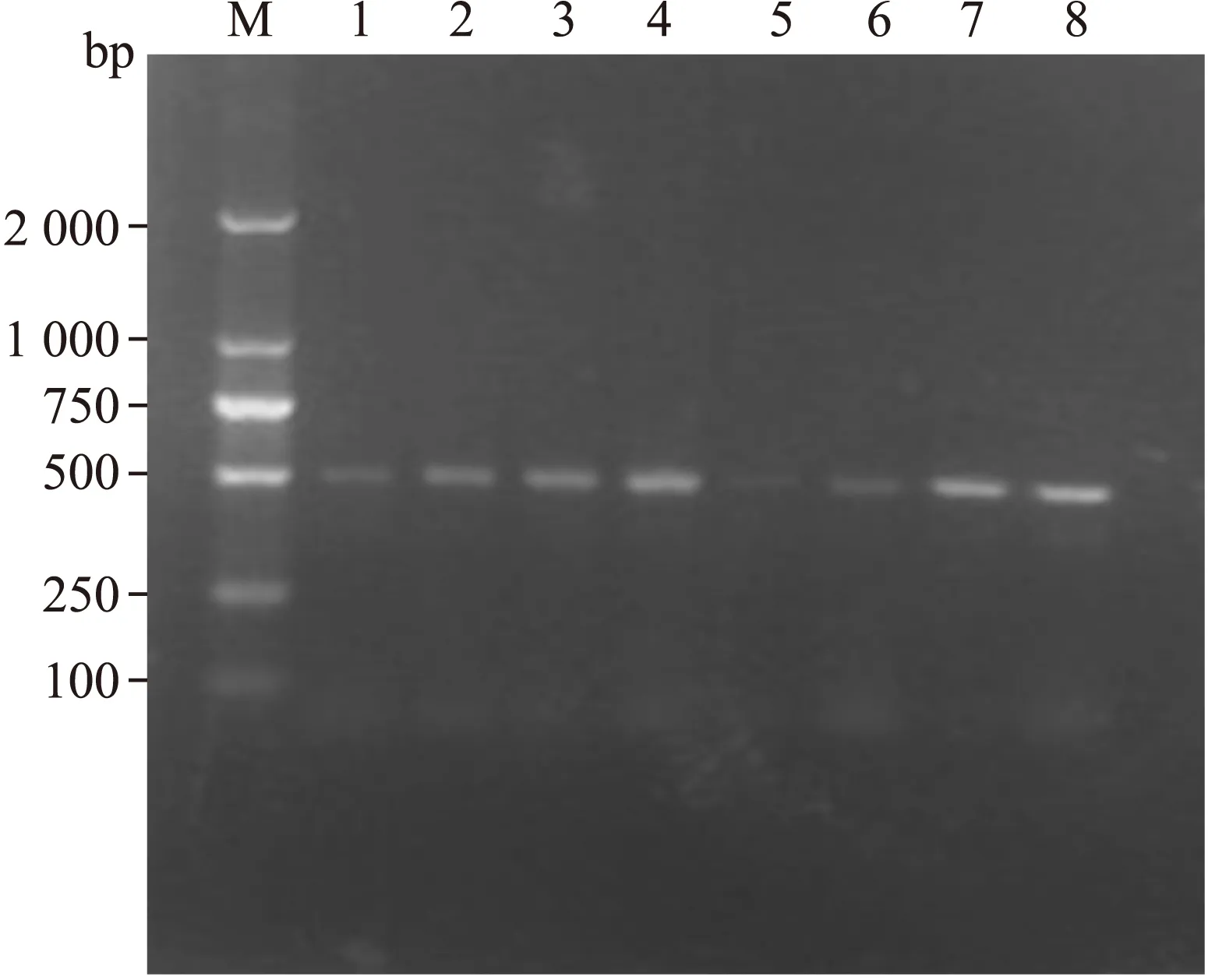
图2CRISPR检测大肠埃希菌O157∶H7在模拟样品中的应用
Fig.2TheapplicationwithCRISPR1todetectE.coliO157∶H7inthemilksample
M:DL2000marker;1~4、5~8分别为原菌液浓度为23、2.3CFU/mL的样品培养8、12、16、20h。
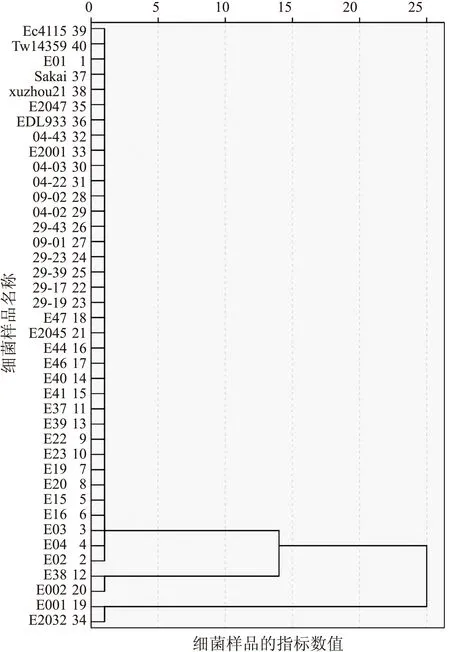
图340株大肠埃希菌O157∶H7的CRISPR1和CRISPR2样品聚类分析结果
Fig.3Theclusteranalysisfor40 Escherichia coliO157∶H7strainswithCRISPR1andCRISPR2
3 讨 论
大肠埃希菌O157∶H7作为大肠埃希菌的一种特定血清型,1982年首次发现于美国[1],1986年我国徐州首次检出,随后沿海地区有散发病例报道,至上世纪末江苏和上海爆发,感染患者超过2万人,死亡达177人[13]。大肠埃希菌O157∶H7一般经污染的食物和水源感染人类,人被感染后轻者表现腹泻,严重后果为出血性结肠炎、出血性尿毒症,甚至死亡[14],重症患者尚无有效的治疗方法。因此,高毒株的大肠埃希菌O157∶H7和新发细菌的感染检测非常重要。标准法检测大肠埃希菌O157∶H7需要3~5d,时间长;分子生物学检测大肠埃希菌O157∶H7需同时扩增rfbEO157基因和鞭毛抗原H7特异基因flic,而毒力因子stx、hly、eaeA只能作为辅助鉴定因子。
CRISPR/Cas广泛分布于细菌和古生菌中。1987年,ISHINO等[15]在对大肠埃希菌的碱性磷酸酶同工酶所对应的核酸序列进行分析时,发现其下游存在29个碱基组成的重复序列和其间的spacer。2002年,JANSEN等[16]其正式命名为ClusteredRegularlyInterspacedShortPalindromicRepeats,即成簇的规律间隔短回文重复序列。本文通过CRISPRFinder对CRISPRdatabase数据库52株大肠埃希菌(5株大肠埃希菌O157∶H7)、9株志贺菌、39株沙门菌的CRISPR和实验室保存的345株大肠埃希菌(包括35株大肠埃希菌O157∶H7)、89株志贺菌和9株沙门菌的CRISPR位点进行检测分析,结果显示有38株大肠埃希菌O157∶H7的CRISPR1可以检测到3条独特spacer,36株大肠埃希菌O157∶H7的CRISPR2可以检测到1条独特spacer,其余4株大肠埃希菌O157∶H7的CRISPR2分别检测到4条spacer,与CRISPR2的1条独特spacer均不一致,但不影响基于CRISPR1对大肠埃希菌O157∶H7的检测。4株大肠埃希菌O157∶H7的CRISPR2的spacer不一致,这是首次在大肠埃希菌O157∶H7发现,或许可以为大肠埃希菌O157∶H7的分型提供依据。至于不一致的原因,有可能与其毒力水平有关,还需要进一步实验研究。
建立基于CRISPR对大肠埃希菌O157∶H7的新型检测方法特异性好。通过ClustalX比对和分析CRISPRdatabase数据库47株非O157∶H7大肠埃希菌、9株志贺菌、39株沙门菌和实验室310株非O157∶H7大肠埃希菌、89株志贺菌、9株沙门菌的spacer,结果发现,CRISPR1的3条独特spacer和CRISPR2的1条独特spacer对大肠埃希菌O157∶H7的检测的特异性分别是100%和99.6%(CRISPRdatabase数据库中有2株大肠埃希菌O55∶H7含有S2-1)。SABINE等研究显示,基于CRISPR定量PCR特异性检测大肠埃希菌O26∶H11、O45∶H2、O103∶H2、O111∶H8、O121∶H19、O145∶H28、O157∶H7[17]和O104∶H4,提示CRISPR与大肠埃希菌的血清型有一定联系,但CRISPR与大肠埃希菌的血清型的内在联系机制有待继续研究。
CRISPRdatabase数据库中有2株大肠埃希菌O55:H7都含有spacerS2-1,提示spacer能反映出大肠埃希菌O157∶H7与O55:H7有很近的亲缘关系。研究显示,大肠埃希菌O55:H7丢失O55抗原簇,基因组插入stx噬菌体,捕获大质粒pO157后,可以改变O抗原形成O157∶H7血清型。因此,推测S2-1spacer是O55:H7向O157∶H7进化过程中保留下来的,且spacerS1-1和S2-1的存在早于S1-2,S1-3。那么spacerS1-2、S1-3在O157∶H7形成过程中是逐个插入还是作为整体插入染色体,仍需继续研究。但spacer或许可以作为新型菌株出现的靶标。S1-1、S1-2、S1-3、S2-1与已知质粒pO157、stx噬菌体和LEE毒力岛均没有匹配,或许提示O157∶H7的形成过程中有可能还有其他未知可移动元件的参与,或者spacer的形成存在其他机制,这尚有待进一步研究。
本实验选用543株肠道菌株进行标准法和PCR扩增CRISPR1(S1-1+S1-2+S1-3)、CRISPR2 (S2-1)比较,结果显示CRISPR1、CRISPR2与标准法检测大肠埃希菌O157∶H7的一致性分别达到99.6%和99.3%,说明PCR扩增CRISPR检测大肠埃希菌O157∶H7与标准法一致性很好。基于CRISPR检测大肠埃希菌O157∶H7在模拟样品中应用,结果显示原样品大肠埃希菌O157∶H7菌液浓度分别为23、2.3CFU/mL的样品经培养12h后均能扩增出预期的目的片段,灵敏性较高,可进行临床推广使用。
大肠埃希菌O157∶H7的CRISPR1和CRISPR2的样本聚类分析显示,40株O157∶H7大肠埃希菌可分为3类。大肠埃希菌O157∶H7菌株EDL933、EC4115、Sakai、TW14359、xuzhou21分别引发了美国、日本、中国暴发性感染性腹泻。实验室菌株是在2000年河南省某县首次大肠埃希菌O157∶H7感染性腹泻疫情中收集的,其中31株属于此类,这提示分离于河南的大肠埃希菌O157∶H7与高毒株大肠埃希菌O157∶H7亲缘关系较近。
基于CRISPR的大肠埃希菌O157∶H7新型检测方法,积极发展并用于O157∶H7的临床诊断和流行病学调查,将成为监测大肠埃希菌O157∶H7感染、高毒株大肠埃希菌O157∶H7或者新型菌株出现的有价值的流行病学工具。
[1]PAICH,GORDONR,SIMSHV,etal.SporadiccasesofhemorrhagiccolitisassociatedwithEscherichia coliO157∶H7.Clinical,epidemiologic,andbacteriologicfeatures[J].AnnInternMed, 1984, 101(6):738-742.
[2]MORIY,WADAH,TAMAKIS,etal.OutcomeofthromboticthrombocytopenicpurpuraandhemolyticuremicsyndromeinJapan[J].ClinApplThrombHemost,1999, 5(2):110-112.
[3]FRANZE,KLERKSMM,DEVOSOJ,etal.PrevalenceofShigatoxin-producingEscherichia colistx1,stx2,eaeA,andrfbEgenesandsurvivalofE.coliO157∶H7inmanurefromorganicandlow-inputconventionaldairyfarms[J].ApplEnvironMicrobiol, 2007, 73(7):2180-2190.
[4]TOUCHONM,ROCHAEP.Thesmall,slowandspecializedCRISPRandanti-CRISPRofEscherichiaandSalmonella[J].PLoSOne, 2010, 5(6):e11126.
[5]NOZAWAT,FURUKAWAN,AIKAWAC,etal.CRISPRinhibitionofprophageacquisitioninStreptococcuspyogenes[J].PLoSOne, 2011, 6(5):e19543.
[6]MARRAFFINILA,SONTHEIMEREJ.CRISPRinterferencelimitshorizontalgenetransferinstaphylococcibytargetingDNA[J].Science, 2008, 322(5909):1843-1845.
[7]MOJICAFJ,GARCA-MARTNEZJ,SORIAE.Interveningsequencesofregularlyspacedprokaryoticrepeatsderivefromforeigngeneticelements[J].JMolEvol, 2005, 60(2):174-182.
[8]MAKAROVAKS,HAFTDH,BARRANGOUR,etal.EvolutionandclassificationoftheCRISPR-Cassystems[J].NatRevMicrobiol, 2011, 9(6):467-477.
[9]GUOX,WANGY,DUANG,etal.DetectionandanalysisofCRISPRsofShigella[J].CurMicrobiol, 2015, 70(1):85-90.
[10] 郭向娇,王颖芳,段广才,等. 临床分离志贺菌中CRISPR/Cas系统的分布及其与毒力基因的关系[J]. 微生物学通报, 2015, 55(3):543-549.
[11] 王琳琳,王颖芳,段广才,等. 志贺菌CRISPR的检测及其与耐药的关系[J].微生物学报, 2015, 55(4):476-483.
[12]TOUCHONM,CHARPENTIERS,CLERMONTO,etal.CRISPRdistributionwithintheEscherichia colispeciesisnotsuggestiveofimmunity-associateddiversifyingselection[J].JBacteriol, 2011, 193(10):2460-2467.
[13]FABREL,ZHANGJ,GUIGONG,etal.CRISPRtypingandsubtypingforimprovedlaboratorysurveillanceofSalmonellainfections[J].PLoSOne, 2012, 7(5):e36995.
[14]LIH,JINGH,PANGB,etal.StudyondiarrheadiseasecausedbyenterohemorrhagicEscherichia coliO157∶H7inXuzhoucity,Jiangsuprovincein2000[J].ChinJEpidemiol, 2002, 23(2):119-122.
[15]ISHINOY,SHINAGAWAH,MAKINOK,etal.Nucleotidesequenceoftheiapgene,responsibleforalkalinephosphataseisozymeconversioninEscherichia coli,andidentificationofthegeneproduct[J].JBacteriol, 1987, 169(12):5429-5433.
[16]JANSENR,EMBDENJD,GAASTRAW,etal.IdentificationofgenesthatareassociatedwithDNArepeatsinprokaryotes[J].MolMicrobiol, 2002, 43(6):1565-1575.
[17]DELANNOYS,BEUTINL,FACHP.UseofclusteredregularlyinterspacedshortpalindromicrepeatsequencepolymorphismsforspecificdetectionofenterohemorrhagicEscherichiacolistrainsofserotypesO26:H11,O45:H2,O103:H2,O111:H8,O121:H19,O145:H28,andO157∶H7byreal-timePCR[J].JClinMicrobiol, 2012, 50(12):4035-4040.
(编辑国荣)
DetectionofEscherichiacoliO157∶H7basedonCRISPRlocus
LIANGWen-juan1,2,ZHANGRong-guang1,DUANGuang-cai1,2,WANGYing-fang3,HONGLi-juan1,ZHANGBing1,WANGPeng-fei1,XIYuan-lin1,YANGHai-yan1
(1.DepartmentofEpidemiology,CollegeofPublicHealthofZhengzhouUniversity,Zhengzhou450001; 2.HenanInnovationCenterofMolecularDiagnosisandLaboratoryMedicine,XinxiangMedicalUniversity,Xinxiang453003; 3.DepartmentofPreventiveMedicine,HenanUniversityofScienceandTechnology,Luoyang471023,China)
ObjectiveToestablishamethodfordetectionandidentificationofEscherichia coliO157∶H7withtheClusteredRegularlyInterspacedShortPalindromicRepeats(CRISPR)locusasatargetbyPCR.MethodsPCRamplificationwasusedtodetecttheCRISPRlocusof443strains(310strainsofnon-O157∶H7 Escherichia coli, 35strainsofEscherichia coliO157∶H7, 89Shigellastrains,and9Salmonellastrains).TheCRISPRFinderwasusedtoanalyzethesequencesincludingthe100strains(47strainsofnon-O157∶H7 Escherichia coli, 5strainsofEscherichia coliO157∶H7, 9Shigellastrains,and39Salmonellastrains)intheCRISPRdatabase.ThespacerswerealignedwithClustalX.ThentheconsistencywasanalyzedwiththestandardmethodandPCRamplificationCRISPR1detectionofE.coliO157∶H7.ResultsWefoundthat75.6%ofnon-O157∶H7 Escherichia coli, 75.5%ofShigella, 91.7%ofSalmonellaand95%ofO157∶H7 Escherichia colicontainedCRISPR1.Thenumberofthespacerscouldbe3-26, 2-9, 2-32and3amongthe543strains,respectively. 57.1%ofnon-O157∶H7 Escherichia coli, 77.6%ofShigella, 85.4%ofSalmonellaand100%ofO157∶H7 Escherichia colicontainedCRISPR2;thenumberofthespacerswas1-20, 1-6, 2-27,and1or4,respectively.Threeuniquespacers(S1-1,S1-2,S1-3)andoneuniquespacer(S2-1)couldbedetectedinCRISPR1andCRISPR2oftheEscherichia coliO157∶H7.ThespaceralignmentresultsshowedthatthespecificityoftheEscherichia coliO157∶H7withtheS1-1+S1-2+S1-3andS2-1was100%and99.6%,respectively.TheconsistencyforCRISPR1andCRISPR2detectingO157∶H7 Escherichia coliwas99.6%and99.3%comparedwiththestandardmethod.TheEscherichia coliO157∶H7wasdetectedafter12hourswithasensitivityof2.3CFU/mLmilksample.Theclusteranalysisshowedthat40strainsofO157∶H7 E.colicouldbedividedintothreegroups.ConclusionThedetectionofE.coliO157∶H7basedontheCRISPRlocusbyPCRmaybecomeavaluableepidemiologictoolforthesurveillanceofE.coliO157∶H7infections.
Escherichia coliO157∶H7;clusteredregularlyinterspacedshortpalindromicrepeat(CRISPR);PCR;detection
2015-10-27
2016-04-24
国家科技重大专项(No.2013ZX100046070),新乡医学院分子诊断与医学检验技术河南省协同创新中心(No.XTCX-2015-PY4)
段广才.E-mail:gcduan@zzu.edu.cn
R37
A
10.7652/jdyxb201605027
SupportedbytheNationalScienceandTechnologyMajorProject(No.2013ZX100046070)andHenanInnovationCenterofMolecularDiagnosisandLaboratoryMedicine(No.XTCX-2015-PY4)
优先出版:http://www.cnki.net/kcms/detail/61.1399.R.20160805.1022.016.html(2016-08-05)

