景观破碎化对植物种群的遗传学效应及其研究方法进展
丁兆红, 卢君婷, 卢剑波,2
(1.杭州师范大学生命与环境科学学院,浙江 杭州 310036;2.杭州师范大学钱江学院,浙江 杭州 310036)
景观破碎化对植物种群的遗传学效应及其研究方法进展
丁兆红1, 卢君婷1, 卢剑波1,2
(1.杭州师范大学生命与环境科学学院,浙江 杭州 310036;2.杭州师范大学钱江学院,浙江 杭州 310036)
景观破碎化在减小物种生境大小的同时增加了生境的空间隔离度,直接或间接影响种群遗传多样性和空间遗传结构.要明确景观特征的改变对种群生存能力的影响,不仅需要收集大量的实例研究来描述一般的发展规律,更要形成标准化的研究试验设计,对生境和物种的变化在时间和空间尺度上进行量化,从而制定更为科学合理的应对措施.文章首先综述了景观破碎化对植物种群遗传学效应的影响,然后分别从分子标记、统计分析方法和取样策略三方面介绍了该领域研究方法的进展,最后指出今后此方面的研究亟需加强多学科间的交流,以期发现和创造新的研究领域和方法.
景观破碎化;植物种群;遗传多样性;空间遗传结构;研究方法
景观破碎化(landscape fragmentation)相关研究一直是景观生态学和保护生物学领域的研究热点之一[1-2].国内外研究学者对于景观破碎化的概念有着不同的表述,总体是指由于自然或人为因素干扰所导致的景观由简单趋向于复杂的过程,即景观由单一、均质和连续的整体趋向于复杂、异质和不连续的斑块镶嵌体[3].景观破碎化对陆地景观生态系统带来的影响主要是以下三个方面:1)增加斑块间隔离程度;2)加大斑块数量及斑块的边缘效应;3)降低平均斑块面积[4].造成的后果则是破碎化生境中的物种将会受到更为明显的生存威胁[5].
景观遗传学(landscape genetics)作为景观生态学和种群遗传学的交叉学科,研究景观特征与微进化过程(如基因流、遗传漂变与选择等)之间的相互作用,在个体和种群水平探讨地理空间和环境特征影响遗传变异的规律与机理[6].近年来,随着景观遗传学理论与技术的不断发展,景观破碎化对植物种群遗传效应的研究也引起了许多科学家的关注[7].本文结合理论与实例研究,从遗传变异和种群空间遗传结构两个方面综述了景观破碎化对植物种群遗传效应的影响及其研究方法的进展.景观破碎化对种群遗传分化影响的机制见图1.
1 景观破碎化对植物种群遗传多样性的影响
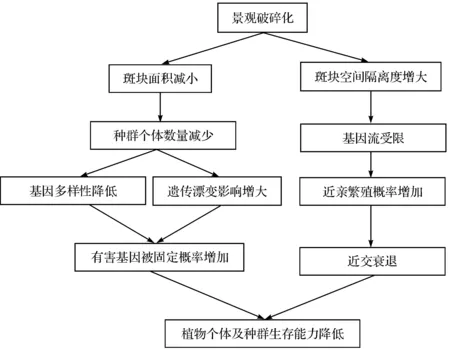
图1 景观破碎化对植物种群遗传效应影响的简易模型Fig. 1 A simple model for the effects of habitat fragmentation on genetic effects of plant populations
遗传多样性(genetic diversity, GD)作为生物多样性的核心和基础,组成了生物进化的本质,代表了物种适应环境变异的能力[8-9].任何一个物种都具有其独特的基因库和遗传结构,同时在不同的个体间往往又存在着丰富的遗传变异,这些变异构成了生物的遗传多样性[10].遗传多样性作为评价物种进化过程的综合指标,其最直接的表达形式就是遗传变异的高低及遗传变异的分布格局,即种群的空间遗传结构[11].
1.1景观破碎化对植物种群遗传变异的影响
景观破碎化过程造成植物种群生存面积的缩小,导致一个完整的种群变为几个大小不同的残存种群,形成复合种群(metapopulation)[12].由于每一个单独残存种群只保留原始种群的一部分基因,从而造成了等位基因多样性(allelic diversity)的直接丢失[13].相关学者利用微卫星分子标记研究中国北方的松口蘑(Tricholomamatsutake)遗传多样性,主成分分析发现4个不同种群中分别隶属于3个基因簇,表明4个种群中由于生境破碎化导致各个种群之间容易因地理隔离形成遗传隔离[14].在具有世代间隔离且面积较小的种群中会遭遇更为明显的遗传漂变(genetic drift),从而导致位点多样性的降低和种群间遗传分化(genetic differentiation)的增加[15]. Ye等对Psilopeganumsinense6个不同面积大小种群的遗传分化状况进行分析,由于三峡大坝的修建导致其生存面积进一步减少使得原本种群数量较少的该物种在小种群中表现出较强的遗传分化[16].同时在水生克隆植物Menyanthestrifoliata和墨西哥东部地区的Cyclopogonluteoalbu均验证了有效生存面积大小对于其种群遗传多样性方面存在负面影响[17-18].一般情况下,种群生物个体越小,遗传漂变的影响越大,而且在对物种适应特殊环境改变有很大作用的稀有等位基因中变现尤为突出[19].同时,由于遗传漂变对中性和选择性基因的作用效果是一样的,因此,有害基因可能会被随机固定下来,最终危害植物个体和种群的长期健康生存[20].
1.2景观破碎化对植物种群遗传结构的影响
种群遗传结构(population genetic structure)指遗传多样性在种群内和种群间的分布,可以综合反映种群大小、组成、繁育系统、隔离程度和迁移格局的时空动态[21].景观破碎化状态下的种群,植株间空间距离增加,由花粉或种子介导的基因流会受到相应的限制,从而使种群遗传分化程度加重,使种群处于近亲繁殖的危害中[22].而近交衰退的严重性似乎在异交种群中更大,因为有害隐性等位基因的影响可以被个体间高的杂合性掩盖.也有假说认为,如果小的残余种群能够持续存在,那么随着时间推移,有害突变可能因选择作用而累积,近交衰退的影响也可以得到缓冲性减小[23].
然而从目前的实例证据来看,景观破碎化对植物种群的遗传效应还不明确,有研究报道说景观破碎化会降低种群的遗传变异,增加斑块种群间的遗传分化[24],但也有相关研究指出其并没有发现这种遗传效应[13].Psaroudaki 等在希腊的克里特岛区域内利用随机扩增片段长度多态性(random amplified polymorphism DNA, RAPD)技术对11种可食用野生草本植物的遗传结构和种群多样性作调查时,在10个不同的种群中分别进行采样,发现物种Anagallisarvensis和Sonchusasperssp.glaucescens的遗传多样性显著低于其他9个物种,原因是由于种群生境的破碎化导致的基因流受到限制.另一方面也说明,同等水平下的生境破碎化对不同物种的影响程度也存在差别[25].这些对比性的结果往往是由物种特有的生物学特性如物种间繁殖模式、生活史长度和生命周期间的差异等因素引起.在景观破碎化初期,较长世代周期或较大的基因流扩散距离等生物学特性有助于物种维持其种群的遗传多样性.当研究中取样的时间尺度、形成破碎化格局所经历的时间以及生境改变程度完全不同时,物种表现出的适应性机制也存在差异性[26].研究揭示,即使长距离的花粉扩散能力和物种长的生命周期能够抵消预期遗传多样性的丢失,人类活动引起种群结构的改变对面积较小隔离种群的遗传变异造成的影响也是不可避免的.Rosas等对濒危树种桃花心木(Swieteniahumilis)花粉流和遗传多样性的研究发现,虽然桃花心木在隔离种群和连续种群中都有强烈的花粉扩散能力(>2 000 m),显示花粉介导的基因流不因破碎化隔离而被限制,但由于高的遗传多样性保存在父本植株中且雄株缺乏,景观破碎化还是带来了一定的有害繁殖成效和负面遗传效应,说明长距离的花粉扩散事件并不足以维持种群的遗传多样性,但对于来自花粉池中的基因流还是必需的[27].
2 景观破碎化对植物种群空间遗传结构的影响
空间遗传结构(spatial genetic structure, SGS)指等位基因在空间尺度上的非随机分布,是种群的生态学过程和遗传学过程相互作用的结果[28].由于不同物种之间的生活史特征、世代时间、传播能力等自身因素以及外界生态系统连接度改变、演替改变、气候变化等因素影响.景观破碎化过程或格局对植物种群遗传多样性的影响往往存在时滞性.因此对已经遭受破碎化潜在影响的植物种群不能采取及时有效的保护措施[29].然而,相关研究提出景观破碎化对植物种群早期的遗传效应可能表现在改变种群空间遗传结构[30].
2.1景观破碎化对基因流的影响
植物种群空间遗传结构的形成是一个受多因素影响的多阶段发展过程,影响因素包括由种子和花粉扩散介导的基因流、种群密度、现有植株的时空分布以及微生境的异质性等[16].然而在小的居群中,影响种群空间遗传结构最主要的因素是有限的基因流及随机遗传漂变导致的地理距离隔离.在热带森林中,有90%以上的树种是靠以果实为食的动物散播种子,种子的扩散在很大程度上决定了种群的更新和空间分布模式[31].景观破碎化导致的斑块隔离直接限制了食果动物的活动范围,降低了种子的扩散距离,因此种子可能会在母株周围沉积性扩散,增加短距离内个体间的遗传相似性和空间遗传强度.同时由于植株高密度的分布会在一定程度上限制花粉流的传播,从而进一步提高其空间遗传结构的显著性.Harata等通过选择10种具有相对生态特征(传粉者、种子扩散距离、种群密度和空间分布模式)的龙香脑科(dipterocarp)植物,分析这些因子与小尺度空间遗传结构(fine-scale spatial genetic structure, FSGS)形成的关系,结果显示:在每个研究对象中,小尺度空间的遗传结构相比大尺度更明显;多重回归分析显示,在小的空间尺度上,种子扩散距离对小尺度空间遗传结构的形成相对比较重要,而在较大的空间尺度上,传粉者和成株的空间分布更重要;在10个物种中,以体积小、运动慢的甲壳虫为传粉者的物种的小尺度空间遗传结构比以体积大、运动快的蜜蜂作为传粉者的物种中更为显著;成株分布更聚集的物种,小尺度空间遗传结构也更强.说明在植物种群微生境中,空间尺度、基因流传播者及种群密度都与其空间遗传结构的形成有一定的联系[32].
2.2景观破碎化对微生境异质性的影响
景观破碎化在改变种群斑块面积和空间隔离度的同时,对于种群生存微生境的异质性也会有所改变.微生境的异质性往往也是形成某种遗传学特性不可忽视的原因.Sezen等研究了哥斯达黎加的棕榈植物(Iriarteadeltoidea)在过熟林和次生林中的空间遗传结构,发现过熟林中的斑块直径为96 m,而次生林中的斑块直径为125 m,说明景观基质的不同将影响到植物群落的更新格局[33].景观破碎化引起微生境的物理性质或化学性质在一定程度上的改变,进而影响种子的萌发与生长,降低成株的繁殖成效或改变其繁殖方式,如通过植株基部和根系萌蘖新植株,进而可能导致植株个体在短距离内呈现聚集分布模式.Ortego等通过对地中海长寿物种麻栎(Quercusilex)的研究发现,相比较森林种群,过度采伐的破碎化麻栎斑块中具有大量的克隆性结构个体,显示了在人为干扰和生境波动下,此物种偏向于无性繁殖[24].相关学者对片断化和非片断化森林中白花延龄草(Trilliumcamschatcens)种群各两个年龄级(成株、幼苗)的小尺度遗传结构进行分析,结果表明:在非片断化种群中,两个年龄级都有着显著的空间遗传结构;而在片断化种群中,显著的空间遗传结构只出现在成株,推测可能是由于片断化生境不利于幼苗萌发、生长,该研究结果提示我们在物种保护和恢复中需要重视保护区域的景观基质[34].
3 植物种群遗传效应和空间遗传结构的研究方法及其进展
在景观遗传学发展过程中,植物遗传效应研究遵循的原则一般为利用高度变异性的分子遗传标记提取基于个体的遗传信息,结合个体和种群的空间分布信息,利用间接的(如种群遗传分化指标、杂合度等)和直接的(如亲子分析、邻域界定、基因流模型等)种群遗传结构分析指标和模型,从不同角度探测种群遗传变异程度和空间遗传结构,并采用多种空间统计方法推断景观格局特征与种群遗传结构的因果关系[35].
在数据库Web of Science中利用关键字landscape fragmentation,genetic diversity,spatial genetic structure在植物科学和生态环境科学两个研究领域中搜索2005年1月至2014年12月出版的相关文献总计约501篇(图2),该领域研究论文数量逐年增加,说明该研究领域越来越被学术界所重视.以相关性从高至低排序,选择前100篇对分子标记应用情况进行统计(图3).由于利用关键字搜索文献,遗漏掉一些文献是在所难免的,所以统计结果只能概括一般趋势.
3.1分子标记
分子标记以个体间遗传物质(核苷酸序列)的变异为基础,能直接反映DNA水平遗传多态性.目前,在景观遗传学中应用的分子标记有18种之多,其中在植物研究中比较普遍的包括微卫星DNA,也称简单重复序列(microsatellite, simple sequence repeats, SSR)、扩增片段长度多态性(amplified fragment length polymorphism, AFLP)、RAPD及线粒体DNA(mtDNA) 序列、叶绿体DNA (cpDNA )序列等,而应用最多的是微卫星标记[36](图3).微卫星由核心串联重复序列和两端保守序列组成,其多态性主要来源于核心序列串联重复数目的不同.同时由于微卫星位点在属内种间甚至在科内属间是保守的,因此其微卫星引物可以在近缘物种之间通用.近些年来,基于第二代测序技术中RAD测序的限制位点相关的DNA(restriction site associated DNA, RAD)标记也逐渐在遗传多样性分析中得到应用[37-38](RAD应用统计归类于其它选项).
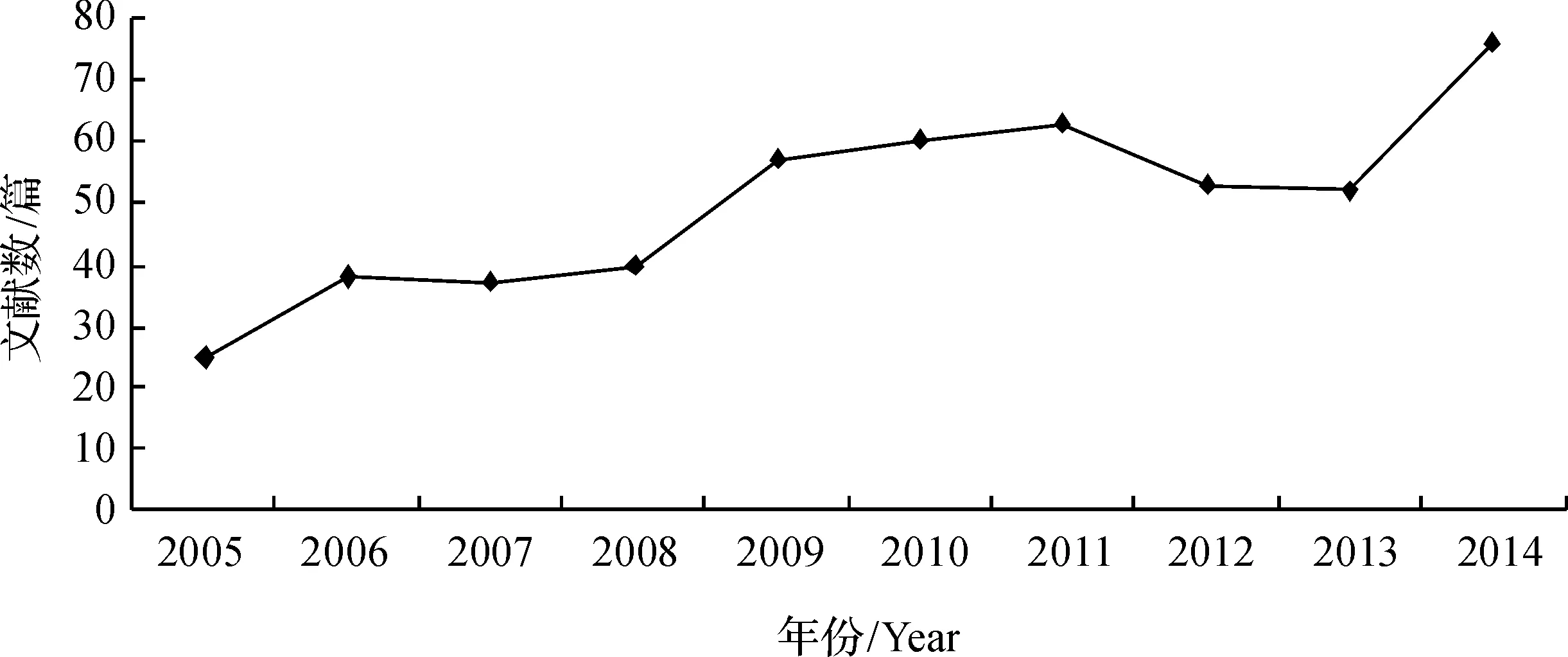
图2 2005-2014年景观破碎化对植物遗传效应影响相关文献统计(检索自Web of Science)Fig. 2 Statistics of literature about plant genetic effect of landscape fragmentation from 2005 to 2014(results based on searches using the Web of Science)
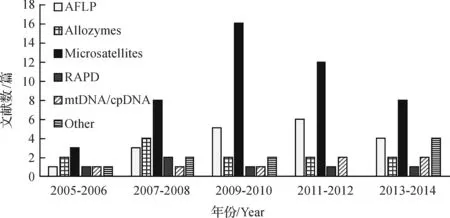
图3 2005-2014年景观破碎化对植物遗传效应影响相关文献中分子标记使用情况统计(数据来源为检索文献总数的前100篇)Fig. 3 Statistics of the using molecular markers in study of plant genetic effect of landscape fragmentation from 2005 to 2014(Source from top 100 of total)
3.2统计分析方法
研究景观破碎化对植物遗传学效应影响时,选择任何一种分析方法都需要结合具体景观格局形成过程和种群发展历史,为景观破碎化对种群遗传分化的影响方式和强度做出合理的结论和解释[39].
在遗传多样性数据分析方面,常用的软件包括HP-Rare、GenePop、FSTAT、Cervus、Genalex 等,其中HP-Rare在统计等位基因多样性时可以消除样本量的差异.通常采用的统计量包括等位基因数(A)、期望杂合度(He)、观察杂合度(Ho)、近交系数(Fis)、遗传分化系数(Fst)以及基因流(Nm)等.一般在分析之前,还需对数据进行连锁不平衡检测、哑基因(null alleles)检测及Hardy-Weinberg检测,确保研究位点的独立性和可行性.在空间遗传结构数据分析方面,常用的软件包括GenAIEx、SPAGeDi、STRUCTURE等[40].分析方法包括空间自相关分析、对应矩阵间的Mantel 检验、异质性检验、空间遗传强度统计以及主成分分析(PCA)、Bayesian聚类分析等.其中空间自相关分析在植物居群的空间遗传结构研究中应用最广泛,此方法通过检测一个位置上的变异是否依赖于邻近位置的变化, 来判断该变异是否存在空间自相关性, 即是否存在空间结构[41].
3.3取样策略
实际研究中,考虑到人力和物力的有限,通常不可能对研究物种的所有种群和个体进行采样,这就面临着如何在可操作的前提下采集包含尽量多变异样品的问题,这关系到所得研究结果是否可靠,是否能诊断出破碎化所引起的遗传效应[42].
制定取样策略时,一般包含取样种群数和每个种群取样个体数两方面.通常会要求样本量应包含居种群95%以上的遗传变异,或要求样本包含稀有等位基因的基因频率小于0.05[43].Brown等根据近交系数F变动在-1~1之间提出的计算模型计算的样本数应在19~58之间;当F值为0时,随机交配的个体数是30[44].同样,Sjögren等根据检测到频率为5%稀有等位基因的要求提出的计算模型,计算结果为取样数至少要达到30个个体[45].另外,Hamrick等提出利用基因分化系数Gst来估算一个物种应取样的种群数,得出的结论是6个种群基本就能够包含该物种95%的变异[46].所以,在对研究对象的生物学特性或是生境基质特征不太明确的前提下,将采样种群定在6个左右,每个种群30个左右的个体数基本上可以满足试验需求,同时由于破碎化导致的生境异质性,所以取样时,还得考虑样本个体的空间分布.此外,针对破碎化效应,许多研究在取样的同时,将取样植株分为不同等级,如破碎前与破碎后或幼苗、幼株及成株等,以便亚种群间的比较[47-48].总之,我们应根据研究物种的生物特异性(包括繁殖方式、基因流扩散机制、分布范围)和研究目的,制定合理有效的取样策略.
4 展 望
由于不同植物的生活史差距很大,对景观破碎化的响应会因物种的生物学特性和具体生境的不同而不同,因此在理解景观破碎化形成机制并预测其结果之前,需要对植物的生态学特性和历史演替过程有一个彻底的了解.因此,当前面临的挑战是实验研究的标准化设计,对生境和物种的变量在时间和空间尺度上的精准量化,以便在未受干扰和干扰后的景观之间进行相互比较.在空间尺度上可以结合3S技术,利用GIS将景观变量叠加于遗传数据上,使空间遗传格局可视化,并对相关的遗传格局做出假设[49-50].同时,相关学者提出从植株个体的角度研究驱动其繁殖与更新的过程,利用多学科方法研究基因扩散时空动态过程新的观点[51].
因此,在研究景观破碎化过程或格局对植物种群造成影响的过程中,必须加强多学科之间的相互交流,提出并相互采用可行的研究方法,推导出一般的适用于破碎化植物景观遗传学研究的原则,从而对因破碎化影响而产生实际动态过程有一个合理的评估与预测.
[1] POGSON, B. Habitat fragmentation reduces biodiversity[J]. Science,2015,347(6228):1325-1325.
[2] TILMAN D, MAY R M, LEHMAN C L, et al. Habitat destruction and the extinction debt[J]. Nature,1994,371(6492):65-66.
[3] MITCHELL M G, SUAREZ-CASTRO A F, MARTINEZ-HARMS M, et al. Reframing landscape fragmentation’s effects on ecosystem services[J]. Trends in Ecology & Evolution,2015,30(4):190-198.
[4] FAHRIG L. Effects of habitat fragmentation on biodiversity[J]. Annual Review of Ecology Evolution and Systematics,2003,34(487):487-515.
[5] WATLING J I, NOWAKOWSKI A J, DONNELLY M A. Meta-analysis reveals the importance of matrix composition for animals in fragmented habitat[J]. Global Ecology and Biogeography,2011,20(2):209-217.
[6] ORTEGO J, AGUIRRE M P, NOGUERALES V, et al. Consequences of extensive habitat fragmentation in landscape-level patterns of genetic diversity and structure in the Mediterranean esparto grasshopper[J]. Evolutionary Applications,2015,8(6):621-632.
[7] MIAO Y C, LANG X D, ZHANG Z Z, et al. Phylogeography and genetic effects of habitat fragmentation on endangeredTaxusyunnanensisin southwest China as revealed by microsatellite data[J]. Plant Biology,2014,16(2):365-374.
[8] MAY M R, GODFREY J. Biological diversity:differences between land and sea(and discussion)[J]. Philosophical Transactions of the Royal Society Biological Sciences,1994,343(1303):105-111.
[9] PAULS S U, NOWAK C, BáLINT M, et al. The impact of global climate change on genetic diversity within populations and species[J]. Molecular Ecology,2013,22(4):925-946.
[10] HUGHES A R, INOUYE B D, JOHNSON M T, et al. Ecological consequences of genetic diversity[J]. Ecology Letters,2008,11(6):609-623.
[11] 武晶,刘志民.生境破碎化对生物多样性的影响研究综述[J].生态学杂志,2014,33(7):1946-1952.
[12] MéNDEZ M, VÖGELI M, TELLA J L, et al. Joint effects of population size and isolation on genetic erosion in fragmented populations: finding fragmentation thresholds for management[J]. Evolutionary Applications,2014,7(4):506-518.
[13] WANG Y, ZHU Y, WANG Y. Differences in spatial genetic structure and diversity in two mosses with different dispersal strategies in a fragmented landscape[J]. Journal of Bryology,2012,34(1):9-16.
[14] ZENG D F, CHEN B. Genetic variability and bottleneck detection of fourTricholomamatsutakepopulations from northeastern and southwestern China[J]. Environmental Microbiology,2015,17(8):2870-2881.
[15] 陈小勇.生境片断化对植物种群遗传结构的影响及植物遗传多样性保护[J].生态学报,2000,20(5):884-892.
[16] YE Q, TANG F, WEI N, et al. Molecular and quantitative trait variation within and among small fragmented populations of the endangered plant speciesPsilopeganumsinense[J]. Annals of Botany,2014,113(1):79-86.
[18] RAABOVA J, VAN ROSSUM F, JACQUEMART A . Population size affects genetic diversity and fine-scale spatial genetic structure in the clonal distylous herbMenyanthestrifoliata[J]. Perspectives in Plant Ecology, Evolution and Systematics,2015,17(3):193-200.
[19] CRISTOFANI-YALY M, NOVELLI V M, BASTIANEL M, et al. Transferability and level of heterozygosity of microsatellite markers in citrus species[J]. Plant Molecular Biology Reporter,2011,29(2):418-423.
[20] FINGER A, KETTLE C J, KAISER-BUNBURY C N, et al. Forest fragmentation genetics in a formerly widespread island endemic tree:Vateriopsisseychellarum(Dipterocarpaceae)[J]. Molecular Ecology,2012,21(10):2369-2382.
[21] 葛颂.植物群体遗传结构研究的回顾和展望[M].北京:高等教育出版社,1997:1-15.
[22] TEE K L, WONG T S. Polishing the craft of genetic diversity creation in directed evolution[J]. Biotechnology Advances,2013,31(8):1707-1721.
[23] MOREIRA P A, FERNANDES G W, COLLEVATTI R G. Fragmentation and spatial genetic structure inTabebuiaochracea(Bignoniaceae)a seasonally dry Neotropical tree[J]. Forest Ecology and Management,2009,258(12):2690-2695.
[24] ORTEGO J, BONAL R, MUOZ A. Genetic consequences of habitat fragmentation in long-lived tree species: the case of the Mediterranean Holm Oak (Quercusilex,L.)[J]. The Journal of Heredity,2010,101(6):717-726.
[25] PSAROUDAKI A, NIKOLOUDAKIS N, SKARACIS G, et al. Genetic structure and population diversity of eleven edible herbs of Eastern Crete[J]. Journal of Biological Research,2015,22(1):1-11.
[26] SORK V L, WAITS L. Contributions of landscape genetics-approaches, insights, and future potential[J]. Molecular Ecology,2010,19(17):3489-3495.
[27] ROSAS F, QUESADA M, LOBO J A, et al. Effects of habitat fragmentation on pollen flow and genetic diversity of the endangered tropical treeSwieteniahumilis(Meliaceae)[J]. Biological Conservation,2011,144(12):3082-3088.
[28] EPPERSON B K. Spatial distributions of genotypes under isolation by distance[J]. Genetics,1995,140(4):1431-1440.
[29] ESSL F, DULLINGER S, RABITSCH W, et al. Delayed biodiversity change: no time to waste[J]. Trends in Ecology & Evolution,2015,30(7):375-378.
[30] VAN ROSSUM F, TRIEST L. Fine-scale genetic structure of the commonPrimulaelatior(Primulaceae) at an early stage of population fragmentation[J]. American Journal of Botany,2006,93(9):1281-1288.
[32] HARATA T, NANAMI S, YAMAKURA T, et al. Fine-scale spatial genetic structure of ten dipterocarp tree species in a bornean rain forest[J]. Biotropica,2012,44(5):586-594.
[33] SEZEN U U, CHAZDON R L, HOLSINGER K E. Genetic consequences of tropical second-growth forest regeneration[J]. Science,2005,307(5711):891.
[34] YAMAGISHI H, TOMIMATSU H, OHARA M. Fine-scale spatial genetic structure within continuous and fragmented populations ofTrilliumcamschatcense[J]. The Journal of Heredity,2007,98(4):367-372.
[35] 沈泽昊,吉成均.景观遗传学原理及其在生境片断化遗传效应研究中的应用[J].生态学报,2010,30(18):5066-5076.
[36] STORFER A, MURPHY M A, SPEAR S F, et al. Landscape genetics: where are we now?[J]. Molecular Ecology,2010,19(17):3496-3514.
[37] 王冠杰,周波,李玉花.RAD标记测序及其在分子育种中的应用[J].中国生物化学与分子生物学报,2012,28(9):797-803.
[38] EMERSON K J, MERZ C R, CATCHEN J M, et al. Resolving postglacial phylogeography using high-throughput sequencing[J]. Proceedings of the National Academy of Sciences,2010,107(37):16196-16200.
[39] EWERS R M, DIDHAM R K, PEARSE W D, et al. Using landscape history to predict biodiversity patterns in fragmented landscapes[J]. Ecology Letters,2013,16(10):1221-1233.
[40] MANEL S, SCHWARTZ M K, LUIKART G, et al. Landscape genetics: combining landscape ecology and population genetics[J]. Trends in Ecology & Evolution,2003,18(4):189-197.
[41] LEGENDRE P. Spatial autocorrelation:trouble or new paradigm?[J]. Ecology,1993,74(6):1659-1673.
[42] ANDERSON C D, EPPERSON B K, FORTIN M J, et al. Considering spatial and temporal scale in landscape-genetic studies of gene flow[J]. Molecular Ecology,2010,19(17):3565-3575.
[43] MARSHALL D R, BROWN A. Optimum sampling strategies in genetic conservation[M]//FRANKEL O H, HAWLES J G. Crop Genetic Resource for Today and Tomorrow. London: Cambridge University Press,1975:53-70.
[44] BROWN A, MORAN G F. Isozymes and the genetic resources of forest trees[M]// Conkle M T.Isozymes of north American forest trees and forest insects pacific south west forest and range experiment station technical report No.48. Washington, D C:US Department of Agriculture,1981:1-10.
[45] SJÖGREN P, WYÖNI P I. Conservation genetics and detection of rare alleles in finite populations[J]. Conservation Biology,1995,8(1):267-270.
[46] HAMRICK J K, GODT M J W, MURAWSKI D A, et al .Correlation between species traits and allozyme diversity: implication for conservation biology[M]// FALK D A,HOLSINGER K E. Genetics and conservation of rare plants. New York: Oxford University Press,1991:75-86.
[47] HAMRICK J L. Response of forest trees to global environmental changes[J]. Forest Ecology and Management,2004,197(1/2/3):323-335.
[48] 金燕,卢宝荣.遗传多样性的取样策略[J].生物多样性,2003,11(2):155-161.
[49] SEGELBACHER G, CUSHMAN S A, EPPERSON B K, et al. Applications of landscape genetics in conservation biology: concepts and challenges[J]. Conservation Genetics,2010,11(2):375-385.
[50] MILLETTE K L, KEYGHOBADI N. The relative influence of habitat amount and configuration on genetic structure across multiple spatial scales[J]. Ecology and Evolution,2015,5(1):73-86.
[51] BACLES C F, JUMP A S. Taking a tree’s perspective on forest fragmentation genetics[J]. Trends in Plant Science,2011,16(1):13-18.
On the Genetic Effects of Landscape Fragmentation on Plant Populations and Research Methods
DING Zhaohong1, LU Junting1, LU Jianbo1,2
(1.College of Life and Environmental Sciences, Hangzhou Normal University, Hangzhou 310036, China; 2.Qianjiang College,Hangzhou Normal University, Hangzhou 310036, China)
Landscape fragmentation decreases habitat scale, intensifies spatial isolation of habitats and directly or indirectly affects the population genetic diversity and spatial genetic structure. To clear the effects of the changes of landscape features on population viability, it needs not only collect quantities of empirical researches to describe the general rules, but also quantify the changes of habitats and species in time and space scales to standardize experimental study design. This paper summarizes genetic effects of landscape fragmentation on plant populations and reviews the research methods commonly used in this filed. Furthermore, interdisciplinary exchanges should be strengthened in order to discover and create new research areas and methods.
landscape fragmentation; plant population; genetic diversity; spatial genetic structure; research methods
2015-09-14
国家自然科学基金项目(31170448);浙江省大学生科技创新活动计划暨新苗人才计划项目(2014R421076).
卢剑波(1963-),男,教授,博士,主要从事景观生态学、遥感应用等研究.E-mail: jianbo.lu@hznu.edu.cn
10.3969/j.issn.1674-232X.2016.05.010
Q948.1
A
1674-232X(2016)05-0502-07

