小麦胁迫相关基因TaSF3B2的克隆及表达特性分析
黄 鑫,夏百成,龙翔宇
(1 海南大学 农学院, 海口 570228 ;2 中国热带农业科学院 橡胶研究所,海南儋州 571737)
小麦胁迫相关基因TaSF3B2的克隆及表达特性分析
黄 鑫1,夏百成1,龙翔宇2*
(1 海南大学 农学院, 海口 570228 ;2 中国热带农业科学院 橡胶研究所,海南儋州 571737)
为研究小麦剪接因子在逆境胁迫中的作用,通过分析小麦抗性相关EST数据库,筛选并克隆获得1条2 327 bp的核苷酸序列。该序列包含了一个1 854 bp的开放阅读框,编码一个由617个氨基酸残基组成的蛋白,氨基酸同源比对发现,该蛋白含有GUS1结构域(Splicing Factor 3B, Subunit 2),属于剪接因子3b亚基中的一员,将该蛋白命名为TaSF3B2。生物信息学分析显示,TaSF3B2平均亲水系数(GRAVY)为-0.895,不稳定系数为43.92,且在细胞核中发挥作用的可能性最大。实时荧光定量PCR分析表明,TaSF3B2基因在不同组织中存在差异表达,不同发育时期其表达也存在差异;在幼苗叶片中,该基因表达受高盐、低温、条锈菌、干旱及ABA激素胁迫明显下调表达,而在幼苗根中变化不明显。推测TaSF3B2基因参与了小麦正常条件下的生长发育,同时在小麦应对逆境胁迫的过程中具有重要的作用。
小麦;TaSF3B2;基因克隆;生物信息学;基因表达
在真核生物中,前体RNA(pre-mRNA)需要经过剪接加工,去除内含子序列,并将外显子序列连接才能成为成熟的有功能的mRNA分子。pre-mRNA的剪接有组成性剪接和选择性剪接两种方式,其剪接是在剪接体中进行。剪接体是由多个小分子核内核糖体蛋白(snRNP: U1、U2、U4/U6和U5)和50~100多种非snRNP剪接因子组成的大分子复合体[1]。在RNA剪接过程中,U2稳定地结合到分支点序列形成复合体A是剪接过程中至关重要的一步,而剪接因子(splicing factor,SF)3是复合体A组装所必需的成分[2]。SF3被分为SF3A和SF3B两种,其中SF3B在剪接体组装和识别内含子的分支点中起重要作用[3]。
选择性剪接是从同一个pre-mRNA选择性地将不同外显子连接起来产生多种不同成熟mRNA的过程,从而可以将同一个基因转录翻译形成许多结构和功能不同的蛋白质[4]。早期的研究结果显示,脊椎动物为了适应外界环境,选择性剪接多发生在免疫和神经等复杂系统,剪接体的研究也主要集中在脊椎动物中[5-6]。近些年,越来越多的研究表明,选择性剪接同样是植物适应逆境胁迫的机制之一,植物可通过选择性剪接调控抗逆基因的表达以适应逆境[7]。目前,对植物剪接体的组成及选择性剪接的调控机理研究相对减少,本实验以小麦为材料,在小麦胁迫相关EST数据库中分离并拼接得到一个剪接因子(TaSF3B2),了解该基因的结构及其蛋白的理化特性,同时分析该基因在不同组织及胁迫环境下的表达模式,为了解小麦剪接因子3B亚基的功能及其调控机理提供一定理论依据。
1 材料和方法
1.1 植物材料及材料处理
本实验使用的材料是六倍体普通小麦‘中国春’,由四川农业大学小麦研究所提供。
选取籽粒饱满的小麦种子用流水冲洗干净后播种于装有干净石英砂的塑料盆内,每盆3粒,然后将其放置到Hoagland营养液中进行水培,每隔24 h更换1次营养液,在光照培养箱中培养约3周,待其长到两叶一心后,选择长势基本一致的植株进行处理,每个处理设有3盆重复。处理1:将待处理材料放入含有200 mmol/L NaCl的Hoagland营养液中,盐胁迫48 h;处理2:将待处理材料放入含有50mmol/L ABA的Hoagland营养液中,激素诱导48 h;处理3:将待处理材料放入含有150 mmol/L PEG的Hoagland营养液中,干旱胁迫48 h;处理4:将待处理材料放入Hoagland营养液中,4 ℃低温胁迫24 h;处理5:供试菌为‘条中32’(购于甘肃省农科院),采用抖粉接种方法,接种时先往接种点喷水雾,然后将菌粉(新鲜孢子与滑石粉1∶250混合)抖落到麦叶上,接种后再喷1次水,盖上塑料薄膜,于15 ℃黑暗培养12 h后,将薄膜取下正常生长,条锈病接种72 h;对照CK:未处理材料在Hoagland营养液中继续水培,光照培养箱参数条件与处理前保持一致,与处理采样时间同步,处理1、2、3和对照,取其根及叶片,处理4和5取其叶片。
在田间,待小麦长到两叶一心后,取苗期叶片、茎及根等不同组织。待开花20 d后取其灌浆中期的叶片、茎、根及种子。所有样品均用液氮冷冻,在-70 ℃保存以备RNA提取。
1.2 方 法
1.2.1 cDNA合成及TaSF3B2基因的克隆 利用TRNzol试剂盒提取不同材料总RNA(购自天根生化科技有限公司),反转录采用PrimeScriptTM1st Strand cDNA Synthesis Kit试剂盒(购自宝生物工程有限公司),提取及合成方法按照略有修改的试剂盒说明书进行。
分析小麦EST数据库(HAWheat152和Plant Expression Database),筛选并拼接小麦胁迫候选基因,根据找到的TaSF3B2(登录号GAJL01260647)序列设计引物TaSF3B2-F1(5′-GAGAGAGCCATTGAGCACCGCTT-3′)和TaSF3B2-R1(5′-ACACCAAGACTCTGATTTGTCCGGAAAG-3′),以小麦幼苗cDNA为模版进行PCR扩增,反应条件为: 94 ℃预变性3 min,然后94 ℃变性 30 s,62 ℃退火30 s,72 ℃延伸1 min 30 s,35个循环;72 ℃延伸10 min,琼脂糖电泳回收目的片段,并将其连接到克隆载体(pMD18-T Vector, TaKaRa,宝生物工程有限公司)上,转化大肠杆菌DH5α(上海生工)送至上海生物工程公司测序。
1.2.2TaSF3B2基因的序列分析TaSF3B2核苷酸翻译及多序列比对采用DNAMAN5.22软件;氨基酸成分、不稳定系数、亲水系数、蛋白分子量、等电点分析采用在线软件http://web.expasy.org/protparam/;信号肽和亚细胞定位采用POSORT预测采用在线软件http://www.cbs.dtu.dk/services/SignalP/及http://psort.hgc.jp/form.html;蛋白质亲水性图谱在线构建使用在线软件http://web.expasy.org/cgi-bin/protscale/protscale.pl;跨膜结构域分析使用在线软件http://www.ch.embnet.org/software/TMPRED_form.html;其他物种的氨基酸序列来源于NCBI数据库,采用MEGA5.10软件,以邻近法构建进化树。
1.2.3 实时荧光定量PCR检测TaSF3B2基因的表达 以小麦不同生长时期、不同组织以及不同胁迫处理组织的cDNA为模版,使用特异性引物TaSF3B2-F2(5′-ACTTCGGGAAATGAAGCCAGGTA-3′)和TaSF3B2-R2(5′-GATAAGAGGGCGGAGGACCATA-3′)进行定量分析。以小麦延长因子TaEF1α基因作为内参基因,对样品进行均一化,其扩增引物为TaEF1α-F(5′-CAGATTGGCAACGGCTA-
CG-3′)和TaEF1α-R(5′-CGGACAGCAAAACGACCAAG-3′)。采用天根公司实时荧光定量PCR试剂盒RealMasterMix(SYBR Green,购自天根生化科技有限公司),在Chromo4TM实时荧光定量PCR检测仪(Bio-Rad)上进行特异性表达分析。反应总体系20 μL,包含模板2 μL、上下游引物混合物0.6 μL和2×SYBR Premix 10 μL、ddH2O 7.4 μL;反应程序为95 ℃预变性30 s;94 ℃变性5 s,60 ℃退火20 s,72 ℃延伸 20 s,42个循环;每个样品设置3个重复,Cq值转换,标准曲线以及相关稀释等数据处理采用其自带软件。
2 结果与分析
2.1 TaSF3B2基因的克隆及其编码蛋白的生物信息学分析
分析小麦EST数据库,筛选并获得与小麦抗性相关的候选基因片段,在NCBI数据库中搜索到1条与其相似的小麦cDNA序列(登录号GAJL01260647),相似度为99%,同源比对与候选基因片段相似的cDNA序列并设计引物,采用RT-PCR技术分离得到1条2 327 bp核苷酸序列(图1),与候选基因序列一致,该序列包含了一个1 854 bp开放阅读框,编码1个由617个氨基酸残基组成的蛋白,蛋白分子量大小为70.31 kD。
在NCBI氨基酸数据中进行保守结构域分析,发现该蛋白含有保守的GUS1结构域(Splicing Factor 3B, Subunit 2),位于第181~429位(COG5182,E-value:1.64e-67),同时该蛋白在N端含有RT_like结构域(reverse transcriptase_like family,cl02808)。进一步Blast结果表明,该蛋白氨基酸序列与乌拉图小麦(Triticumurartu,EMS56988.1)、二穗短柄草(Brachypodiumdistachyon,XP_003573003.1)、水稻(OryzasativaJaponicaGroup,NP_001048592.1)、大豆(Glycinemax,XP_003541544.1)、番茄(Solanumlycopersicum,XP_004234125.1)、蓖麻(Ricinuscommunis,XP_002519025.1)、拟南芥(Arabidopsislyratasubsp.lyrata,XP_002867835.1)和SF3B2(Splicing factor 3b, subunit2)氨基酸序列的一致性分别达到99%、89%、87%、74%、72%、75%、71%和69%(图2)。据此表明该蛋白为小麦剪接因子3B亚基2家族的一员(SF3B2),命名为TaSF3B2。
氨基酸组成分析结果显示(http://web.expasy.org/protparam/),该蛋白富含谷氨酸Glu(77,12.5%),赖氨酸Lys(69,11.2%),天冬氨酸Asp(49,7.9%),亮氨酸Leu(47,7.6%),而组氨酸His(8,1.3%),色氨酸Trp(6,1.0%)和半光氨酸Cys(3,0.5%)含量较低。该蛋白理论等电点(PI)为4.94,不稳定系数为(instability index)43.92,说明该蛋白是一个不稳定的酸性蛋白。蛋白二级结构分析结果显示(SOPMA软件),TaSF3B2蛋白中40.84%氨基酸参与了随机卷曲(random coil),37.93%的氨基酸残基参与了α-螺旋(alpha helix),14.26%的氨基酸参与了延伸链(extended strand),仅有6.97%的氨基酸参与了β-转角(beta turn)(图3),由此可见,该蛋白二级结构最大原件是随机卷曲,α-螺旋、延伸链及β-转角均匀地散布于整个蛋白结构中,其二级结构相对简单。导肽的预测和分析结果表明其信号肽分值较低,无氨基酸残基位点,此蛋白可能不存在导肽酶切位点,不具有导肽。蛋白质疏水性的预测和分析结果显示,最低值为-3.867(位于第602位氨基酸),最高值为1.644(位于第7位氨基酸),平均亲水系数(GRAVY)为-0.895,总体表现为亲水性,同时发现该蛋白也不存在明显的跨膜结构域。TaSF3B2亚细胞定位分析结果显示(http://psort.hgc.jp/form.html),该蛋白定位于细胞核的可能性最大,得分为0.980。
2.2 TaSF3B2氨基酸同源性比对及聚类分析
根据TaSF3B2基因编码的氨基酸序列,在大麦、山羊草、短柄草、玉米、水稻、高粱、黄瓜、杨树、蓖麻、番茄、可可树、苜蓿、大豆、拟南芥、草莓及碧桃中,搜索得到16条与TaSF3B2相似的SF3B2氨基酸序列。Clustalx多序列比对,JTT模型估算系统进化距离矩阵,Mega5.10软件邻接法(100次自展检验,其他参数取软件默认值)进行聚类分析。结果显示17条SF3B2氨基酸序列被明显分为双子叶和单子叶两类,第一类包括了橡胶树、白杨树、蓖麻及拟南芥在内的共10条双子叶SF3B2氨基酸序列,第二类包括了小麦、大麦、及水稻在内的共7条单子叶SF3B2氨基酸序列(图4)。氨基酸同源性显示TaSF3B2与亲缘关系较近的山羊草SF3B2的蛋白序列一致性高达83%,与其亲缘关系较远的拟南芥SF3B2的蛋白序列也有较高的相似性(69%)。多序列比对分析显示这两类植物SF3B2在C端存在高度的保守性,尤其在GUS1结构域,而不同物种SF3B2的N端氨基酸序列之间存在明显差异,氨基酸序列的多样性也主要存在于N端特定的区域。聚类、氨基酸同源性及序列比对结果表明,SF3B2蛋白分子进化相对保守,且其分子进化与物种间的生物进化关系趋于一致。

M.Marker; 1. PCR扩增产物图1 TaSF3B2基因cDNA的PCR扩增片段M.Marker; 1.PCR productFig.1 Electrophoresis of TaSF3B2 cDNA fragment

图2 植物SF3B2氨基酸序列比对Fig.2 Alignment of amino acid sequences of SF3B2 in plants

蓝线,红线,绿线和紫线各代表α-螺旋,延伸链,β-转角和随机卷曲图3 小麦TaSF3B2蛋白质的二级结构分析The blue, red, green and purple lines represent the alpha helix, extended strand, beta turn and random coil, respectivelyFig. 3 Secondary structure of wheat TaSF3B2 protein

图4 与TaSF3B2相似的SF3B2蛋白进化分析Fig.4 Phylogenetic analysis of SF3B2 from plants
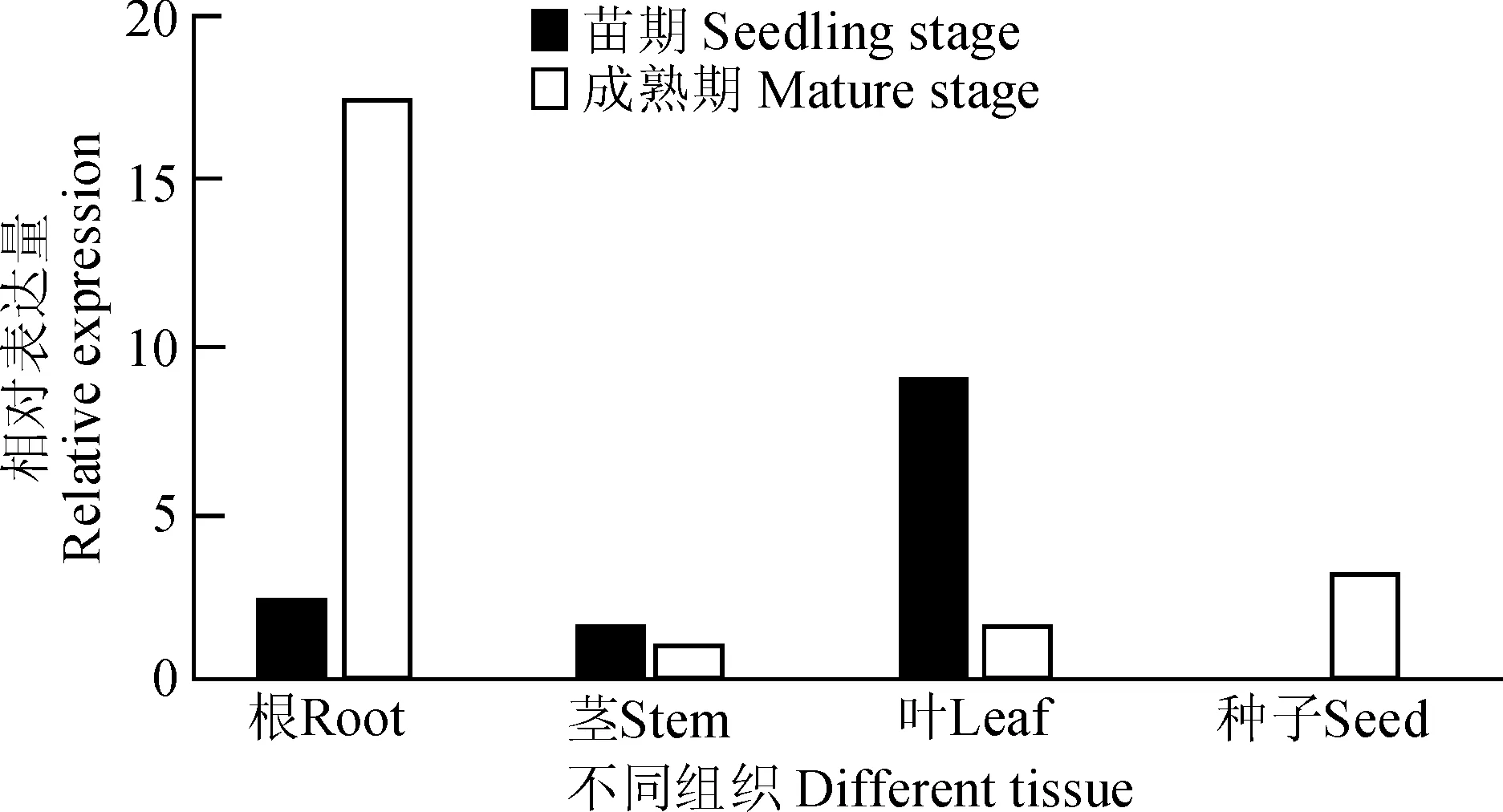
图5 TaSF3B2基因在不同时期及组织中的表达模式Fig.5 Expression pattern analysis of TaSF3B2 in different periods and tissues

图6 不同胁迫下TaSF3B2基因在苗期根中的表达模式Fig.6 Expression pattern analysis of TaSF3B2 in root under stress
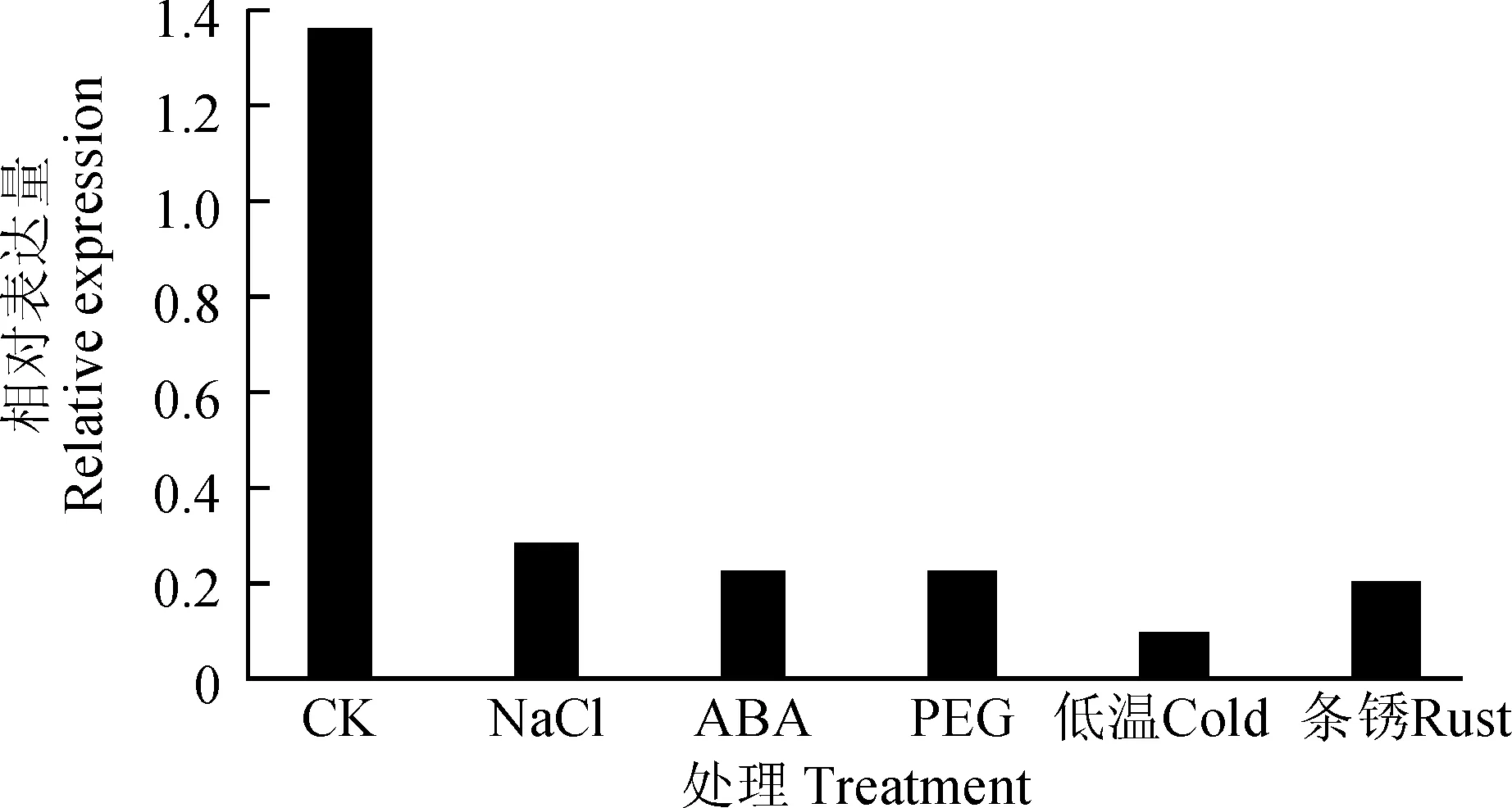
图7 不同胁迫下TaSF3B2基因在苗期叶片中的表达模式Fig.7 Expression pattern analysis of TaSF3B2 in leaf under stress
2.3 TaSF3B2基因的组织特异性表达分析
采用实时荧光定量PCR技术分析TaSF3B2基因在小麦不同发育时期、不同组织中的表达特性,在苗期的根、茎及叶子,成熟期的根、茎、叶子及种子中,均能检测到TaSF3B2基因的表达,表明该基因在小麦的不同发育时期的不同组织中均表达(图5)。在小麦的苗期,TaSF3B2基因在叶中表达量最高,根次之,茎中表达量最低;在小麦成熟时期,TaSF3B2基因在根中表达量最高,种子中次之,叶子及茎中表达量最低。综合组织特异表达结果,尽管TaSF3B2基因在所检测的组织中均表达,但是该基因仍存在组织特异性表达,同时该基因的表达模式受不同发育时期的影响。
2.4 TaSF3B2基因在各种胁迫下的表达模式分析
为了进一步了解TaSF3B2基因的功能,分析该基因在小麦苗期各种胁迫下的表达模式,在盐、ABA及干旱胁迫下,TaSF3B2基因在根中表达变化不明显,这可能与该基因在小麦苗期根中的表达量相对较低有关(图6);在小麦苗期的叶片中,该基因受盐、ABA、干旱、低温及条锈菌的诱导下调表达,且下调倍数均在6倍以上,其中低温诱导该基因下调最为明显,其次为条锈菌、ABA、干旱及盐胁迫(图7)。以上的结果表明,TaSF3B2基因受高盐、低温、病菌、激素及干旱等胁迫的调控,推测可能参与了小麦生物与非生物胁迫应答。
3 讨 论
RNA剪接广泛存在于真核生物中,转录形成的pre-mRNA需经过剪接体的加工形成成熟的mRNA,剪接体组装是一个高度有序的动态过程,剪接因子在此过程中发挥着重要作用,如剪接点位点识别,外显子保留以及剪接位点的相互作用。本研究在小麦胁迫相关的EST数据库中分离并克隆得到一个SF3B亚基的一员,其氨基酸序列分析显示该基因为哺乳动物SF3B2或SAP145的同源基因。在哺乳动物的剪接体中,SAP145与SAP49形成复合体, VPr(HIV-1的附属基因产物)可以通过干扰SAP145-SAP49复合体的形成从而阻止剪接体的组装,表明SAP145在剪接体的组装中发挥着重要作用,推测TaSF3B2在小麦剪接体组装中具有相同作用[8-12]。
在植物中,选择性剪接可调节基因表达,从而维持蛋白质的多样性,大多数发生选择性剪接的基因与调控功能相关,如植物抗逆相关基因容易发生选择性剪接[1,13-14]。当植物受到逆境胁迫时,信号转导分子可感受逆境信号,通过影响剪接因子的活性及浓度,从而影响剪接活性和剪接位点识别,最终调节抗逆基因的表达,使植物进入抵抗逆境的生理状态[15-17]。本研究分析了剪接因子TaSF3B2基因在不同组织及各种胁迫下的表达模式,结果显示该基因在不同发育时期存在组织特异性表达,并且在幼苗叶片中的表达量受多种胁迫影响,推测TaSF3B2通过调节剪接体的组装进而在小麦发育及生物与非生物胁迫过程中发挥重要作用。本研究结果为揭示小麦剪接体的组装,及剪接因子3B各亚基在此过程中的作用提供理论依据,有助于进一步了解小麦选择性剪接在其抗逆过程中的作用机理。
[1] HASTINGS ML, KRAINER AR. Pre-mRNA splicing in the new millennium[J].CurrentOpinioninCellBiology, 2001, 13(3):302-309.
[2] BROSI R, HAURI HP,etal. Separation of splicing factor SF3 into two components and purification of SF3a activity[J].JournalofBiologicalChemistry, 1993, 268(23):17 640-17 646.
[3] 袁丽华, 罗小洋, 张吉翔. 剪接因子——SF3b[J]. 生命的化学, 2009, 5:746-749.
YUAN L H, LUO X Y, ZHANG J X. SF3b: a splicing factor[J]. Chemistry of Life, 2009, 5:746-749.
[4] CHEAH M T, WACHTER A,etal. Control of alternative RNA splicing and gene expression by eukaryotic riboswitches[J].Nature, 2007, 447(7 143):497-500.
[5] CARNINCI P,etal.(194 coauthors). The transcriptional landscape of the mammalian genome[J].Science, 2005, 309(5 740):1 559-1 563.
[6] MODREK B, LEE C. A genomic view of alternative splicing[J].Naturegenetics, 2002, 30(1):13-19.
[7] KAZAN K. Alternative splicing and proteome diversity in plants: the tip of the iceberg has just emerged[J].Trendsinplantscience, 2003, 8(10):468-471.
[8] BRYANT H E, WADD S E,etal. Herpes simplex virus IE63 (ICP27) protein interacts with spliceosome-associated protein 145 and inhibits splicing prior to the first catalytic step[J].JournalofVirology, 2001, 75(9):4 376-4 385.
[9] DAS B K, XIA L,etal. Characterization of a protein complex containing spliceosomal proteins SAPs 49, 130, 145, and 155[J].MolecularandCellularBiology, 1999, 19(10):6 796-6 802.
[10] HASHIZUME C, KURAMITSU M,etal. Human immunodeficiency virus type 1 Vpr interacts with spliceosomal protein SAP145 to mediate cellular pre-mRNA splicing inhibition[J].MicrobesandInfection, 2007, 9(4):490-497.
[11] RYMOND B C. Rds3p is required for stable U2 snRNP recruitment to the splicing apparatus[J].MolecularandCellularBiology, 2003, 23(20):7 339-7 349.
[12] WILL C L, SCHNEIDER C,etal. A novel U2 and U11/U12 snRNP protein that associates with the pre-mRNA branch site[J].TheEMBOJournal, 2001, 20(16):4 536-4 546.
[13] 曾纪晴, 张明永. 选择性剪接在植物逆境相关基因表达调控中的作用[J]. 植物生理学通讯, 2006, 42(6):1 005-1 014.
ZENG J Q, ZHANG M Y. The role of alternative splicing in the regulation of plant stress-associated gene expression[J].PlantPhysiologyCommunications2006, 42(6):1 005-1 014.
[14] IIDA K, SEKI M, SAKURAI T,etal. Genome-wide analysis of alternative pre-mRNA splicing inArabidopsisthalianabased on full-length cDNA[J].NucleicAcidsResearch, 2004, 32(17):5 096-5 103.
[15] 林婵婵. 17S U2 snRNP中不可或缺的剪接因子SF3a的研究进展[J]. 安徽农业科学, 2014, 41(15): 6 604-6 607.
LIN C C. Research progress of indispensable splice factor SF3a in 17S U2 snRNP[J].JournalofAnhuiAgri.Sci, 2014, 41(15): 6 604-6 607.
[16] 曲瑞莲, 吴春霞, 冯献忠. mRNA选择性剪切在植物发育中的作用[J]. 植物生理学报, 2014, 50(6): 717-724.
QU R L, WU C X, FENG X Z.Function of mRNA alternative splicing in plant development[J].PlantPhysiologyJournal, 2014, 50(6): 717-724.
[17] 周新成, 王海燕, 卢 诚,等. 植物功能基因选择性剪接研究进展[J].热带农业科学, 2012, 32(2): 36-41.
ZHOU X C, WANG H Y, LU C,etal. Research advances in alternative splicing of functional genes.[J].ChineseJournalofTropicalAgriculture, 2012, 32(2): 36-41.
(编辑:宋亚珍)
Molecular Cloning and Expression Analysis ofTaSF3B2 from Wheat
HUANG Xin1, XIA Baicheng1, LONG Xiangyu2*
(1 Colleges of Agronomy, Hainan University, Haikou 570228, China; 2 Rubber Research Institute,Chinese Academy of Tropical Agricultural Sciences,Danzhou, Hainan 571737, China)
To study the function of splicing factor responses to abiotic stress, a cDNA sequence ofTaSF3B2 was isolated and analyzed from wheat EST database. The results showed thatTaSF3B2 contained a 1 854 bp ORF encoding 617 amino acids. Using the methods of bioinformatics to predict and analyze TaSF3B2, the protein has classic domain of splicing factor 3B subunit 2, high hydrophilia (GRAVY, -0.895) and heat stability (Instability index, 43.92). The results of real-time PCR revealed that the expression ofTaSF3B2 had different levels in different developments and tissues. In addition, the expression was induced markedly by drought, salt, low-temperature, ABA and yellow rust (CY32),with down-regulation in leaves of seedling, but no change in root. In summary, it is speculated that TaSF3B2 could play a role not only in growth and development conditions but also in biotic and abiotic stresses of wheat.
wheat;TaSF3B2; gene clone; bioinformatic analysis; gene expression
1000-4025(2016)08-1528-06
10.7606/j.issn.1000-4025.2016.08.1528
2016-03-15;修改稿收到日期:2016-07-18
国家转基因生物新品种培育重大专项(2011ZX08002001)
黄 鑫(1997-),女,本科生。E-mail:15501852343@163.com
*通信作者:龙翔宇,副研究员,主要从事植物分子生物学研究。E-mail:xiangyulong@gmail.com
Q785; Q786
A

