食醋中污染菌的分离与鉴定
翟磊 苏姣姣 刘洋 曹艳花 姚粟 程池
(中国食品发酵工业研究院 中国工业微生物菌种保藏管理中心,北京 100015)
食醋中污染菌的分离与鉴定
翟磊 苏姣姣 刘洋 曹艳花 姚粟 程池
(中国食品发酵工业研究院 中国工业微生物菌种保藏管理中心,北京 100015)
首次关于耐酸乳杆菌是引起食醋变质的报道,旨在为食醋生产企业进行微生物污染控制提供理论依据。采用PCRDGGE技术以及纯培养技术,对正常醋样和污染醋样的微生物群落结构和种类进行了研究,通过对比分析以及回接实验发现了引起食醋变质的污染菌为耐酸乳杆菌(Lactobacillus acetotolerans)。
PCR-DGGE;纯培养技术;耐酸乳杆菌(Lactobacillus acetotolerans)
食醋在中国拥有上千年的历史,因其具有色泽红棕,酸味柔和,醇香回甜,久陈不腐等特点,成为深受人们喜爱的一种调味品。食醋主要是以含淀粉、糖或乙醇的原料发酵酿造而得,通过微生物的代谢作用产生的众多酶系催化实现醋酸等基本风味物质的转化,本质上是一个微生物菌种混合发酵的复杂过程。目前国内关于食醋微生物的研究主要集中在传统食醋酿造过程中微生物群落的多样性及功能性方面。食醋中醋酸的形成包括淀粉转化为乙醇和乙醇转化为乙酸两个过程,其中淀粉质原料的糊化、糖化及酒化主要是通过霉菌和酵母菌以及分泌的多种水解酶系的共同催化完成的,醋酸发酵阶段是在包括醋酸菌在内的众多细菌以及分泌的水解酶系作用下生成醋酸,形成食醋的主体风味[1-5]。
虽然微生物在酿醋过程中发挥着重要作用,但是食醋酿造过程中的微生物污染是亟待解决的难题。微生物污染后的食醋产生异味、出现颜色变浅、醋中固形物减少等现象,大大降低了食醋的营养价值。本研究采取PCR-DGGE 技术对正常醋样和污染醋样的微生物群落结构进行比较,研究发现引起食醋变质的潜在污染菌;在此基础上,采用可培养的方法对正常醋样和污染醋样的微生物进行分离纯化及鉴定,结合污染菌回接试验以期分离得到引起食醋变质的污染菌,旨为食醋生产企业进行微生物污染控制提供参考。
1 材料与方法
1.1 材料
某食醋生产企业食醋生产过程中的正常醋样和污染醋样。
1.2 方法
1.2.1 样品理化特征比较 从醋样颜色、气味、可溶性固形物、pH值和CO2含量方面对正常醋样和污染醋样的理化特征进行比较。
1.2.1.1 可溶性固形物含量测定 取正常醋样和污染醋样各20 mL,10 000×g,离心10 min后弃掉上清,比较固形物含量。
1.2.1.2 pH值测定 取正常醋样和污染醋样各20 mL至于灭菌的烧杯中,使用pH计直接测定。
1.2.1.3 CO2含量测定 取正常醋样和污染醋样瓶中的气体,使用气相色谱法测定。检测条件为:柱温箱170℃,进样器温度120℃,检测器温度120℃,分析柱为TDX-01,载气为He。
1.2.2 样品微生物群落结构分析 利用PCR-DGGE对正常醋样和污染醋样的微生物群落进行比较分析[6-8]。
1.2.2.1 样品中微生物总DNA提取 微生物总DNA提取采用试剂盒方法进行,试剂盒购自天根生化科技(北京)有限公司。提取获得的总DNA用0.8%琼脂糖凝胶电泳检测,并置于-20℃保存备用。
1.2.2.2 PCR扩增 用通用引物341f-GC和534r进行16S rRNA基因V3可变区片段的扩增。341f-GC:5'-CGCCCGCCGCGCCCCGCGCCCGTCCCGCCGCCCC CGCCCGCCTACGGGAGGCAGCAG-3'(40个碱基GC夹);534r:5'-ATTACCGCGGCTGCTGG-3'。PCR反应50 μL体系包括10×PCR buffer(不含Mg2+)5.0 μL,dNTPs(2.5 mmol/L)4 μL,Taq酶(2.5 U/μL)1.2 μL,模板各1 μL,上下游引物10 mmol/L各1 μL,无菌双蒸水补齐至25 μL。PCR反应程序如下94℃预变性5 min 后进入循环,94℃变性30 s,54℃退火30 s,72℃延伸45 s,35个循环,72℃再次延伸10 min。
1.2.2.3 变性梯度凝胶电泳(DGGE) 16S rRNA基因V3可变区片段的PCR扩增产物通过DGGE系统进行分离。丙烯酰胺凝胶浓度为8%,变性梯度为40%-60%。60℃恒温,80 V下电泳16 h,电泳完毕后用SYBR green I(1×TAE,1∶10 000)染色45 min,电泳结果通过凝胶成像系统(英国UVI)及Quantity one分析软件进行分析。
1.2.2.4 DGGE条带的回收、重新扩增、克隆、转化、序列测定及分析 在波长为 354 nm 紫外线下,切取DGGE 凝胶上的主要条带,溶于 50 μL 无菌去离子水中,4℃过夜溶解。重扩增,扩增产物连接到载体pEASY-T1(TRANS),转入 E. coli 感受态细胞,在LB 固体培养基上 37℃ 筛选培养 16 h,挑取白色转化子用含有氨苄青霉素抗性的 LB液体培养基 37℃震荡培养 8 h。T-载体通用引物进行菌液 PCR,经琼脂糖凝胶电泳检测后,每个样品3个阳性克隆交由生工生物工程(上海)有限公司完成测序。所测得的16S rRNA基因V3可变区序列用DNASTAR软件去除载体序列和GC夹后,将有效序列在NCBI上进行比对,以其中同源性最高的序列为参考序列,相似性≥97%的序列归为同一操作分类单元(OTU)。
1.2.3 样品中可培养微生物的分离纯化与鉴定
1.2.3.1 样品中微生物的分离纯化 在限菌条件下量取25 mL正常醋样,放入盛有225 mL无菌水带玻璃珠的三角瓶中,充分振荡20 min,之后静置5 min。采用梯度稀释法制备10-2到10-6的系列稀释液,分别涂布到NA和MRS培养基中在37℃下培养2-7 d。挑单菌落转接到新鲜的上述培养基斜面上于4℃保藏。采取同样的方法对污染醋样中微生物进行分离纯化[9]。
1.2.3.2 样品中微生物的鉴定 以基因组DNA为模板,利用通用引物对16S rRNA基因进行扩增。引物序列为:正向引物799f(5'-AACAGGATTAGATACCCTG-3'),反向引物1492r(5'-GGTTACCTTGTTACGACTT-3')[10]。50 μL PCR反应体系:50 ng DNA模板,1×Taq reaction buffer,引物各20 pmol,20 μmol dNTP,1.5单位Taq酶(Ferments)。反应程序为:94℃预变性5 min,94℃变性1 min,52℃复性1 min,72℃延伸1 min,30个循环后72℃延伸10 min。PCR扩增产物用1%的琼脂糖进行检测。纯化后的PCR产物用ABI3700基因测序仪测序。测序由北京诺赛基因组研究中心有限公司完成。测序结果用Chromas软件参照正反序列图谱人工校对。将测序得到的结果在EzTaxon server 2.1进行比对[11],确定与已知序列的同源关系。
1.2.4 食醋中污染菌的回接实验 将分离得到的食醋污染菌接种到正常醋样中,37℃厌氧培养7 d,观察醋样变化。
2 结果
2.1 正常醋样和污染醋样理化特征
正常醋样和污染醋样的理化特征对比结果如表1所示。
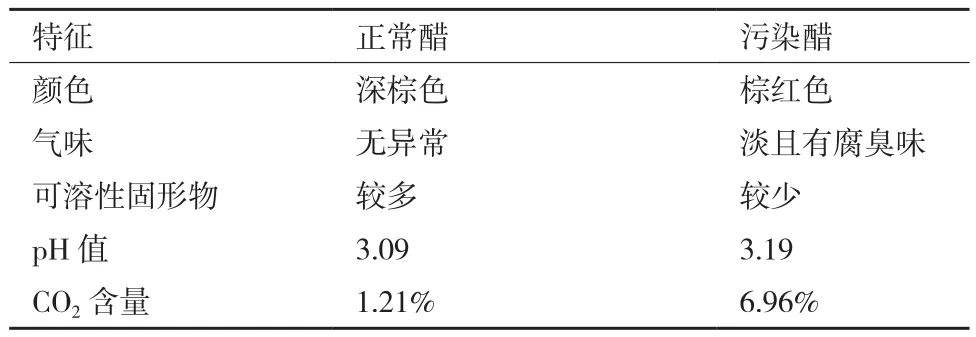
表1 正常醋样和污染醋样特征对比
2.2 正常醋样和污染醋样PCR-DGGE图谱分析
将正常醋样和污染醋样的总DNA经PCR扩增获得大小约为200 bp的16S rRNA基因V3可变区的序列。将PCR扩增产物进行DGGE检测,获得正常醋样和污染醋样中微生物群落结构的DGGE指纹图谱(图1),每条泳带代表一种样品中微生物DGGE指纹图谱,不同位置的条带理论上应代表不同种类的细菌,条带的荧光强度则反应了该细菌的含量,条带信号越亮,表示该种细菌的相对数量越多。如图1表明,正常醋样和污染醋样中之间微生物群落结构存在一定的差异,但优势菌条带相对比较稳定。

图1 正常醋样和污染醋样中微生物DGGE分离图谱及分析示意图

表2 优势条带测序比对分析结果
2.3 正常醋样和污染醋样微生物群落结构比较分析
为进一步研究正常醋样和污染醋样中微生物群落结构组成成分,经DGGE电泳后,对其中6个优势条带进行切胶、回收、克隆、测序(表2)。测序结果经GenBank数据库比对后,序列相似性均大于97%,比对结果显示正常醋样中出现的条带1和4均鉴定为木葡糖酸醋杆菌(Gluconacetobacter xylinus),葡糖酸醋杆菌是制醋过程中产生醋酸的功能微生物之一,能够将酵母菌分解产生的葡萄糖和果糖转化为醋酸、葡萄糖酸等产物,同时分解醋酸酶,将乙醇氧化为醋酸。而污染醋样中出现的条带2,3和5,6尽管电泳的位置有所差别,但是均鉴定为耐酸乳杆菌(Lactobacillus acetotolerans),这可能是由于少数碱基变异造成的。在生产过程中,耐酸乳杆菌能够将葡萄糖等底物转化为醋酸和乳酸,丰富醋的风味。一旦其出现在成品醋中就会引起醋的变质和腐败。通过PCR-DGGE分析发现,污染醋样中存在的耐酸乳杆菌(Lactobacillus acetotolerans)是引起食醋的潜在污染菌。
2.4 正常醋样和污染醋样可培养微生物的分离纯化与鉴定
采用PCR-DGGE技术分析微生物群落结构发现耐酸乳杆菌(Lactobacillus acetotolerans)是潜在的引起食醋变质的污染菌,为了进一步确认,需要采用稀释梯度平板法对正常醋样和污染醋样中的微生物进行纯培养。通过平板划线纯化和分子生物学鉴定,从正常醋样中分离得到了葡糖酸醋杆菌属(Gluconacetobacter sp.)菌株,蜡样芽孢杆菌群(Bacillus cereus group)菌株以及纤维微菌属(Cellulosimicrobium sp.)菌株;而污染的样品中分离得到了耐酸乳杆菌(Lactobacillus acetotolerans),短杆菌属(Brevibacterium sp.)菌株以及赖氨酸芽孢杆菌属(Lysinibacillus sp.)菌株(表3)。样品中可培养微生物的分离纯化与鉴定结果发现,污染醋样中含有引起醋样变质的潜在污染菌-耐酸乳杆菌,这也与非培养研究的结果一致。目前耐酸乳杆菌(Lactobacillus acetotolerans)已经保藏于中国工业微生物菌种保藏管理中心,编号为CICC 10774。
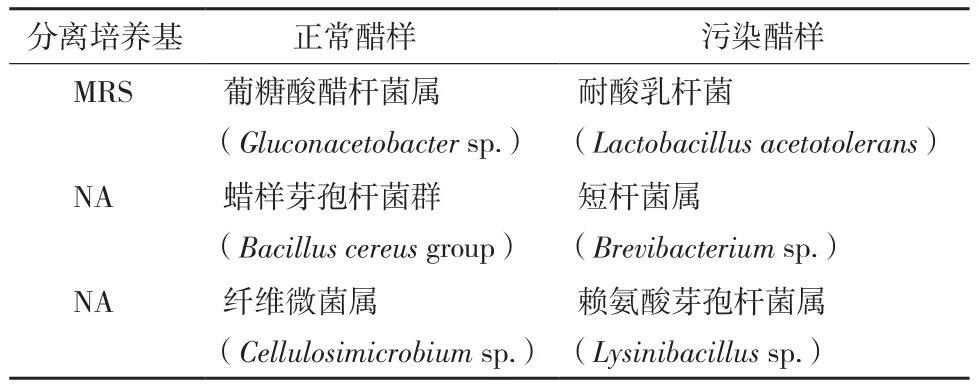
表3 正常醋样和污染醋样中可培养微生物
2.5 回接实验
为了进一步确认耐酸乳杆菌就是引起食醋变质的污染菌,将耐酸乳杆菌接种到正常醋样中进行回接试验。回接实验结果表明,耐酸乳杆菌的接入会引起正常醋样出现产气,淡腐,臭味颜色变浅,可溶性固形物减少以及pH值升高等变质现象。因此我们确定耐酸乳杆菌就是引起食醋变质的污染菌。
3 讨论
非培养技术是指借助于分子生物学方法,直接对样品的微生物结构进行分析,其中变性梯度凝胶电泳(DGGE)技术是最为常见的研究微生物群落结构组成的非培养分析方法,其基本原理为在含有浓度线形递增的变性剂的聚丙烯酰胺凝胶电泳中对PCR产物分离,部分解链的双链DNA分子的电泳迁移率降低,而序列不同的DNA 分子有着不同的解链行为,它们在凝胶的不同位置停止迁移从而使长度相同而序列不同的DNA 片段分离。广泛应用于研究湖泊、海洋、活性污泥、发酵食品及各种土壤等生态环境的微生态研究中[6-8]。可培养技术是根据样品特征,设计一系列合适的培养基从样品中分离得到微生物菌种的研究方法,包括微生物菌种的分离、纯化、鉴定等多种方法,是研究微生物菌种最为常用的分析方法。
本研究结合微生物的非培养技术和可培养技术,比较分析了正常醋样和污染醋样的微生物结构和种类,通过回接试验确定了耐酸乳杆菌(Lactobacillus acetotolerans)就是引起食醋变质的污染菌。Entan[12]在1986年首次从食醋发酵液中分离得到了耐酸乳杆菌。该菌革兰氏染色呈阳性,兼性厌氧,最适生长为37℃,这也是食醋生产企业夏季易发生微生物污染的原因。耐酸乳杆菌能够代谢产生醋酸并且适应高酸环境,在醋酸发酵生产过程中发挥着关键的作用,能够产生醋酸和乳酸等风味物质,是主要生产菌之一。然而,成品醋都需要经过高温灭菌工艺,如果灭菌不彻底耐酸乳杆菌出现在成品醋中,会使正常醋样出现产气、淡腐、臭味颜色变浅、可溶性固形物减少,以及pH值升高等变质现象,降低食醋的营养价值。此外,有关研究发现耐酸乳杆菌还是引起啤酒变质的主要污染菌,该菌能够引起啤酒失光、异味、变酸、形成浑浊和沉淀[13]。
4 结论
本研究采用PCR-DGGE技术以及纯培养技术,发现了导致食醋变质污染菌。通过分子生物学将污染菌鉴定为耐酸乳杆菌(Lactobacillus acetotolerans)。
[1] Haruta S, Ueno S, Egawa I, et al. Succession of bacterial and
fungal communities during a traditional pot fermentation of ricevinegar assessed by PCR-mediated denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology,2006, 109:79-87.
[2]许伟, 张晓君, 许泓瑜, 等. 镇江香醋醋酸发酵过程中细菌群落组成分析[J]. 微生物学通报, 2007, 34(4):646-649.
[3]聂志强, 汪越男, 郑宇, 王敏. 传统食醋酿造过程中微生物群落的多样性及功能研究进展[J]. 中国酿造, 2012, 31(7):1-6.
[4] 苏敬东. 传统酿醋过程中醋醅微生物时空动态变化规律研究[J]. 甘肃科技, 2011, 27(18):70-72.
[5] 崔云, 卢红梅, 张义明, 刘佳. 食醋中微生物的分离与鉴定[J].中国酿造, 2008, 22:29-31
[6]朱扬玲. 采用PCR-DGGE方法研究浙江玫瑰醋酿造过程中的微生物多样性[D]. 杭州:浙江工商大学, 2009.
[7]刘琳, 刘洋, 宋未. PCR-DGGE技术及其在植物微生态研究中的应用[J]. 生物学通报, 2009(3):54-56.
[8]梁新乐, 朱扬玲, 蒋予箭, 等. PCR-DGGE法研究泡菜中微生物群落结构的多样性[J]. 中国食品学报, 2008, 8(3):133-137.
[9]东秀珠, 蔡妙英, 等. 常见细菌系统鉴定手册[M]. 北京:科学出版社, 2001.
[10]Baker GC, Smith JJ, Cowan DA . Review and re-analysis of domainspecific 16S primers[J]. J Microbiol Methods, 2003, 55:541-555.
[11]Chun J, Lee JH, Jung Y, et al. EzTaxon:a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J]. Int J Syst Evol Micr, 2007, 57:2259-2261.
[12]Entan E, Masa H, Suzuki KI. Lactobacillus acetotolerans, a New Species from Fermented Vinegar Broth[J]. Int J Syst Evol Bact,1986, 36(4):544-549.
[13]田小群, 周世宁. 一株啤酒污染菌的分离鉴定及快速检测方法[J]. 中山大学学报:自然科学版, 2006 45(4):70-73.
(责任编辑 李楠)
Isolation and Identification of Microorganisms from Spoilage Vinegar
ZHAI Lei SU Jiao-jiao LIU Yang CAO Yan-hua YAO Su CHENG Chi
(China Center of Industrial Culture Collection,China National Research Institute of Food and Fermentation Industries,Beijing 100015)
The structure and diversity of microbial community in normal and spoilage vinegar samples were studied by PCR-DGGE technology combined with pure culture technology. By comparative analysis and tie-back test,we found that Lactobacillus acetotolerans was the polluting strain causing vinegar spoilage. This is the first domestic reports regarding that L. acetotolerans was the cause of the vinegar spoilage and provided theoretical basis for the enterprises of vinegar production to control the microbial pollution.
PCR-DGGE;pure culture technology;Lactobacillus acetotolerans
10.13560/j.cnki.biotech.bull.1985.2016.03.031
2015-05-15
国家微生物资源平台专项(NIMR2015-4),中国食品发酵工业研究院科技发展基金(博士基金)项目(2015KJFZ-BS-04)
翟磊,男,博士,研究方向:微生物学;E-mail:zhailei@china-cicc.org
程池,男,教授级高工,研究方向:微生物学;E-mail:cheng100027@163.com

