基于不同测序技术的生物群落结构及功能菌分析
蔡言安,李 冬,毕学军,曾辉平,张 杰,3
基于不同测序技术的生物群落结构及功能菌分析
(1.青岛理工大学环境与市政工程学院,山东 青岛 266033;2.北京工业大学水质科学与水环境恢复工程北京市重点实验室,北京 100124;3.哈尔滨工业大学城市水资源与水环境国家重点实验室,黑龙江 哈尔滨 150090)
分别采用克隆文库和高通量测序技术,解析生物去除铁锰氨滤池内微生物的群落结构和功能菌多样性,并探讨不同测序手段的差异.高通量测序获得15057条有效序列、共32个分类纲,克隆文库测序涵盖9个具有明确分类地位的纲(75条序列),前者能揭示更为丰富的细菌群落结构多样性.功能菌(铁锰氧化细菌和硝化细菌)分析过程中,一些功能菌属在克隆文库中出现,而在高通量测序中未检测到,反之亦然.与单一的测序手段相比,二者相结合能更好地揭示功能细菌的分布特点.
生物滤池;硝化作用;高通量测序;克隆文库
在生物水处理领域,生物反应器内的功能微生物是研究的热点.分子生物学技术手段的应用,使研究者对生物反应器的运行效果、内在反应机理、微生物群落演替等有了深入地了解.
聚合酶链式扩增反应(PCR)是分子生物技术分析中至关重要的一步,其目的是采用引物扩增目标细菌的16S rRNA分子片段或功能基因片段,实现对微生物样品中功能细菌的快速定性定量分析.分子片段特异引物可定向扩增某一菌属内的细菌,而功能基因扩增引物却能扩增生态功能相近、在亲缘关系较远的多个种属内的细菌[1].例如,功能基因引物Amoa1F/Amoa2R可扩增好氧氨氧化菌AOB菌属(、)的功能基因片段;NIT3r/338f可定向扩增的16S rRNA基因片段、Ntspa685r/338f定性扩增的16S rRNA基因片段[2-3].
对于铁锰氧化细菌,采用16S rRNA分子片段特异性引物或功能基因扩增引物研究其结构多样性均存在一定的困难.首先,所设计的16S rRNA分子片段扩增引物(PS-1/PSP-6)多针对于属,无法同时分析其他的铁锰氧化菌属(包括以及等),并且该引物可非特异性扩增非属的细菌[4].其次,多铜氧化酶基因()被认为是参与生物锰氧化的功能基因,但其扩增引物(CumAF/CumARdg)同样存在非特异性扩增的缺陷[5].
因此,考虑到上述铁锰氧化菌扩增引物存在的一系列问题,本文基于细菌通用引物,分别采用克隆文库和高通量技术分析生物滤池内铁锰氧化菌和硝化细菌群落结构多样性,并对比探讨了二者的各自分析特点.
1 材料方法
1.1 生物滤池反应器
生物滤池反应器由圆柱体有机玻璃制成,总高度3m、内径185mm;石英砂为滤料、滤层高度130cm,重力流过滤、液上水头保持30~40cm.生物滤池已成熟运行数年[6],本实验过程中,模拟进水铁、锰、氨氮浓度分别为1.5~2.5mg/L、0.6~ 0.8mg/L、1.0~1.2mg/L,生物滤池对污染物去除率均保持在95%以上.
1.2 基因组DNA提取
DNA提取过程:自生物滤层内取10g泥砂混合滤料,置于10mL灭菌离心管中,分别加入2.7mLDNA提取液、50μL蛋白酶K (Proteinase K,30g/L)、溶菌酶(lysozyme,20g/L);将离心管在37℃水浴振荡器中震荡30min、转速22;随后加入0.3mL浓度20%的SDS、水浴65℃、2h.
将上述离心管室温冷却后8000r/min离心10min,取上清液转移至另一离心管、记录体积;加入等体积氯仿/异戊醇(24:1)混合、8000r/min离心10min;吸取上清液至另一离心管.继续加入0.6倍体积的异丙醇,–20℃过夜后10000r/min离心5min;去上清液后继续10000r/min、离心5min;再次尽量吸去上清液,将离心管内沉淀物置于通风橱内晾干;最后,以300~500μLTE缓冲液溶解后,收集至1.5mL离心管保存.
琼脂糖凝胶电泳检测得到的基因组DNA溶液,若DNA分子跑胶拖带严重,则采用基因组纯化试剂盒予以纯化.
1.3 PCR扩增过程
以上述所得基因组DNA溶液为模板、1492R(5‘-TACCTTGTTAYGACTT-3’)和27F (5‘-AGAGTTTGATYMTGGCTCAG-3’)为特异引物,进行PCR扩增.按照TaKaRa酶说明书配制PCR反应体系50μL,扩增采用降落式PCR程序:初始94℃变性5min、30个循环扩增:94 ℃变性45s、58℃退火45s(每个循环退火温度降低0.1℃)、72℃延伸1min,72℃延伸7min.
扩增均设3次平行,所获得PCR反应产物片段经1.5%琼脂糖凝胶电泳检测后合并于一只离心管内.采用纯化试剂盒对PCR产物进行切胶回收后,再次采用1.5%琼脂糖凝胶电泳检测回收片段.最终的目的基因片段于4℃条件下保存,并于2d内完成后续克隆测序试验.
1.4 克隆文库构建
克隆采用的试剂盒与感受态细胞试剂盒均购自TaKaRa公司,按照操作说明书对上述所获得的PCR产物进行克隆连接.克隆菌株经蓝白斑后,选取阳性菌株过夜摇菌培养、菌液送至商业生物公司测序(生工,上海).
将测序序列采用软件RDP classifier version 2.6进行分类地位划分,相似度大于97%的序列被划分为一个(OTU),同时与基因数据库中序列比对分类地位最接近的菌株(http://blast.ncbi.nlm.nih.gov/Blast.cgi).Shannon指数通过EstimateS软件计算获得.
1.5 高通量测序
Illumina Miseq测序平台测序通量高、快速经济,其单个微生物样品分析的商业成本与克隆文库相当.高通量库容通常可提供上万条序列数,而克隆文库的库容却含有数十条或数百条序列,高通量的单个序列成本远低于克隆文库.
高通量测序由商业服务公司完成(生工,上海).基于MiSeq测序平台(美国Illumina公司),以所提取基因组DNA为模板,341F (5‘-CCTACG- GGNGGCWGCAG-3’)和805R (5‘-GACTACH- VGGGTATCTAATCC-3’)为引物,扩增细菌16S rRNA基因 V3-V4区序列.共获得15057条有效序列,将相似度大于97%的序列划分为一个OUT,采用mothur软件(http://www.mothur.org/)计算Shannon指数并在线比对(http://www.arb- silva.de) 分类地位最接近的菌株.
2 结果与讨论
2.1 生物群落结构对比分析
指标通常用以估算生物群落结构多样性,其值高低代表群落结构多样性的高低.克隆文库分析过程中,通过EstimateS软件分析得到的指标曲线相对比较平滑,表明序列条数已覆盖样品内的大部分微生物,其Shannon值为3.29;高通量分析过程中, Shannon曲线不再显著变化时,其值为5.27.由Shannon值分析可知,采用高通量测序技术更能体现微生物群落结构的多样性.
图1(a)中,在纲的OTU分类水平上,克隆文库中克隆子序列(75条)共覆盖12个纲、其中9个纲有明确的分类地位:α变形纲(Alphaproteobacteria,30.7%)、β变形纲(Betaproteobacteria,16%)、γ变形纲(Gammaproteobacteria,13.3%)、硝化螺旋菌纲(Nitrospira,12%)、δ变形纲(Deltaproteobacteri- a,5.3%)、黄杆菌纲(Flavobacteria,4%)、酸杆菌纲(Acidobacteria,4%)、丰佑菌纲(Opitutae,2.7%)、鞘脂杆菌纲(Sphingobacteria,1.3%).
图1(b)中,高通量所得序列(15057条)涵盖32个纲,其中15个纲的序列均比例大于0.6%、占总纲的96.1%,可认为此15类纲是微生物的优势类群.分析发现,这15个分类纲中包含了克隆文库所获得的9个分类纲;其中α变形纲占23.3%、β变形纲20.5%、γ变形纲6.2%、硝化螺旋菌纲20.2%、δ变形纲1.3%、黄杆菌纲1.2%、酸杆菌纲3.2%、丰佑菌纲0.9%、鞘脂杆菌纲12.4%,占总纲比例为89.2%,表明上述9类纲在15个主导纲中占有主体地位.因此,与克隆文库技术相比,高通量技术在读取序列方面具有信息量大的优点,使群落结构展现出更为丰富的多样性、且Shannon值显著提高,但克隆文库所获得的微生物群落结构同样能体现出微生物群落的主体结构,尽管两者的具体比例值有所不同.
生物滤池内的功能细菌,包括硝化细菌、铁锰氧化细菌,是滤池发挥效能的关键因子.对比分析克隆文库和高通量测序技术在解析微生物群落结构方面差异的同时,仍需探究不同分析方法对功能菌检测的影响作用.
2.2 硝化细菌分析
硝化细菌包括好氧氨氧化细菌(AOB)和亚硝酸盐氧化细菌(NOB),是生物滤池内氨氮去除的功能细菌,表1统计了2种分析方法对AOB和NOB的检测情况.
AOB常见的菌属包括亚硝化单胞菌属()和亚硝化螺旋菌属(),二者均出现在克隆文库内,在高通量分析库中却并未出现与亚硝化螺旋菌属相关的细菌序列.Li等[7]同样采用克隆文库和T-RFLP技术手段分析了铁锰氨净化滤池内生物群落结构,并没有检测到AOB,且认为氨氮的氧化归功于其他种类的微生物.一方面,本文试验结果进一步证明了生物滤池内存在与氨氮氧化相关的AOB菌;另一方面,结果表明不同测序技术在功能菌分析方面存在差异.
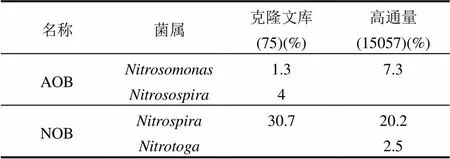
表1 基于克隆文库与高通量测序的硝化细菌分析Table 1 Analysis of nitrifiers in the processes of clone library and high throughout sequencing
分析NOB可知,硝化螺旋菌属()在克隆文库和高通量测序库中均占有极高的比例,分别高达30.7%、20.2%,而常见的硝化菌属()却未检测到.有研究表明这是由于不同类型反应器对菌群不同的优势选择造成的[8-9].此外,是一种新型的亚硝酸氧化菌属[10],在高通量序列中有2.5%的序列划分在此属内;而克隆文库中未检测到此类菌群.
此外,克隆文库分析过程中NOB序列数所占比例显著高于AOB,二者比例分别为30.7%、5.3%;高通量分析过程中二者比例分别为22.7%、7.3%. 由于氨氧化古菌(AOA)亦能将氨氮氧化为亚硝酸盐氮[11], AOA可能存在于生物滤池内, 而本研究中未对AOA予以分析.因此,今后研究AOB、AOA、NOB三者的相对比例能更好的体现参与硝化过程的功能微生物.
2.3铁锰氧化菌分析
中性pH值水体条件下,铁在滤层内以化学溶解氧接触氧化去除为主,而锰仅可由生物氧化作用去除,表2统计了与铁锰氧化相关的菌群.
嘉利翁氏菌属()是一类铁氧化相关的菌,仅能氧化铁离子.铁极易被溶解氧化学接触氧化,有研究发现高达94%以上的铁是通过化学途径去除[12],但克隆文库中检测到嘉利翁氏菌属,表明生物氧化作用不可忽视,与Michalakos等[13]的反应器运行数据表现一致;但该类菌的序列在高通量序列中未检测到.
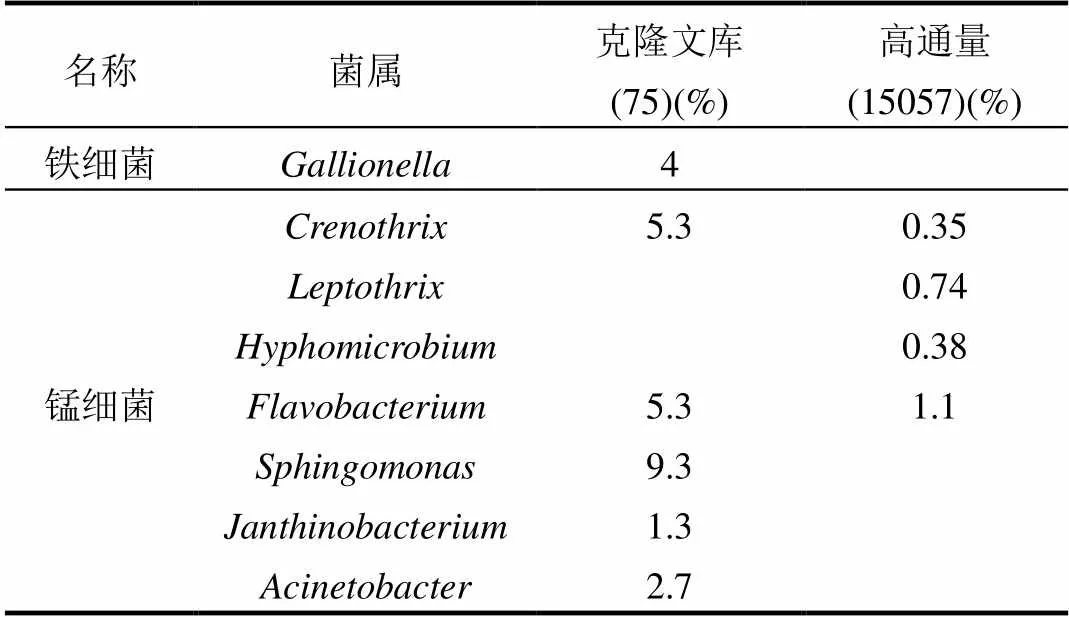
表2 基于克隆文库与高通量测序的铁锰细菌分析Table 2 Analysis of Fe- and Mn-related bacteria in the processes of clone library and high throughout sequencing
泉发菌属()、纤发菌属()与生丝微菌属()是常见的锰氧化菌属,三者同时出现在高通量序列中,而仅有泉发菌属出现克隆文库中.Hope等[14]认为纤发菌属是反应器内锰氧化的主体优势菌属,但Burger等[15]基于四座实际规模生物滤池分析了锰氧化细菌,认为锰的去除与纤发菌属的存在没有直接联系、其他菌属亦可完成锰的去除,这与本文高通量测序结果相一致,即泉发菌属、纤发菌属、生丝微菌属共同参与了锰氧化过程.
另外,一些与鞘氨醇单胞菌属()、黄杆菌属()、紫色杆菌属()、不动杆菌属()相关的细菌被发现具有锰氧化能力,并且这些细菌多被发现在锰沉积物或锰矿环境中[16-17],在净水领域中鲜有报道.与这些菌属相关的细菌均出现在克隆文库中,而仅有黄杆菌出现在高通量序列中.
总体而言,在功能菌分析过程中,一些菌属在克隆文库中出现,而在高通量测序中未出现,反之亦然.分析其原因,这两种测序技术不能完全将其表达;其次,由PCR扩增过程的随机性和扩增效率不同造成的.因而,采用克隆文库和高通量测序相结合的技术手段可以相互弥补、更全面的解析功能菌的多样性.
3 结论
3.1 与克隆文库技术相比,高通量技术测序数量大、微生物分类丰富,更能体现微生物群落结构多样性,但前者仍能体现微生物的优势群落结构.单一的技术手段在解析功能细菌(硝化细菌和铁锰氧化细菌)方面存在差异,克隆文库与高通量技术相结合可更全面了解功能细菌的分布.
3.2 亚硝化单胞菌与亚硝化螺旋菌参与了氨氮氧化,硝化螺旋菌属与新型的硝化菌属参与了亚硝酸盐的氧化.一些在净水领域鲜有报道的铁锰氧化菌属,如鞘氨醇单胞菌属、黄杆菌属、紫色杆菌属、不动杆菌属等在本研究中均被检测到.
梁丽华,左剑恶.反硝化功能基因-检测反硝化菌种群结构的分子标记 [J]. 微生物学通报, 2009,(4):627-633.
刘 涛.基于亚硝化的全程自养脱氮工艺(CANON)效能及微生物特征研究 [D]. 哈尔滨:哈尔滨工业大学, 2013.
Regan J M, Harrington G W, Noguera D R.-and nitrite-oxidizing bacterial communities in a pilot-scale chloraminated drinking water distribution system [J]. Applied and Environmental Microbiology, 2002,68(1):73-81.
Siering P, Ghiorse W.-targeted probes for detection of iron-and manganese- oxidizing sheathed bacteria in environmental samples [J]. Applied and Environmental Microbiology, 1997,63(2):644- 651.
Francis C A, Tebo Bmulticopper oxidase genes from diverse Mn(II)-oxidizing and non-Mn(II)-oxidizingstrains [J]. Applied and Environmental Microbiology, 2001,67(9): 4272-4278.
蔡言安,李 冬,曾辉平,等.[J]. 中国环境科学, 2014,34(8):1993-1997.
Li X K, Chu Z R, Liu Y J, et al.-scale biofilters treating iron, manganese and ammonia containing groundwater in Harbin, China [J]. Bioresource Technology, 2013(147):234-239.
Regan J M, Harrington G W, Baribeau H, et al.-scale chloraminated distribution systems [J]. Water Research, 2003,37(1):197-205.
Liu T, Li D, Zeng H, et al. Distribution and genetic diversity of functional microorganisms in different CANON reactors [J]. Bioresource Technology, 2012(123):574-580.
Alawi M, Lipski A, Sanders T, et al. Cultivation of a novel cold-adapted nitrite oxidizing betaproteobacterium from the Siberian Arctic [J]. ISME Journal, 2007,1(3):256-264.
鲍林林,陈永娟,王晓燕.北运河沉积物中氨氧化微生物的群落特征 [J]. 中国环境科学, 2015,35(1):179-189.
Tekerlekopoulou A G, Vayenas D V. Ammonia, iron and manganese removal from potable water using trickling filters [J]. Desalination, 2007,210(1-3):225-235.
Michalakos G D, Nieva J M, Vayenas D V, et al. Removal of iron from potable water using a trickling filter [J]. Water Research, 1997,31(5):991-996.
Hope C K, Bott T R. Laboratory modelling of manganese biofiltration using biofilms of[J]. Water Research, 2004,38(7):1853-1861.
Burger M S, Krentz C A, Mercer S S, et al. Manganese removal and occurrence of manganese oxidizing bacteria in full-scale biofilters [J]. Journal of Water Supply Research and Technology, 2008,57(5):351-359.
Carmichael M J, Carmichael S K, Santelli C M, et al. Mn (II)-oxidizing bacteria are abundant and environmentally relevant members of ferromanganese deposits in caves of the upper Tennessee River Basin [J]. Geomicrobiology Journal, 2013,30(9): 779-800.
Miller A Z, Hernández-Mariné M, Jurado V, et al. Enigmatic reticulated filaments in subsurface granite [J]. Environmental Microbiology Reports, 2012,4(6):596-603.
* 责任作者, 教授, cya_yanan@163.com
Analysis of microbial community structure and functional bacteria in a biofilter with different sequencing technologies
CAI Yan-an1, LI Dong2*, BI Xue-jun1, ZENG Hui-ping2, ZHANG Jie2,3
(1.School of Environmental and Municipal Engineering, Qingdao University of Technology, Qingdao 266033, China;2.Key Laboratory of Beijing Water Quality Science and Water Environment Recovery Engineering, Beijing University of Technology, Beijing 100124, China;3.State Key Laboratory of Urban Water Resource and Environment, Harbin Institute of Technology, Harbin 150090, China)., 2016,36(6):1830~1834
Effects of the technologies of clone library and high throughout sequencing for revealing microbial community structure and functional bacteria in a biofilter which simultaneously removes iron, manganese and ammonia were compared. 15057 sequences with 32 classifications in class level were obtained after the high throughout sequencing process, while there are 9classified groups in clone library. The former revealed more diversity of bacterial community structure. However, in the functional bacteria analysis process, some nitrifiers or iron and manganese bacteria which were detectable in one sequencing analysis process were not appeared in the other process. Comparing with using these two technologies individually in the analysing process, the combined using can compensate for each other and reveal the functional bacteria better.
biofilter;nitrification;high throughout sequencing;clone library profiling
X522
A
1000-6923(2016)06-1830-05
蔡言安(1986-),男,山东高密人,讲师,博士,主要从事水污染控制与水质保障技术研究.发表论文13篇.
2015-11-28
国家自然科学基金优秀青年科学基金(51222807)

