基于PCR技术的木材腐朽菌鉴定方法的研究
冯 璐,戚大伟
(东北林业大学 理学院,哈尔滨 150040)
基于PCR技术的木材腐朽菌鉴定方法的研究
冯璐,戚大伟*
(东北林业大学 理学院,哈尔滨 150040)
木材腐朽菌侵染木材而造成的木材腐朽现象会对木材的质量有着很大的影响,为了鉴定木材腐朽的病原菌,基于PCR技术设计了一种快速鉴定木材腐朽病原菌的方法。首先从腐朽木材上分离纯化得到两种病原菌,之后对两种病原菌进行实验室培养,通过观察其培养特性对其进行形态学鉴定。随后设计了7组引物,对两种病原菌进行了PCR扩增,并通过BLAST对比鉴定出这两种木材腐朽病原菌。研究结果表明,木材腐朽菌A为彩绒革盖菌,木材腐朽菌B属于多孔菌属真菌。分子生物学鉴定结果与形态学鉴定结果一致,而基于PCR技术的的鉴定方法更为简便快捷且精确,证明了该方法的可行性。
木材腐朽病原菌;PCR;引物设计;分子水平鉴定
0 引 言
木材在人类生产生活的各个方面有着非常重要的作用,但是随着经济的发展木材资源已经供不应求,保证木材的质量显得尤为重要。在影响木材质量的众多因素中,由木材腐朽菌侵染木材而引起的木材腐朽现象十分常见。木材腐朽发生频率高并且影响范围广,对木材质量造成不可逆的破坏,每年由木材腐朽引起的木材资源流失数目庞大,对人们的生产和生活也带来了严重的影响。为了合理使用木材资源,减小由木材腐朽菌造成的大面积木材腐朽,人们对木材腐朽和木材腐朽菌作了深入的研究[1]。目前鉴定木材腐朽菌的方法主要有两种,一种是传统的形态学鉴定方法,即通过观察菌落宏观培养特性和微观培养特性的特征来对菌种进行鉴定和分类;另一种是近年来发展起来的分子水平的鉴定方法[2],该方法依赖于PCR技术,通过扩增未知菌种的DNA序列,测序后与基因库内已知菌种进行比对来鉴定木材腐朽菌。PCR在物种鉴定上的应用集中在医学病原菌的鉴定、农副产品的鉴定上,而将PCR技术运用到木材腐朽的病原菌上的研究才刚刚开始[3-9]。
1 木材腐朽菌形态学鉴定
1.1木材腐朽菌的采集和筛选
菌种来源:菌种采集于东北林业大学林场,从人工水曲柳林病株上采集新鲜子实体,并在实验室内进行培养。
用75%的酒精对新鲜的子实体进行消毒后在无菌环境下将其切割成0.2 cm3的组织块,用70%酒精对组织块表面进行消毒,之后放入0.1%升汞消毒液中浸泡2 min,立即用无菌水冲洗3次,然后用镊子接种到PDA斜面培养基上,将斜面培养基放置于25℃生物培养箱中进行培养并每天观察菌种的生长状况,当菌丝布满斜面时即完成菌种的分离[10-11]。经过分离获得了A、B两种木材腐朽菌。
1.2木材腐朽菌的生物学特性
1.2.1温度
选取5种不同温度对两种真菌进行培养,分别为15、20、25、30、35℃[12],每组设置5个重复,控制其他培养基成分相同(马铃薯200 g,葡萄糖20 g,琼脂粉15 g,蒸馏水1 000 mL,pH自然,121℃灭菌20 min),之后将已接种的各组培养皿置于不同温度的生物培养箱中自然光条件下进行培养,观察并记录A、B两菌种的生长情况,记录第7天时每组菌落的平均直径。
1.2.2pH值
选取5种不同酸碱度的PDA培养基对两种真菌进行培养,分别为pH 6、pH 6.5、pH 7、pH 7.5、pH自然(经测pH=6.8)[13],每组设置5个重复,控制其他培养基成分相同(马铃薯200 g,葡萄糖20 g,琼脂粉15 g,蒸馏水1 000 mL,121℃灭菌20 min),之后将已接种的各组培养皿放置于25℃生物培养箱中进行培养,观察并记录A、B两木材腐朽菌菌种的生长情况,记录第7天时每组菌落的平均直径。
1.2.3光照强度
分别将生物培养箱的光照强度设置为0、1 000、2 000、3 000 lux[12],每组设置5个重复,控制其他培养基成分相同(马铃薯200 g,葡萄糖20 g,琼脂粉18 g,蒸馏水1 000 mL,温度25℃,pH自然,121℃灭菌20 min),之后将接种的培养基置于不同温度的生物培养箱中自然光条件下进行培养,观察并记录A、B两菌种的生长情况,记录第7天时每组菌落的平均直径。
1.3木材腐朽菌的形态学特性
1.3.1菌落特征
当菌丝生长至接近培养皿边缘10 mm时打开培养皿上盖[7],将培养皿至于水平台上,用普通数码相机拍摄菌落此时的生长状况。
1.3.2菌丝特征
制作菌丝体玻片,置于光学显微镜下进行观察。
2 基于PCR技术的分子生物学鉴定
2.1PCR引物的设计
引物的优劣直接关系到PCR扩增的成功与否。对引物的设计不可能有一种包罗万象的规则确保PCR的成功,但遵循某些原则,则有助于引物的设计。用特异性引物扩增ITS区段这种方法允许所扩增ITS区段的测序结果存在一定的误差,由于木材腐朽菌ITS区段的长度一般都在400bp以上,所以少量误差并不会影响最终鉴定结果[14-17]。在对ITS区段的DNA序列进行分析时,即使DNA序列数据库中没有被鉴定菌株所属物种的DNA序列,通过BLAST软件搜索出的物种也应是数据库中与被鉴定物种最近缘的物种,结合经典鉴定方法也可以对菌种做出正确的鉴定。
本文共设计了7对引物对供试材料的rDNA ITS序列进行PCR扩增。这7对引物分别是:Fung / ITS4,ITS1 / ITS4,NS1 / NS4,OPAA11 / OPAA18,OPD18 / OPD15,OPAA17 / OPO15,OPAA11 / OPD18。
各引物序列如下:


2.2反应条件的设计与优化
PCR反应体系(20 μl)如下:

注:不足20 μl的体积用dd H2O补足。每次反应均以无菌去离子水替代模板DNA作为空白对照[18]。
PCR扩增反应PCR仪上进行,反应时间大概两小时,热循环参数设定如下:

3 结果与分析
3.1形态学鉴定
3.1.1生物学特征
木材腐朽菌A的适宜生长温度为20~25℃,最适生长温度为25℃。15~25℃时,菌种的生长速率随着温度的升高而增高,25℃是达到最高,之后生长速率急速降低。木材腐朽菌B生长速率与木材腐朽菌A基本相同。不同温度下两种木材腐朽菌的生长速率如图1和图2所示。

图1 不同温度下菌落A的生长速率Fig.1 The growth rate of colony A under different temperature
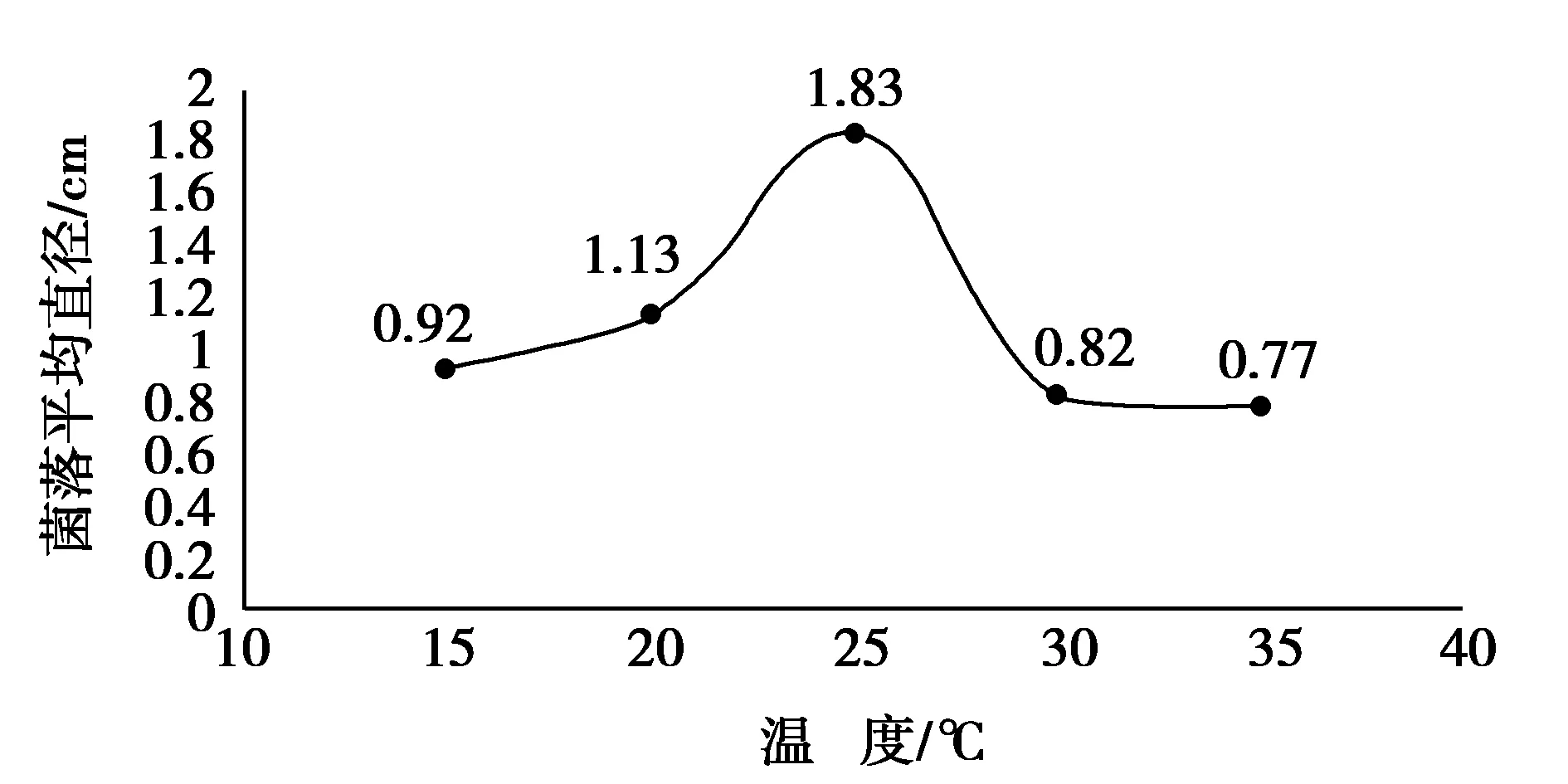
图2 不同温度下菌落B的生长速率图Fig.2 The growth rate of colony B under different temperature
两种木材腐朽菌的最适生长pH值均为7左右,但由于PDA培养基自然状态下(不用酸碱调节pH的情况下)pH约为6.8,此时两菌种的生长速率和pH=7时的生长速率非常接近,为了方便操作、节省资源,可以选择pH自然状态下进行培养。不同温度下两种木材腐朽菌的生长速率如图3和图4所示。
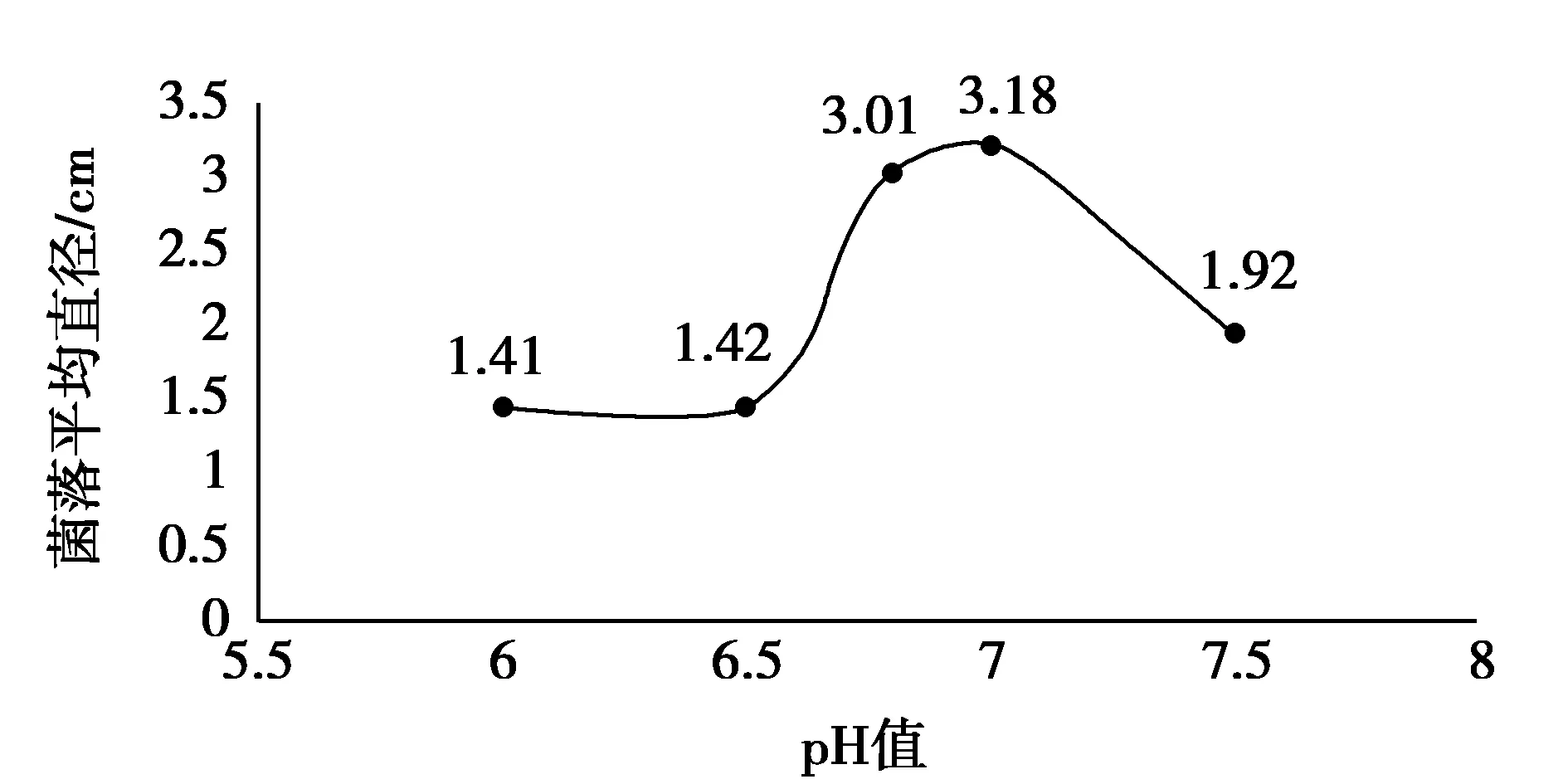
图3 不同pH下菌落A的生长速率Fig.3 The growth rate of colony A under different pH

图4 不同pH下菌落B的生长速率Fig.4 The growth rate of colony B under different pH
两种木材腐朽菌对光照不敏感,在不同的光照条件下生长速率基本相同,说明光照强度对两种木材腐朽菌的生长基本没有影响。
3.1.2形态学特征
对两种木材腐朽菌进行实验室培养,通过数码照相机照相后结果如图5和图6所示。

图5 木材腐朽菌A的菌落生长状况Fig.5 The colony’s growth condition of wood-decaying fungi A
菌种A:生长速率快,1~2周覆盖平板;菌落边缘为锯齿状;菌丝体紧贴于琼脂培养基表面生长;菌丝短而纤细,疏松地排列在培养基表面,形成一个绒毛状菌落;菌落保持白色或奶油色;表面较平滑;在6周前反面部分变褐;生长新区的边缘菌丝为升起的羊毛状;培养皿对光观察,不能分清单个菌丝的尖端。

图6 木材腐朽菌B的菌落生长状况Fig.6 The colony’s growth condition of wood-decaying fungi B
菌种B:生长速率快,2周覆盖平板;菌落边缘为锯齿状;菌丝体在培养基内部生长,在表面之下;菌丝紧密扭结,形成一个白色外皮壳,较硬且不易剥落;菌落保持白色;在6周前反面部分变褐;生长新区的边缘菌丝平伏在培养基表面;培养皿对光观察,能够分清单个菌丝的尖端。
挑取两种木材腐朽菌做成临时装片,分别置于光学显微镜下观察,得到结果如图7和图8所示。

图7 光学显微镜下木材腐朽菌A的菌丝照片Fig.7 The mycelium photos of wood-decaying fungi A under optical microscope
菌种A:生长新区的菌丝无色,节状分隔;气生菌丝像生长新区的菌丝,具有垂直分枝;纤维菌丝较多,壁厚,时常分枝,弯曲和交织在一起;分生节有孢子。
菌种B:生长新区的菌丝无色,多分枝,有内含物,节状分隔很多,柔软;气生菌丝像生长新区的菌丝;纤维菌丝多,颜色深,分枝较明显,中空或有内含物;担孢子透明,均匀,圆柱形。
经过试验观察菌种A、B的宏观和微观特征,包括菌种的最适生长环境,菌落的颜色、质地、生长速率、边缘特征,显微镜下各个阶层菌丝的形状、生长状况等,可以大致判断出两种木材腐朽菌所属的科属情况。真菌A属于担子菌纲、多孔菌目、多孔菌科、栓菌属;木材腐朽菌B属于担子菌纲、多孔菌目、多孔菌科、多孔菌属。
3.2分子生物学鉴定
对两种木材腐朽菌7对引物扩增后得到的DNA序列分别登陆GeneBank,在DNA序列数据库中进行同源DNA序列的搜索工作,并进行BLAST对比,根据BLAST搜索和比较的结果,判断菌丝体菌种的物种或与其近缘的物种[19-20]。
登录GenBank,在GenBank核酸序列数据库中进行BLAST序列相似性检索,引物1扩增序列对比得到与菌种A相似率为96%的为Trametes versicolor(L.)Lloyd.彩绒革盖菌,引物2扩增序列检测到与被试样品序列相似性为85%以上的菌种3种,引物3、5、6、7扩增产物并未在genebank上找到与其相似性达到50%以上的已知序列,引物4扩增序列检测到与被试样品相似率为83%以上的4种菌。而之前经典形态学鉴定方法对该菌种的子实体照片、菌落照片、菌丝照片的鉴定结果是木材腐朽菌A为采绒革盖菌。
在GenBank核酸序列数据库中进行BLAST序列相似性检索,未检测到木材腐朽菌B这个种,但与菌种B相似率在85%以上的同源物种序列共有9个,将这9个序列同测得的菌种B序列一起进行系统发育分析。从图中可以看出,10个序列形成3个大的聚类组,木材腐朽菌B与多孔菌属其他种同属于一个聚类组,每个分支间的自举支持率都很高(>87%),序列差异小,亲缘关系近。菌种B与另两个聚类组的自举支持率都很低(<50%),序列差异大,说明其亲缘关系远。由此可以确定木材腐朽菌B属于多孔菌属真菌,如图9所示。

图9 用ITS序列构建的木材腐朽菌B系统发育树Fig.9 Wood-decaying fungi B phylogenetic tree constructed of ITS sequence
4 结 论
本文用PCR方法快速鉴定了两种木材腐朽菌,并结合传统的形态学鉴定方法,通过对比分析两种鉴定结果发现,两种鉴定方法得到的结果一致。但对木材腐朽菌A进行鉴定时,分子水平的鉴定精确到了种的水平,而传统形态学方法只能判定该菌种是卧孔菌属,并不能判定它是哪种菌种。因此在基于PCR技术的木材腐朽菌快速鉴定方法更加精确。
然而,基于PCR技术的木材腐朽菌快速鉴定方法也存在一定的局限性,由于ITS序列的高度保守性,种间关系相近的种的ITS序列之间的差异非常微小,所以这种分子鉴定方法不适合于属内种及种群间的鉴定[14-15],只有把传统形态学鉴定方法和基于PCR技术的鉴定方法想结合,才能得到更加可靠地鉴定结果。
[1]池玉杰.木材腐朽菌培养特性的研究综述[J].菌物学报,2004,23(1):158-164.
[2]王立海,孙天用.木材腐朽检测及防治的研究进展[J].黑龙江大学工程学报,2011,2(2):63-69.
[3]卢君辉,孙佳佳,董世龙,等.乌翅真鲨弗氏柠檬酸杆菌的分离与鉴定[J].海洋湖沼通报,2015,(4):83-88.
[4]Salazer O,Julian M C,Rubio V.Primers based on specific rDNA-ITS sequences for PCR detection of Rhizoctonia solani,R.solani AG 2 subgroups and ecological types and binucleate Rhizoetonia[J].Mycological Research,2000,104(3):281-285.
[5]Renske L,Paula L,Thom W K.Molecular identification of ectomycorrhizal mycelium in soil horizons[J].Applied and Environmental Microbiology,2003,69(1):327-333.
[6]White T J,Bruns T,Lee S.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M].New York:Academic Press,1990.
[7]Alba Z,Marc D,Odile D.In situ molecular detection of some white-rot and brown-rot basidiomycetes infecting temperate and tropical woods[J].Canadian Journal of Forest Research,2005,35(5):1256-1259.
[8]Erwin S H,Takemoto W J,Hwang M,et al.Anatomical characterization of decayed wood in standing light red meranti and identification of the fungi isolated from the decayed area[J].The Japan Wood Research Society,2008,54(3):233-241.
[9]郑小龙,王群,岳志芹,等.普氏野马物种的PCR鉴定[J].动物医学进展,2013,34(11):81-84.
[10]池玉杰.木材腐朽与木材腐朽菌[M].北京:科学出版社,2003.
[11]魏玉莲,戴玉成.木材腐朽菌在森林生态系统中的功能[J].应用生态学报,2004,15(10):1935-1938.
[12]Shinzato N,Namihira T,Tamaki K,et al.Application of random amplified polymorphic DNA(RAPD)analysis coupled with microchip electrophoresis for high-resolution identification of Monascus strains[J].Applied Microbiology and Biotechnology,2009,82(1):1187-1193.
[13]高国平,信雯,郑雅楠,等.刺槐心材腐朽菌的鉴定及其生物学特性[J].东北林业大学学报,2010,38(8):60-62.
[14]Jung H Y,Bae I Y,Lee S Y,et al.Effect of the degree of sulfation on the physicochemical and biological properties of Pleurotus eryngii polysaccharides[J].Food Hydrocolloid,2011,25(5):1291-1295.
[15]陈凤毛.真菌ITS区序列结构及其应用[J].林业科技开发,2007,21(2):5-7.
[16]Chenna R,Sugawara H,Koike T,et al.Multiple sequence alignment with the clustal series of programs[J].Nucleic Acids Research,2003,31(13):3497-3500.
[17]刘主,蔡爱群,刘静华,等.基于ITS区序列的疑似野生“南华”草菇菌株的分子鉴定[J].北方园艺,2016(1):126-130.
[18]Landeweert R,Leeflang P,Kuyper T W,et al.Molecular identification of ectomycorrhizal mycelium in soil horizons[J].Appl.Environ.Microbiol.,2003 ,69(1):327-333.
[19]Morin N,Vallaeysc T,Hendrickx L,et al.An efficient DNA isolation protocol for filamentous cyanobacteria of the genus Arthrospira[J].Journal of Microbiological Methods,2010,80(2):148-154.
[20]周贤轩,杨波,陈新华.几种分子生物学方法在菌种鉴定中的应用[J].生物技术,2004(6):35-38.
Study on Identification of Wood-decayingFungi Based on PCR Technique
Feng Lu,Qi Dawei*
(College of Science,Northeast Forestry University,Harbin 150040)
Wood decay caused by wood-decaying fungi will affect the wood quality.In order to investigate the pathogens resulted in wood decay,a new method was designed to rapidly identify wood-decaying fungi based on PCR(Polymerase chain reaction)technique.Two pathogens separated and depurated from decayed wood were cultivated in the laboratory,and then wood-decaying fungi was identified by morphological identification method.Seven groups of primer were designed to refine and amplify the PCR conditions.Two wood-decaying pathogens were compared and identified with BLAST.Results showed that wood-decaying fungi A wasTrametesversicolor(L.)Lloydwood-decaying fungi and fungi B belonged toPolyporaceaePolyporus.The results obtained by molecular identification were consistent with that by morphological identification.It is proven that the identification method based on molecular is feasible due to its rapid and accurate characteristics.
wood-decaying fungi;PCR;primer design;molecular identifications
2016-05-20
国家自然科学基金(31570712)
冯璐,硕士研究生。研究方向:生物物理。
戚大伟,博士,教授。研究方向:生物物理。
冯璐,戚大伟.基于PCR技术的木材腐朽菌鉴定方法的研究[J].森林工程,2016,32(5):35-39.
S 782.3
A
1001-005X(2016)05-0035-05
E-mal:qidw9806@126.com

