基于多序列比对的甜瓜SSR标记开发及应用
王盼乔,周亚峰,许彦宾,胡建斌,杨路明,孙守如
(河南农业大学园艺学院,河南 郑州 450002)
基于多序列比对的甜瓜SSR标记开发及应用
王盼乔,周亚峰,许彦宾,胡建斌,杨路明,孙守如
(河南农业大学园艺学院,河南 郑州 450002)
采用多序列比对的方法,从甜瓜EST序列中发现约1 100个SSR差异位点,并随机挑选50个位点设计成SSR引物,对47份遗传背景不同的甜瓜种质进行PCR扩增。结果表明,41对引物可产生清晰的带谱,有效扩增率达82.00%,且均具有多态性。共检测到130个等位基因,平均3.17个。Na、Ne、Ho、He、PIC等参数表明,这些标记具有一定程度的多态性。本研究开发的SSR标记可用于甜瓜的遗传多样性和群体结构分析,基于多序列比对的标记开发方法亦可在基因组信息丰富的物种中应用。
甜瓜;序列比对;EST-SSR;遗传多样性;群体结构
甜瓜(CucumismeloL.)是世界重要的园艺作物,其生产规模在世界10大水果中居第9位[1]。甜瓜是中国重要的设施蔬菜种类之一,其生产规模仅次于西瓜,播种面积和产量历年稳居世界第一[2]。近年来,甜瓜基因组领域的研究进展十分迅速,目前已经构建了至少20张遗传图谱,基因组信息不断增加[3]。2007年,西班牙、美国、中国等14个实验室联合成立了国际葫芦科基因组计划(International Cucurbit Genomics Initiative, ICuGI),启动了包括甜瓜在内的葫芦科植物全基因组测序工作。2012年,甜瓜全基因组测序结果公开发表[4],是继黄瓜、西瓜之后完成全基因组测序的葫芦科植物。甜瓜基因组学的快速发展产生了大量的数据信息,目前GenBank中甜瓜的EST序列已经超过13万条,为甜瓜的遗传研究(如标记开发)提供了丰富的数据信息。植物EST序列中一般含有SSR位点,其出现频率一般为1/10~1/5[5],这些SSR位点可用来开发SSR标记。通过SSR搜寻工具(如SSRHunter软件)可将含有SSR的EST序列找出,在SSR位点侧翼设计引物,便可开发成SSR标记。这种方法充分利用了现有的基因组信息,无需构建文库和测序,被认为是一种“免费”开发SSR标记的途径,目前已经应用于多种植物[6]。然而,许多研究表明,通过这种方法开发出的SSR标记,其多态性标记的比例不高。如白菜中多态性EST-SSR标记的比例为46.7%[7],萝卜中多态性EST-SSR标记的比例为35.0%[8],黄瓜中多态性EST-SSR标记比例仅为24.3%[9]。本研究采用多序列比对的方法开发甜瓜SSR标记,期望进一步提高多态性标记的比例,并对这些标记的可用性进行分析。
1 材料与方法
1.1材料
供试材料选自河南农业大学园艺学院瓜类育种实验室已构建的甜瓜核心种质库[10],共47份,其中厚皮种质(subsp.melo) 28份,薄皮种质(subsp.agrestis) 19份。所有材料均已培育成高代自交系,材料名称及来源见表1。
1.2方法
1.2.1 DNA提取 取各种质幼叶约300 mg,采用CTAB法[11]提取基因组DNA,用无菌ddH2O稀释至250 mg·L-1,备用。
1.2.2 SSR位点发掘及引物设计 从国际葫芦科基因组(ICuGI)网站(http://www.icugi.org)下载含有SSR位点的Unigene(MU43177~MU67602),摒弃现有报道中已用于开发SSR标记的序列,将剩余的Unigene与GenBank中甜瓜EST序列进行同源性比对,选取适宜的序列进行引物设计。选择含有SSR的序列的条件为:1) 序列比对的相似性为85%以上;2) 至少有2条EST序列与该序列存在SSR位点差异。
采用Primer 5在符合条件的目标序列(含有SSR差异位点的序列)侧翼设计引物,引物设计的标准为:Tm 值为55~60 ℃,引物长度为18~24 bp,PCR产物大小为100~350 bp,GC含量为40%~60%。引物由上海生工生物工程有限公司合成。

表1 试验材料和来源Table 1 Experimental materials and their origins
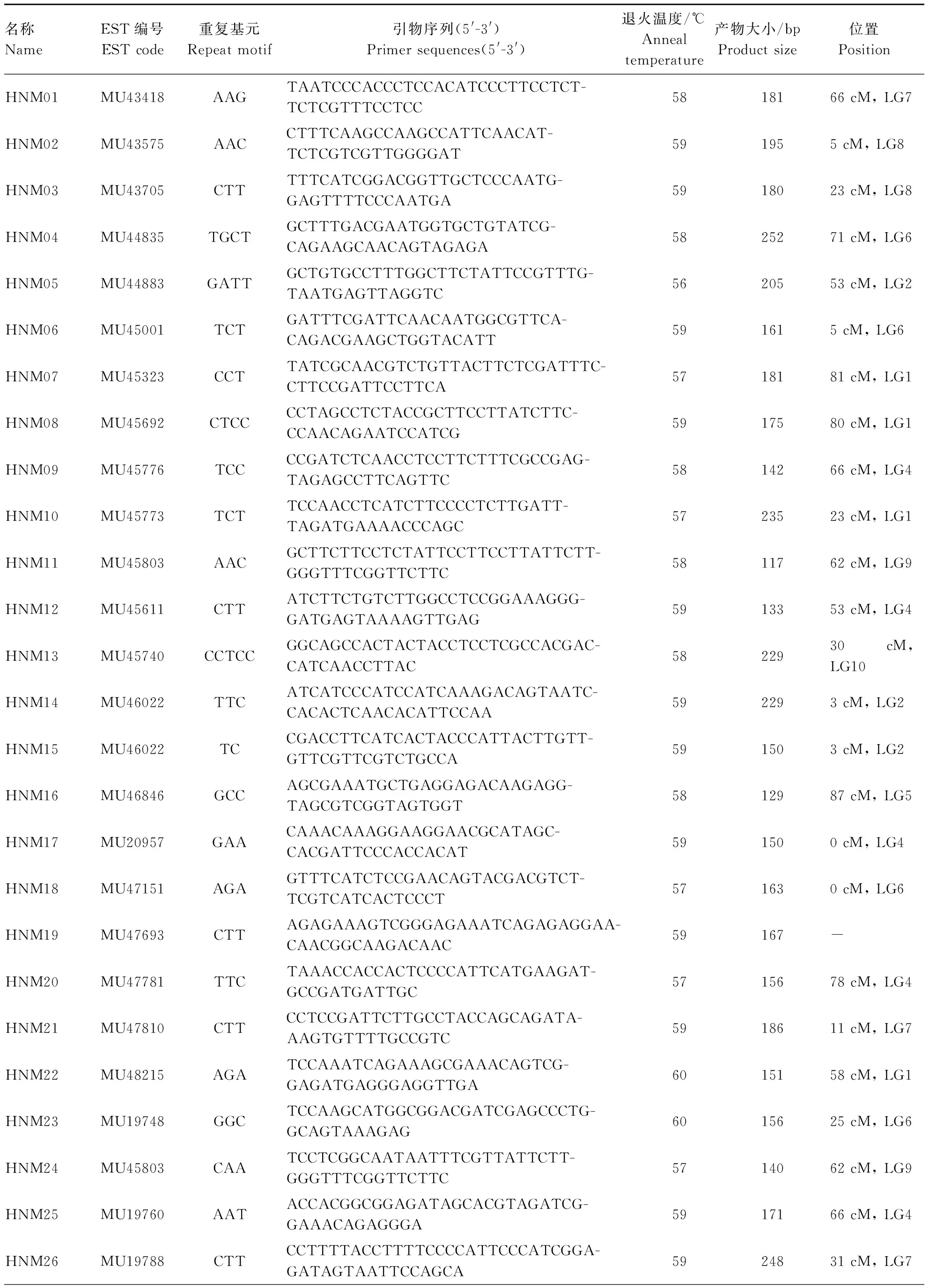
表2 试验所设计的50对引物的信息Table 2 Information of 50 primer pairs designed in the present study

续表Continuing Table
注:‘-’ 不能确定标记的位置。
Note: ‘-’indicates that the marker is not located.
1.2.3 PCR扩增及电泳检测 以47份甜瓜种质基因组DNA为模板,采用新合成的50对引物进行PCR扩增。扩增体系为15 μL,其中2 μL 50 mg·L-1模板DNA,1.5 μL 10×PCR buffer (含Mg2+),0.4 μL 2.5 mmol·L-1dNTPs,0.2 μL 2 500 U·L-1TaqDNA聚合酶,0.2 μL 10 μmol·L-1引物,8.6 μL ddH2O。PCR扩增在Mastercycler PCR 仪上进行,反应程序为:94 ℃预变性4 min;94 ℃变性40 s,55~60 ℃退火40 s,72 ℃延伸1 min,共循环35次;最后72 ℃延伸8 min。采用本实验室建立的PAGE 凝胶制备方法和银染的技术[12],对PCR扩增产物进行检测、记录。
1.3SSR的多态性检测与数据统计
根据电泳带谱,在相同迁移位置有带记作1,无带记作0,建立基因分型的0/1矩阵。采用PopGene 1.32软件计算等位基因数(Na)、有效等位基因数(Ne)、期望杂合度(He)和观测杂合度(Ho)。根据BOTSTEIN等[13]的方法计算多态性信息量(Polymorphism information content, PIC),
(1)
式中:Xi为第i种基因型出现的频率。
采用NTSYSpc 2.10软件进行数据分析,计算Jaccard相似系数,采用UPGMA法对47份材料的基因型数据进行聚类分析,并通过Treeplot程序生成聚类图。为了估测供试种质的群体遗传结构,应用STRUCTURE 2.3.1软件对多态性位点的分子数据进行分析,计算各种质的Q值(第i个材料基因组变异源于第K亚群的概率)。先设定群体数(K)为1~10,将MCMC(markov chain monte carlo)开始时的不作数迭代(length of burn-in period)设为10 000次,再将不作数迭代后的MCMC设为100 000次,依据似然值最大的原则选取1个合适的K值,绘制群体遗传结构图。
2 结果与分析
2.1SSR位点识别及扩增效率
在ICuGI网站中发现有3 263个unigene至少含有1个SSR位点,约 70% 的unigene能在GenBank中找到同源序列。采用Clustalx软件对unigene及其同源序列进行多序列比对,结果发现约1 100个SSR位点在多序列比对中表现出位点差异。其中,unigene MU45323的序列比对结果见图1,该序列的重复基元CTC和TCT与其他EST序列存在差异。本试验随机选取了50个符合条件的差异SSR位点,设计50对EST-SSR引物(表2),根据ICuGI网站公布的最新的甜瓜遗传图谱信息,将除HNM09和HNM27外的48个标记定位在除LG3和LG11之外的所有连锁群上。采用这些标记对47份遗传背景不同的甜瓜种质进行基因分型,结果发现,41对引物产生清晰可见的带谱,引物的有效扩增率为82.00%。在这41对引物中,4对引物(HNM01、HNM10、HNM35和HNM50)扩增出非预期大小的片段,实际片段比期望长度多出50 ~ 200 bp。编号为HNM27的引物同时检测到2个SSR位点的变异,因而可分为HNM27a (~300 bp)和HNM27b (~ 200 bp) 2个标记。剩余36对引物扩增出预期产物大小的片段,其中引物HNM08的扩增带谱见图2。

图1 MU45323的多序列比对结果Fig.1 The result of multiple sequence alignment of MU45323
2.2SSR标记的多态性验证
41对引物共检测到130个等位基因,每对引物检测到的等位基因数为2~9,平均3.167个(表3)。由于每对引物在47份种质中均能检测到等位变异,因而多态性引物的比例为100%。所有引物的有效等位基因数为1.403~5.111,HNM7最大,HNM49最小,平均2.410。期望杂合度为0.088~0.250,HNM01最大,HNM15最小,平均0.191。观测杂合度为0.000~0.095,平均0.023。多态性信息量为0.246~0.775,HNM07最大,HNM49最小,平均0.470。这些参数说明,供试种质的纯合度较高,新开发的引物均具有一定程度的多态性。所有引物的扩增产物呈“single-locus”变异,表型出典型的共显性特征(图2),为电泳数据记录提供了便利。

M:分子标量marker pUC18 DNA/Msp1;泳道1~ 47为甜瓜种质,序号同表1。 M: marker pUC18 DNA/Msp1; Lanes 1~ 47 mean the melon germplasms and their codes are the same to what is in table 1.

标记名称Name等位基因数/个Allelenumber有效等位基因数/个Effectiveallelenumber期望杂合度Expectedheterozygosity观测杂合度Observedheterozygosity多态性信息量PolymorphisminformationcontentHNM122.0000.2500.0210.375HNM242.9520.1650.0000.593HNM421.5410.1760.0000.290HNM521.9380.2420.0000.367HNM775.1110.1150.0650.775HNM821.6670.2000.0000.320HNM921.9960.2490.0210.375HNM1043.4050.1770.0230.653HNM1132.5960.2050.0430.533HNM1274.4490.1110.0950.742HNM1342.2890.1410.0000.503HNM1432.3180.1900.0000.477HNM1594.7300.0880.0000.761HNM1642.6010.1540.0650.549HNM1721.9130.2390.0210.363HNM1821.9770.2470.0440.372HNM1921.9570.2440.0000.369HNM2021.9570.2440.0000.369HNM2231.9530.1630.0430.388HNM2321.6580.1980.0000.318HNM2432.7360.2120.0430.558HNM2521.9960.2500.0210.375HNM2632.6650.2080.0000.547HNM27a42.4680.1490.0000.543HNM27b32.3490.1910.0210.502HNM2821.5840.1840.0000.301HNM3153.1050.1360.0640.629HNM3221.9410.2420.0000.367HNM3353.3730.1410.0460.646HNM3421.9760.2470.0000.372HNM3521.9860.2480.0210.373HNM3632.0060.1670.0000.412HNM3732.5880.2050.0640.533HNM3832.0620.1720.0210.459HNM3921.9570.2440.0000.369HNM4042.7740.1600.0000.589HNM4132.1460.1780.0480.469HNM4232.2020.1820.0250.484HNM4532.1200.1760.0510.441HNM4632.5580.2030.0430.526HNM4921.4030.1440.0440.246HNM5032.1980.1820.0000.486平均Mean3.1672.4100.1910.0230.470
2.3遗传多样性分析
为了进一步评价所开发的SSR标记的可用性,对41对SSR引物在47份甜瓜种质中的扩增数据进行聚类分析。所有种质的Jaccard相似系数为0.41~0.94,相似系数变化范围较广,说明参试种质遗传背景差异较大。内蒙古的‘金巴齿’和河南的‘牛角蜜’的相似系数最大,两者在果实性状上存在明显的相似性;河北的薄皮种质‘小翠瓜’和土耳其厚皮种质‘PI-169358’相似系数最小,两者在果实性状和地理分布上差异明显(表1)。
47份种质在相似系数为0.52的水平上可明显分为2类(I和II) (图3)。第I类包括26份厚皮种质,第II类包括剩余的19份薄皮种质和2份厚皮种质。在第I类中,不同地理来源地厚皮种质聚在一起,说明它们的遗传关系紧密,可能具有相同的起源中心;但是河南的‘Horneseoneen’与其他种质遗传关系较远。 所有的薄皮种质(中国17份,韩国和日本各1份)均聚在第II类,平均相似系数高达0.78,其遗传关系十分紧密。然而2份厚皮种质(中国台湾的‘PI-321004-S2’和伊朗的‘PI-137852’)也被划分在第II类,可能由于它们含有薄皮甜瓜的血缘,或因所用标记数量的限制而不能将它们分开;但在0.77的相似系数水平上,它们可与薄皮种质完全区别开。该研究结果表明,通过序列比对开发的SSR标记能较准确地将甜瓜厚皮和薄皮种质区分开,可用于甜瓜种质资源的遗传多样性研究。

图3 基于SSR数据构建的47份甜瓜种质的聚类图Fig.3 Dendrogram of 47 melon germplasms based on the SSR data
2.4群体结构分析
为了进一步分析参试种质的群体结构,试验采用STRUCTURE软件对47份种质的基因型数据进行基于Bayesian算法的群体结构划分,以推定的K所对应的最大似然值LnP(D)来选择合适的群体数目。由于LnP(D)随K的增大持续增加,难以确定真实的K值,因此参照EVANNO等[14]的方法通过ΔK来确定K值。当K=2时,ΔK出现明显的峰值(图4),即参试种质明显存在2个亚群(图5)。19份薄皮种质和一份厚皮种质(‘PI-321004-S2’)被划分为亚群Pop1,27份厚皮种质被划分为亚群Pop2,基于Bayesian算法基本能将参试种质按厚皮和薄皮亚种区分开。供试种质中,39份(95.12%)的种质的Q值大于0.90,表明其血缘组成相对单一,2份厚皮种质(‘PI-321004-S2’和‘PI-137852’)的Q值为0.30~0.70,具有混合血缘。本研究结果与UPGMA聚类方法一致。

K,群体数目。K,The number of population.

图5 47份甜瓜种质的群体遗传结构Fig.5 Population structure of 47 melon germplasms
3 结论与讨论
随着新一代的测序技术快速发展,海量的序列信息(主要是EST序列)被释放到公共数据库中,成为开发SSR、InDel、SNP等标记的宝贵资源。由于SSR标记具有共显性、稳定性、多等位变异等特点,是基于EST序列开发的主要对象。目前,基于EST序列的SSR标记开发在植物[6]、动物[15]、微生物[16]中均有报道,所开发的EST-SSR标记已广泛用于遗传多样性分析[17]、遗传图谱构建[18]、比较作图[19]、基因定位[20]等领域。然而,众多的研究表明,从EST序列中开发的SSR标记主要存在两个问题:一是标记的有效扩增率不高;二是标记的多态性较低[21]。
从基因组文库开发SSR标记是传统方法,由于该方法是在基因组DNA上搜寻SSR位点,因而所开发的标记对基因组的扩增是有效的,有效扩增率可达100%[22]。而EST-SSR标记是来自于基因组的编码区,如果EST-SSR引物恰好落在2个外显子上,或者上下游引物之间存在一个较长的内含子(超过了DNA聚合酶的扩增长度),标记则不会有扩增产物。因此,EST-SSR标记的有效扩增率一般不会达到100%。本试验中,甜瓜SSR标记的有效扩增率为82.00%,略低于黄瓜的89.19%[9]和萝卜的86.88%[8],高于辣椒的60.44%[23]。随着一些植物的全基因组序列的公开释放,标记开发者可先将目的EST序列与该物种的全基因组序列进行比对,排除内含子存在引物结合处或引物之间的情况,可提高标记的扩增效率。
从差异位点的检测效率来讲,EST-SSR的多态性一般低于基因组SSR。HU等[24]比较分析了黄瓜基因组SSR和EST-SSR的多态性,发现基因组SSR检测到的等位基因数和PIC值均明显高于EST-SSR。本试验开发的41个SSR标记平均检测到的等位基因3.17个,PIC值0.470,而萝卜的EST-SSR标记仅能检测2.5个等位基因[7]。与基因组的非编码区相比,编码区在植物进化过程中承受的选择压力较大,其序列变异相对较小,因而EST-SSR标记检测到的等位变异的几率不会很高[21]。TEMNYKH等[25]认为SSR的多态性与重复基元的长度有关,选择长度大于20 bp的SSR可提高标记的多态性。但在百合[26]、甜瓜[27]、刺梨[28]等植物中,一些长度超过20 bp的EST-SSR位点也不具有多态性,本研究中一些低重复的SSR (长度小于20 bp)却在不同种质间表现等位变异。本研究是针对具有SSR差异的序列(簇)开发标记,从源头上保证了标记的多态性潜能,在以遗传背景差异明显的材料作为试材的前提下,表现多态性的标记比例可达到100%。而针对单个EST序列开发的SSR标记,如在上述萝卜、黄瓜、辣椒等植物中,并非全部具有多态性。因此,在基因组信息丰富的物种中,通过序列比对法开发SSR标记,其优势十分明显。目前,基因组测序的成本越来越低,一些植物还完成了不同遗传背景材料的重测序。在这种情况下,电子PCR (Electronic PCR, e-PCR)技术的优势十分突出。它通过借助相应的序列分析软件,将设计的PCR引物与所查询样品的序列进行比对,以评价引物的扩增效率和多态性,可用于PCR 引物辅助筛选[29]。
此外,本研究还将新开发的SSR标记用于47份甜瓜种质的遗传多样性和群体结构分析,结果发现,基于新开发标记的聚类结果和群体划分与甜瓜分类基本一致。由于薄皮甜瓜主要分布在东亚地区(中国中、东部,朝鲜半岛和日本),厚皮种质主要分布在中亚、西亚、欧洲和美洲[30],因此聚类分析和群体划分的结果也与甜瓜的地理分布基本相符,说明所开发的标记可用于甜瓜遗传多样性和群体结构研究。
本研究采用多序列比对的方法从甜瓜EST序列中发现约1 100个SSR差异位点,并开发了50对SSR引物,48个标记分布在除LG3和LG11之外的其他连锁群。对47份遗传背景不同的甜瓜种质进行分子标记分析,41对引物产生清晰的带谱,且均具有多态性,共检测到130个等位基因。Na、Ne、Ho、He、PIC等遗传参数显示,这些标记具有一定程度的多态性。UPGMA聚类方法和Bayesian群体划分方法均能按照甜瓜植物学分类和地理分布将所有种质分为2类或亚群。本研究开发的甜瓜SSR标记不仅进一步丰富了甜瓜DNA标记资源,而且对于基因组丰富物种的标记开发,也具有十分重要的启示意义。
[1] 刘君璞, 许勇, 孙小武, 等. 我国西瓜甜瓜产业“十一五”的展望及建议[J]. 中国瓜菜, 2006, 19(1): 1-3.
[2] 王志丹. 中国甜瓜产业经济发展研究[D].北京:中国农业科学院, 2014.
[3] ARGYRIS J M, PUJOL M. MARTIN-HERNANDEZ A M, et al. Combined use of genetic and genomics resources to understand virus resistance and fruit quality traits in melon[J]. Physiologia plantarum, 2015, 155(1): 4-11.
[4] GARCIA-MAS J, BENJAK A, SANSEVERINO W, et al. The genome of melon (CucumismeloL.) [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(29): 11872-11877.
[5] GAO L, TANG J, LI H, et al. Analysis of microsatellites in major crops assessed by computational and experimental approaches[J]. Molecular Breeding, 2003, 12(3): 245-261.
[6] KALIA R K, RAI M K, KALIA S, et al. Microsatellite markers: an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3): 309-334.
[7] 忻雅, 崔海瑞, 卢美贞, 等. 白菜EST-SSR信息分析与标记的建立[J]. 园艺学报,2006, 33(3): 549-554.
[8] 崔娜, 邱杨, 李锡香, 等. 萝卜EST资源的SSR信息分析及EST-SSRs标记开发[J]. 园艺学报, 2012, 39(7): 1303-1312.
[9] 关媛, 李效尊, 潘俊松, 等. 黄瓜果实EST-SSR标记的开发[J]. 分子植物育种, 2007, 5(5): 725-728.
[10] 胡建斌, 马双武, 王吉明, 等. 基于表型性状的甜瓜核心种质构建[J]. 果树学报, 2013, 30(3): 404-411.
[11] PATERSON A H, BRUBAKER C L, WENDEL J F. A rapid method for extraction of cotton (Gossypium spp.) genomic DNA suitable for RFLP or PCR analysis[J]. Plant Molecular Biology Reporter, 1993, 11(2): 122-127.
[12] 胡建斌, 李静, 袁鸣, 等. PAGE凝胶制备方法和银染方法的改进[J]. 江西农业大学学报, 2009, 31(4): 742-745.
[13] BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American journal of human genetics, 1980, 32(3): 314-331.
[14] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J]. Molecular Ecology, 2005, 14(8): 2611-2620.
[15] 王解香, 于凌云, 白俊杰, 等. 草鱼EST-SSR标记及5个不同地域群体的遗传结构分析[J]. 动物学杂志, 2011, 46(5): 24-32.
[16] 朱振东, 霍云龙, 王晓鸣, 等. 从大豆疫霉菌ESTs中发掘SSR标记[J]. 科学通报, 2004, 49(17): 1749-1754.
[17] 刘俊青, 李佩芳, 胡建斌, 等. 用EST-SSR标记评价21份黄瓜种质的遗传多样性[J]. 河南农业大学学报, 2011, 45(6): 657-661.
[18] 陈海梅, 李林志, 卫宪云, 等. 小麦EST-SSR标记的开发、染色体定位和遗传作图[J]. 科学通报, 2005, 50(20): 2208-2216.
[19] YU J K, DAKE T M, SINGH S, et al. Development and mapping of EST-derived simple sequence repeat markers for hexaploid wheat[J].Genome, 2004,47(5): 805-818.
[20] 张宁, 张显, 张勇, 等. 甜瓜果实糖含量相关性状QTL分析[J]. 西北植物学报, 2015, 35(2): 252-257.
[21] VARSHNEY R K, GRANER A, SORRELLS M E. Genic microsatellite markers in plants: features and applications[J]. Trends in Biotechnology, 2005, 23(1): 48-55.
[22] 张增翠, 侯喜林. SSR分子标记开发策略及评价[J]. 遗传, 2004, 26(5): 763-768.
[23] 刘峰, 王运生, 田雪亮, 等. 辣椒转录组SSR挖掘及其多态性分析[J]. 园艺学报, 2012, 39(1): 168-174.
[24] HU J, WANG L, LI J. Comparison of genomic SSR and EST-SSR markers for estimating genetic diversity in cucumber[J]. Biologia Plantarum, 2011, 55(3): 577-580.
[25] TEMNYKH S. Computational and Experimental Analysis of Microsatellites in Rice (Oryzasativa L.): Frequency, Length Variation, Transposon Associations, and Genetic Marker Potential [J]. Genome Research, 2001, 11(8): 1441-1452.
[26] 杨素丽, 明军, 刘春, 等. 基于EST信息的百合SSR标记的建立[J]. 园艺学报, 2008, 35(7): 1069-1074.
[27] 胡建斌, 李建吾. 甜瓜EST-SSR位点信息及标记开发[J]. 园艺学报, 2009, 36(4): 513-520.
[28] 鄢秀芹, 鲁敏, 安华明. 刺梨转录组SSR信息分析及其分子标记开发[J]. 园艺学报, 2015, 42(2): 341-349.
[29] 张洁, 陆海峰, 李有志. 电子PCR [J]. 分子植物育种, 2004, 2(1): 139-145.
[30] 林德佩. 中国栽培甜瓜植物的起源、分类及进化[J]. 中国瓜菜, 2010, 23(4): 34-36.
(责任编辑:李莹)
DevelopmentofSSRmarkersbasedonmultiplesequencealignmentandtheirapplicationsinmelon
WANG Panqiao, ZHOU Yafeng, XU Yanbin, HU Jianbin, YANG Luming, SUN Shouru
(College of Horticulture, Henan Agricultural University, Zhengzhou 450002, China)
Multiple sequence alignment was used to develop SSR markers in melon, which identified about 1 100 different SSR loci from the melon ESTs. Of these loci, 50 were randomly selected for primer design and the next PCR amplification. The genomic DNA of 47 melon germplasms with different genetic backgrounds was analyzed with these primers. The results showed that 41 primer pairs generated clear band patterns, with the amplification efficiency of 82.00%, and all the products were polymorphic. A total of 130 alleles were detected with a mean of 3.17. The genetic parameters, Na, Ne, Ho, He, and PIC, verified a certain level of polymorphism of these newly-developed markers. Therefore, the SSR markers developed herein can be used to analyze genetic diversity and population structure in melon. Also, the method of SSR development based on multiple sequence alignment can be well applied in the species with abundant genomic information.
melon; sequence alignment; EST-SSR; genetic diversity; population structure
S652
:A
2015-09-07
国家自然科学基金项目(31101544);河南省高等学校重点科研项目(15A210011)
王盼乔(1990-) ,男,河南郏县人,硕士研究生,从事甜瓜育种研究。
胡建斌(1976-) ,男,湖北武汉人,副教授,博士。
1000-2340(2016)02-0189-09
——栽培注意事项

