基于RIL群体和IF2群体的玉米开花期相关性状QTL分析
石超男,张战辉,唐贵良,薛亚东
(河南农业大学农学院,河南 郑州 450002)
基于RIL群体和IF2群体的玉米开花期相关性状QTL分析
石超男,张战辉,唐贵良,薛亚东
(河南农业大学农学院,河南 郑州 450002)
利用一套来源于玉米杂交种农大108的RIL群体及其IF2群体,对玉米开花期相关性状进行比较QTL分析。表型分析结果表明,杂交种农大108和IF2群体具明显的杂种优势,其玉米开花期相关性状在不同环境间较双亲和RIL群体更加稳定。通过QTL分析,RIL群体中定位到17个开花期相关的QTLs,IF2群体中定位到15个QTLs,主要分布在染色体区域bin 1.02~1.03,bin 4.00~4.01,bin 4.07~4.08,bin 9.04和bin 10.03。但仅qDS1在2个群体中同时被检测到,表明玉米杂交种和自交系具有截然不同的开花期遗传调控机制。本研究检测到的环境间、性状间保守QTLs可能含有调控玉米开花期的主效基因,在育种过程中可用于筛选开花期适合的优良自交系,并指导适于玉米机械化收获优良品种的选育。
玉米;IF2群体;RIL;开花期;QTL分析
玉米开花期标志着玉米植株由营养生长向生殖生长的转变,不仅决定着玉米的株高和叶片总数,还决定着玉米的产量[1]。玉米开花期受植物生长发育的自发性、光周期和激素等因素的调控[2-5],干旱[6-8]和害虫[1]等非生物和生物胁迫也是影响开花期的重要因素。对玉米开花期进行研究,不仅有利于剖析开花期性状的遗传机制和分子调控机理,而且可为玉米品种的改良和优良品种的选育提供重要的理论依据。研究人员挖掘和鉴定了一系列开花期相关基因和数量性状位点(QTLs),并剖析了相关的分子调控网络[9-10]。这些研究在一定程度上阐述了模式植物开花期的分子调控机制,但包括玉米在内的主要粮食作物开花期相关的遗传机理和分子调控机制还不清楚。玉米开花期性状属于复杂数量性状,受控于少数主效基因的同时还受到一定数量微效基因的调控[11]。已经鉴定到的玉米开花期相关的基因包括:vgt1(Vegetativetogenerativetransition1)[12-15],ZCN8(ZeamaysCENTRORADIALIS8)[4, 16],ZCN1(ZeamaysCENTRORADIALIS8)[17],Dwarf8[18],id1(Indeterminate1)[19-20],ids1(Indeterminatespikelet1)[21],sid1(Sisterofindeterminatespikelet1)[22],ts4(Tasselseed4)[22],gl15(Glossy15)[23],cg1(Corngrass1)[24],zfl1(Zmfloricaula/Leaf1)[12, 25],dlf1(Delayedflowering1)[8],AN1(Antherears1)[26]和EPC1(Earlyphasechange1)[27]。研究者还对玉米开花期性状进行了一系列QTL分析[27-28],鉴定了一定数量的一致性QTLs,这些QTLs主要分布在bin 1.03,bin 1.05~1.06,bin 2.03,bin 3.04,bin 4.04~4.05,bin 4.08,bin 6.01~6.02,bin 6.06~6.08,bin 7.02,bin 7.04~7.05,bin 8.04~8.06,bin 9.04,bin 10.03~10.04和bin 10.06~10.07。这些研究结果进一步证实了玉米开花期性状受控于大量的微效QTLs[11]。然而,多环境条件下利用杂交种群体对开花期进行QTL分析还鲜见报道。本研究利用一套玉米杂交种农大108的RIL群体与IF2群体对玉米开花期相关性状进行QTL分析,比较了自交系和杂交种玉米开花期性状的遗传差异;定位玉米开花期相关的QTL;为玉米开花期相关QTL的应用提供依据。
1 材料与方法
1.1材料
本研究利用一套包含166个家系的RIL群体和一套包含249个家系的ImmortalizedF2(IF2)群体对玉米开花期进行QTL分析。这两个群体来源于中国一个大面积推广的优良玉米杂交种农大108(黄C×许178)。IF2群体具有高度杂合的基因型,能够实现永久分离群体(RIL)和临时性分离群体(F2)的有机结合,是进行QTL定位和杂种优势分析的理想分离群体。
1.2田间试验与性状调查
(1)
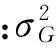
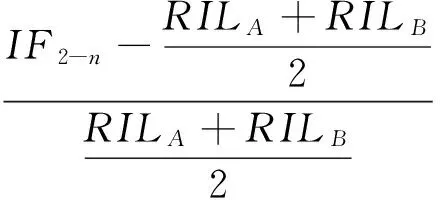
(2)
式中:RILA和RILB为IF2群体的双亲优势。
1.3遗传连锁图谱与QTL分析
在本研究中,利用RIL群体166个家系构建的遗传连锁图谱包含均匀分布在玉米基因组10条染色体上的217个SSR标记,标记区间总长为2 438.2 cm,平均标记区间为11.2 cm[32]。由于IF2群体是由成对RIL家系杂交而来的杂交种群体,因而由RIL群体构建的遗传连锁图谱同样也可应用于IF2群体[30-31, 33]。
利用软件Windows QTL Cartographer 2.5的复合区间作图法(Composite interval mapping,CIM)[34],不同环境条件下的RIL和IF2群体开花期、吐丝期和开花吐丝间期的表型数据作为输入数据,分别进行QTL分析。在P=0.05显著水平下,1 000次排列测验(Permutation test)计算每个性状LOD值。QTL分析结果中,大于对应计算得到的LOD值即认为存在一个QTL。定位到的QTL加性效应为正值表示加性效应来自黄C的等位基因;相反,负值表示加性效应来自许178的等位基因。
2 结果与分析
2.1RIL和IF2群体玉米表型分析
2009年和2010年,2个试点农大108开花期和吐丝期均显示明显的杂种优势(表1),而开花吐丝间期没有表现出杂种优势。不同环境条件之间,农大108及其双亲(许178和黄C)的开花期和吐丝期存在明显的差异,尤其是在不同年份间,但开花吐丝间期在不同环境间表现较为稳定。不同环境条件下,RIL群体开花期最小值均大于杂交种农大108,而RIL群体吐丝期最小值与农大108差异较小。除2010年郑州试点外,RIL群体开花期和吐丝期均大于2个亲本。RIL群体3个开花期性状在基因型间存在极显著的差异,基因型与环境互作也存在极显著差异。IF2群体(表1和表2),3个开花期性状均表现出杂种优势。与杂交种农大108相比,IF2群体3个开花期性状的最小值均小于农大108,表明杂交种农大108 3个开花期性状尚未达到最大杂种优势。IF2群体3个开花期性状在基因型和基因型与环境互作间均存在极显著差异。3个开花期性状在RIL和IF2群体间均呈正态分布(表1、表2、图1和图2),且3个性状在2个群体间均表现出较高的遗传力(85.8%~ 90.2%)。
红牌:表示脚手架未经验收合格,通常是指脚手架在搭设、整改或拆除等过程,仅脚手架工可以使用,其他人员不得使用该脚手架。
2.2IF2群体玉米开花期性状的杂种优势
IF2群体玉米开花期性状杂种优势表现如表3所示,不同环境条件下,3个开花期相关性状均表现出杂种优势,开花期杂种优势为-8.34%~-4.78%,吐丝期为-8.42%~-4.96%,开花吐丝期为-40.68%~-11.20%。3个开花期相关性状的群体最大平均杂种优势出现在2009年郑州试点,2010年安阳试点开花期和散粉期表现出最小平均杂种优势,2009年安阳试点开花吐丝间期的表现出最小平均杂种优势。开花期杂种优势最小变幅出现在2009年安阳试点和2010年郑州试点,最大变幅出现在2010年安阳试点;吐丝期杂种优势最小变幅和最大变幅分别出现在2010年安阳试点和2009年郑州试点;而开花吐丝间期杂种优势的最小变幅和最大变幅分别出现在2009年和2010年郑州试点。
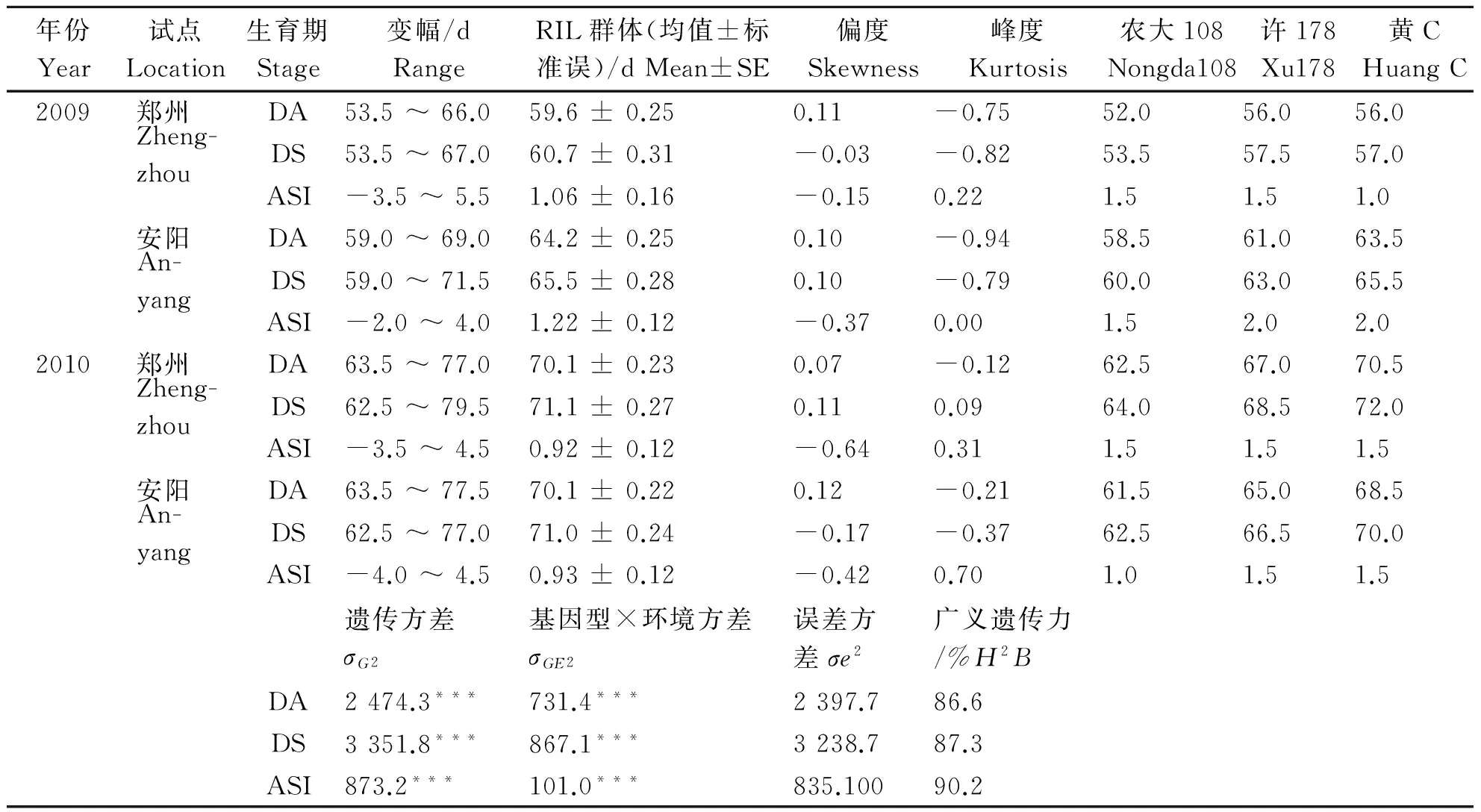
表1 RIL群体玉米开花期、吐丝期和开花吐丝间期的表型分析Table 1 Phenotypic analysis of days to anthesis, days to silking and anthesis silking interval in a set of RILs at two locations over two years in maize
注:DA,开花期;DS,吐丝期;ASI,开花吐丝间期。单位:天。***表示P值≤0.001,呈极显著水平。
Note: DA, Days to anthesis; DS, Days to silking; ASI, Anthesis silking interval. The unit of DA, DS and ASI is days. *** , represents the significance level atP≤ 0.001.
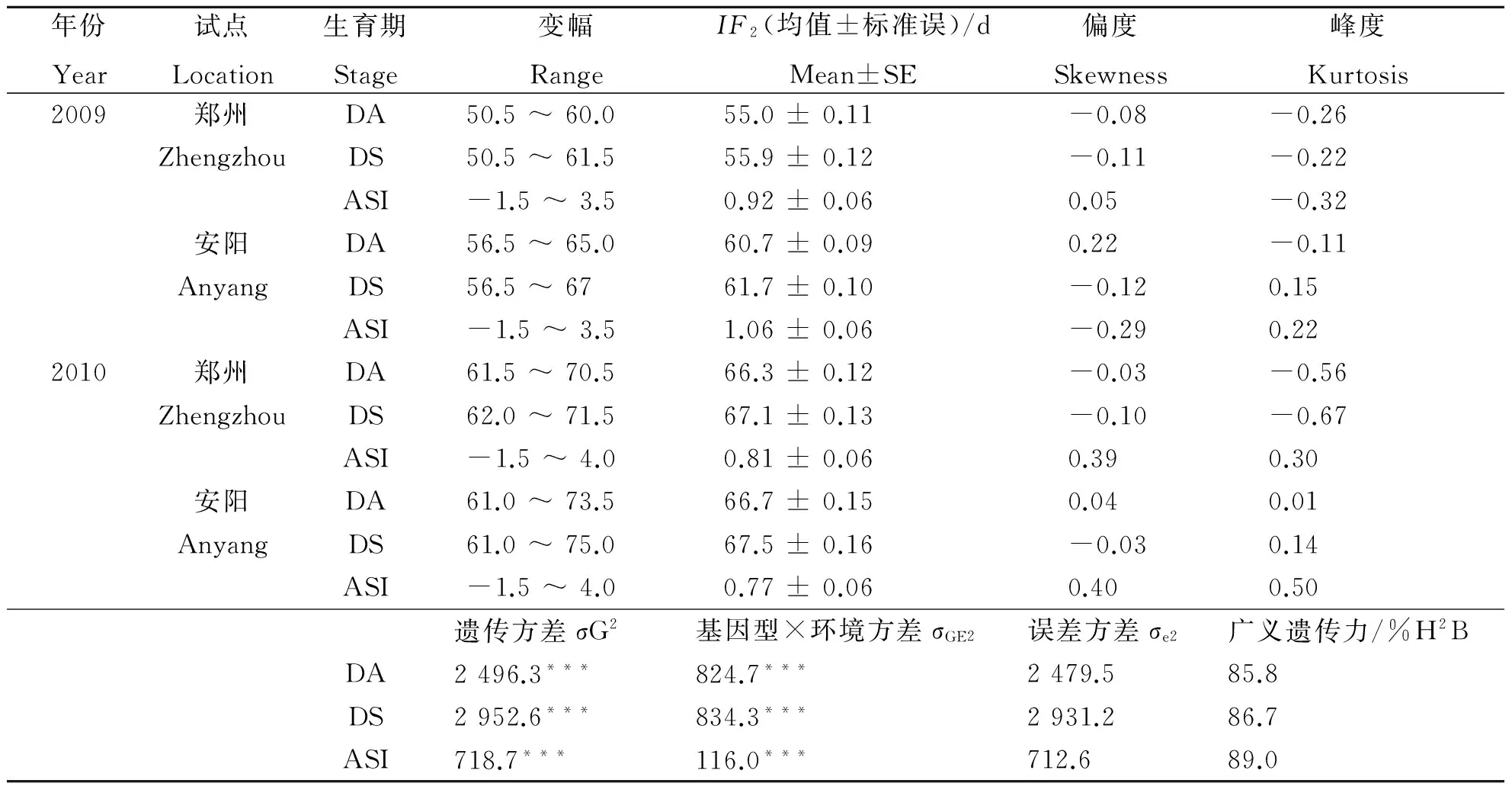
表2 IF2群体玉米开花期、吐丝期和开花吐丝间期的表型分析Table 2 Phenotypic analysis of days to anthesis, days to silking and anthesis silking interval in a set of IF2 population in maize
注:DA,开花期;DS,吐丝期;ASI,开花吐丝间期。单位:天。***表示P值≤0.001,呈极显著水平。
Note: DA, Days to anthesis; DS, Days to silking; ASI, Anthesis silking interval. The unit of DA, DS and ASI is days. *** , represent the significance level isP≤ 0.001.

表3 IF2群体两年间两个试点玉米开花期、吐丝期和开花吐丝间期杂种优势分析Table 3 The heterosis of days to anthesis, days to silking and anthesis silking interval in an IF2 population in maize
注:DA,开花期;DS,吐丝期;ASI,开花吐丝间期。
Note: DA, Days to anthesis; DS, Days to silking; ASI, Anthesis silking interval.
2.3RIL群体玉米开花期性状QTL分析
RIL群体3个开花期性状共定位到17个QTLs,分别分布在第1、2、3、4、6、8、9和10染色体上(表4、图3)。其中,qASI4在4个环境中重复检测到,其加性效应来自于亲本许178,贡献率分别为16.03%、12.56%、9.87%和11.74%。在第4染色体的标记区间umc 1 847~umc 1 808,分别检测到开花期,散粉期和开花吐丝间期相关的QTLs,而且qDA4在2010年两个试点均检测到,贡献率分别为10.95% 和10.26%。此外,检测到的其它QTLs均为环境特异性QTLs。除了以上的环境保守性QTLs,2009年郑州试点分别检测到1个开花期和1个散粉期相关的QTLs,这2个QTLs均临近标记 umc1 078,贡献率分别为10.41%和10.98%;2009年安阳试点检测到5个QTLs,2个开花期相关QTLs,一个散粉期相关的QTLs和2个开花吐丝间期相关的QTLs;2010年郑州试点还检测到2个散粉期相关的QTLs,其中qDS4临近标记umc1847,贡献率为14.22%;2010年安阳试点分别检测到一个散粉期相关的QTLs和一个开花吐丝间期相关的QTLs,分别位于第1和第8染色体上。
2.4IF2群体玉米开花期性状QTL分析
IF2群体3个玉米开花期相关性状共定位到15个QTLs,分别在第1、5、6、7和10染色体上(表5)。其中,qDS1和qASI1分别在4个环境中被检测到,其贡献率变幅分别为9.00%~17.97%和13.90%~19.74%,其加性效应均来自亲本许178。此外,qDS10b和qDS5分别在2009年的郑州和安阳试点被检测到,其加性效应来自亲本黄C,贡献率分别为7.10%和7.23%。4个环境条件下,分别检测到1个开花期相关的QTLs,其中qDA1b和qDA1c分布在第1染色体的相邻标记区间。IF2群体3个开花期相关性状检测到的15个QTLs中,8个QTLs表现部分显性效应,5个QTLs表现显性效应,仅有2个QTLs表现超显性效应。
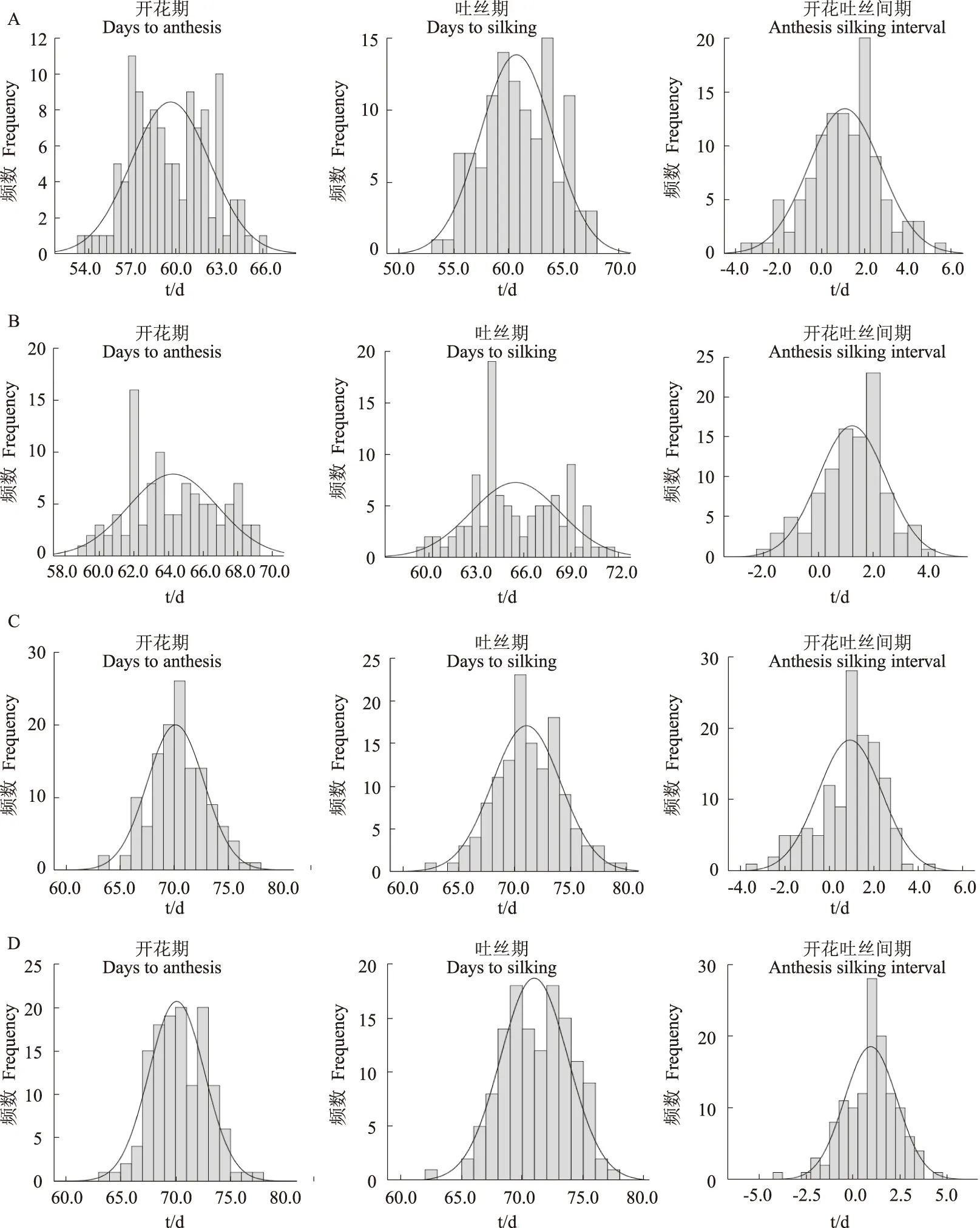
图1 2009年和2010年郑州(A和C)和安阳(B和D)试点RIL群体玉米开花期、吐丝期和开花吐丝间期的频率分布Fig.1 The frequency distribution of days to anthesis, days to silking and anthesis silking interval in a set of RILsat Zhengzhou location in 2009 and 2010(A and C) and Anyang location in 2009 and 2010(B and D)
2.5RIL和IF2群体间保守的QTL
4个环境条件下,RIL群体和IF2群体共定位到32个开花期相关的QTLs,但仅qDS1在两个群体间同时被检测到。这些QTLs成簇分布于染色体区域bin 1.02~1.03,bin 4.00~4.01,bin 4.07~4.08,bin 9.04和bin 10.03~10.04。
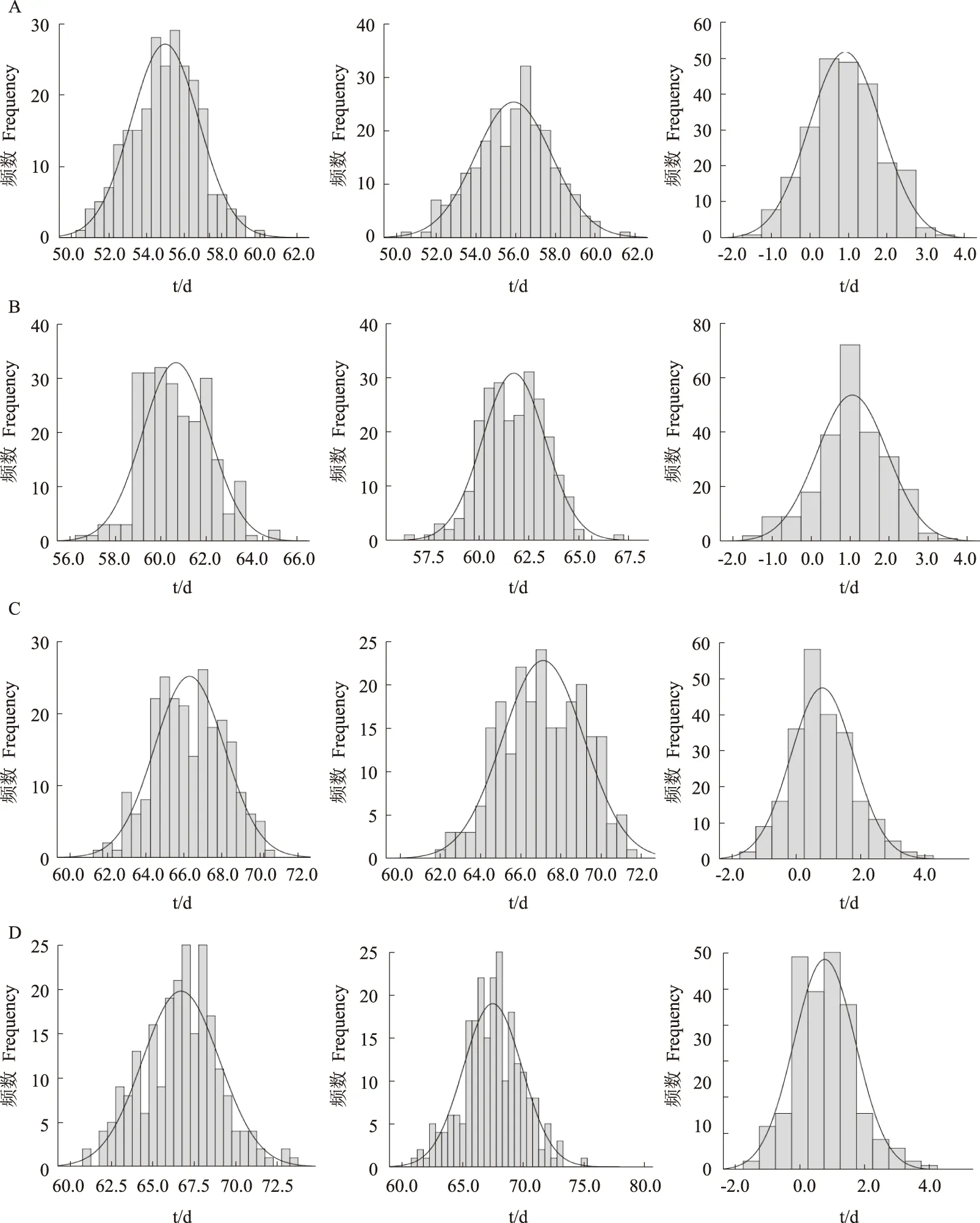
图2 2009年和2010年郑州(A和C)和安阳(B和D)试点IF2群体玉米开花期、吐丝期和开花吐丝间期的频率分布Fig.2 The frequency distribution of days to anthesis, days to silking and anthesis silking interval in IF2population at Zhengzhou location in 2009 and 2010(A and C) and Anyang location in 2009 and 2010(B and D)
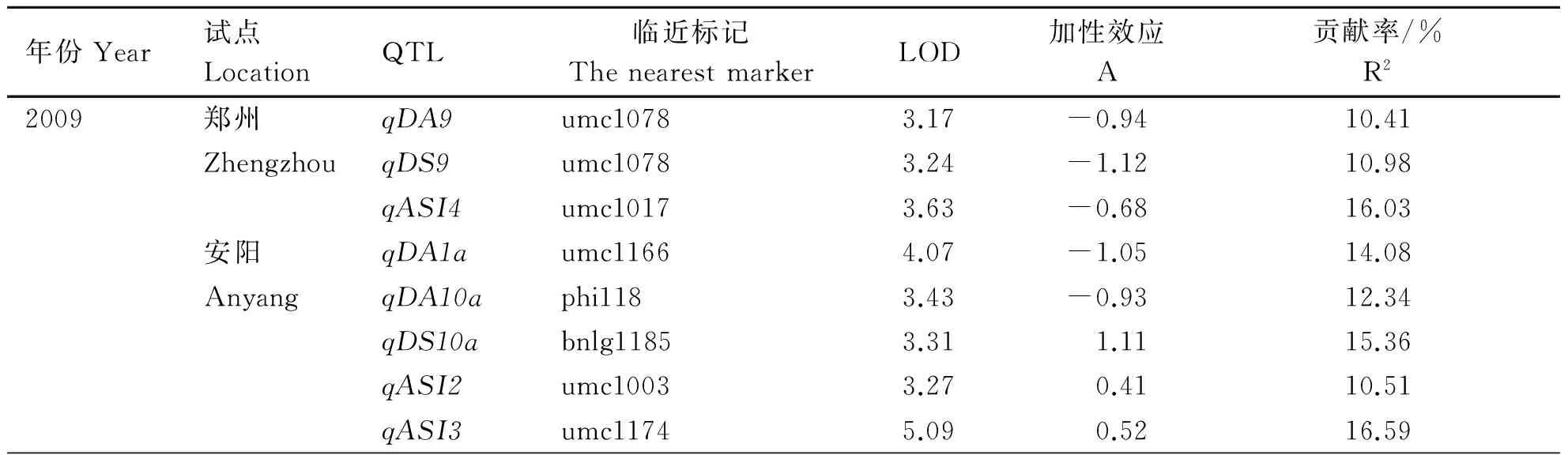
表4 RIL群体定位到的开花期性状相关QTLTable 4 QTLs for three flowering time related traits detected in a set of RIL
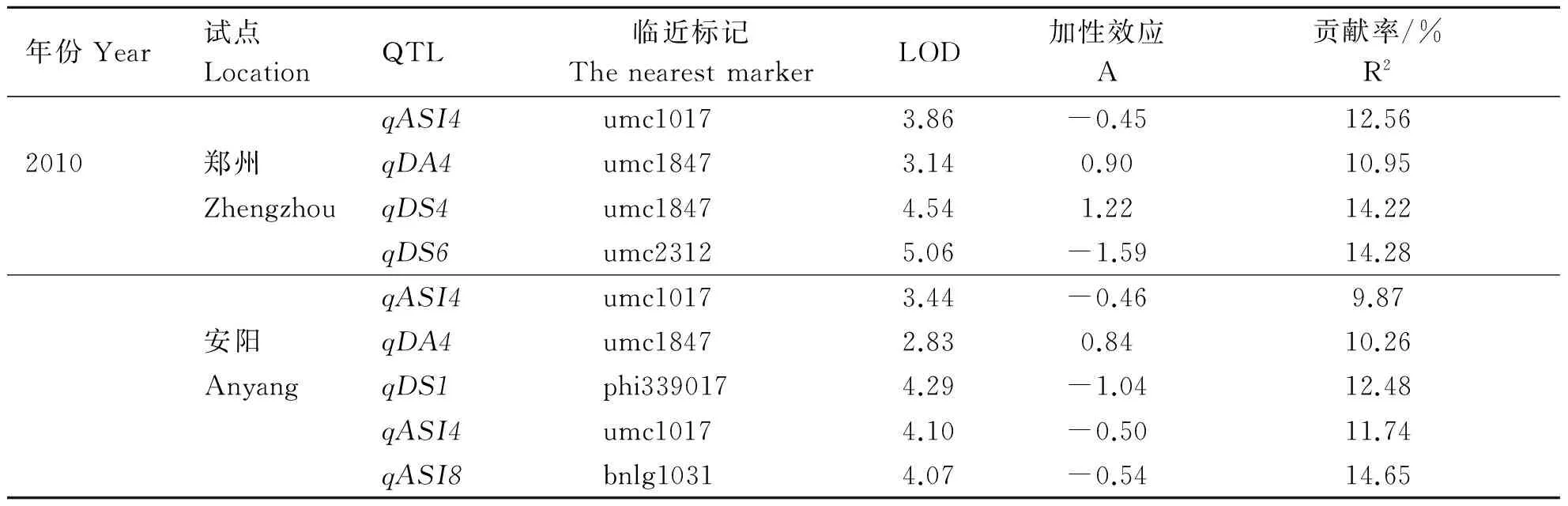
续表4 Continuing Table 4
注: RIL群体中定位到的玉米开花期性状相关QTLs命名按照q+性状缩写+染色体号+QTL编号;LOD为每个QTL的LOD值;A,加性效应值;R2,贡献率。
Note: QTLs for flowering time related traits in the RILs are named as q + trait abbreviation + chromosome number + QTL number; Logarithm of odds for each QTL; A, additive values; R2, contribution rate.

图3 RIL群体和IF2群体3个开花期相关性状QTLs在染色体上的分布Fig.3 Chromosomal locations of QTLs for three flowering time related traits in a RIL and an IF2 maize populations
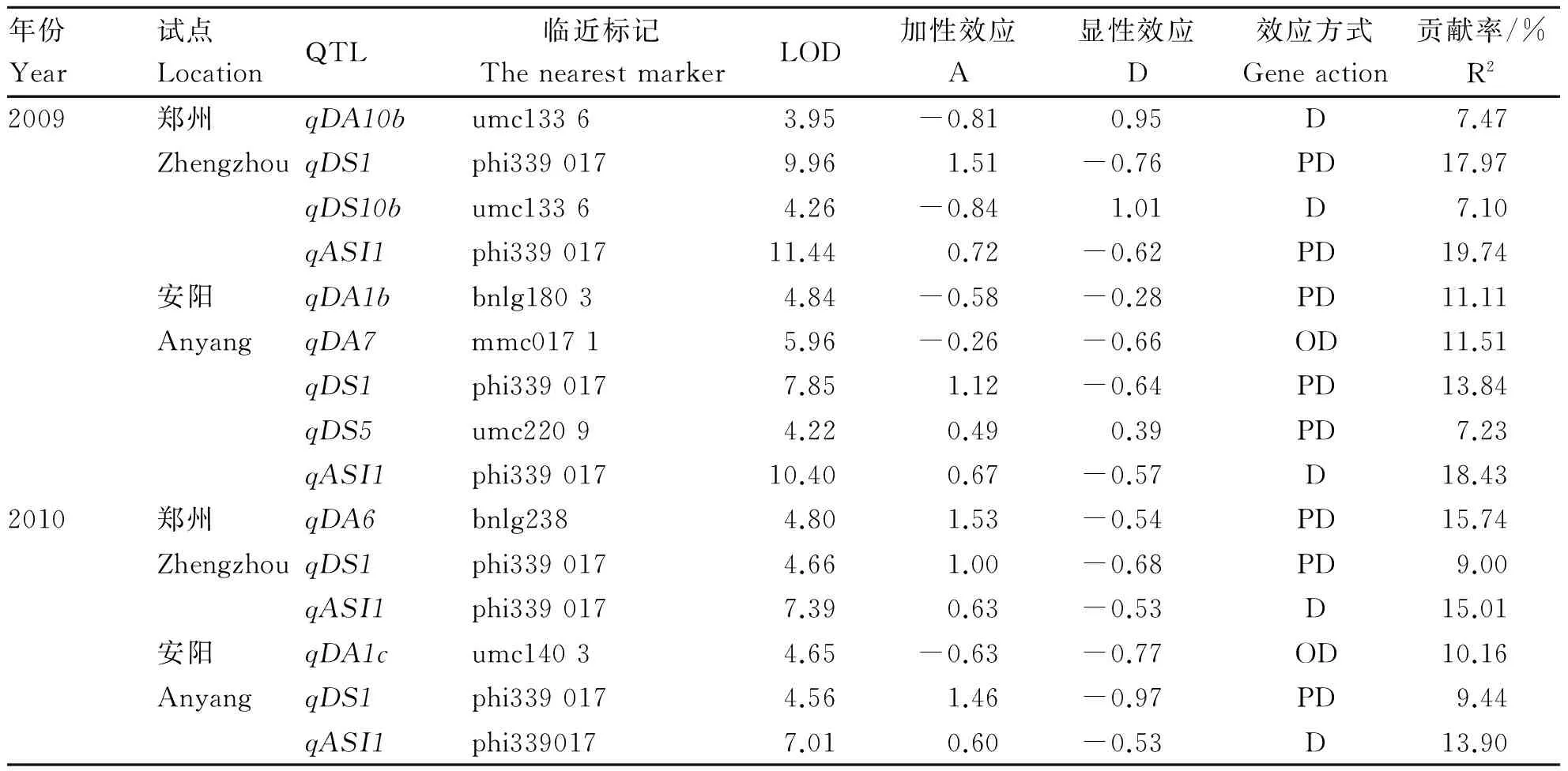
表5 IF2群体定位到的开花期性状相关QTLsTable 5 QTLs for three flowering time related traits detected in a set of IF2 population
注:IF2群体中定位到的玉米开花期性状相关QTLs命名按照q+性状缩写+染色体号+QTL编号;LOD,每个QTL的LOD值;A,加性效应值;D,显性效应值;A,加性效应;PD,部分加性效应;D,显性效应;OD,超显性效应R2,贡献率。
Note: QTLs for flowering time related traits in theIF2population are named as q+trait abbreviation + chromosome number + QTL number;Logarithm of odds for each QTL;A, additive values; D, dominant values;The effect of each QTL; A, additive; PD, partial dominance; D, dominance; OD, overdominance;R2, contribution rate.
3 结论与讨论
3.1自交系和杂交种开花期性状间的遗传差异
玉米开花期性状在玉米对外界环境变化的适应过程中具有极其重要的意义,决定玉米株高、叶片总数和产量[8]。玉米开花期性状具有典型的数量性状特点,鉴定玉米开花期相关的基因需要多环境试验,虽然QTL分析鉴定到了一些环境一致性QTLs[27-28],但这些QTL或鉴定于自交系群体中,或鉴定于在单一环境条件下的F2/F2:3群体中,所以玉米杂交种开花期相关性状的基因和QTLs仍有待研究挖掘。本研究,利用基于玉米杂交种农大108的RIL群体和IF2群体对玉米开花期相关性状在多环境条件下进行比较QTL分析,期望进一步解析玉米开花期在自交系和杂交种间的遗传差异。
杂种优势是指杂交子代在生长活力、育性和种子产量等方面都优于双亲的现象。本研究中,杂交种和IF2群体与两个亲本和RIL群体相比,3个玉米开花期相关性状均表现出明显的杂种优势现象。而且,杂交种农大108和IF2群体3个开花期相关性状在不同环境因素间表现的更加稳定。RIL群体和IF2群体分别定位到17个和15个开花期相关的QTLs,但仅有qDS1在这2个群体间同时检测到。而且,RIL群体在染色体区域bin 1.02~bin 1.03检测到开花期和吐丝期相关的QTLs,IF2群体在该区域检测到吐丝期和开花吐丝间期相关的QTLs,表明该区域存在调控玉米开花期的主要基因。但是,RIL群体中检测到的QTLs主要分布在bin 4.01,bin 4.07~4.08和bin 9.04,而IF2群体中检测到的QTLs主要分布在bin 1.02~1.03和bin 10.03,这表明玉米自交系和杂交种在开花期相关性状的遗传调控方面具有截然不同的机制。玉米作为研究杂种优势的模式作物,研究者开展了大量的研究,并提出显性[35-36]和超显性[37]两个假说来解释杂种优势现象,这表明显性和超显性为杂种优势的重要组成部分。杂种优势较高的性状,检测到的QTLs会有更高比率表现显性或超显性,而杂种优势较低性状检测到的QTLs表现出较低比率的显性/超显性效应[38-39]。本研究中,开花期,吐丝期和开花吐丝间期3个开花期相关性状4个环境条件下IF2分别检测到5、6和4个QTLs,显性/超显性效应QTLs分别占40%、16.6%和75%。而3个开花期性状的杂种优势表现为:开花吐丝间期>开花期≈吐丝期。虽然,杂种优势最大的性状开花吐丝间期检测到最高比率的显性/超显性QTLs,但开花期和吐丝期相关的QTLs却没有表现出与前人结果相同的结论。这表明,该结论具有群体和性状的特异性。
3.2调控玉米开花期相关性状的重要基因和QTLs
本研究共检测到32个QTLs,分布在玉米基因组的10条染色体上,其中在染色体区间bin 1.02~1.03,bin 4.01,bin 4.07~4.08,bin 9.04和bin 10.03呈簇状分布。CHARDON等[27]对玉米开花期相关的QTL Meta-analysis鉴定了62个一致性QTLs,其中一定数量的一致性QTLs在染色体区域bin 4.08,bin 9.04和bin 10.03~10.04集中分布。此外,XU等[28]对玉米开花期和光周期敏感性的QTL Meta-analysis鉴定到25个一致性QTLs,这些QTLs在染色体区域bin 1.03,bin 1.05,bin 8.05和bin 10.04集中分布,本研究定位到的QTLs在bin 1.02~1.03和bin 10.03集中分布。DUCROCP等[12]在染色体区域bin 10.04检测到调控开花期的主效QTL,该主效QTL的候选基因为水稻Ghd7的同源基因,编码CCT-Domain蛋白,调控水稻抽穗期。本研究中定位到的qASI2与zfl2基因处于同一染色体区域,该基因调控花器的形态结构[12, 25];qASI3,qDS6和qDA6与CHARDON[27]等人对玉米开花期的QTL Meta-analysis的结果一致;qASI3与ts4基因处于同一染色体区域,该基因编码microRNA172,调控玉米由营养生长向生殖生长的转变和花器的性别决定[22];qDA9和qDS9与gl15基因处于同一染色体区域,该基因为microRNA172的靶基因,调控玉米由营养生长向生殖生长的转变[23];qDS10a与zfl1基因处于同一染色体区域,该基因调控玉米开花期[12, 25]。
[1] IRISH E E, NELSON T M. Identification of multiple stages in the conversion of vegetative to floral development [J]. Development, 1991, 112(3):891-898.
[2] BORTIRI E, HAKE S. Flowering and determinacy in maize [J]. J Exp Bot, 2007, 58(5):909-916.
[3] WANG C, CHENY, KU L, et al. Mapping QTL associated with photoperiod sensitivity and assessing the importance of QTL×environment interaction for flowering time in maize [J]. PloS one, 2010, 5(11):e14068.
[4] DANILEVSKAYA O N, MENG X, MCGONIGLE B, et al. Beyond flowering time: pleiotropic function of the maize flowering hormone florigen [J]. Plant Signal Behav, 2011, 6(9):1267-1270.
[5] CHANDLER JW.Floral meristem initiation and emergence in plants [J]. Cellular and molecular life sciences, 2012, 69(22):3807-3818.
[6] 金林峰,于海燕,吕艳杰.干旱胁迫下不同施氮量对玉米开花期叶片生理指标的影响 [J]. 吉林农业科技学院学报, 2015, 24(2): 9-11.
[7] 高世斌,赵茂俊,潘光堂,等.干旱胁迫和正常环境下控制玉米开花期性状的QTL鉴定 [J]. 西南农业学报,2005, 18(5):593-597.
[8] ALMEIDA G D, MAKUMBI D, MAGOROKOSHO C, et al. QTL mapping in three tropical maize populations reveals a set of constitutive and adaptive genomic regions for drought tolerance [J]. TheorAppl Genet,2013, 126(3):583-600.
[9] KOORNNEEF M, ALONSO-BLANCO C, PEETERS A J, et al. Genetic control of flowering time in Arabidopsis [J]. Annu Rev Plant Physiol, 1998, 49:345-370.
[10] BLAZQUEZ M A. Flower development pathways [J]. Journal of cell science, 2000, 113 (20):3547-3548.
[11] BUCKLER E S, HOLLAND J B, BRADBURY P J, et al. The genetic architecture of maize flowering time [J]. Science, 2009, 325(5941):714-718.
[12] DUCROCQ S, GIAUFFRET C, MADUR D, et al. Fine mapping and haplotype structure analysis of a major flowering time quantitative trait locus on maize chromosome 10 [J]. Genetics, 2009, 183(4):1555-1563.
[13] DUCROCQ S, MADUR D, VEYRIERAS J B, et al. Key impact of Vgt1 on flowering time adaptation in maize: evidence from association mapping and ecogeographical information [J]. Genetics, 2008, 178(4):2433-2437.
[14] SALVI S, SPONZA G, MORGANTE M, et al. Conserved noncoding genomic sequences associated with a flowering-time quantitative trait locus in maize [J]. Proc Natl AcadSci USA, 2007, 104(27):11376-11381.
[15] CHARDON F, HOURCADE D, COMBES V, et al. Mapping of a spontaneous mutation for early flowering time in maize highlights contrasting allelic series at two-linked QTL on chromosome 8 [J]. Theor Appl Genet, 2005, 112(1):1-11.
[16] BOUCHET S, SERVIN B, BERTIN P, et al. Adaptation of maize to temperate climates: mid-density genome-wide association genetics and diversity patterns reveal key genomic regions, with a major contribution of the Vgt2 (ZCN8) locus [J]. PloS one, 2013, 8:e71377.
[17] DANILEVSKAYA O N, MENG X, ANANIEV E V. Concerted modification of flowering time and inflorescence architecture by ectopic expression of TFL1-like genes in maize [J].Plant Physiol, 2010, 153(1):238-251.
[18] ANDERSEN J R, SCHRAG T, MELCHINGER A E, et al. Validation of Dwarf 8 polymorphisms associated with flowering time in elite European inbred lines of maize (ZeamaysL.) [J]. Theor Appl Genet, 2005, 111(2):206-217.
[19] COLASANTI J, TREMBLAY R, WONG A Y, et al. The maize INDETERMINATE1 flowering time regulator defines a highly conserved zinc finger protein family in higher plants [J]. BMC genomics, 2006, 19(7):158.
[20] KOZAKI A, HAKE S, COLASANTI J. The maize ID1 flowering time regulator is a zinc finger protein with novel DNA binding properties [J]. Nucleic Acids Res, 2004, 32(5):1710-1720.
[21] CHUCK G, MEELEY R, HAKE S. Floral meristem initiation and meristem cell fate are regulated by the maize AP2 genes ids1 and sid1 [J]. Development, 2008, 135(18):3013-3019.
[22] CHUCK G, MEELEY R, IRISH E, et al. The maize tasselseed 4 microRNA controls sex determination and meristem cell fate by targeting Tasselseed6/indeterminate spikelet1 [J]. Nat Genet, 2007, 39(12):1517-1521.
[23] LAUTER N, KAMPANI A, CARLSON S, et al. micro RNA172 down-regulates glossy15 to promote vegetative phase change in maize [J]. Proc Natl AcadSci USA, 2005, 102(26):9412-9417.
[24] CHUCK G, CIGAN AM, SAETEURN K, et al. The heterochronic maize mutant Corngrass1 results from overexpression of a tandem microRNA [J]. Nat Genet, 2007, 39(4):544-549.
[25] BOMBLIES K, WANG R L, AMBROSE B A, et al. Duplicate FLORICAULA/LEAFY homologs zfl1 and zfl2 control inflorescence architecture and flower patterning in maize [J]. Development, 2003, 130(11):2385-2395.
[26] BENSEN R J, JOHAL G S, CRANE V C, et al. Cloning and characterization of the maize An1 gene [J]. Plant Cell, 1995, 7(1):75-84.
[27] CHARDON F, VIRLON B, MOREAU L, et al. Genetic architecture of flowering time in maize as inferred from quantitative trait loci meta-analysis and synteny conservation with the rice genome [J]. Genetics, 2004, 168(4):2169-2185.
[28] XU J, LIU Y, LIU J, et al. The genetic architecture of flowering time and photoperiod sensitivity in maize as revealed by QTL review and meta-analysis [J]. Journal of Integrative plant Biology, 2012, 54(6):358-373.
[29] KNAPP S J, STROUP W W, ROSS W M. Exact confidence intervals for heritability on a progeny mean basis [J]. Crop Science, 1985, 25(1): 192-194.
[30] HUA J P, XING Y J, XU C G, et al. Genetic dissection of an elite rice hybrid revealed that heterozygotes are not always advantageous for performance [J]. Genetics, 2002, 162(4):1885-1895.
[31] HUA J, XING Y, WU W, et al. Single-locus heterotic effects and dominance by dominance interactions can adequately explain the genetic basis of heterosis in an elite rice hybrid [J]. Proc Natl AcadSci USA, 2003, 100(5):2574-2579.
[32] LIU Z, JI H, CUI Z, et al. QTL detected for grain-filling rate in maize using a RIL population [J]. Mol Breed, 2010, 27(1):25-36.
[33] TANG J, YAN J, MA X, et al. Dissection of the genetic basis of heterosis in anelite maize hybrid by QTL mapping in an immortalized F2 population[J]. TheorAppl Genet, 2010, 120(2):333-340
[34] ZENG Z B. Precision mapping of quantitative trait loci [J]. Genetics, 1994, 136(4):1457-1468.
[35] BRUCE A B. The Mendelian theory of heredity and the augmentation of vigor [J]. Science,1910, 32(827):627-628.
[36] JONES D F. Dominance of linked factors as a means of accounting for heterosis [J]. Genetics,1917,2(5):466-479.
[37] EASTEM. Heterosis [J]. Genetics,1936, 21(140):375-397.
[38] FRASCAROLI E, CANEMA, LANDIP, et al. Classical genetic and quantitative trait loci analyses of heterosis in a maize hybrid between two elite inbred lines [J]. Genetics,2007,176(1):625-644.
[39] GUO T, YANG N, TONG H, et al. Genetic basis of grain yield heterosis in an “immortalized F2” maize population [J]. TheorAppl Genet, 2014, 127(10):2149-2158.
(责任编辑:李 莹)
ComparativeQTLanalysisformaizefloweringtimerelatedtraitsbetweenanimmortalizedF2populationanditscorrespondingRILs
SHI Chaonan,ZHANG Zhanhui,TANG Guiliang,XUE Yadong
(College of Agronomy,Henan Agricultural University,Zhengzhou 450002,China)
Flowering time is an important agronomy trait for maize, which determines maize plant height, plant leaf number and grain yield. In the present study, a set ofRILandIF2(immortalized F2) populationboth come from Nongda108 were employed to identify QTLs for maize flowering time. And, 17 and 15 QTLs were identified for three flowering time related traits, including days to anthesis, days to silking and anthesissilking interval (ASI). These QTLs were mainly distributed in chromosomal regions bin 1.02~1.03, bin 4.00~4.01, bin 4.07~4.08, bin 9.04 and bin 10.03. However, onlyqDS1 was identified across the two populations. Most of these detected QTLs were consistent with several previous studies, and these QTLs probably contain genes involving in flowering time regulation. In maize breeding procedure, these QTLs can be used to screen elite inbred lines with proper flowering time via molecular assistant selection and thereby, to select new maize hybrids fit to mechanized harvest.
maize;IF2population;RIL; flowering time; QTL analysis
S513
:A
2015-10-10
小麦玉米作物学国家重点实验室开放课题 (39990002)
石超男(1992-),女,河南封丘人,硕士研究生,主要从事玉米分子遗传研究。
薛亚东(1978-),男,江苏盐城人,讲师,博士。
1000-2340(2016)02-0147-10

