第三芽孢梭菌致颅内感染的检测与分析
张爱平, 邹玖明, 尹艳华
第三芽孢梭菌致颅内感染的检测与分析
张爱平, 邹玖明*, 尹艳华
目的 对分离的1株耐氧生长的厌氧菌进行鉴定及药敏分析。方法 利用API 20A进行生化鉴定、琼脂稀释法进行药物敏感性试验、聚合酶链反应(PCR)和测序技术分析该菌的16SrRNA基因。头孢硝噻吩纸片法检测细菌β内酰胺酶,PCR扩增β内酰胺酶cfxA耐药基因。结果 经鉴定,该株厌氧菌为第三芽孢梭菌,经16SrRNA分析与美国国家生物技术信息中心数据库中第三芽孢梭菌相似性达100%,药敏结果显示该菌对亚胺培南、四环素、甲硝唑敏感,对氨苄西林、阿莫西林-克拉维酸、头孢替坦耐药。结论 该株厌氧菌为第三芽孢梭菌,携带cfxA耐药基因。
第三芽孢梭菌; 基因; cfxA; 药敏试验
第三芽孢梭菌是革兰染色阳性的芽孢杆菌,属于芽孢梭菌属。该菌寄生于哺乳动物及人类口腔、肠道中,可随粪便排出体外,在外界土壤中广泛存在。由于该菌不产毒素,一直以来认为没有致病意义。1917年HENRY[1]从战伤患者坏疽组织首次分离出第三芽孢梭菌,但不能肯定其致病性;直到1936年KING等[2]从败血症患者血液中分离出该菌,才首次阐明了其在人类疾病中的致病意义。2014年12月我院神经外科1例颅内感染患者脑脊液中分离出1株第三芽孢梭菌,现将检测过程和药敏试验结果报道如下。
1 材料与方法
1.1材料
1.1.1一般资料 患者,男,52岁。因“头痛、头晕伴发热2 d”入院。患者2 d前头皮外伤,已在我院门诊行清创缝合,但之后出现头痛、头晕伴体温升高。颅内穿刺显示颅内压增高(2.8 kPa),头皮伤口缝合处微红、肿,有渗出液。入院初即采集伤口渗出液、脑脊液及血液标本进行微生物学检查。
1.1.2试剂与仪器 API 20A试剂条购自法国生物梅里埃公司,聚合酶链反应(PCR)引物、DNA标记、Taq酶、各种限制性内切酶均购自大连宝生物公司,其他生化试剂购自美国SIGMA公司,序列分析在上海生工生物工程有限公司进行,Prism 7300型PCR扩增仪为美国ABI公司产品。血培养瓶,哥伦比亚血琼脂干粉、布氏琼脂为生物梅里埃公司产品。
1.2方法
1.2.1生化鉴定及药敏试验 将血培养瓶分离到的菌株转种至哥伦比亚血平皿,35 ℃培养48~72 h,按照API 20A试剂条操作程序进行生化鉴定。抗菌药物敏感性试验采用琼脂稀释法检测,具体操作及判断折点参考2012 CLSI M11-A7文件,厌氧环境孵育48 h后读取最低抑菌浓度(MIC)值。
1.2.216SrRNA鉴定 试验菌株接种哥伦比亚血平皿,35 ℃培养48~72 h,吸取1 mL菌液,采用细菌基因组DNA提取试剂盒制备模板,-20 ℃冰箱保存备用。16SrRNA的PCR扩增采用通用引物序列,产物大小944 bp,扩增产物测序后进行BLAST序列比对分析,见表1。

表1 16SrRNA、cfxA基因引物序列Table 1 Primers used in PCR assay for 16SrRNA and cfxA genes
1.2.3β内酰胺酶及其耐药基因检测 头孢硝噻吩(OXOID公司产品)纸片法检测细菌β内酰胺酶,PCR扩增β内酰胺酶cfxA耐药基因,引物参照文献合成[3-4]。核酸提取: 取布氏琼脂上生长48 h菌落悬于100 μL无菌去离子水,调整浊度至1.5麦氏单位,盖紧离心管于震荡器上震荡混匀,加热96 ℃,10 min,立即转移冰浴5 min后,12 000 r/min离心5 min,上清液作为扩增模板液。反应条件:94 ℃5 min预变性,94 ℃1 min,58 ℃1 min,72 ℃90 s,25个循环,最后72 ℃延伸5 min。PCR产物经1%琼脂糖凝胶电泳,Goldenview染色,凝胶成像仪读取结果。
2 结果
2.1培养结果菌落特征及生化鉴定
血液培养显示无细菌生长,伤口渗出液及脑脊液培养均分离出革兰阳性杆菌。
脑脊液及伤口渗出液培养各分离到1株细菌在哥伦比亚血平皿上生长缓慢,24 h后菌落针尖样大小,48~72 h方可见直径1~2 mm菌落,扁平透明,边缘光整,不溶血。厌氧和需氧环境培养,菌落革兰染色为阳性杆菌,但易脱色,厌氧培养条件下可见未染色芽孢,而需氧培养条件下未见,见图1。API 20A生化鉴定均为第三芽孢梭菌,生物编码一致,为47746023,符合率99.99%,该2株菌判断为同一株细菌。药敏试验结果显示对亚胺培南、四环素、甲硝唑敏感,而对氨苄西林、阿莫西林-克拉维酸、头孢替坦耐药,见表2。
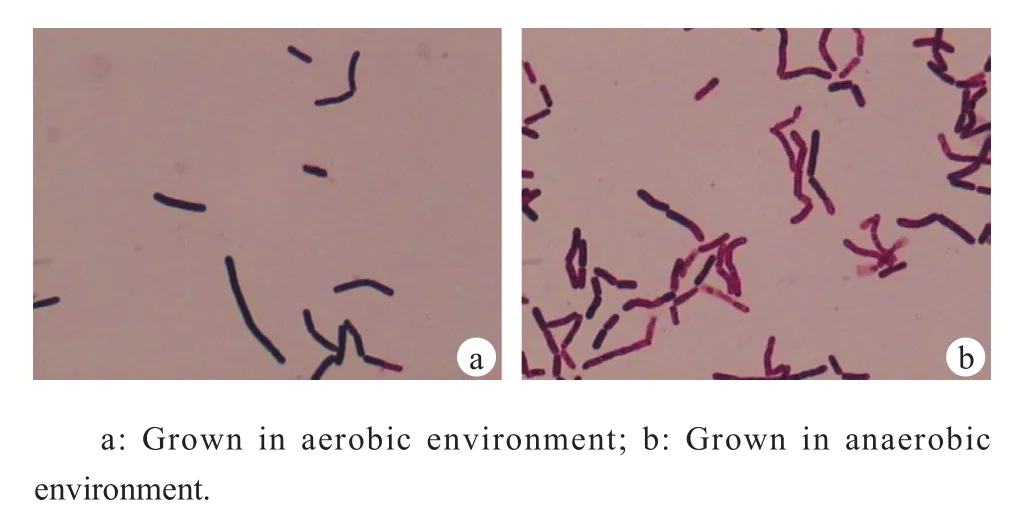
图1 第三芽孢梭菌革兰染色图(1000×)Figure 1 Representative gram stain images of Clostridium tertium(1000×)
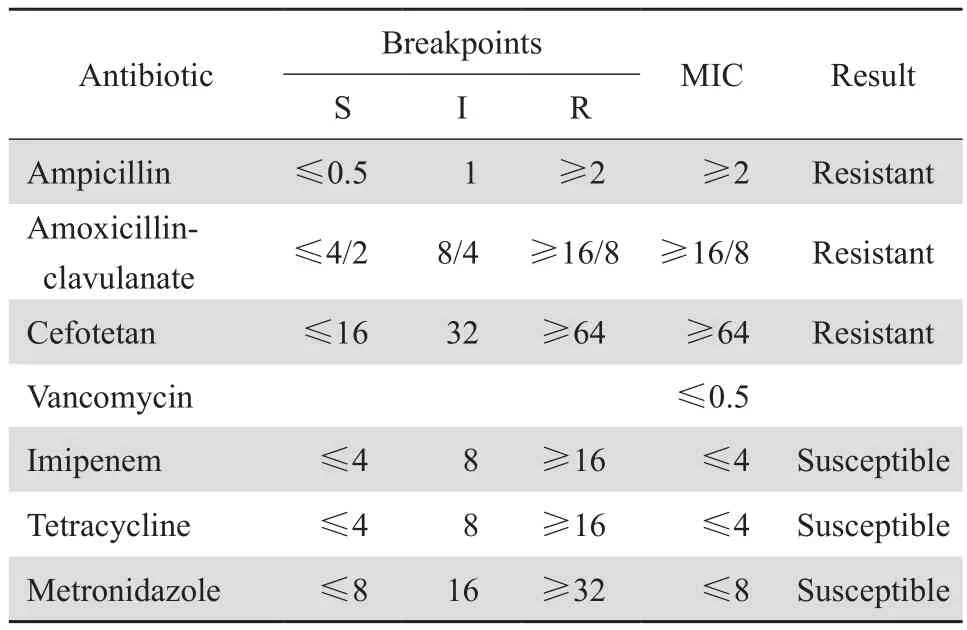
表2 第三芽孢梭菌抗菌药物敏感试验结果Table 2 Susceptibility of the Clostridium tertium strain to antimicrobial agents (mg/L)
2.216SrRNA种属鉴定
经过16SrRNA保守区基因扩增,电泳结果显示950 kb处有一清晰对应条带,见图2。将扩增后产物测序,BLAST序列比对显示与NCBI数据库中第三芽孢梭菌相似性达100%,为同一种属。

图2 16SrRNA保守区基因电泳结果Figure 2 Electrophoretogram of 16SrRNA gene amplified by PCR
2.3β内酰胺酶及其基因检测
来源于伤口渗出液及脑脊液的2株第三芽孢梭菌的β内酰胺酶检测均为阳性。PCR结果显示cfxA基因均阳性,见图3。
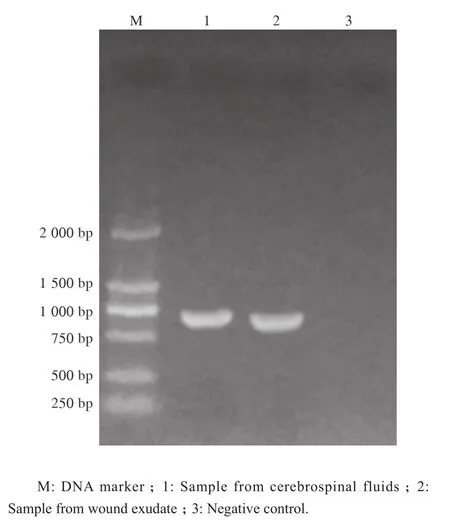
图3 cfxA基因检测电泳结果Figure 3 Electrophoretogram of cfxA gene amplified by PCR
3 讨论
依靠细菌的保守区基因设计引物对细菌进行分子生物学鉴定是准确可靠的方法之一。16SrRNA基因序列分子在结构与功能上具有高度的保守性,可以实现快速、准确地对微生物进行分类鉴定。本研究中,2株不同来源分离菌株通过生化反应均鉴定为第三芽孢梭菌,16SrRNA 分析进一步验证了表型结果。
由于该菌的生物学特性有异于其他种类细菌,因此很容易被错误鉴定或忽略。例如在革兰染色的过程中该菌很容易被脱色而误认为革兰阴性杆菌,在鉴别上可以观察在其他革兰阴性菌选择性平皿上(如麦康凯、含万古霉素巧克力平皿)的生长情况来综合判定,该菌不会在这类选择性平皿上生长。此外,该菌不同于其他的厌氧芽孢梭菌,在有氧环境下也可以缓慢生长,具有一定的耐氧性,这是很重要的一点,因为在需氧环境分离的第三芽孢梭菌很容易被误认为是乳杆菌或棒状杆菌而被忽略。它们的区别在于乳杆菌不会形成芽孢,第三芽孢梭菌在厌氧条件下可以形成芽孢;棒状杆菌属触酶试验阳性,而第三芽孢梭菌触酶试验阴性;其他的芽孢梭菌往往在有氧环境下形成芽孢,而第三芽孢梭菌与之恰恰相反。
以往认为第三芽孢梭菌不产生毒素,因此难以肯定它的致病意义[5]。但近期越来越多的报道显示,该菌在人类多种疾病中起着重要的作用。2011年,STEENSMA等[6]在1例糖尿病患者伴发坏死性软组织感染病灶中分离出第三芽孢梭菌;2013年SALVADOR等[7]报道第三芽孢梭菌在急性白血病伴中性粒细胞减少发热患者中引起急性血流感染;2015年,VIROT等[8]报道1例两伊战争后30年由于体内金属碎片残留而引起继发的第三芽孢梭菌骨关节感染。本例中,从患者头皮伤口渗出液、脑脊液均分离出第三芽孢梭菌,提示该菌与患者伤口愈合不良、颅内感染有关。
研究显示,第三芽孢梭菌感染主要与3个因素有关:肠黏膜屏障受损、中性粒细胞减少和β内酰胺类抗生素的过度使用(尤其是第三代广谱头孢菌素的使用)[9]。究其原因可能是因为第三芽孢梭菌定植于肠道,肠黏膜屏障受损有利于细菌侵入肠壁,随血流侵入远端部位,造成移位感染;中性粒细胞减少会降低机体的防御功能,不利于免疫系统对入侵细菌的吞噬杀灭;而广谱抗生素的使用会造成肠道菌群失衡,间接损伤肠黏膜屏障。尽管第三芽孢梭菌感染的患者多伴有发热,使用抗生素治疗一定程度上亦利于降低体温,但迄今为止没有直接证据显示发热是因为该菌感染引起。本研究中,患者并不伴中性粒细胞减少,抗生素的使用距离发病间隔时间短暂,因此病原菌很可能不是来自于肠道定植细菌移位,而是来自于外环境:患者头部缝合伤口感染,细菌经此途径侵入,造成颅内感染。本研究中分离的第三芽孢梭菌对甲硝唑、氟喹诺酮类、亚胺培南敏感,而对青霉素、头孢菌素类耐药,基因检测结果提示均携带cfxA基因。cfxA基因主要介导厌氧菌对β内酰胺类抗生素的耐药性,在厌氧菌耐药基因的水平传递中起着重要作用,在拟杆菌属、普雷沃菌属及二氧化碳嗜纤维菌中均有报道[10]。本例患者采用万古霉素治疗,病情好转。
目前有关第三芽孢梭菌研究的临床病例太少,尽管已经意识到其致病性,但该菌的毒力因子、致病机制、感染易感因素均不详,还有待于进一步探讨。
[1]HENRY H. An investigation of the cultural reactions of certain anaerobes found in wounds[J]. J Pathol Bacteriol, 2005,21(3):344-385.
[2]KING BM, RANCK BA, DAUGHERTY FD, et al.Clostridium tertium septicemia[J]. N Engl J Med, 1963,269:467-469.
[3]CHAUDHRY R, VERMA N, BAHADUR T, et al. Clostridium sordellil as a cause of constrictive pericarditis with pyopericardium and tamponade[J]. J Clin Microbiol, 2011,49 (10):3700-3702.
[4]FERNANDEZ-CANIGIA L, CEJAS D, GUTKIND G, et al. Detection and genetic characterization of β-lactamases in Prevotella intermedia and Prevotella nigrescens isolated from oral cavity infections and peritonsillar abscesses[J]. Anaerobe,2015,33:8-13.
[5]VANDERHOFSTADT M, ANDRE M, LONCHAY C, et al. Clostridium tertium bacteremia: contamination or true pathogen? A report of two cases and a review of the literature[J]. Int J Infect Dis, 2010,14(Suppl 3):e335-e337.
[6] STEENSMA EA, ERTL CW, BURKE LH.Clostridium tertium isolated from a necrotizing soft tissue infection in a diabetic but otherwise nonimmunocompromised patient[J]. J Am Col Certif Wound Spec,2011,3(2):42-44.
[7]SALVADOR F, PORTE L, DURAN L, et al. Breakthrough bacteremia due to Clostridium tertium in a patient with neutropenic fever, and identification by MALDI-TOF mass spectrometry[J]. Int J Infect Dis, 2013,17(11):e1062-e1063.
[8]VIROT E, SERVIEN E, LAURENT F, et al. Reactivation of Clostridium tertium bone infection 30 years after the Iran-Iraq war[J]. BMJ Case Rep,2015,pii:bcr2014209169.
[9]MILLER DL, BRAZER S, MURDOCH D,et al. Significance of Clostridium tertium bacteremia in neutropenic and nonneutropenic patients: review of 32 cases[J]. Clin Infect Dis,2001,32(6):975-978.
[10]GARCIA N, GUTIERREZ G, LORENZO M, et al. Genetic determinants for cfxA expression in Bacteroides strains isolated from human infection[J]. J Antimicrob Chemother,2008,62(5):942-947.
Characterization of a strain of Clostridium tertium causing intracranial infection
ZHANG Aiping, ZOU Jiuming, YIN Yanhua. (Intensive Care Unit, Xiangyang Central Hospital, Xiangyang Hubei 441021, China)
Objective To identify an anaerobic strain isolated from a patient with intracranial infection, and characterize the strain in terms of antibiotic resistance and relevant genes. Methods The strain was identified by API 20A. Susceptibility to antimicrobial agents was tested by agar dilution method. PCR and sequencing analysis was used to analyze 16SrRNA gene. Nitrocefin test was used to determine the production of lactamases. Beta-lactamase cfxA gene was detected by PCR. Results This anaerobic isolate was confirmed as Clostridium tertium. Susceptibility testing showed that this strain was susceptible to imipenem, tetracycline,metronidazole, and vancomycin, but resistant to ampicillin, amoxicillin-clavulanate, and cefotetan. The sequence of 16SrRNA gene showed 100% homologous with that in NCBI database. Conclusions This anaerobic isolate is confirmed as Clostridium tertium carrying cfxA gene, which mediates resistance to beta-lactam antibiotics.
Clostridium tertium; resistance gene; cfxA; antimicrobial susceptibility testing
·论著·
R378.8
A
1009-7708(2016)01-0071-04
10.16718/j.1009-7708.2016.01.016
湖北文理学院附属医院 襄阳市中心医院重症医学科,湖北襄阳 441021;*医学检验部。
张爱平(1982—),女,本科,护师,主要从事医院感染工作。
尹艳华,E-mail: 5650722273@qq.com。
2015-03-27
2015-07-21

