不同氮处理茶树实时定量PCR内参基因筛选和验证
刘圆,王丽鸳,韦康,成浩,张芬,吴立赟,胡娟
不同氮处理茶树实时定量PCR内参基因筛选和验证
刘圆,王丽鸳*,韦康,成浩*,张芬,吴立赟,胡娟
中国农业科学院茶叶研究所 国家茶树改良中心,浙江 杭州 310008
为筛选不同氮处理下茶树()实时荧光定量PCR(qRT-PCR)试验体系中最佳内参基因,以茶树幼叶、成熟叶和幼根为材料,应用qRT-PCR分析、、、、-tubulin、等6个内参基因在不同氮浓度和氮源处理下的表达变化,借助GeNorm和NormFinder程序对候选内参基因稳定性进行评价。结果表明,在不同氮浓度和氮源处理下,、-actin和表达稳定性较好,+组合稳定性最佳,可用作茶树基因表达研究的内参基因;而-tubulin和表达稳定性较差,不适合做内参基因。为进一步证实上述结果,分别以、-tubulin 、+组合为内参基因,分析茶树幼叶中硝酸根转运蛋白基因()和铵根转运蛋白基因()的表达水平,发现以和+组合为内参基因时,目的基因的相对表达量随处理时间延长变化规律基本一致;而以-tubulin为内参基因时,目的基因的变化规律与前两者存在明显差别。因此,根据特定试验体系选择合适的内参基因对于qRT-PCR定量结果的准确性和可靠性具有重要意义。
茶树;内参基因;氮处理;qRT-PCR
实时荧光定量PCR(Quantitative real-time reverse-transcriptase PCR, qRT-PCR)因其定量准确、灵敏度高、重复性好、高通量等特点已广泛应用于基因表达水平检测[1-3]。在qRT-PCR相对定量检测中,为避免不同样品在RNA质量、逆转录效率和PCR反应条件上可能存在的差异,通常选择相应的内参基因进行校正和标准化[4-5]。然而,近年来的研究表明,在不同组织、器官、发育时期和不同试验条件下都稳定表达的内参基因并不存在[6-7],因此,根据特定试验体系选择稳定表达的内参基因,已成为qRT-PCR结果准确可靠的重要前提。
目前,不同氮处理下内参基因的筛选及定量表达研究在黄瓜[8]、玉米[9]、番茄[10]、咖啡[11]和盐角草[12]上已有报道,而茶树中则主要集中在病害、虫害、冷害、干旱和盐胁迫等方面[13-17],不同氮素浓度和氮源处理下内参基因筛选研究未见报道。氮素作为茶树生长的必需元素,与茶树新梢产量及品质密切相关[18]。不同氮素处理下,植物生理功能会受到不同程度的影响,同时mRNA和蛋白质水平也会发生相应的改变[19-20]。因此,筛选不同氮处理下稳定表达的内参基因对茶树功能基因研究具有重要意义。
本研究选取6个候选内参基因,包括核糖体、甘油醛-3-磷酸脱氢酶基因、肌动蛋白基因-actin、微管蛋白基因、1,5-二磷酸核酮糖脱羧酶/加氧酶基因、核糖体蛋白基因,采用qRT-PCR技术,结合GeNorm和NormFinder程序对它们在不同氮处理下茶树不同组织间的表达稳定性进行评价,以期筛选出不同氮处理下稳定表达的内参基因,为后续茶树氮吸收和代谢相关的功能基因研究提供参考。
1 材料与方法
1.1 试验材料与处理
供试茶树为生长在自然光照温室下的龙井43半年生实生苗。选取生长良好、大小一致的茶苗,用蒸馏水清洗后转移至不透光蓝色周转箱(长45 cm,宽30 cm,高12 cm),每箱28株。营养液参照Ruan等[21-22]的配方,氮源为1 mmol·L-1NH4NO3,调节pH 6.0,每3 d更换营养液并持续通气,预培养30 d。预培养结束后,对部分茶苗进行氮饥饿7 d,然后转至不同氮处理的营养液中。
不同氮浓度处理:设置高氮(5 mmol·L-1NH4NO3)、正常氮(1 mmol·L-1NH4NO3)和缺氮3个梯度。
不同氮源处理:设置混合态氮(1 mmol·L-1NH4NO3)、硝态氮(2 mmol·L-1NaNO3)和铵态氮[1 mmol·L-1(NH4)2SO4]3种氮源。
处理期间,每2 d更换一次营养液。样品处理后7 d和15 d,分别取一芽二叶、成熟叶和幼根,每个样品设3个生物学重复,液氮速冻后-80℃保存备用。
1.2 总RNA的提取和cDNA合成
取50~100 mg茶树样品,TissuelyzerⅡ(QIAGEN)充分研磨,使用RNeasy®Plant Mini Kit提取总RNA,利用NanoDrop 2000c(USA)和1.2%琼脂糖凝胶电泳检测核酸质量。每个样品取1mg总RNA,使用FastQuant RT Super Mix试剂盒反转录合成cDNA第1链,-20℃保存备用。
1.3 引物设计和qRT-PCR分析
本研究选取等6个基因为候选内参基因,其中、、引物参照孙美莲等[13]的方法,其余基因利用Primer5.0软件设计引物,并利用Primer-BLAST(http://ncbi.nlm.nih.gov./tools/primer-BlAST)检测引物序列的特异性。所有引物由英俊生物技术有限公司合成(表1)。
qRT-PCR反应体系为:SYBR®Premix Ex TaqTM(2×)10.0mL,上、下游引物(10mmol·L-1)各0.8mL,ROX DyeⅡ(50×) 0.4mL,cDNA模板2.0mL,加ddH2O至终体积20.0mL,每个样品3次技术重复。反应在ABI PRISM®7500 PCR仪(USA)上进行,PCR扩增反应程序:95℃预变性30 s;95℃变性5 s,60℃退火延伸34 s,40个循环;扩增完毕后,温度提升至95℃获取样品熔解曲线,以确定扩增产物特异性。

1.4 数据分析
根据公式QEΔCt及扩增样品的Ct值,计算内参基因的相对表达量。其中E为基因扩增效率,ΔCt=Ctmin-Ct样品(Ctmin为样品中最低Ct值;Ct样品为每个样品Ct值)[23]。采用ΔCt法[24]分析内参基因的表达丰度,使用GeNorm[25]和NormFinder[26]程序对候选基因表达稳定性进行评价,并通过标准化因子的配对变异系数Vn/n+1值确定最佳内参基因数目。一般认为当Vn/n+1值小于0.15时,无需引入新的内参基因;反之,则需引入第n+1个基因[25]。
2 结果与分析
2.1 样品总RNA质量检测
茶树各组织样品的总RNA经NanoDrop 2000c和1.2%琼脂糖凝胶电泳检测,A260/280均在1.8~2.1,A230/260在2.0左右,RNA电泳图谱带型清晰,28 S亮度约为18 S的两倍,表明RNA样品纯度较高,完整性较好,可进行后续实验。
2.2 内参基因引物特异性
以幼叶总RNA反转录产物为模板进行普通PCR扩增,经过2%琼脂糖凝胶电泳检测,各个样品扩增片段大小与预测的结果一致。进一步以茶树不同组织样品cDNA为模板进行qRT-PCR反应(图1),结果显示所有基因的熔解曲线均为明显单一峰,且样品的PCR扩增曲线重复性较好,表明所设计的引物均能特异性扩增各个基因的相应产物,不存在引物二聚体,因此可证实qRT-PCR的分析结果准确可靠。
2.3 内参基因的表达分析
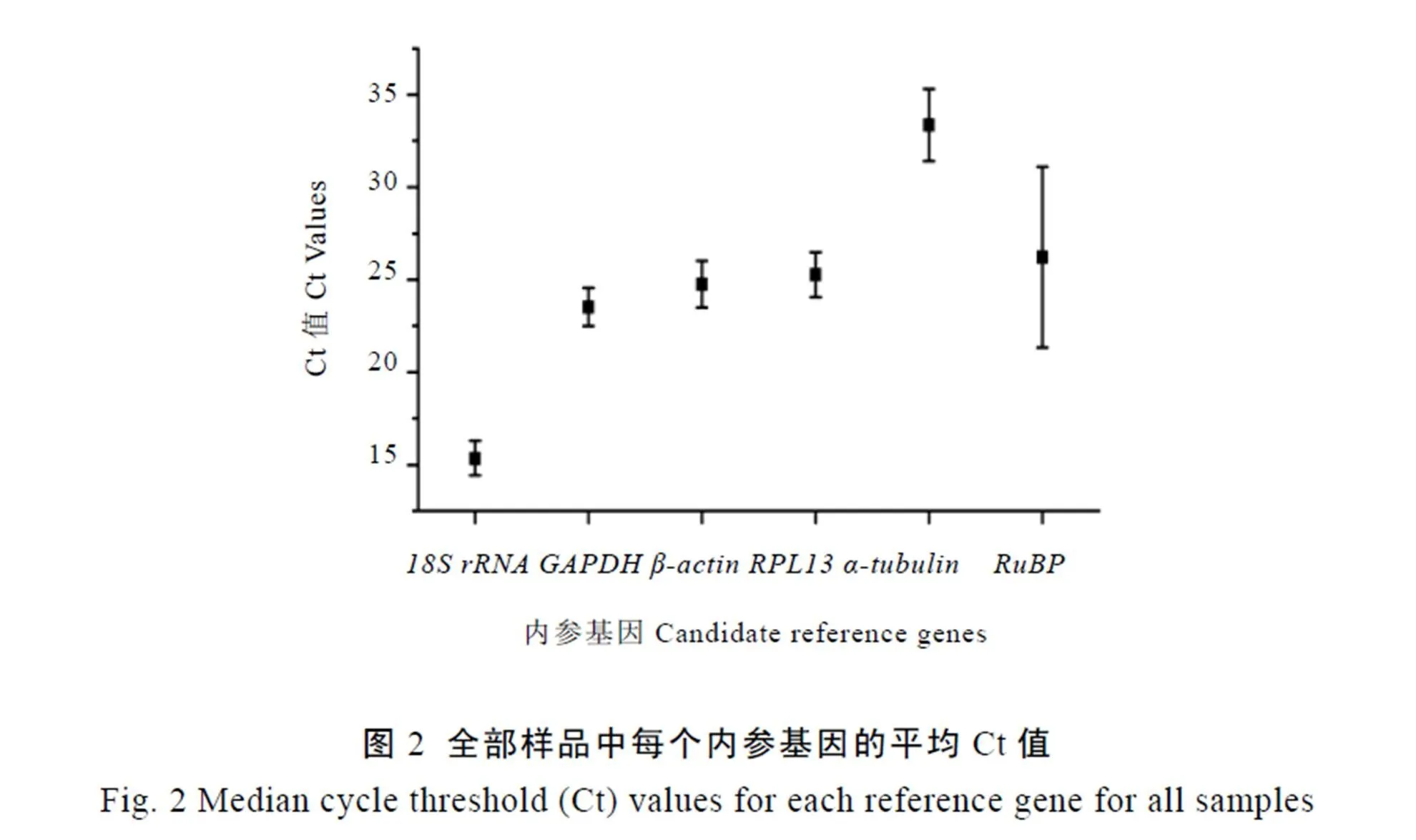
qRT-PCR分析了6个内参基因在所有处理样品中的表达丰度(图2),结果显示,所有内参基因Ct值处于15(,SD±0.92)至33(-tubulin,SD±2.0)之间,大部分内参基因的Ct值分布在20~26之间;其中的平均Ct值最高,达33.4,相对于其他内参基因在所有处理样品中表达丰度最低。的平均Ct值最低,为15.4,相对于其他内参基因在所有处理样品中表达丰度最高;6个内参基因在所有处理样品中的转录表达均存在一定变化,其中、和的Ct值标准差较小,表达丰度相对稳定。而的Ct值标准差最大,表达稳定性最差。

2.4 内参基因稳定性分析
2.4.1 GeNorm软件分析
GeNorm通过分析内参基因在不同样品中表达稳定性值(Expression stability value,M)并进行排序,以确定最稳定的内参基因。M值越大,稳定性越差;反之,则越高。逐步排除高M值的基因,直至剩下M值最低的一对基因。将每次排除高M值基因后所剩候选基因重新计算M值,最后按每次计算后的M值的平均值进行排序即可得到基因稳定性趋势图[20,25]。本研究将处理时间7 d和15 d样品分成两组,分别计算它们在所有处理样品中基因的表达稳定性(图3)。从GeNorm分析后的数据看,相较7 d处理,15 d处理后所有基因的M值都呈下降趋势,这表明可能随着处理时间的延长,植物体内代谢和生理活动变化减弱,整体基因表达趋于稳定。

GeNorm分析表明,6个内参基因表达稳定性各异。在不同氮浓度处理下,幼叶中和-actin稳定性最好(图4-A),成熟叶中和表达最稳定(图4-B),幼根中-actin和稳定性最好(图4-C);不同氮浓度处理下不同组织间,-actin和表达最稳定(图4-D);不同氮源处理下不同组织间,和-actin稳定性最好(图4-E);所有处理样品池中,和-actin表达最稳定(图4-F)。此外,在大多数处理下和-tubulin稳定性较差。

本研究中各个处理的V2/3均小于0.15阈值(图5),表明进行基因表达分析时最佳内参基因数为2个。不同氮浓度处理下幼叶、成熟叶和幼根中,分别可选择+-actin+和-actin+组合为内参基因。不同氮源处理下各个组织及所有处理样品池中,可使用+-actin组合作为内参基因。

2.4.2 NormFinder软件分析
NormFinder主要基于方差分析对内参基因稳定性进行排序,并引入不同样品组间表达差异[26]。结果表明(表2),不同氮浓度处理下,在幼叶中表达最稳定,-actin在成熟叶和幼根中表达最稳定。不同氮浓度处理各个组织间,单基因-actin表达最稳定,+组合表达稳定性最好。不同氮源处理各个组织和所有处理样品池中单基因表达最稳定,而稳定性最佳的组合分别为+和+。此外,除不同氮浓度处理成熟叶外,NormFinder的分析结果与GeNorm基本一致。

2.4.3 综合评价
综合比较2种软件评价结果(表3),在所有处理样品池中、-actin和表达稳定性较好,可用作氮处理下内参基因。不同氮浓度处理下,成熟叶中表达最稳定的组合为+;不同氮浓度处理幼根和各个组织间,-actin+为最佳组合;不同氮浓度处理幼叶、不同氮源处理各个组织间和所有处理样品池中表达稳定性最好的组合为+-actin。
2.5 内参基因稳定性验证
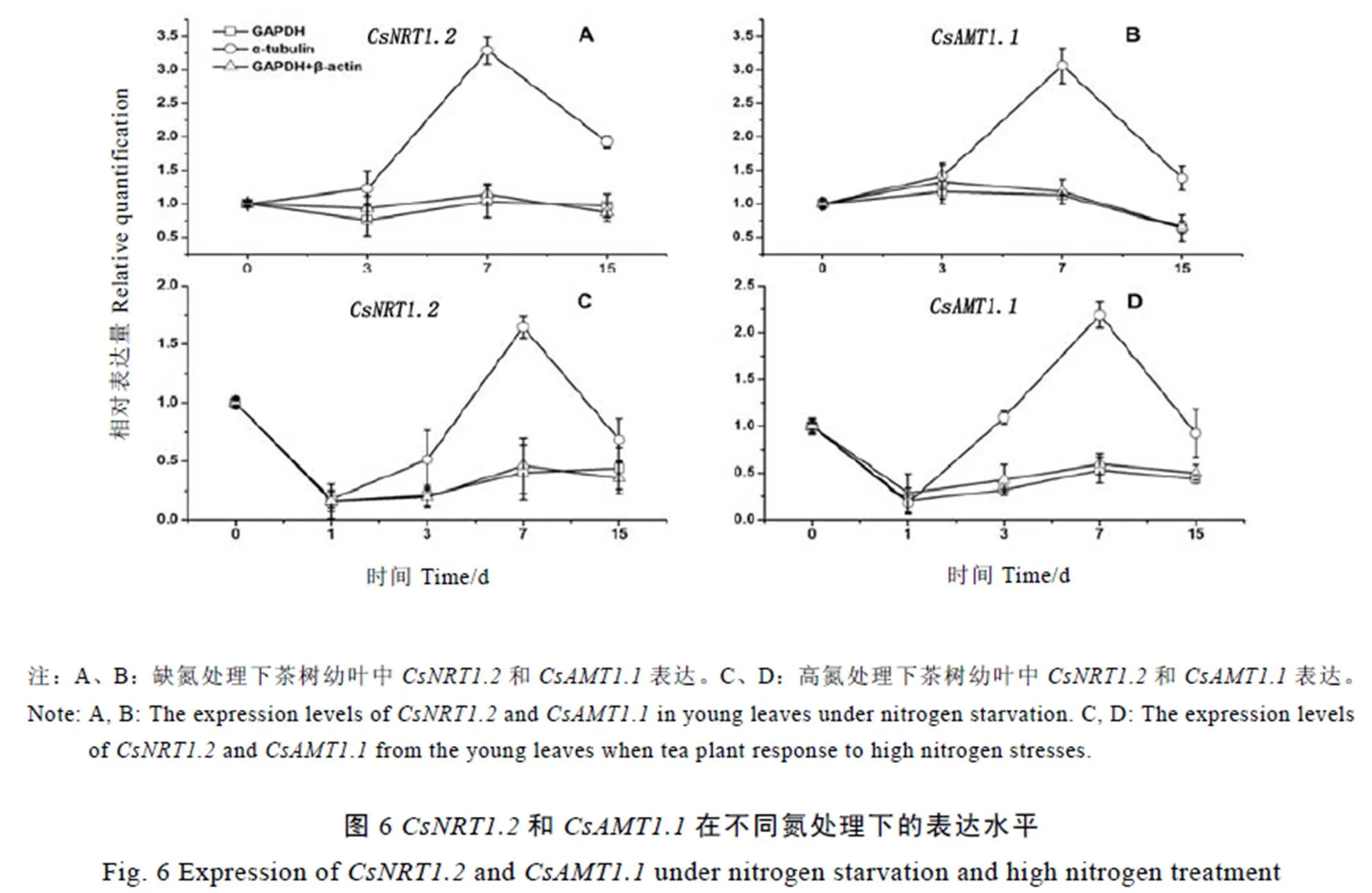
硝酸根转运蛋白(Nitrate transporter, NRT)和铵根转运蛋白(Ammonium transporter,AMT)主要参与植物根系对硝酸盐(NO3-)和铵盐(NH4+)的吸收及在不同组织和器官间转运,其中和分别参与植物根系对硝态氮低亲和吸收和铵态氮高亲和吸收[27-29]。为验证内参基因对qRT-PCR结果的影响,根据不同氮浓度处理下茶树幼叶中内参基因稳定性评价结果,分别以稳定性较好的、稳定性较差的-tubulin和稳定性最佳的组合+-actin为内参基因,分析和在幼叶中的表达模式。由图6可知,以或+-actin为内参基因时,目的基因的相对表达量随处理时间延长变化规律基本一致,表明这两个基因表达稳定性较好;而以-tubulin为内参基因时,目的基因的相对表达量变化规律与前两者存在明显差别,表明-tubulin在不同氮处理幼叶中表达稳定性较差,不能对目的基因的相对表达量进行准确校正。
3 讨论
本研究所有处理样品池中、-actin和3个内参基因稳定性最好,可能由于这些基因参与生物体基本生命活动。是糖酵解、糖异生及光合作用碳固定循环过程中的关键酶,表达量较高[30]。-actin为生物体维持生命活动所必需细胞骨架的基本成分。真核生物中与核内rRNA结合构成60S大亚基,在蛋白质生物合成中起重要作用,且具有较强的保守性[31-32]。孙美莲等[13]研究表明,在茶树不同成熟度叶片和愈伤组织中较稳定,而在不同器官中-actin最稳定。郝姗[16]对不同逆境条件下茶树幼叶研究表明,在冷害、干旱、伤害和盐胁迫下表达稳定性较好。Jain等[33]研究发现,在冷害、盐害、脱水等胁迫处理下水稻幼苗中转录表达水平不变。Ye等[34]对樱桃不同基因型和不同组织、器官中基因表达研究发现,表达较稳定。作为传统内参基因之一,在茶树qRT-PCR中已经得到普遍应用[35-36]。然而在本研究中,虽然在所有处理样品中表达丰度很高,但其除在不同氮浓度处理下成熟叶和幼根中稳定性较好外,它在其他处理下稳定性并不理想。这可能因为在qRT-PCR中表达丰度过高,而目的基因丰度较低,导致定量结果准确性降低[25]。和-tubulin在所有处理样品中表达稳定性均较差,然而Gohain等[14]对茶树不同试验条件下4个常用内参基因稳定性研究发现,稳定性较好。在灵芝[37]不同组织间、盐角草[12]不同氮胁迫下-tubulin表达最稳定。可见,同样的内参基因在不同作物品种或试验条件下表达稳定性存在很大差异。这可能因为其所归属的基因家族中不同基因间具有组织特异性或对不同试验条件响应不同[1,38]。本研究中内参基因稳定性验证的结果进一步证实根据特定试验体系选择合适的内参基因是qRT-PCR定量结果准确可靠的关键。此外,在进行目的基因表达分析前,有必要对内参基因在某一具体试验条件下的表达稳定性进行评价[39]。
本研究中、-actin和3个内参基因在氮处理下稳定性均较好,其中作为新的内参基因首次在茶树内参基因研究中得到验证。-tubulin和稳定性较差,不适宜用作氮处理下内参基因。为消除单一内参基因所带来的定量偏差,增加结果的可靠性和准确度,建议选择+-actin组合进行氮处理下目的基因校正。此外,随着茶树全基因组测序研究的深入,内参基因的获得将不再局限于几个传统内参基因,除了EST数据库外还可以像拟南芥[40]、水稻[41]等模式生物一样,利用基因芯片数据进行全基因组搜索并经试验验证来获得更多稳定可靠的新内参基因[42]。
[1] Bustin S A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): Trends and problems [J]. Journal of Molecular Endocrinology, 2002, 29(1): 23-39.
[2] Bustin S A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays [J]. Journal of Molecular Endocrinology, 2000, 25(2): 169-193.
[3] Radonić A, Thulke S, Mackay I M, et al. Guideline to reference gene selection for quantitative real-time PCR [J]. Biochemical and Biophysical Research Communications, 2004, 313(4): 856-862.
[4] Gutierrez L, Mauriat M, Guenin S, et al. The lack of a systematic validation of reference genes: A serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR) analysis in plants [J]. Plant Biotechnology Journal, 2008, 6(6): 609-618.
[5] Brunner A M, Yakovlev I A, Strauss S H. Validating internal controls for quantitative plant gene expression studies [J]. BMC Plant Biol, 2004, 4:14.
[6] Gutierrez L, Mauriat M, Pelloux J, et al. Towards a systematic validation of references in real-time RT-PCR [J]. Plant Cell, 2008, 20(7): 1734-1735.
[7] Thellin O, Zorzi W, Lakaye B, et alHousekeeping genes as internal standards: Use and limits [J]. Journal of Biotechnology, 1999, 75(2/3): 291-295.
[8] Warzybok A, Migocka M. Reliable reference genes for normalization of gene expression in cucumber grown under different nitrogen nutrition [J]. PLoS One, 2013, 8(9): e72887.
[9] Manoli A, Sturaro A, Trevisan S, et al. Evaluation of candidate reference genes for qPCR in maize [J]. Journal of Plant Physiology, 2012, 169(8): 807-815.
[10] Lovdal T, Lillo C. Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen, cold, and light stress [J]. Analytical biochemistry, 2009, 387(2): 238-242.
[11] De Carvalho K, Bespalhok Filho J C, Dos Santos T B, et al. Nitrogen starvation, salt and heat stress in coffee (L.): Identification and validation of new genes for qPCR normalization [J]. Molecular Biotechnology, 2013, 53(3): 315-325.
[12] Xiao X L, Ma J B, Wang J R, et al. Validation of suitable reference genes for gene expression analysis in the halophyteby real-time quantitative PCR [J]. Frontiers in Plant Science, 2015, 5: 788.
[13] 孙美莲, 王云生, 杨冬青, 等. 茶树实时荧光定量PCR分析中内参基因的选择[J]. 植物学报, 2010, 45: 579-587.
[14] Gohain B, Bandyopadhyay T, Borchetia S,et al. Identification and validation of stable reference genes inSpecies [J]. Journal of Biotechnology and Pharmaceutical Research, 2011, 2(1): 9-18.
[15] Gohain B, Bandyopadhyay T, Bhorali P,et al. Rubisco-bis-phosphate oxygenase ()-a potential housekeeping gene for qPCR assays in tea [J]. African Journal of Biotechnology, 2012, 11(51): 11193-11199.
[16] 郝姗. 茶树不同逆境条件下qRT-PCR适宜内参基因的筛选[D]. 南京: 南京农业大学, 2012: 47-55.
[17] Hao X, Horvath D, Chao W, et al. Identification and evaluation of reliable reference genes for quantitative real-time PCR analysis in tea plant ((L.) O. Kuntze) [J]. International Journal of Molecular Sciences, 2014, 15(12): 22155-22172.
[18] Okano K, Chutani K, Matsuo K. Suitable level of nitrogen fertilizer for tea (L.) plants in relation to growth, photosynthesis, nitrogen uptake and accumulation of free amino acids [J]. Japanese Journal of Crop Science, 1997, 66(2): 279-287.
[19] Urbanczyk-Wochniak E, Fernie A R. Metabolic profiling reveals altered nitrogen nutrient regimes have diverse effects on the metabolism of hydroponically-grown tomato () plants [J]. Journal of Experimental Botany, 2005, 56(410): 309-321.
[20] 魏毅东, 陈玉, 郭海萍, 等. 水稻缺素胁迫下实时荧光定量RT-PCR的内参基因的选择[J]. 农业生物技术学报, 2013, 21: 1302-1312.
[21] Ruan J Y, Gerendas J, Hardter R,et al. Effect of nitrogen form and root-zone ph on growth and nitrogen uptake of tea () plants [J]. Annals of Botany, 2007, 99(2): 301-310.
[22] Ruan J, Gerendás J, Härdter R, et al. Effect of root zone pH and form and concentration of nitrogen on accumulation of quality-related components in green tea [J]. Journal of the Science of Food and Agriculture, 2007, 87(8): 1505-1516.
[23] 蒋晓梅, 张新全, 严海东, 等. 柳枝稷根组织实时定量PCR分析中内参基因的选择[J]. 农业生物技术学报, 2014, 22: 55-63.
[24] Silver N, Best S, Jiang J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR [J]. BMC Molecular Biology, 2006, 7(1): 33.
[25] Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J]. Genome Biology, 2002, 3(7): research0034.1-0034.11.
[26] Andersen C L, Jensen J L, Orntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets [J]. Cancer Research, 2004, 64(15): 5245-5250.
[27] 汪进, 添先凤, 江昌俊, 等. 茶树硝酸盐转运蛋白基因的克隆和表达分析[J]. 植物生理学报, 2014, 50: 983-988.
[28] Wang YY, Hsu PK, Tsay YF. Uptake, allocation and signaling of nitrate [J]. Trends in Plant Science, 2012, 17: 458-467.
[29] Wu X, Yang H, Qu C,. Sequence and expression analysis of the AMT gene family in poplar [J]. Frontiers in Plant Science, 2015, 6: 337.
[30] Faccioli P, Ciceri G P, Provero P, et al. A combined strategy of ′in silico′ transcriptome analysis and web search engine optimization allows an agile identification of reference genes suitable for normalization in gene expression studies [J]. Plant Mol Biol, 2007, 63(5): 679-688.
[31] Olvera J, Wool I G. The primary structure of rat ribosomal protein L13 [J]. Biochemical and Biophysical Research Communications, 1994, 201(1): 102-107.
[32] Martinez-Guitarte J L, Planello R, Morcillo G. Characterization and expression during development and under environmental stress of the genes encoding ribosomal proteins L11 and L13 in[J]. Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology, 2007, 147(4): 590-596.
[33] Jain M, Tyagi S B, Thakur J K,et al. Molecular characterization of a light-responsive gene, breast basic conserved protein 1 (), encoding nuclear-localized protein homologous to ribosomal protein L13 from[J]. Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression, 2004, 1676(2): 182-192.
[34] Ye X, Zhang F M, Tao Y H, et al. Reference gene selection for quantitative real-time PCR normalization in different cherry genotypes, developmental stages and organs [J]. Scientia Horticulturae, 2015, 181: 182-188.
[35] Wang X C, Zhao Q Y, Ma C L, et al. Global transcriptome profiles ofduring cold acclimation [J]. BMC Genomics, 2013, 14(1): 415.
[36] Wei K, Wang L Y, Wu L Y, et al. Transcriptome analysis of indole-3-butyric acid-induced adventitious root formation in nodal cuttings of(L.) [J]. PLoS One, 2014, 9(9): e107201.
[37] Lim F-H, Fakhrana I N, Rasid O A, et al. Isolation and selection of reference genes forgene expression study using quantitative Real-time PCR (qPCR) [J]. Journal of Oil Palm Research, 2014, 26(2): 170-181.
[38] An Y Q, Mcdowell J M, Huang S R,et al. Strong, constitutive expression of theactin subclass in vegetative tissues [J]. Plant Journal, 1996, 10(1): 107-121.
[39] 周晓惠, 刘军, 庄勇. 喀西茄内参基因实时荧光定量PCR表达稳定性评价[J]. 园艺学报, 2014, 41: 1731-1738.
[40] Czechowski T, Stitt M, Altmann T, et al. Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis [J]. Plant Physiol, 2005, 139(1): 5-17.
[41] Jain M. Genome-wide identification of novel internal control genes for normalization of gene expression during various stages of development in rice [J]. Plant Science, 2009, 176(5): 702-706.
[42] 周兰, 张利义, 张彩霞, 等. 苹果实时荧光定量PCR分析中内参基因的筛选[J]. 果树学报, 2012, 29(6): 965-970.
Screening and Validation of Reference Genes for Quantitative Real-time PCR Analysis in Tea Plant () under Different Nitrogen Nutrition
LIU Yuan, WANG Liyuan*, WEI Kang, CHENG Hao*, ZHANG Fen, WU Liyun, HU Juan
Tea Research Institute of the Chinese Academy of Agricultural Sciences, National Center for Tea Improvement, Hangzhou 310008, China
The objective of this study was to select the most reliable reference genes for qRT-PCR analysis of target tea plant genes under varying nitrogen source and availability. We chose 6 housekeeping genes which included five commonly used and one new candidates to systematically assess their expression levels at three different tissues (young leaves, mature leaves and roots) under different nitrogen regimes by qRT-PCR. GeNorm and NormFinder software were used to analyze and evaluate the data for reference genes. The results indicated that,andare the best reference genes for normalizing target gene expression in tea plant under different nitrogen nutrition, whereasandare not suitable in many experimental conditions and the best combination (+) was recommended. Meanwhile, the expression levels ofandin young leaves of tea plants were analyzed. The results showed that the variation tendency ofandare exactly consistent when usingand+as reference genes. However, the expression levels of these genes are showed significant differences whenwas used as a reference gene. Thus, validation of
suitable reference genes for specific condition can guarantee the accurate quantification of the target genes in qRT-PCR analysis.
tea plant (), reference gene, nitrogen treatment, qRT-PCR
S571.1;S143.1
A
1000-369X(2016)01-092-10
2015-04-14
2015-07-27
浙江省茶树农业新品种选育重大科技专项(2012C12905)、国家茶叶产业技术体系(nycytx-23)。
刘圆,男,硕士研究生,主要从事茶树分子生物学研究。*通讯作者:chenghao@mail.tricaas.com

