核糖体展示研究进展
郭园
(内蒙古农业大学职业技术学院,包头 014109)
核糖体展示研究进展
郭园
(内蒙古农业大学职业技术学院,包头 014109)
核糖体展示是一种无细胞系统,可以从文库中筛选蛋白质和多肽。翻译的蛋白质及其mRNA同时结合在核糖体上形成mRNA-核糖体-蛋白质三聚体,通过配体亲和分离得到功能性蛋白及其编码的mRNA,转换成对应的DNA后进行相关蛋白的表达,可用于抗体及蛋白质文库选择、蛋白质体外改造等,而且其可以展示较大的文库而不受细菌转化的限制,可对毒蛋白、蛋白酶敏感和不稳定的蛋白质进行筛选,也可在特定位点进行氨基酸修饰。就核糖体展示技术的研究进展及其在蛋白质进化和筛选方面的应用进行综述。
核糖体展示;抗体;选择;蛋白质进化
展示系统可以从蛋白和肽为表型的文库中,筛选与它们基因型相关的DNA或RNA,同时也可以通过多轮突变、筛选、复制进行进化,最终改变表型特征。而成功的展示则依赖于功能蛋白恢复其遗传信息的能力。目前,已设计和验证出一些基于细胞的展示方法,如噬菌体展示[1,2]和细胞表面展示[3-5]。另外,还有无细胞方法,如核糖体展示[6,7]和mRNA 展示[8,9]。
核糖体展示技术是在体外进行的,可避免一些基于细胞展示系统的限制。通过在无细胞系统中形成mRNA-核糖体-蛋白质三聚体(PRM),将新生蛋白与它们的相关mRNA分子联系起来,通过蛋白质的功能特性选择遗传物质。mRNA可进行扩增或通过RT-PCR反转录成DNA,再进行循环、突变或克隆。近些年,研究人员利用核糖体展示技术进行广泛的研究,对传统的核糖体展示技术进行了改进,成功筛选到多种对靶分子有特异性亲和力的抗体、酶和多肽等大分子及一些小分子。
1 核糖体展示原理
核糖体展示技术的本质是利用形成的mRNA-核糖体-蛋白质三聚体复合物,通过核糖体把新生蛋白和其mRNA联系起来。最早的核糖体展示系统利用E. coli S30进行肽的筛选[10],合成的DNA文库编码随机肽序列通过加入氯霉素终止翻译,产生多核糖体复合物。展示了肽表位的特定复合物通过固定化抗体进行捕获,得到的多核糖体加入EDTA使其分解释放绑定的mRNA,进而通过RT-PCR反转录成cDNA(图1)。之后发展出的真核核糖体展示系统ARM(antibody-ribosome-mRNA)则被用于功能性单链抗体片段的筛选[7,11]。通过删除PCR片段的终止密码子产生真核PRM复合物,产生的PRM复合物随后通过抗原包被的磁珠进行捕获,mRNA通过RT-PCR无需解离核糖体复合物就可得到相应的DNA。而特异的复合物可以从突变文库中进行富集,改进的ARM展示还可在mRNA中引入突变,用于体外蛋白质定向进化和酶的筛选[12]。

图1 核糖体展示原理
转化效率限制了基于细胞系统的文库多样性,核糖体展示相对噬菌体和细胞表面展示有很多优势。较大的文库可以通过每个循环进行筛选,PCR产物可以直接用作模板,避免克隆过程,很容易就可得到巨大多样性的PCR文库,展示文库的大小仅取决于反应中的功能性核糖体的数量,可以高达1014/mL[7]。因此,更大的文库给核糖体展示提供一个绝佳的机会,可用于选择稀有的序列和高亲和性结合位点。利用PCR方法还可在选择后将更多的多样性引入DNA库中,这就提供了一个有效的蛋白质进化途径。此外,无细胞系统可以表达有毒的、对蛋白酶敏感或不稳定的蛋白,这些都是细菌表达系统通常达不到的[13]。真核无细胞系统也可进行翻译后修饰,扩展了展示功能相关蛋白的可能性。此外,核糖体展示还能将修饰的氨基酸,如化学标记的或是非天然氨基酸引入蛋白质的特定位置[14]。
2 PURE核糖体展示技术
核糖体展示技术虽然已出现一段时间,但是传统的展示技术存在一定的缺陷。核糖体展示中一些内在组分的存在,如无细胞蛋白合成系统中细胞提取液中的核酸酶等,会减弱mRNA-核糖体-蛋白质三聚体的稳定性,影响实验结果。Kanamori等[15]开发了一种高效可控的核糖体展示系统,即基于重组元件的蛋白合成系统PURE(protein synthesis using recombinant elements)。该系统中的mRNA-核糖体-蛋白质三聚体高度稳定,核酸酶和其他抑制因子的活性在这个系统中非常低,筛选到的mRNA可以轻易被反转录成cDNA。PURE系统已应用到抗体工程中,如利用该系统进行表位定位和抗体亲和力成熟度研究等。这些研究结果均表明PURE系统比传统核糖体展示方法更高效。
高效稳定的三聚体对核糖体展示至关重要,PURE系统不同于以往传统的基于细胞提取物的无细胞翻译系统,因为它含有明确的组分,主要包含转录、翻译和能量再生的必需因子,而其他与蛋白合成过程不相关的物质,包括氨基酸代谢酶、能量消耗因子和核酸酶、蛋白酶等的含量则很少等。这种优势使科研人员能够绕过蛋白质合成反应过程中的障碍,在一个可重复、可靠的系统中进行核糖体展示实验。此外,在细胞中形成的mRNA-核糖体-蛋白质三聚体复合物有细胞毒性,因此,原核和真核生物对于停滞的核糖体通常都有相应的补救系统,如在E. coli中,tmRNA(ssrA)和SmpB蛋白介导的反式翻译系统能在合成不完整的蛋白加上标签,使核糖体从RNA上滑落,且新合成的带标签的蛋白也会被SmpB等蛋白降解。基于大肠杆菌S30提取物的核糖体展示也含有这样的停滞核糖体补救系统,该系统的存在不利于PRM复合物的稳定,因此需要添加ssrA的反义寡核苷酸以抑制反式翻译系统的活性。但PURE系统组分中并不含有SmpB蛋白,因而其PRM复合体更加稳定。此外,这个系统的另一个优点是其中的组分可以根据实验目的进行调整,如释放因子、tRNA或者是非天然的氨基酸等。利用PURE系统无释放因子的mRNA终止密码子可以很容易地形成稳定的mRNA-核糖体-蛋白质三聚体。
3 IVC技术用于密码子抑制tRNA的筛选
筛选和扩增是蛋白质和核酸定向进化的常用工具,筛选和鉴定需要催化剂和产物在一个独立的区域中配对。体内筛选使用活细胞,而体外筛选与体内选择相比更简单、更容易操作,同时可以解决细胞毒性的问题,可从丰富、多样的文库开始进行。Griffiths和Tawfik 设计了一个体外划分技术 IVC(in vitro compartmentalization),用于体外生物催化剂等生物大分子的进化,催化剂在一个区划的油包水乳液中与催化剂产物配对[16],该技术已经成功用于如核酶和普通酶等生物催化剂的改进中[17,18]。
Ogawa等[19]利用IVC技术结合通读核糖体展示原理提出了体外抑制tRNA选择的概念。他们使用区划油包水乳液微胞中的双链DNA作为模板库(图2),模板包含转录和翻译的触发器,一个琥珀终止密码子和另一个转录触发器用于反密码子或反密码子环随机丝氨酸tRNA基因。转录生成两种类型的RNA:tRNA和可翻译的mRNA(mRNA-tRNA)。当tRNA 抑制mRNA终止密码子UAG的时候,完整的蛋白质翻译后仍附着在mRNA上,其中利用的即是通读核糖体展示技术。通过这种方式,有活性的抑制tRNA能够被选择出来并读出它们的序列。经过充分的改进后,这种方法可以成为一个很好的体外tRNA进化工具。
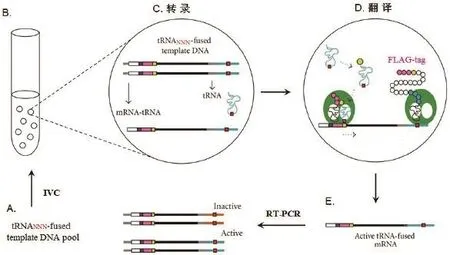
图2 IVC通读核糖体展示扩增循环
4 单链可变片段筛选
抗独特型抗体是临床前和临床开发过程中的免疫原性和药代动力学实验发展的关键试剂。Chin等[20]将噬菌体展示和核糖体展示结合起来,用以分离单克隆抗独特型抗体。抗独特型抗体通过噬菌体展示的scFv文库进行富集,利用生化表位竞争测试鉴定中和IgE 结合的抗独特型抗体。采用随机点突变的方法在核糖体展示中使噬菌体衍生的抗独特型抗体迅速亲和成熟。通过2轮筛选使抗独特型抗体的中和活性提高近20倍,并保持了对病人抗体的特异性,可用于抗药抗体和药代动力学分析中,实现了快速生产抗IgE 的抗独特型抗体。
此外,开发高亲和性、强特异性的试剂对于监测环境中的毒素和激素十分重要。Zhao等[21]利用核糖体展示,在非免疫小鼠的脾细胞的文库中筛选抗双酚A的scFv片段。研究揭示出核糖体展示可以从非免疫天然文库中分离出小分子的结合剂,而无需任何体内步骤,可高效快速的产生重组抗体。Luo等[22]构建了抗杀螟松单链可变片段的cDNA文库,通过核糖体展示筛选特异性scFv,用于快速精确检测食品样本中的杀螟松,显示出核糖体展示在杀虫剂抗体片段筛选方面的潜力。
在疾病治疗和诊断方面,核糖体展示技术也有一定的应用。Chen等[23]利用核糖体展示技术进行乙肝病毒HCV外壳E2连接肽的功能筛选。他们利用核糖体展示技术亲和筛选,筛选到的PE2D肽对HCV E2蛋白显示出高特异性和亲和性,可阻断E2结合到干细胞上,抑制乙肝病毒HCV的侵入,可作为潜在的抗病毒药物。Zhao等[24]同样利用核糖体展示技术,从人的单链可变片段scFv基因文库中筛选抗狂犬病病毒的抗体片段。其他一些课题组也相继利用核糖体展示技术对单链可变片段进行了筛选[25,26]。此外,核糖体展示技术已发展成为一个新的免疫检测策略[27,28],对于药物免疫检测发展具有重要意义。
5 酶的定向进化
核糖体展示在体外酶的进化应用中受到酶的范围和催化效率的限制。核糖体展示用于酶活的筛选只有在三聚体完整情况下可行,酶分子催化的反应产物连接到mRNA 上,否则基因型-表型的关联将会消失。最有效的进化酶的方式是利用定向选择压力作用于酶的各个方面,如底物结合、产物形成和速率加速等,表型和基因型相关的筛选结果将被保留下来。到目前为止体外方法中只有IVC方法最佳,在油包水乳液中,油分散了液滴,阻止基因、体外合成蛋白及反应产物形成交叉污染,因此能够保持表型和基因型的关联。
研究人员在酶与底物间的自由分子间相互作用的条件下进行酶催化活性的筛选[29]。突变文库中,不稳定的mRNA-核糖体-蛋白质三聚体复合物以油包水乳液的方式被区划在体外,每一个液滴平均含有不超过一个单一的复合物。之后复合体在液滴中分解,释放的酶分子酶学特性在选择性压力下自由地与底物作用,提供了一个高效选择催化活性的机会。定向进化是用于蛋白质在特定应用中进行定制化的一种手段,虽然已经取得一定的成果,但是其潜力才刚开始展现出来。未来生成定制蛋白的发展,在很大程度上依赖于改进现有的进化方法和新方法的出现。而经过改进的区划核糖体展示则可被用于酶活性的筛选中,可在单分子水平下筛选任何一种酶。
6 展望
虽然早在20年以前就开始有关于核糖体展示技术的报道,但是这项技术并没有像噬菌体展示技术一样被广泛应用,原因是在细胞提取液中准备稳定的三聚体复合物比较困难,而无细胞蛋白合成系统中含有许多抑制因子抑制了有效的核糖体展示。最近发展出的PURE系统作为无细胞蛋白合成系统,可以使目的基因通过核糖体展示技术分离得到。同时正是由于如文库多样性丰富这类优于噬菌体展示的特点,使得核糖体展示技术有更加广阔的应用前景。未来随着技术的发展,核糖体展示技术除了进行选择目的蛋白或抗体外,也可以用于生产筛选的蛋白。体外展示筛选表位候选物时经常会遇到假阳性的情况,而核糖体展示筛选到的肽可直接进行合成,通过Western blot进行分析,这样就不需过表达和纯化步骤。通过PCR扩增DNA加入到展示系统中,可以对大量的肽同时进行研究。因此,未来利用简单、高效的核糖体展示策略就可以完成低成本、高通量的表位筛选工作。
除了生物学领域的研究外,在化学生物学和材料化学领域中核糖体展示技术还将更多的被用于肽适配体的筛选,从由亲水氨基酸组成的导电聚合物文库中进行筛选,结合的适配体通过石英晶体微量天平进行测定,进而进行圆二色性解析其二级结构。为构建新型功能材料,还可以将不同种类的成分结合到一起。目前,研究人员已开始设计可结合有机和无机材料的分子,这些分子的筛选多使用体外选择技术,肽适配体则用于不同种类的非生物材料的筛选,如金属氧化物、金属、量子点和合成聚合物等[30,31]。Li等[32]使用核糖体展示技术,从随机文库中筛选导电聚合物3-己基噻吩-2,5-二基(P3HT)的肽适配体。P3HT属于半导体聚合物家族,常用于太阳能电池、晶体管和传感器中。核糖体展示可成功用于亲水肽适配体的筛选,而这些肽均显示出了特异的亲和活性,亲水氨基酸组成的肽对于疏水聚合物具有非常好的结合能力。通过核糖体展示得到的肽适配体未来也会用于生物传感器的制备中,如单壁碳于碳纳米管的筛选[33]。可以预见的是改进的核糖体展示技术将会有更加广泛的应用前景,将从蛋白质的筛选扩展到小分子筛选中。
[1]Smith GP. Filamentous fusion phage:novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985,228(4705):1315-1317.
[2]Santich BH, Liu H, Liu C, et al. Generation of TCR-like antibodies using phage display[J]. Methods Mol Biol, 2015, 1348:191-204.
[3]Shusta, EV, VanAntwerp, J, Wittrup KD. Biosynthetic polypeptide libraries[J]. Curr Opin Biotechnol, 1999, 10(2):117-122.
[4]Van Deventer JA, Yuet KP, Yoo TH, et al. Cell surface display yields evolvable, clickable antibody fragments[J]. Chembiochem, 2014,15(12):1777-1781.
[5]Soga K, Abo H, Qin SY, et al. Mammalian cell surface display as a novel method for developing engineered lectins with novel characteristics[J]. Biomolecules, 2015, 5(3):1540-1562.
[6]Hanes J, Pluckthun A. In vitro selection and evolution of functional proteins by using ribosome display[J]. Proc Natl Acad Sci, USA,1997, 94(10):4937-4942.
[7]He M, Taussig MJ. ARM complexes as efficient selection particles for in vitro display and evolution of antibody combining sites[J]. Nucleic Acids Res, 1997, 25:5132-5134.
[8]Chen L, Kutskova YA, Hong F, et al. Preferential germline usage and VH/VL pairing observed in human antibodies selected by mRNA display[J]. Protein Eng Des Sel, 2015, 28(10):427-435.
[9]Tokunaga M, Shiheido H, Hayakawa I, et al. Hereditary spastic paraplegia protein spartin is an FK506-binding protein identified by mRNA display[J]. Chem Biol, 2013, 20(7):935-942.
[10]Mattheakis LC, Bhatt RR, Dower WJ. An in vitro polysome display system for identifying ligands from very large peptide libraries[J]. Proc Natl Acad Sci USA, 1994, 91(19):9022-9026.
[11]Taussig MJ, Groves MAT, Menges M, et al. ARM complexes for in vitro display and evolution of antibody combining sites[M]// Monoclonal Antibodies:A Practical Approach. In Shepherd P,Dean C Eds. Oxford:Oxford University Press, 2000:91-109.
[12]Irving RA, Coia G, Roberts A, et al. Ribosome display and affinity maturation:from antibodies to single V-domains and steps towards cancer therapeutics[J]. J Immunol Methods, 2001, 248:31-45.
[13]He M, Taussig MJ. Ribosome display:Cell-free protein display technology[J]. Brief Funct Genomic Proteomic, 2002, 1(2):204-212.
[14]Ellman J, Mendel D, Anthony-Cahill S, et al. Biosynthetic method for introducing unnatural amino acids site-specifically into proteins[J]. Methods Enzymol, 1991, 202:301-336.
[15]Kanamori T, Fujino Y, Ueda T. PURE ribosome display and its application in antibody technology[J]. Biochimica et Biophysica Acta, 2014, 1844:1925-1932.
[16]Griffiths AD, Tawfik DS. Man-made enzymes—from design to in vitro compartmentalization[J]. Cur Opin Biotech, 2000, 11(4):338-353.
[17]Levy M, Ellington AD. Directed evolution of streptavidin variants using in vitro compartmentalization[J]. Chemi Biol, 2008, 15(9):979-989.
[18]Tay Y, Ho C, Droge P, et al. Selection of bacteriophage lambda integrases with altered recombination specificity by in vitro compartmentalization[J]. Nucleic Acids Res, 2010, 38(4):e25.
[19]Ogawa A, Hayami M, Sando S, et al. A concept for selection of codon-suppressor tRNAs based on read-through ribosome display in an in vitro compartmentalized cell-free translation system[J]. J Nucleic Acids, 2012, 2012:538129.
[20]Chin SE, Ferraro F, Groves M, et al. Isolation of high-affinity,neutralizing anti-idiotype antibodies by phage and ribosome display for application in immunogenicity and pharmacokinetic analyses[J]. J Immunol Methods, 2015, 416:49-58.
[21]Zhao L, Ning BA, Bai JL, et al. Selection of bisphenol A-singlechain antibodies from a non-immunized mouse library by ribosome display[J]. Analytical Biochemistry, 2015, 488:59-64.
[22]Luo YH, Xia YX. Selection of single-chain variable fragmentantibodies against fenitrothion by ribosome display[J]. Anal Biochem, 2012, 421:130-137.
[23] Chen F, Zhao Y, Liu M, et al. Functional selection of hepatitis C virus envelope E2-binding peptide ligands by using ribosome display[J]. Antimicrob Agents Chemother, 2010, 54(8):3355-3364.
[24]Zhao XL, Chen WQ, Yang ZH, et al. Selection and affinity maturation of human antibodies against rabies virus from a scFv gene library using ribosome display[J]. J Biotech, 2009, 144:253-258.
[25]Zhou L, Mao WP, Fen JA, et al. Selection of scFvs specific for the HepG2 cell line using ribosome display[J]. J Biosci, 2009, 34 (2):221-226.
[26]Sun YN, Ning BA, Liu M, et al. Selection of diethylstilbestrolspecific single-chain antibodies from a non-immunized mouse ribosome display library[J]. PLoS One, 2012, 7(3):e33186.
[27]Qi YH, Wu CM, Zhang SX, et al. Selection of anti-sulfadimidine specific ScFvs from a hybridoma cell by eukaryotic ribosome display[J]. PLoS One, 2009, 4(7):e6427.
[28]Cheng HW, Chen YF, Yang Y, et al. Characterization of anticitrinin specific ScFvs selected from non-immunized mouse splenocytes by eukaryotic ribosome display[J]. PLoS One, 2015,10(7):e0131482.
[29]Skirgaila R, Pudzaitis V, Paliksa S, et al. Compartmentalization of destabilized enzyme-mRNA-ribosome complexes generated by ribosome display:a novel tool for the directed evolution of enzymes[J]. Protein Eng Des Sel, 2013, 26(7):453-461.
[30]Whaley SR, English DS, Hu EL, et al. Selection of peptides with semiconductor binding specificity for directed nanocrystal assembly[J]. Nature, 2000, 405:665-668.
[31]Ejima H, Matsumiya K, Sawada T, et al. Conjugated polymer nanoparticles hybridized with the peptide aptamer[J]. Chem Commun, 2011, 47:7707-7709.
[32] Li Z, Uzawa T, Zhao H, et al. In vitro selection of peptide aptamers using a ribosome display for a conducting polymer[J]. J Biosci Bioeng, 2014, 117(4):501-503.
[33] Li Z, Uzawa T, Tanaka T, et al. In vitro selection of peptide aptamers with affinity to single-wall carbon nanotubes using a ribosome display[J]. Biotechnol Lett, 2013, 35:39-45.
(责任编辑 马鑫)
Current Progress on Ribosome Display
GUO Yuan
(Vocational and Technical College,Inner Mongolia Agricultural University,Baotou 014109)
Ribosome display is a cell-free system for the selection of proteins and peptides from large libraries. It uses the principle of coupling the translated proteins(phenotypes)and their corresponding mRNA(genotypes)to ribosome and the stable protein-ribosomemRNA complexes are formed. Then,the functional proteins and the corresponding encoding mRNAs are obtained by the simultaneous affinity separation of ligands,and they are converted and amplified as DNA for further expressions of related proteins,as well as selection of antibody and protein libraries,in vitro modification of proteins. It may display very large libraries while it is not limited by bacterial transformation. It is also suitable for screening the toxic,proteolytically sensitive,and unstable proteins,and allows the incorporation of modifying amino acids at defined loci. In this paper,research progress on ribosome display systems and their applications in the selection and evolution of proteins are reviewed.
ribosome display;antibody;selection;protein evolution
10.13560/j.cnki.biotech.bull.1985.2016.08.004
2015-10-15
郭园,女,讲师,研究方向:微生物与免疫;E-mail:guoyuan_2003@163.com

