番茄褪绿病毒RT-PCR检测技术的优化及河南分离物的鉴定
冯 佳, 孙晓辉, 高利利, 刘灵芝, 乔 宁, 刘永光, 竺晓平*
(1. 山东省蔬菜病虫生物学重点实验室, 山东农业大学植物保护学院, 泰安 271018;2. 潍坊科技学院蔬菜花卉研究所, 潍坊 262700)
番茄褪绿病毒RT-PCR检测技术的优化及河南分离物的鉴定
冯佳1,孙晓辉1,高利利1,刘灵芝1,乔宁2,刘永光2,竺晓平1*
(1. 山东省蔬菜病虫生物学重点实验室, 山东农业大学植物保护学院, 泰安271018;2. 潍坊科技学院蔬菜花卉研究所, 潍坊262700)
为获得高效、灵敏和稳定的番茄褪绿病毒(Tomatochlorosisvirus, ToCV)RT-PCR检测体系,选取文献报道的和重新设计的共9套ToCV的RT-PCR引物,经过一系列测试和比较,筛选出了灵敏度、特异性均较高的引物组合,并对反应条件、参数进行了优化,确定了最优反应条件。应用优化的ToCV RT-PCR检测体系对从河南设施蔬菜生产区采集的疑似感病番茄样品进行检测,扩增产物测序后经BLAST比对发现,河南分离物的CP和HSP70基因序列与GenBank上注册的ToCV典型分离物序列的相似性均达94%以上,通过对ToCV病毒粒子的电镜观察而进一步确定ToCV已在河南侵染设施番茄。
番茄褪绿病毒;RT-PCR;多重PCR;序列分析
番茄褪绿病毒(Tomatochlorosisvirus, ToCV)隶属长线形病毒科(Closteroviridae)毛形病毒属(Crinivirus),能通过2属4种粉虱进行传播,但不能通过摩擦接种传毒[1]。自1998年美国首次发现ToCV后,其他多个国家陆续报道了该病毒的发生[2-7];国内ToCV最早在台湾被发现[8],2012年后北京、山东、河北、天津等地区均有该病毒危害的报道[9-12]。2013年至2014年该病毒病害在山东等地大面积流行,造成番茄严重减产[13]。植株感染ToCV后首先表现为下部叶片叶脉间褪绿黄化,后逐渐向上部叶片蔓延,随着病情加重,叶脉变成深绿色,感病叶片变厚、变脆,且易折,植株长势变弱,果实变小且提前转色。
高效、灵敏的ToCV检测技术对病害的早期预警及发病规律的了解至关重要。目前国际上主要采用RT-PCR技术检测番茄褪绿病毒[14-17],血清学方法应用得较少[18]。ToCV的分子鉴定通常需要同时检测其外壳蛋白(coat protein, CP)基因和热激蛋白(heat shock protein 70 family homologue, HSP70)基因。已报道的检测ToCV的引物各不相同,针对的靶标序列也各有侧重,有的仅针对CP基因或HSP70基因的部分片段,有的针对其全长,反应条件也差别不一。鉴于此,本研究对已报道的ToCV分子检测技术体系进行了系统测试、比较和重新设计、优化、筛选,建立了快速、稳定、高效的ToCV RT-PCR检测技术,并应用该技术对河南地区设施番茄病样进行了检测,确定了ToCV在河南省的发生情况。
1 材料和方法
1.1材料
1.1.1样品采集
2014年7月至8月在河南省设施番茄产区共采集疑似感染番茄褪绿病毒的病样46份,其中中牟县10份,滑县8份,内黄县10份,北庄县5份,马村区9份,武陟县4份。以本实验室从河北、天津采集并测序鉴定的感病叶片和山东省农业科学院植物保护研究所提供的山东淄博感病叶片为阳性对照;以健康番茄植株叶片作为阴性对照。所有待测样品和阳性样品均保存于-20℃冰箱,用于RNA提取等后续试验。
1.1.2菌株、载体及试剂
大肠杆菌(Escherichiacoli)DH5α由山东省蔬菜病虫生物学重点实验室提供;克隆载体pMD18-T、RNase Inhibitor、RTase M-MLV(RNase H-)、T4 DNA连接酶购自TaKaRa公司;快捷型琼脂糖凝胶DNA回收试剂盒Ⅱ(离心柱型)购自北京百泰克生物技术有限公司;EasyTaqDNA聚合酶、dNTPs、DNA分子量标准Trans2K Plus购自北京全式金生物技术有限公司;其他生化试剂及普通化学试剂均为进口或国产分析纯。
1.2方法
1.2.1疑似病样总RNA的提取及cDNA合成
称取疑似感病植物叶片0.1 g,加液氮研磨成粉末,采用TRIzol法提取叶片总RNA,用于后续cDNA的合成。反转录步骤参照文献[15]进行。
1.2.2引物设计与合成
本研究所用引物共9套,其中5套来自已发表文献[19-22];利用Premier 5重新设计了针对HSP70和CP基因的引物4套(表1)。9套引物中针对CP基因4套、热激蛋白HSP70 基因5套,引物由铂尚生物技术(上海)有限公司合成。

表1 本研究所用引物信息
1.2.3RT-PCR检测体系的优化
采用RT-PCR对表1中的引物进行对比和筛选优化,在优化时,针对影响PCR扩增的模板浓度、引物浓度和退火温度进行检测和调整[22-25]。以山东淄博ToCV阳性样品RNA (1 μg)为模板合成cDNA。分别应用ToCV特异性引物(表1)进行PCR扩增,引物初始浓度均设为10 pmol/μL。cDNA的初始浓度为1 600 ng/μL,分别稀释至25、50、75、100、125、150、180倍,通过比较扩增效果,选择灵敏度高的引物组合,分别设置其浓度为7、5、4、3、2和1 pmol/μL进行灵敏度测试;同时对退火温度进行优化,分别在30、33.8、37.4、42.2、48.4、52、59.5、64.2和68 ℃下进行扩增。最后对结果进行比较,选定最优引物组合、最佳引物浓度和退火温度。应用优化的扩增体系对河南设施番茄产区的番茄样品进行RT-PCR检测。
1.2.4双重PCR体系设计
ToCV的鉴定需要同时检测CP和HSP70基因,采用双重PCR扩增可以提高效率。分别用To-CP-F/R和To-HSP-F/R的下游引物对山东淄博阳性对照样品叶片总RNA进行反转录,然后同时扩增CP基因和HSP70基因,试验方法参考文献进行[26-30]。扩增体系:10× PCR buffer 2.5 μL,2.5 mmol/L dNTPs 1.0 μL,To-CP-F/R和To-HSP-F/R各1.0 μL,cDNA 1.0 μL,TaqDNA 聚合酶 0.5 μL,ddH2O 补足 25 μL。扩增条件为:94℃ 3 min;94℃ 30 s,52℃ 30 s,72℃ 2 min,循环25次;72℃ 10 min。PCR扩增产物通过1.0%琼脂糖凝胶电泳检测并拍照记录结果。
1.2.5PCR产物回收及测序
参照凝胶回收试剂盒说明书对检测结果呈阳性的样品进行回收和纯化,将纯化的PCR产物连接到pMD18-T载体上并转化DH5α。阳性克隆经验证后,将菌液送上海铂尚生物科技有限公司测序。
1.2.6序列分析
利用NCBI中的BLAST程序比较病毒样品的CP和HSP70基因序列。从GenBank中下载已注册的ToCV序列,用DNAStar中的MegAlign对选定的序列进行相似性比对,再利用MEGA 4.0构建系统进化树。
1.2.7ToCV病毒粒子的提纯和电镜观察
取300 g感病番茄叶片置于-20℃冰箱过夜(尽量去除粗大叶柄),参照Fribourg等[31]提取马铃薯Y病毒的方法并略加改进提取ToCV病毒粒子,最后回溶病毒沉淀时,适当减少0.01 mol/L磷酸盐缓冲液的用量并增加回溶时间,以增加溶液中病毒粒子的浓度。
利用磷钨酸负染色技术对提纯的ToCV病毒粒子进行染色观察[32-34],选用3%的磷钨酸染色液,将样品滴加在Formvar膜的铜网上,室温晾干后进行电镜观察。
2 结果与分析
2.1PCR扩增条件优化
RT-PCR检测体系的优化结果表明,当cDNA模板稀释至8.89 ng/μL时,引物To-HSP-F/R和To-CP-F/R依然可以扩增出可见的目的条带,而其他ToCV特异性引物均未检测出目的条带(图1)。选择引物To-CP-F/R,对其浓度和退火温度进行进一步调整优化。结果表明,退火温度在30~68℃时均有条带出现(图2a),但退火温度过高或过低均会对特异性扩增造成影响,最适退火温度在37~64℃;引物浓度在1~10 pmol/μL时,均能检测到特异性条带(图2b),但引物浓度过低同样会影响特异性扩增,引物浓度在4~10 pmol/μL时,扩增效率最好。
2.2双重PCR扩增体系鉴定及在河南番茄褪绿病毒检测中的应用
以To-CP-F/R和To-HSP-F/R建立的双重PCR检测体系能同时检测到ToCV的CP和HSP基因(图3a),达到了预期的效果。
以从河南省设施番茄种植区采集的46份番茄叶片总RNA为模板,利用引物To-CP-F/R和To-HSP-F/R进行双重RT-PCR扩增。从检测结果(表2)可以看出,仅河南中牟县和滑县样品中扩增得到了970 bp和1 864 bp特异性条带(图3b),测序结果表明,河南中牟县ToCV分离物(HNZZZM1)、滑县地区ToCV分离物(HNAYHX)的CP基因全长为774个核苷酸,GenBank登录号分别为KP264984和KP264983;HSP70基因全长为1 665个核苷酸,GenBank登录号分别为KP264987和KP264986。

图1 不同ToCV引物灵敏度对比结果Fig.1 Sensitivity of different ToCV primers
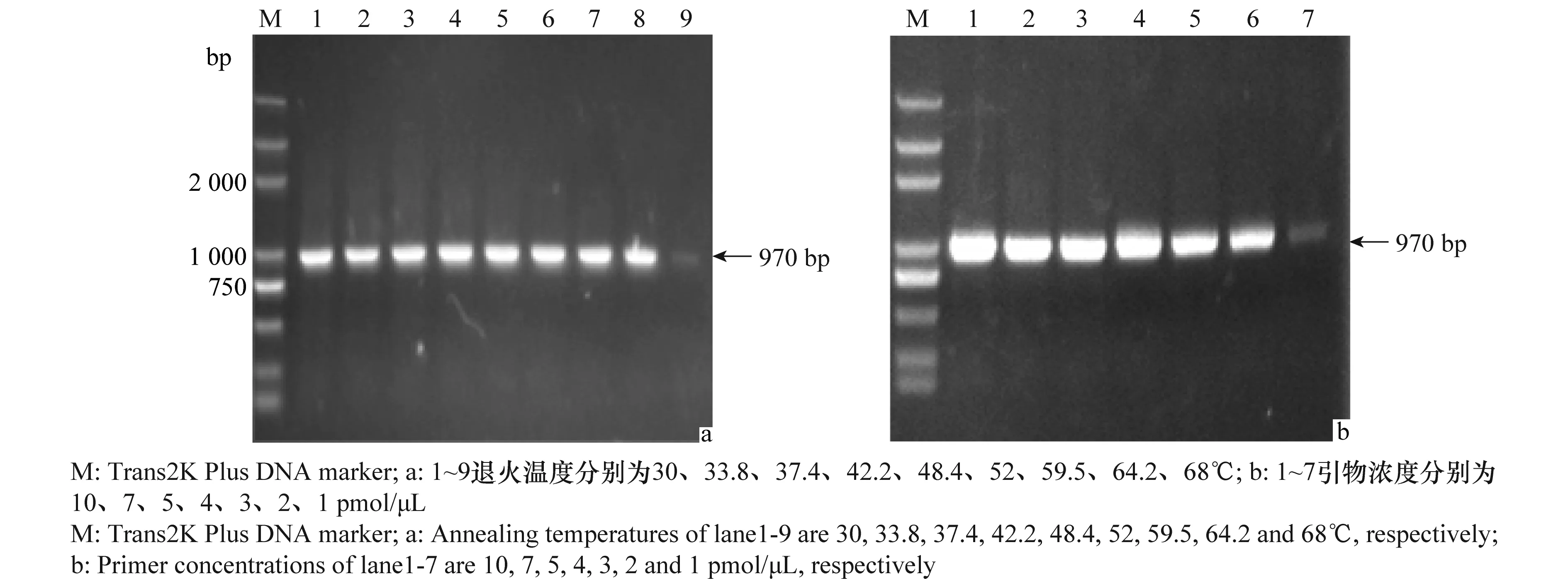
图2 To-CP-F/R的最适退火温度和浓度筛选Fig.2 Screening annealing temperature and primer concentrations of To-CP-F/R for RT-PCR

采集时间/年-月-日Collectiondate采集地点Collectionsite样品症状Symptoms阳性样品数/总样品数Positive/totalsamples2014-07-28河南中牟县ZhongmouCounty,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing2/102014-07-29河南滑县HuaCounty,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing1/82014-07-31河南内黄县NeihuangCounty,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing0/102014-08-01河南北庄县BeizhuangCounty,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing0/52014-08-02河南马村区MacunDistrict,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing0/92014-08-02河南武陟县WuzhiCounty,Henan叶片黄化,植株矮小Leafchlorosis,plantdwarfing0/4

图3 ToCV的双重PCR体系及河南感病番茄的检测Fig.3 ToCV duplex PCR system and detection results of diseased samples from Henan
2.3引物稳定性比较
选取保存于-20℃冰箱的山东淄博、河南中牟、河北衡水和天津武清ToCV番茄阳性样品对优化得到的两对ToCV特异性引物To-CP-F/R和To-HSP-F/R进行稳定性比较。结果发现To-CP-F/R相对稳定,而To-HSP-F/R在扩增不同地区ToCV阳性样品时稳定性存在差异 (图4)。
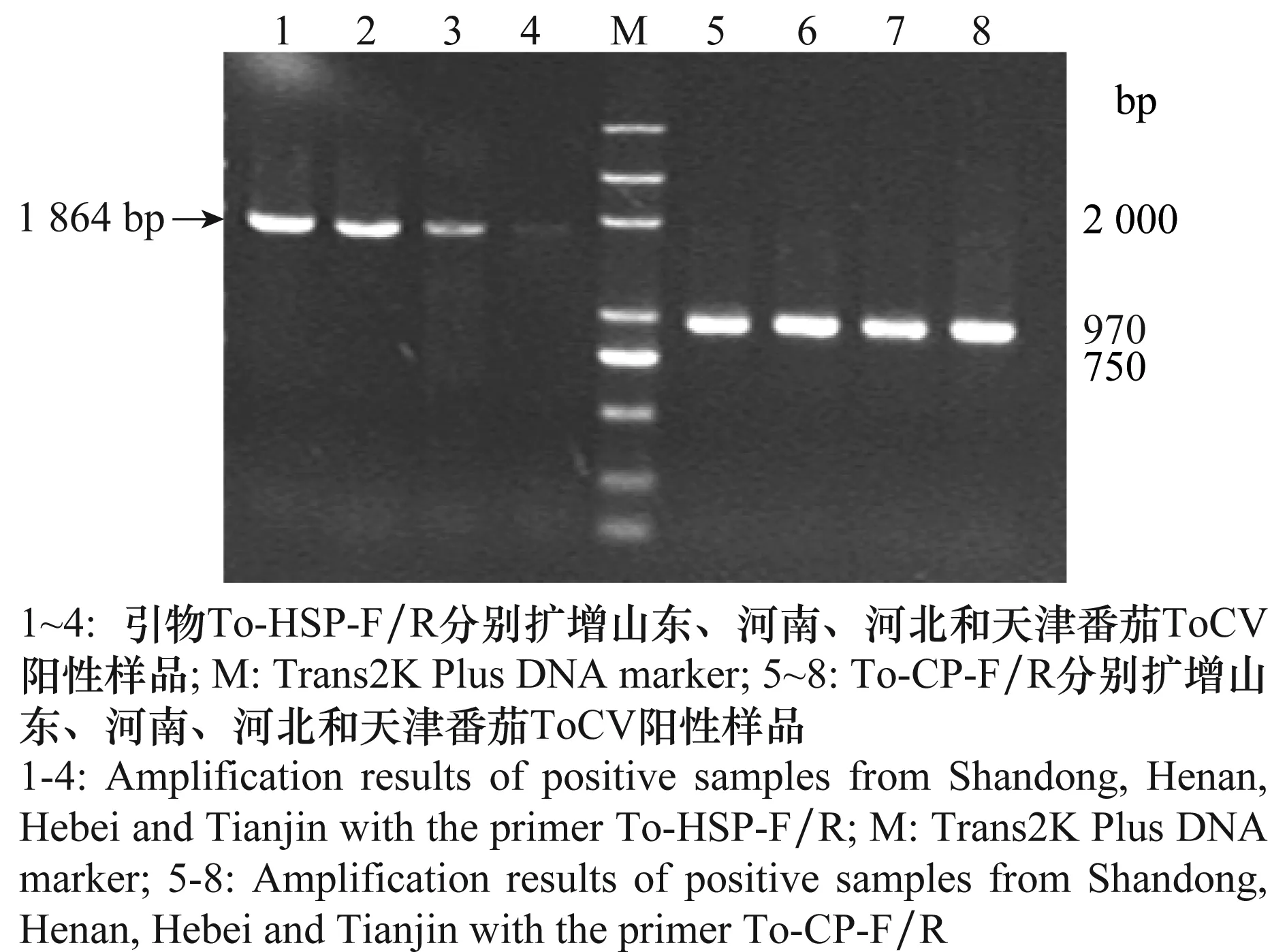
图4 引物To-HSP-F/R和To-CP-F/R对不同地区阳性样品的扩增结果Fig.4 Amplification products of positive samples from different regions using To-HSP-F/R and To-CP-F/R
选取山东淄博阳性样品测定HSP70基因分段扩增引物HSP-1-F/R、HSP-2-F/R的稳定性,结果表明HSP-2-F/R的扩增效率优于HSP-1-F/R(图5a)。进一步选取河北衡水、天津武清、河南中牟和山东淄博ToCV阳性番茄样品测定To-HSP-F/R和HSP-2-F/R的稳定性,结果表明,引物To-HSP-F/R的稳定性低于HSP-2-F/R(图5b)。
2.4序列分析
采用DNAStar中的MegAlign软件分析本试验获得的河南中牟县ToCV分离物(HNZZZM1)和滑县ToCV分离物(HNAYHX)的CP基因(GenBank登录号KP264984和KP264983)和HSP70基因(GenBank登录号KP264987和KP264986)的核苷酸序列。结果显示,中牟县和滑县ToCV分离物的CP基因同源性为99.6%,HSP70基因同源性为99.16%。由于两地分离物同源性较高,因此以河南滑县分离物(HNAYHX)为代表与GenBank中注册的各地区典型ToCV分离物的核苷酸序列进行一致性比较。结果表明,河南滑县ToCV分离物(HNAYHX)的CP和HSP70基因序列与GenBank中代表性的ToCV分离物相似性均在94%以上(表3)。为进一步分析其进化关系及分类地位,选取国内外不同地区代表性的番茄褪绿病毒分离物,分别针对CP和HSP70基因序列构建系统进化树(图6a~b),结果表明ToCV河南分离物(HNZZZM1、HNAYHX)与中国北京(ToCV-BJ)、南京(Nanjing)、山东(SDSG)和日本(Tochigi)分离物亲缘关系处于同一个大分支上,其中与日本分离物的亲缘关系最近。
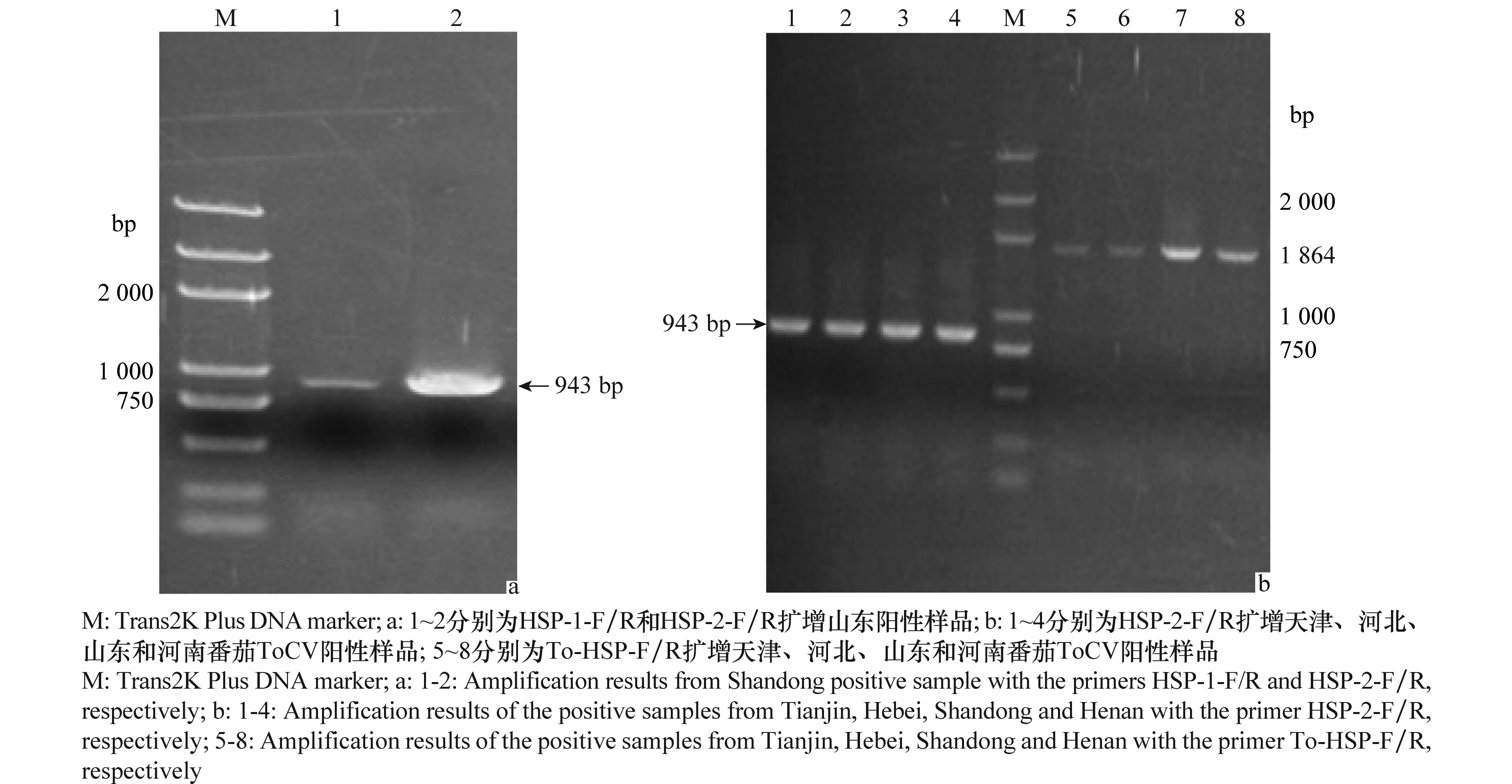
图5 不同引物扩增HSP70基因结果Fig.5 Amplification results of HSP70 gene using different primers

分离物名称Isolatename分离地点Location登录号Accessionno.一致性/% IdentityCP序列HSP70序列Tochigi日本JapanAB513443100-Tochigi日本JapanAB513442-99.7AT80/99-IC西班牙SpainKJ74025798.098.8AT80/99西班牙SpainDQ13614697.998.8SDSG中国ChinaKC70951099.799.8SDTAMZ中国ChinaKC812627-99.7SDLC中国ChinaKC812626-99.7SDLC中国ChinaKC81262299.7-BJ中国ChinaKC31137599.7-ToCV-BJ中国ChinaKC88799999.899.6Nanjing中国ChinaKJ81504599.799.5Florida美国USAAY90344899.599.5Fr法国FranceEU62535098.3-ToC-Br2巴西BrazilJQ95260199.099.4Gr-535希腊GreeceEU28474499.199.3IS以色列IsraelDQ23467398.9-HP韩国KoreaKP11453799.8-Amelia美国USAHQ87984094.6-TICV-CA4美国USAFJ54230630.964.4
2.5ToCV病毒粒子观察
用磷钨酸负染法对安阳番茄样品中提取的ToCV病毒粒子染色并进行电镜观察(图7),电镜结果可以清晰地显示长度为800 nm左右的线条形病毒粒子聚集在一起。
3 讨论与结论
ToCV的鉴定需要同时对CP和HSP70两个基因进行测定,目前文献中针对ToCV的检测方法主要以RT-PCR为主,已报道的文献中检测CP基因的引物有ToCV-F/R[21]和CP-F/R[19];检测HSP70基因的引物有ToC-5/6[20]、HSP-1-F/R和HSP-2-F/R[19],其中引物CP-F/R、HSP-1-F/R、HSP-2-F/R和ToC-5/6引用次数较多[5-9,15-17]。ToCV-F/R和CP-F/R均可扩增全长CP基因,引物ToC-5/6扩增HSP70基因的部分序列,HSP-1-F/R和HSP-2-F/R是通过扩增HSP70基因的上、下两部分序列,通过重叠区拼接得到全长HSP70基因序列,但HSP-2-F/R的扩增效率高于HSP-1-F/R。本研究重新设计合成了检测CP和HSP70全长基因的引物To-CP-F/R和To-HSP-F/R,并与已有的引物进行对比分析。结果表明,本研究设计的引物To-CP-F/R的灵敏度和稳定性均较好,而To-HSP-F/R的稳定性较已发表的分段扩增引物HSP-2-F/R[19]稍差,但该引物可直接一步扩增得到HSP70基因全序列,省去分段扩增和序列拼接等步骤,达到高效、省时的扩增效果。本研究对ToCV常规PCR检测技术进行了优化,对引物的退火温度和浓度进行了调整,同时设计并测试了双重PCR体系。优化后的RT-PCR检测体系对ToCV病毒的检出率相对较高,避免了因引物灵敏度低而出现假阴性现象,同时得到了相对稳定的RT-PCR扩增条件,减少扩增过程中出现的引物浓度和退火温度对PCR结果的影响;设计的双重PCR体系可以同时扩增ToCV的CP和HSP70基因,能有效缩短试验时间。用优化的RT-PCR检测技术对河南番茄种植区的疑似感病番茄样品进行检测,确定河南番茄已受到ToCV的侵染,同时通过提纯病毒和电镜观察,进一步确定了结果。
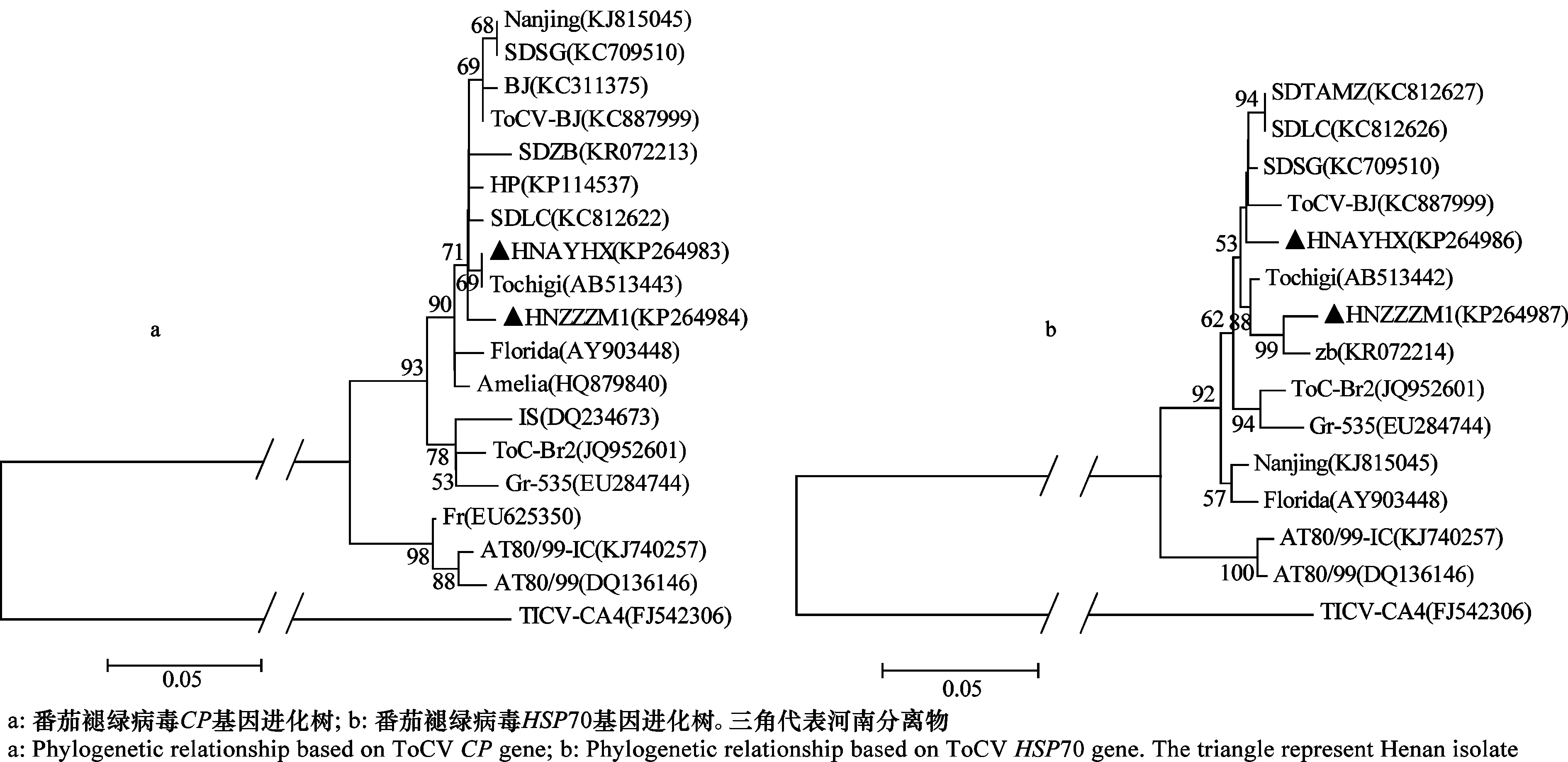
图6 番茄褪绿病毒CP和HSP70基因进化树Fig.6 Phylogenetic relationships based on sequences of the ToCV CP and HSP70 genes

图7 ToCV病毒粒子电镜图片Fig.7 ToCV virus particles electron microscopy image
番茄褪绿病毒是番茄生产上近几年新发生的病毒,该病毒可通过烟粉虱传播,发展蔓延迅速,其引起的症状与生理性的营养缺素症非常相似,给诊断带来一定的难度,造成病情延误,番茄产量下降,给种植户带来巨大的损失。开发和优化高效、准确的分子检测方法,在植株发病的早期检测并及时预警,有利于控制该病害的发生和流行。
[1]Wintermantel W M, Wisler G C. Vector specificity, host range, and genetic diversity ofTomatochlorosisvirus[J]. Plant Disease, 2006, 90 (6): 814-819.
[2]Wisler G C, Li R H, Liu H Y, et al.Tomatochlorosisvirus: A new whitefly-transmitted, phloem-limited, bipartite closterovirus of tomato [J]. Phytopathology, 1998, 88(5): 402-409.
[3]Accotto G P, Vaira A M, Vecchiati M, et al. First report ofTomatochlorosisvirusin Italy[J]. Plant Disease, 2001, 85(11): 1208.
[4]Dalmon A, Bouyer S, Cailly M, et al. First report ofTomatochlorosisvirusandTomatoinfectiouschlorosisvirusin tomato crops in France [J]. Plant Disease, 2005, 89(11): 1243.
[5]Barbosa J C, Teixeira A P M, Moreira A G, et al. First report ofTomatochlorosisvirusinfecting tomato crops in Brazil [J]. Plant Disease, 2008, 92(12): 1709.
[7]Papayiannis L C, Ioannou N, Dovas C I, et al. First report ofTomatochlorosisviruson tomato crops in Cyprus [J]. Plant Pathology, 2006, 55(4): 567.
[8]Tsai W S, Shih S L, Green S K, et al. First report of the occurrence ofTomatochlorosisvirusandTomatoinfectionschlorosisvirusin Taiwan [J]. Plant Disease, 2004, 88(3): 311.
[9]Zhao R N, Wang R, Wang N, et al. First report ofTomatochlorosisvirusin China [J]. Plant Disease, 2013, 97(8): 1123.
[10]赵黎明, 李刚, 刘永光, 等. 番茄褪绿病毒与番茄黄化曲叶病毒复合侵染的分子鉴定[J]. 中国蔬菜, 2014(12): 15-20.
[11]周涛, 杨普云, 赵汝娜, 等. 警惕番茄褪绿病毒在我国的传播和危害[J]. 植物保护, 2014, 40(5): 196-199.
[12]高利利, 孙国珍, 王勇, 等. 天津地区番茄褪绿病毒的分子检测和鉴定[J]. 华北农学报, 2015, 30(3): 211-215.
[13]刘永光, 魏家鹏, 乔宁, 等. 番茄褪绿病毒在山东暴发及其防治[J]. 中国蔬菜, 2014(5): 67-69.
[14]Zhao Liming, Li Gang, Gao Ying, et al. Molecular detection and complete genome sequences ofTomatochlorosisvirusisolates from infectious outbreaks in China [J]. Journal of Phytopathology, 2014, 162(10): 627-634.
[15]赵黎明, 李刚, 刘永杰, 等. 侵染番茄的番茄褪绿病毒山东泰安分离物的分子鉴定和序列分析[J]. 植物保护, 2014, 40(5): 34-39.
[16]赵汝娜, 王蓉, 师迎春, 等. 侵染甜椒的番茄褪绿病毒的分子鉴定[J]. 植物保护, 2014, 40(1): 128-130.
[17]李洁, 李慧, 丁天波, 等. 胶东半岛地区番茄褪绿病毒的快速检测与鉴定[J]. 山东农业科学, 2015, 47(2): 86-89.
[18]Jacquemond M, Verdin E, Dalmon A, et al. Serological and molecular detection ofTomatochlorosisvirusandTomatoinfectionschlorosisvirusin tomato [J]. Plant Pathology, 2009, 58(2): 210-220.
[19]Hirota T, Natsuaki T, Murai T, et al. Yellowing disease of tomato caused byTomatochlorosisvirusnewly recognized in Japan [J]. Journal of General Plant Pathology, 2010, 76(2): 168-171.
[20]Dovas C I, Katis N I, Avgelis A D.Multiplex detection of criniviruses associated with epidemics of a yellowing disease of tomato in Greece [J]. Plant Disease, 2002, 86(12): 1345-1349.
[21]孙国珍, 高利利, 陆文利, 等. 河北省设施番茄褪绿病毒分子检测和鉴定研究[J]. 北方园艺, 2015(9): 95-98.
[22]周丹, 谢晓娜, 陈明辉, 等. 甘蔗宿根矮化病PCR检测技术优化分析[J]. 南方农业学报, 2012, 43(5): 616-620.
[23]尚明照, 何宁, 殷冬梅, 等. 花生基因组SRAP-PCR体系的优化[J]. 中国农学通报, 2007, 23(5): 99-103.
[24]陈静, 胡晓辉, 苗华荣, 等. 花生SRAP-PCR技术体系的优化[J]. 山东农业科学, 2008(5): 24-26.
[25]贾晋, 张鲁刚. 萝卜DDRT-PCR技术体系的优化[J]. 西北农林科技大学学报(自然科学版), 2008, 36(4): 128-134.
[26]Dai Jin, Cheng Julong, Huang Tian, et al. A multiple reverse transcription PCR assay for simultaneous detection of five tobacco viruses in tobacco plants[J]. Journal of Virological Methods, 2012,183(1): 57-62.
[27]Lefeuvre P, Hoareau M,Delatt H, et al. A multiplex PCR method discriminating between the TYLCV and TYLCV-Mld clades ofTomatoyellowleafcurlvirus[J]. Journal of Virological Methods,2007, 144: 165-168.
[28]陈明洁, 刘勇, 涂知明, 等. 多重PCR法快速鉴定转基因小麦植株及后代[J]. 华中科技大学学报(自然科学版), 2004, 32(9): 105-107.
[29]陈浩东,刘方,王为, 等.棉花多重PCR技术及其对杂交棉纯度鉴定的初步研究[J].棉花学报,2011,23(1):22-27.
[30]张显勇,蔡文伟, 杨本鹏,等.甘蔗花叶病和宿根矮化病多重PCR检测方法的建立[J].中国农业科学, 2008, 41(12): 4321-4327.
[31]Fribourg C E, Nakashima J. Characterization of a new potyvirus from potato [J]. Phytopathology, 1984, 74(11): 1363-1369.
[32]韦石泉, 徐慧民. 应用负染色技术电镜观察快速检测植物病毒粒子的研究[J]. 微生物学杂志, 1985, 5(4): 7-10.
[33]洪健,陈集双, 周雪平, 等. 植物病毒的电镜诊断[J]. 电子显微学报, 1999, 18(3): 274-289.
[34]张文丽, 张玉芬, 张秀军. 应用电镜负染色技术检测嗜水气单胞菌[J]. 安徽农业科学, 2009, 37(27): 12910-12911.
(责任编辑:杨明丽)
Technique optimization of RT-PCR for detecting Tomato chlorosisvirusand molecular identification of isolates in Henan Province
Feng Jia1,Sun Xiaohui1,Gao Lili1,Liu Lingzhi1,Qiao Ning2,Liu Yongguang2,Zhu Xiaoping1
(1. Shandong Provincial Key Laboratory for Biology of Greenhouse Vegetable Diseases and Insect Pests,College of Plant Protection,Shandong Agricultural University,Tai’an271018,China; 2. Institute of Vegetables and Flowers, Weifang University of Science & Technology, Weifang262700, China)
To obtain stable, sensitive and specific RT-PCR detection system forTomatochlorosisvirus(ToCV), 9 primer pairs redesigned or selected from those published research papers were compared for their productivity, sensitivity and specificity in the RT-PCR detection system, a series of tests were conducted and the primer pairs that most sensitive and specific among them were selected and confirmed, and the RT-PCR reaction conditions were optimized, The optimized RT-PCR detection system was used to detect the ToCV-suspected tomato samples from greenhouse vegetable-growing area in Henan Province. Sequence analysis revealed that theCPandHSP70 homologue gene of Henan isolates shared 97% nucleotide sequence identity with that of ToCV isolates registered in the NCBI, indicating the tomato plants were infected by ToCV in Henan. This results were also confirmed by electron microscopy observation of the virus particles extracted from the samples.
Tomatochlorosisvirus;RT-PCR;multiplex PCR;sequence analysis
2015-09-10
2015-11-23
公益性行业(农业)科研专项(201303028);山东省科技发展计划项目(2014GNC111008);山东省自然科学基金(ZR2012CM032)
E-mail:zhuxp@sdau.edu.cn
S 436.412.11
A
10.3969/j.issn.0529-1542.2016.04.020

