内蒙古达茂旗牧区牛乳中乳酸菌的鉴定与耐药性分析
吕耀龙,李少英,包丹丹,赵春杰
(1.内蒙古农业大学职业技术学院,内蒙古包头 014109;2.内蒙古农业大学食品科学与工程学院,内蒙古呼和浩特 010018)
内蒙古达茂旗牧区牛乳中乳酸菌的鉴定与耐药性分析
吕耀龙1,李少英2,*,包丹丹1,赵春杰1
(1.内蒙古农业大学职业技术学院,内蒙古包头 014109;2.内蒙古农业大学食品科学与工程学院,内蒙古呼和浩特 010018)
本研究以内蒙古达茂旗牧区牛乳中分离的16株乳酸菌为研究对象,采用形态观察、生物学特性实验和16S rRNA鉴定,并用纸片扩散法对阿莫西林、氨苄西林和红霉素三种抗生素进行了耐药测定。结果显示,除DM8为乳酸乳球菌、DM21为约氏乳杆菌外,其余均为植物乳杆菌;供试菌株对阿莫西林的耐药率为12.5%,对氨苄西林和红霉素表现为敏感。经分析,敏感菌株不完全是安全菌株。
乳酸菌,鉴定,耐药性,分析
内蒙古达茂旗牧区牛乳中含有多种乳酸菌,这些菌种多被应用于食品业、饲料业和医药业等相关领域。乳酸菌有着较长的安全使用历史,人们普遍认为发酵产品中所用的乳酸菌都是安全(GRAS)菌株,并与人及动物肠道固有微生物群落存在着相互的益生作用,对人和动物的健康有益[1]。近年来研究表明,抗生素在医药、饲料等行业得到了广泛甚至泛滥应用,致使微生物耐药问题日趋严重,使人们在应对微生物性疾病时将面临严重挑战[2]。有研究表明,临床分离的细菌中有近50%~70%的乳酸菌对青霉素类药物表现出耐药性[3-4]。
阿莫西林、氨苄西林和红霉素三种抗菌药物,是临床治疗中感染性疾病应用较多的药物。乳酸菌对三种抗菌药物表现为较低的耐药率,但也存在一定的差异性[3-8]。在不明确菌株耐药程度的情况下盲目研究机理及防控乃至用于生产实践,将造成后续工作的不确定性、甚至影响人们的健康。本文旨在研究内蒙古达茂旗牧区牛乳中乳酸菌对阿莫西林、氨苄西林和红霉素三种抗菌药物的耐药性,为进一步研究耐药机理和选育乳酸菌提供方向,为乳酸菌的安全使用提供参考依据。
1 材料与方法
1.1材料与仪器
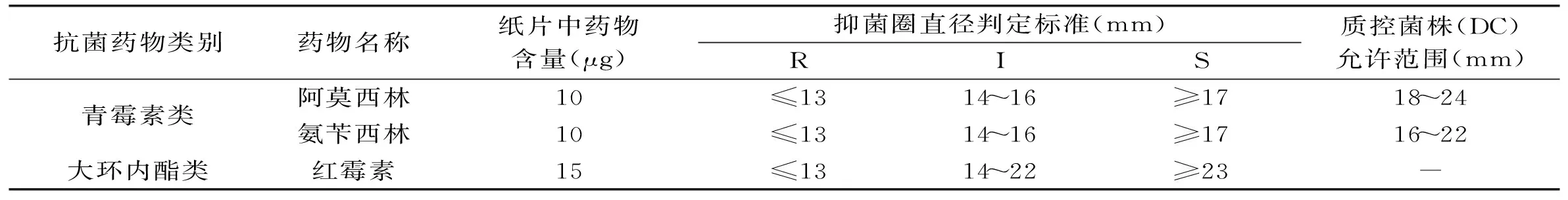
表1 纸片法使用的抗菌药物及药物敏感型标准
注:S(susceptible)为敏感;R(resistant)为耐药;I(intermediate)为中度敏感。
实验菌株来自内蒙古达茂旗牧区牛乳中分离纯化保存的菌种,共16株,分别为:DM1、DM2、DM3、DM4、DM6、DM8、DM9、DM10、DM11、DM13、DM14、DM15、DM17、DM18、DM19、DM21;质控菌株:大肠杆菌(Escherichiacoli,ATCC:25922)标号DC购于中国工业微生物菌种保藏中心;琼脂糖Sigma公司;细菌基因组DNA提取试剂盒天根生化科技(北京)有限公司;2×Easy Tap PCR Supermix、DL2000 DNA Marker、6×Loading buffer北京全式金生物技术有限公司;核酸染料北京赛百盛生物工程公司;16S rRNA序列引物:上游:5′-tgtcgtgagatgttgggttaag-3′,下游:5′-actagcgattccgacttcatgt-3′中美泰和生物技术(北京)有限公司,查阅文献[3]而定;阿莫西林(HTWS-C054,10 μg/片)、氨苄西林(C002,10 μg/片)、红霉素(C023,15 μg/片)杭州天和微生物试剂有限公司;TPY培养基、MRS培养基和脱脂乳培养基按文献[9]进行配制。
荧光分光光度计北京普析通用仪器有限责任公司;超净台哈尔滨市东联公司;离心机Eppendorf公司;PCR扩增仪BIO-RAD公司;电泳仪北京市六一仪器厂。
1.2实验方法
1.2.1菌种活化按照文献[10]中方法进行。
1.2.2供试菌液制备按照文献[3]中方法进行。
1.2.3供试菌株的形态及生物学特性细菌镜检观察、革兰氏染色、运动性实验、过氧化氢酶实验、葡萄糖产气实验、糖发酵实验等具体实验步骤参照文献[11]方法进行。
1.2.4供试菌株16S rRNA鉴定
1.2.4.1供试菌株DNA提取具体操作步骤按照DNA提取试剂盒操作步骤进行。
1.2.4.2供试菌株DNA验证0.5 μL的6×Loading buffer与5 μL提取后供试菌株的DNA混匀,点入1%琼脂糖凝胶进行电泳检测,观察条带。电泳参数:电泳液为1×TBE buffer,时间为30 min,电压为110 V。
1.2.4.3PCR扩增16S rRNA PCR扩增的引物通过查阅文献[3]而定,扩增片段长为275 bp,由中美泰和生物技术(北京)有限公司合成。
扩增体系为50 μL:模板DNA(4 μL)、上游引物(1 μL)、下游引物(1 μL)、2×Easy Tap PCR Supermix(25 μL)、ddH2O(19 μL)。PCR循环参数[5],预变性:94 ℃、3 min,变性:94 ℃、30 s,退火:60 ℃、30 s,延伸:72 ℃、1 min,共45个循环,末端延伸:72 ℃、5 min。
1.2.4.4PCR扩增产物验证将提取的DNA 5 μL点入1%琼脂糖凝胶进行电泳检测,观察条带。电泳参数:电泳液为1×TBE buffer,时间为30 min,电压为110 V。
1.2.4.5供试菌株16S rRNA同源性分析将1.2.4.3中PCR扩增产物送中美泰和生物技术(北京)有限公司进行测序。测序结果与NCBI中BLAST(http://www.ncbi.nlm.nih.gov/BLAST)和GenBank数据库中已有菌株16S rRNA序列进行比对,找出与供试菌株序列同源性最高菌种。将供试菌株序列与已知NCBI数据库中乳酸菌序列用DNASTAR软件中MegAlign进行分析,得到同源性百分比,绘制系统发育树鉴定供试菌株。
1.2.5供试菌株抑菌直径测定采用纸片扩散法测定,用无菌吸管取1 mL供试菌液,分别加入到25 mL未凝固MRS培养液中,充分摇匀后再倒入平皿中待凝固。在凝固好的平皿中均等放置2个药敏纸片、1个空白纸片。同样方法做质控菌株对照。37 ℃恒温培养24 h,测量抑菌圈直径。上述实验每个菌株做3次重复。
1.2.6供试菌株对三种抗菌药的敏感性判断供试菌株对三种抗菌药的敏感性参照美国临床和实验标准协会(CLSI)[10,12]规定标准(详见表1)进行,报告各供试菌株对三种抗菌药物是敏感、中度敏感、耐药分别用S、I和R表示[11-12]。
2 结果与分析
2.1供试菌株的生物学特性
16株供试乳酸菌的形态及部分理化特性见表2。从表2可知,各供试菌株的菌落大小不尽相同,偏小、中等居多,菌落颜色均为乳白色,菌落除DM8和DM11外,均为不透明。供试菌株均为革兰氏阳性,4株球菌,12株杆菌,包括4株短杆菌。理化检测中硝酸盐还原实验均为阴性。运动性实验,9株阳性,7株阴性;阳性菌株3株球菌,6株杆菌,阴性菌株2株球菌,其余为杆菌。葡萄糖产气、过氧化氢酶实验均为阴性,实验结果符合乳酸菌特性。本实验还选取五种代表性的糖:葡萄糖、乳糖、麦芽糖、蔗糖和甘露醇进行糖发酵实验,只有DM17葡萄糖发酵略产气,其他均不产气。糖发酵中有25%种次为阴性,其余均为阳性。致此原因:糖发酵原理、微生物含有酶系不同所致,综上,DM8为乳酸乳球菌、DM21为约氏乳杆菌,其余均为植物乳杆菌。

表2 供试菌株的生物学特性结果
注:—:表示不透明,半:表示半透明,G+:表示革兰氏阳性,-:表示阴性,+:表示阳性,+○:表示阳性产气,=:表示阴性无发酵。
2.2供试菌株16S rRNA鉴定
2.2.1DNA提取条带条带如图1所示。目的条带与预期条带一致,均大于2000 bp。
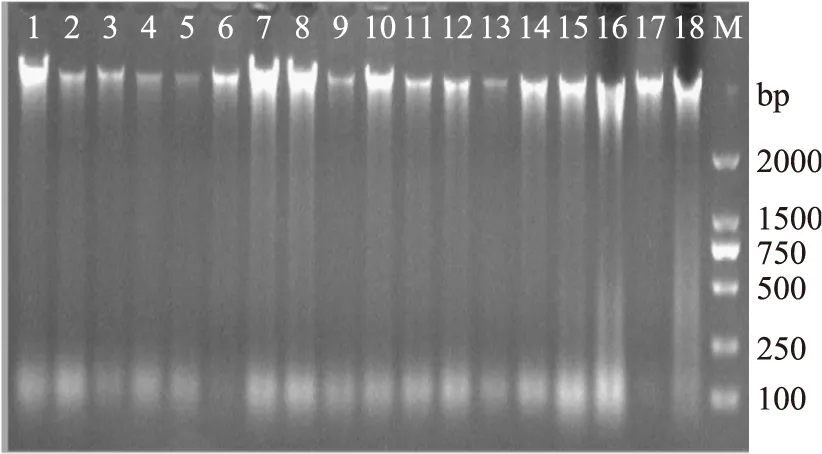
图1 供试菌株总DNA的琼脂糖凝胶电泳图Fig.1 Agarose gel electrophoresis of total DNA of the tested strains注:M:DNA Marker DL2000,1:DM1,2:DM2,3:DM3,4:DM4,5:DM6,6:DM8,7:DM9,8:DM10,9:DM11,10:DM13,11:DM14,12:DM15,13:DM17,14:DM18,15:DM19,16:DM21,17:FC,18:DC。
2.2.216S rRNA扩增条带以试剂盒提取的DNA为模板,对16株供试菌株16S rRNA基因50 μL体系扩增,再用1%的琼脂糖凝胶电泳检测PCR产物,供试菌株、标准菌株都在275 bp处出现特异性的条带,与预期吻合,DM16、DM17、DM18有重复条带现象,分析可能是因退火温度不够高或引物不够特异所致,详见图2。
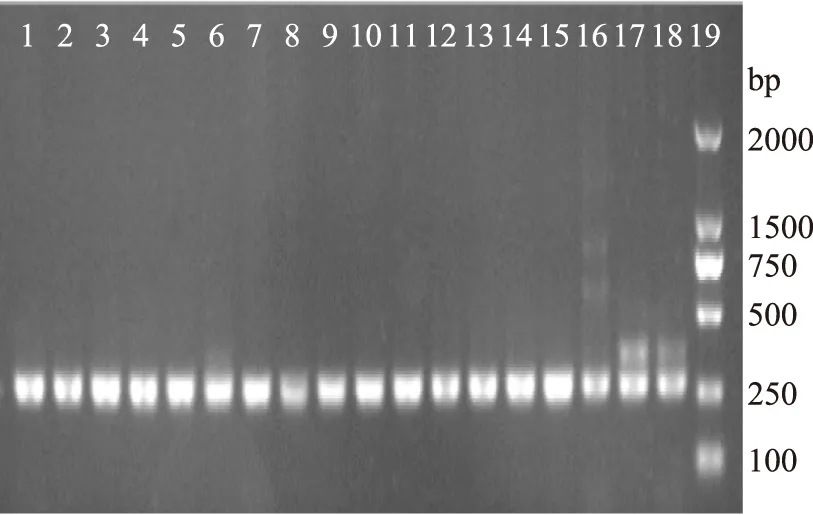
图2 供试菌株的16S rRNA基因扩增电泳图Fig.2 16S rRNA gene amplificationelectrophoresis of the tested strains注:M:DNA Marker DL2000,1:DM1,2:DM2,3:DM3,4:DM4,5:DM6,6:DM8,7:DM9,8:DM10,9:DM11,10:DM13,11:DM14,12:DM15,13:DM17,14:DM18,15:DM19,16:DM21,17:FC,18:DC。
2.2.316S rRNA同源性分析将上述PCR产物与Genbank/EMBL/DDBJ数据库中已知乳酸菌的16S rRNA基因序列同源性分析并出图(见图3)。16株供试菌株中DM2、DM3、DM4、DM6、DM9、DM10、DM11、DM13、DM14、DM15、DM17、DM18和DM19的16S rRNA序列结果与植物乳杆菌(Lactobacillusplantarum)序列的同源性为100%,菌株DM1的16S rRNA序列结果与植物乳杆菌(Lactobacillusplantarum)序列的同源性为99.5%,故判定以上14株供试菌株为植物乳杆菌(Lactobacillusplantarum);DM8的16S rRNA序列结果与乳酸乳球菌(Lactococcuslactis)序列的同源性为99.5%,故判定为乳酸乳球菌(Lactococcuslactis);DM21的16S rRNA序列结果与约氏乳杆菌(Lactobacillusjohnsonii)序列的同源性为97.9%,故判定为约氏乳杆菌(Lactobacillusjohnsonii)。
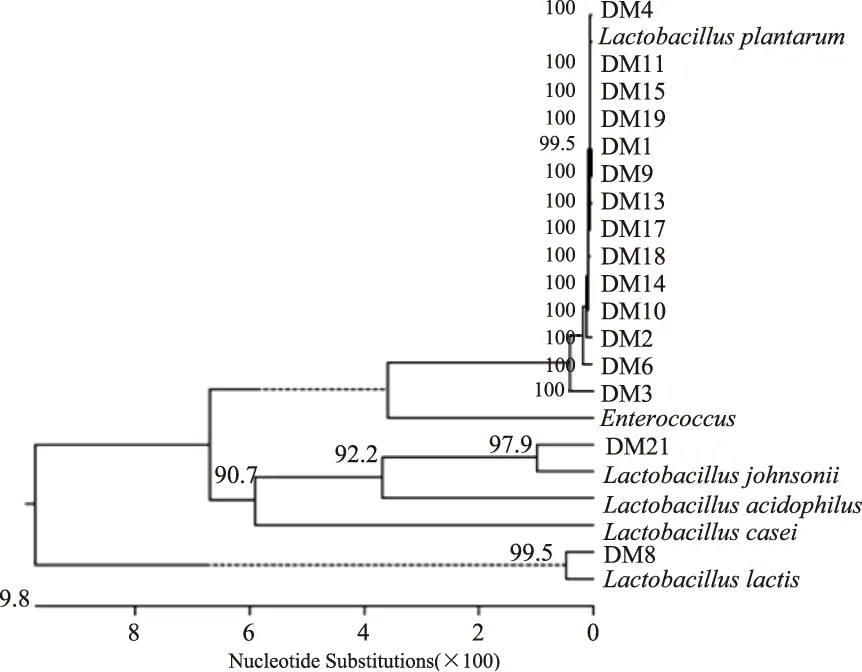
图3 供试菌株的同源性和系统发育树Fig.3 Homology and phylogenetic tree oftested strains
2.3供试菌株的耐药特性
2.3.1供试菌株耐药抑菌圈直径及药敏结果供试菌株培养24 h后,测量培养皿抑菌圈直径大小求均值,得药敏结果见表3,耐药率结果见表4。
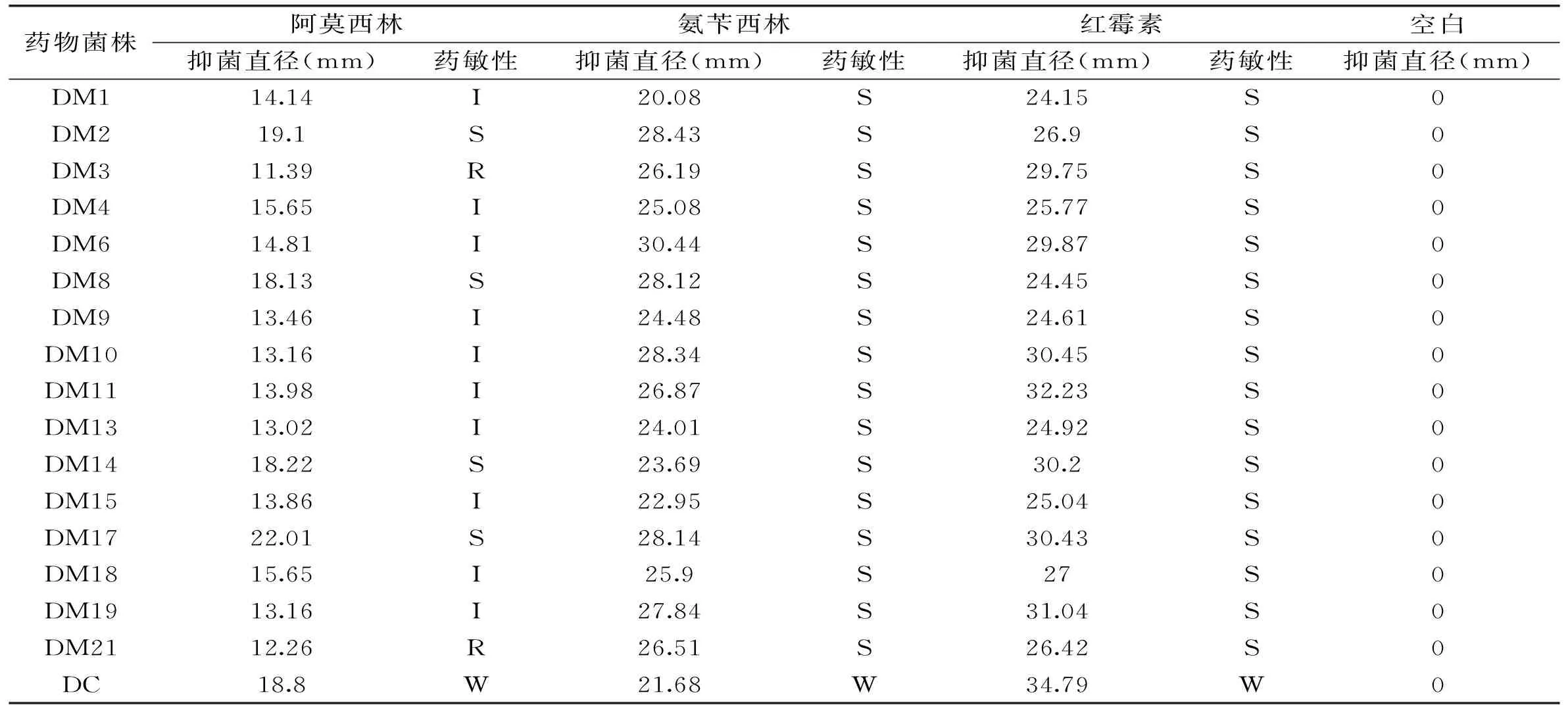
表3 三种抗菌药物对16株乳酸菌的抑菌直径及药敏结果
注:R为耐药,I为中度敏感,S为敏感,W为在CLSI规定值范围之内。
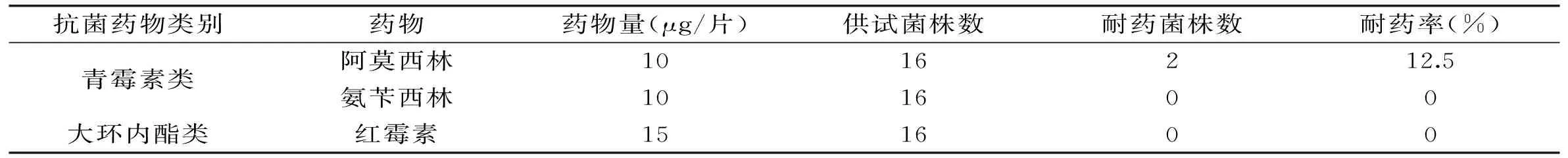
表4 16株供试菌株对三种抗菌药物的耐药率

表5 三种抗菌药物对供试菌株抑菌直径的F检验
表4结果表明,16株菌对氨苄西林、红霉素均为敏感,对阿莫西林有2株为耐药,其余为敏感。16株菌对氨苄西林、红霉素的耐药率为0,对阿莫西林的耐药率为12.5%。
质控菌株大肠杆菌对阿莫西林、氨苄西林和红霉素的耐药直径均在CLSI规定允许范围内,说明供试菌株耐药直径数值有效。
2.3.2供试菌株的耐药性分析结合上述实验结果做三种抗菌药对供试菌株抑制性差异分析,结果见表5。
从表4、表5可以看出,三种抗菌药物对乳酸菌抑制作用差异极显著(p<0.01)。
周宁[7]从全国不同地区市售的酸奶中分离的43乳酸菌中,得出对氨苄西林的耐药率为34.9%,对红霉素的耐药率为44.2%。秦宇轩[8]等人从市售酸奶中分离的100种乳酸菌,对链霉素和庆大霉素的耐药率为100%,对万古霉素的耐药率为42%,对头孢氨苄、四环素、红霉素和土霉素的耐药率为0。Hummel等[13]将40株商用乳酸菌对红霉素、四环素、氯霉素、青霉素G、氨苄西林的耐药性研究得出,耐药率低于7%。MASCO等[14]对100株乳酸菌的耐药性研究得出,对阿莫西林、万古霉素、氯霉素、红霉素的表现为敏感。张丽芳等[15]对34株乳酸菌的耐药性研究得出,对红霉素、氨苄西林、青霉素G均表现为敏感或中度敏感。陈燃等[16]对31株乳酸菌的耐药性研究得出,对红霉素的耐药率为9.7%。本实验研究结果与以上学者实验结果一致,16株乳酸菌对阿莫西林、氨苄西林、红霉素表现为较低的耐药率,分别为12.5%、0、0。但Hummel等人[12,17]在对氯霉素敏感的乳酸菌中检测出氯霉素耐药基因(cat),说明无耐药型的乳酸菌也可能携带耐药基因。秦宇轩等人[8]在对红霉素敏感菌株检测到耐药基因(ermB)。吕耀龙[18]研究内蒙古达茂旗牧区牛乳中乳酸菌对氧氟沙星的表现为较高的耐药率。目前人类大量滥用抗菌药物,乳酸菌对多数抗菌药物表现为不同程度的耐药,但也存在着对部分抗菌药物表现为敏感。对抗菌药物敏感的菌株不完全是安全菌株,也可能含有耐药基因,只是没有表达或表达不充分。FAO/WHO评价益生菌安全的重要指标之一是其耐药性评价[15,19]。
3 结论
3.116株供试菌株中,经生物学特性实验和16S rRNA鉴定,除DM8为乳酸乳球菌、DM21为约氏乳杆菌外,其余均为植物乳杆菌。
3.216株供试菌株中,2株菌对阿莫西林表现为耐药,耐药率为12.5%;对氨苄西林和红霉素均为敏感菌株,耐药率为0。供试菌株对三种抗菌药物表现为较低的耐药率。内蒙古达茂旗牧区牛乳中乳酸菌对不同的抗菌药物呈现不同的耐药性。敏感菌株也可能含有耐药基因,不完全是安全菌株。鉴于此,对食品工业而言,在选育评价乳酸菌菌株时,除了考虑其发酵能力,发酵产物的品质和安全性外,还应检测菌株的耐药性和耐药机理,这样可保证食用菌株的安全性,防范耐药菌株的扩增和耐药基因的转移。
[1]Mathur S,Singh R. Antibiotic resistance in food lactic acid bacteria-a review[J]. International Journal of Food Microbiology,2005,105(3):281-295.
[2]Bronzwaer SLAM,Cars O,Buchholz U,et al. A European study on the relationship between antimicrobial use and antimicrobial resistance[J]. Emerging Infectious Diseases,2002,8
(3):278-282.
[3]李少英.奶牛源性双歧杆菌和乳杆菌的分离鉴定及耐药性研究[D].呼和浩特:内蒙古农业大学,2008:14-28,55-73.
[4]曾振灵,林居纯. 氟喹诺酮类药物耐药性的研究进展[J].广东畜牧兽医科技,2005,30(1):11-15.
[5]霍小琰.乳酸菌拓扑异构酶gyrA基因突变与耐喹诺酮类药物的相关性[D].呼和浩特:内蒙古农业大学,2012:21-35.
[6]张燕燕.乳酸菌对氟喹诺酮类药物耐药机制的研究[D].呼和浩特:内蒙古农业大学,2008:44-50.
[7]周宁.中国不同地区市售酸奶中乳酸菌的药物敏感性分析[D].西安:西北农林科技大学,2012:25-34.
[8]秦宇轩,李晶,王秋涯,等.市售酸奶中乳酸菌的鉴定与耐药性[J].微生物学通报,2013,53(8):889-896.
[9]内村泰,岗田早苗.乳酸菌实验手册(日文版)[M].日本東京都新宿区新小川町,朝仓书店,1992:6-29.
[10]邹立扣,蒲妍君,杨莉,等.四川省猪肉源大肠杆菌和沙门氏菌的分离与耐药性分析[J].食品科学,2012,33(3):203-206.
[11]凡琴,刘书亮,李娟,等.中国市售酸奶乳酸菌的耐药性分析[J].卫生研究,2012,41(3):476-479.
[12]CLSI.抗菌药物敏感性实验执行标准(第十九版信息增刊)[S].2009,1:34-88.
[13]Hummel as,Hertelc,Holzapfel w h,et al. Antibiotic resistances of starter and probiotic strains of lactic acid bacteria[J].Am Soc Microbiol,2007,73:730-739.
[14]Mascoll,Huysg,Swiugsj,et al. Antimicrobial susceptibility of Bifidobacterium strains from humans,animals and probiotic products[J].J Antimicrob Chemother,2006,58:85-94.
[15]张丽芳,田洪涛,张玉兰,等.健康人体及保健品中乳酸菌和双歧杆菌的抗药性分析[J].中国食品学报,2009,9(6):144-151.
[16]Food and Agriculture Organization/World Health Organization. Guidelines for the evaluation of probiotics in food:report of a Joint FAO/WHO Working Group[R].Cordoba:FAO/WHO,2002.
[17]潘露,胡晓清,王小元.食品中乳酸菌危害因子的研究进展[J].食品工业科技,2011,32(6):447-451.
[18]吕耀龙,李少英,马春艳,等.内蒙古达茂旗牧区牛乳中乳酸菌对氧氟沙星的耐药性分析[J].内蒙古农业大学学报,2013,7(4):86-89.
[19]韩俊华,李珊珊,裴家伟,等.益生乳杆菌质粒抗生素抗性基因与其耐药性的相关性探讨[J].食品工业科技,2013,34(12):181-191.
Identification and drug resistance analysis on the lactic acid bacteria of cow milk in the pastoral areas of Damao Banner in Inner Mongolia
LV Yao-long1,LI Shao-ying2,*,BAO Dan-dan1,ZHAO Chun-jie1
(1.Vocational and Technical College of Inner Mongolia Agricultural University,Baotou 014109;2.Food Science and Engineering College of Inner Mongolia Agricultural University,Hohhot 010018)
The sixteen 16 strains of lactic acid bacteria were isolated from the cow milk in the Damao pasture of Inner Mongolia,and were used as research subjects in the study. The method of morphological observation,biological characteristics test and 16S rRNA identification were adopted in the experiment. The disk diffusion method was used to determine drug resistance of three antibiotics of amoxicillin,ampicillin and erythromycin. Results showed that DM8 wasLactococcuslactis,and DM21 wasLactobacillus,and that except DM8 and DM21,the rest were allLactobacillusplantarum. The study also showed that the tested strains resistant to amoxicillin was 12.5%,the tested strains were sensitive to ampicillin and erythromycin,and the sensitive strain was not entirely safety strain through analysis.
lactic acid bacteria;identification;drug resistance;analysis
2015-10-22
吕耀龙(1982-),男,硕士,讲师,主要从事食品微生物方面的研究,E-mail:lylong1982@163.com。
李少英(1961-),女,博士,教授,研究方向:有益微生物方面的研究,E-mail:nmglshy@126.com。
内蒙古高等学校科学研究项目(NJ09059);国家自然科学基金项目(31060014)。
TS201.3
A
1002-0306(2016)12-0178-05
10.13386/j.issn1002-0306.2016.12.026

