赣州市2014年新型甲型H1N1流感病毒NA基因特征及耐药性分析
熊衍峰,李如,李健雄,施勇,胡晓军,龙彩云
(1、赣州市疾病预防控制中心,江西 赣州341000;2、江西省疾病预防控制中心,江西 南昌330029)
赣州市2014年新型甲型H1N1流感病毒NA基因特征及耐药性分析
熊衍峰1,李如1,李健雄2,施勇2,胡晓军1,龙彩云1
(1、赣州市疾病预防控制中心,江西 赣州341000;2、江西省疾病预防控制中心,江西 南昌330029)
目的 分析江西省赣州市2014年新型甲型H1N1流感病毒NA基因的特点,掌握其耐药情况,为临床治疗和疾病控制提供参考依据。方法 随机选择17株新型甲型H1N1流感病毒,经核酸提取和one-step RT-PCR扩增NA基因片段,双向序列测定,采用DNAStar5.0和Mage4.0序列分析软件分析NA基因特征以及耐药性位点。结果 17株毒株的NA基因片段与代表株A/California/07/2009(H1N1)的序列核苷酸序列进行比对,核苷酸序列同源性高达98.4%以上,氨基酸的同源性也高达97.0%以上。17株毒株的NA活性中心位点氨基酸及周围的辅助位点氨基酸均未发生氨基酸替换。结论 17株毒株的NA基因片段保持高度的同源性并均对流感病毒神经氨酸酶抑制剂药物敏感,但仍应加强对流感病毒的耐药性监测,为制定新型甲型H1N1流感的防制措施提供技术支持。
新型甲型H1N1流感病毒;NA;耐药性
始于2009年初,并在全球引起爆发的新型甲型H1N1流感为急性呼吸道传染病,其病原体是一种新型的甲型H1N1流感病毒[1]。该病毒与以往的季节性流感病毒不同,包含有猪流感、禽流感和人流感三种流感病毒的核糖核酸基因片段,8个基因片段分别来自不同分支的流感病毒,其中HA、NP 和NS基因为古典型北美系猪流感病毒;NA和M基因来自欧亚系猪流感病毒;PB2和PA来自禽H1N1病毒;而PB1基因来自季节性H3N2病毒[2,3]。由于新型甲型H1N1流感病毒传染性强,人群对这个新型病毒缺乏天然免疫力,易导致疾病在短期内迅速播散至大规模流行,对人们的健康构成了威胁。而当前,常用于治疗和预防该病毒的有效药物就是NA抑制剂,因此,密切监测流行株及对NA抑制剂的耐药性变化情况非常重要。本研究分析了赣州市2014年新型甲型H1N1流感病毒的NA基因特征以及特异性耐药性位点,结果报告如下。
1 材料与方法
1.1标本来源从赣州市流感国家级哨点医院(南昌大学附属医院赣州市人医院)采集发病≤3d的流感样病例(体温≥38℃,伴咳嗽或咽痛之一者)的鼻咽拭子标本[4]和新型甲型H1N1流感重症病例鼻咽拭子标本。
1.2病毒分离采用狗肾传代细胞(MDCK)进行病毒分离,根据细胞的病变用血细胞凝集实验(HA)和血细胞凝集抑制试验(HI)进行流感病毒型别鉴定 (MDCK和标准血清均由国家流感中心提供)。所获得的毒株均经国家流感中心复核鉴定。
1.3病毒选择选择17株来自不同时间的流感样病例和新型甲型H1N1流感重症病例鼻咽拭子标本中分离到的新型甲型H1N1流感病毒,进行基因扩增和测序。
1.4病毒RNA提取采用德国Qiagen公司的QIAamp Viral RNA Mini Kit试剂对含流感病毒的MDCK细胞培养液进行病毒RNA的提取。
1.5引物NA基因序列参考WHO[5],由上海生工生物工程技术服务有限公司合成。
1.6RT-PCR扩增NA基因[6]采用QIAGEN One Step RT-PCR Kit试剂盒进行RT-PCR反应。RT反应条件:60℃ 1min,42℃ 10min,50℃ 30mim,95℃ 15min。PCR循环如下:94℃ 40s,50℃ 40s,72℃1min 30s,35次循环,72℃10min。
1.7核苷酸序列测定PCR产物送上海生工生物工程技术服务有限公司进行双向测定。
1.8序列分析核苷酸和氨基酸同源性以及遗传进化分析采用 DNAStar5.0、Mage4.0序列分析软件,神经氨酸酶抑制剂药物敏感参比株A/California/07/2009(H1N1)的NA基因序列作为参比分析,序列收录号分别为FJ984386。
2 结果
2.12014年赣州市流感病毒分离结果2014年共对1079份流感样病人的鼻咽拭子标本开展了病毒分离,共分离到流感病毒133株(见表1),其中新型甲型H1N1流感病毒39株,季节性A(H3N2)亚型流感病毒64株和B型流感病毒30株。新型甲型H1N1流感病毒的分离主要集中在第一季度,共占全年总毒株的29.32%(39/133)。(详见表1、图1)。
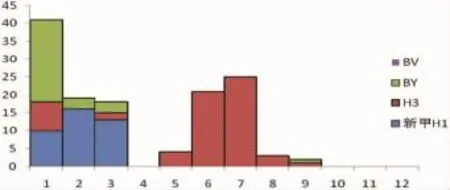
图1 赣州市2014年流感监测标本病毒分离鉴定月分布图

表1 赣州市流感网络实验室监测标本病毒分离鉴定情况
2.22014年赣州市新型甲型H1N1流感病毒NA基因核苷酸同源性及遗传进化分析对PCR获得的NA基因经核苷酸测序、序列拼接后,获得每株病毒NA基因片段序列1248bp。经DNAStar5.0、Mage4.0软件分析,与新型甲型H1N1病毒代表株A/California/07/2009(H1N1)的序列核苷酸序列进行比对,核苷酸序列同源性在98.4%~98.9%之间、氨基酸的同源性在97.0%~97.9%之间;17株病毒之间的NA基因核苷酸序列的同源性高达99.1%~100%;氨基酸序列的同源性也达到98.4%~100%。在NA基因氨基酸序列的种系进化树 (见图2)上可以看出A/JiangxiZhanggong/SWL145/2014(H1)和A/JiangxiZhanggong/SWL1116/2014(H1)与其他毒株不在同一分支上。
2.3NA基因耐药性位点分析对NA蛋白酶催化活性位点 (R-118、D-151、R-152、R-224、E-276,R-292位、R-371和Y-406)氨基酸和辅助位点(E-119、R-156、W-178、S-179、D-198、I-222、E-227、H-274、E-277和N-294位)氨基酸进行了分析,结果表明,17株毒株均未发现有上述氨基酸位点的突变。
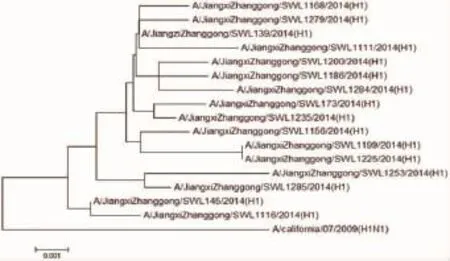
图2 NA基因氨基酸进化树
3 讨论
神经氨酸酶是分布于流感病毒被膜上的一种糖蛋白,它具有抗原性,可以催化唾液酸水解,协助成熟流感病毒脱离宿主细胞感染新的细胞,在流感病毒复制和传播中发挥重要作用。如能抑制神经氨酸酶的活性就可以阻止病毒颗粒的释放,切断病毒的扩散链,因而神经氨酸酶也是一个很重要的治疗流感的药物靶点[7]。流感病毒神经氨酸酶活性中心位点氨基酸及周围的辅助位点氨基酸高度保守,R-118、D-151、R-152、R-224、E-276,R-292、R-371和Y-406是直接与底物结合的活性中心位点,E-119、R-156、W-178、S-179、D-198、I-222、E-227、H-274、E-277、N-294和E-425作为辅助位点具有维持酶活性中心空间构象的作用。如NA发生点突变,特别是关键位点突变,会导致NA与NAI结合减少,NAI对病毒的抑制作用减弱,这是NAI耐药的最主要原因[8,9]。不同NAI对流感病毒的耐药位点可能不同,如奥司他韦耐药株出现H274Y、R292K位的替换,扎那米韦耐药株出现E119V、R292K的替换,帕拉米韦出现R292K位的替换,A-315675出现E119V的替换。而从病人标本中分离的奥司他韦耐药株出现的是E119V(A/ N2),D198N(B/NA),H274Y(A/N1),和R292K(A/N2)氨基酸残基的替换,扎那米韦耐药株出现的是R152K(B/NA)氨基酸残基的替换[10],说明R152、R292、E119、D198和H274氨基酸是突变的重点[6]。本次研究对赣州市2014年新型甲型H1N1流感病毒的NA进行了序列比对和同源性分析。17株新型甲型H1N1流感病毒的NA基因片段与代表株A/California/07/2009(H1N1)的序列核苷酸序列进行比对,核苷酸序列同源性高达98.4%以上,氨基酸的同源性也高达97.0%以上。表明本市流行的新甲型H1N1流感病毒与国内代表株的有较近的亲缘关系,17株毒株的NA片段仍然保持高度的同源性。突变重要位点分析可知,17株毒株的NA蛋白催化活性位点和辅助位点均未发生氨基酸替换。表明通过基因型分析,17株毒株的NA基因片段保持高度的同源性并均对流感病毒神经氨酸酶抑制剂药物敏感,与无锡地区新甲型H1N1流感病毒基因变异分析结果一致[11],仍应加强对流感病毒的耐药性监测。
目前,除了疫苗接种,抗病毒药物是预防和治疗流感的有效手段。现临床上用于抗流感病毒的药物主要有两类,一类是烷胺类药物,即M2离子通道抑制剂,包括金刚烷胺和金刚乙胺,该类药物仅作用于甲型流感病毒,对乙型流感病毒无效,但现在大部分流感病毒已对烷胺类药物耐药[12,13],它已不再是世界卫生组织(WHO)推荐的预防和治疗的首选药物;另一类药物是NA抑制剂,包括扎那米韦(zanamivir)、奥司他韦(oseltamivir)、帕拉米韦(peramivir)和拉尼那米韦(laninamivir)等,该类药物对于甲、乙型流感病毒均有效,因此成为WHO推荐预防和治疗流感的首选药物。然而随着NA抑制剂在全球的广泛使用,病毒发生突变导致抗药性出现的现象已经存在。虽然目前全球报道新型甲型H1N1流感病毒的NA抑制剂耐药株较为少见,2011年全球报道新型甲型H1N1流感病毒奥司他韦耐药株出现比例低于1%。但随着抗病毒药物的广泛应用和病毒的自身变异,出现NA抑制剂耐药株的几率也在增加[14]。当前新型甲型H1N1流感疫情仍然严峻,依然存在新型甲型H1N1流感基因组与高致病性H5N1禽流感基因组整合变异成更毒力更强的可能,耐药性监测不容忽视。研究奥司他韦等药物在预防和治疗季节性流感、新型甲型H1N1流感中发生的耐药性现象,以明确流感病毒对奥斯他韦等药物产生耐药的机制,对于WHO和各国卫生机构如何合理的使用抗流感病毒药物来对抗新型甲型H1N1流感疫情具有指导意义[15,16]。因此,对新型甲型H1N1流感病毒的NA基因NAI耐药性位点监测应长期、持续地进行。
[1]Centers for Disease Control and Prevention(CDC).Outbreak of swineorigin influenza A(H1N1)virus infection-Mexico,March-April 2009 [J].MMWR Morb Mortal Wkly Rep,2009,58(17):467-470.
[2]夏文英,黄吉城,伊怀文.甲型H1N1流感病原学及其治疗药物和疫苗研究进展[J].中国国境卫生检疫杂志,2009,32(4):236-242.
[3]赵红玲,章文,黄涛.甲型H1N1流感病毒基因特征及耐药性研究[J].中国病原生物学杂志,2014,9(9):811-815.
[4]中国疾病预防控制中心.全国流感监测方案(2010年版)[M].2010.
[5]WHO.WHO information for molecular diagnosis of influenza virus in humans-update.2011.
[6]熊英,龚甜,周珺,等.江西省2009年~2011年新型甲型H1N1流感病毒耐药性分析[J].现代预防医学.2014,41(9):1681-1688.
[7]许沙沙,常彦敏,徐霖,等.2010年广州市甲型H1N1流感病毒分离株NA基因变异分析[J].检验医学,2014,29(1):42-49.
[8]黄兰,周剑芳,韦红,等.流感病毒神经氨酸酶抑制剂药物耐药现状及机制研究进展[J].病毒学报,2012,28(5):572-576.
[9]李健雄,熊英,施勇,等.江西省2009年至2012年甲型H3N2流感病毒耐药性分析[J].实验与检验医学.2014,32(6):667-669.
[10]Hay AJ,Zambon MC,Wolstenholme AJ,et al.Molecular basis of resistance of influenza A viruses to amantadine[J].J Ant imicrob Chemother,1986,18(Suppl B):S19-S29.
[11]马广源,尤凤兴,凌霞,等.2009-2013年无锡地区新甲型H1N1流感病毒基因变异分析 [J].中国病原生物学杂志,2013,8(12):1109-1112.
[12]Bright RA,Shay DK,Shu B,et al.Adamantane resistance among influenza A viruses isolated early during the 2005-2006 influenza seas on in the United States[J].JAMA,2006,295(8):891-894.
[13]蓝雨,李梓,董丽波,等.中国H3N2亚型流感病毒烷胺类药物耐药性研究[J].中华实验和临床病毒学杂志,2006,20(20):21-23.
[14]马江涛,詹军,鱼小红,等.宁夏2011-2012年甲型H1N1流感病毒NAI耐药性研究[J].宁夏医学杂志.2015,37(3):235-237.
[15]王晓中.奥斯他韦等抗流感病毒药物的应用现状和其耐药机制研究进展[J].口岸卫生控制,2010,15(1):51-55.
[16]谢剑锋,沈晓娜,王美爱,等.福建省2009年甲型H1N1流感病毒NA基因特征及对达菲的耐药性 [J].中国人兽共患病学报,2010,26(10):904-911.
Analyzing the characteristics of NA genes of pandemic influenza H1N1 virus in Ganzhou city in 2014
XIONG Yanfeng1,LI Ru1,LI Jianxiong2,SHI Yong2,HU Xiaojun1,LONG Caiyun1.1.Ganzhou Center for Disease Control and Prevention,Ganzhou Jiangxi 341000,China;2.Jiangxi Provincial Center for Disease Control and Prevention,Nanchang 330029,China.
ObjectiveTo analyze the characteristics of NA genes of pandemic influenza H1N1 virus in Ganzhou,Jiangxi Provience in 2014.Methods 17 strains of pandemic H1N1 influenza virus were randomly selected for detection and virus RNA were extracted.Fragments of NA genes were amplified by one-step RT-PCR and then were sequenced.The data obtained was analyzed with the software DNAStar5.0 and Mage4.0.Results Compared with the sequences of A/California/07/2009(H1N1),the homology of the nucleotide sequences reached more than 98.4%,and the homology of the amino acid was more than 97%.All strains of pandemic H1N1 influenza virus had no mutation in catalytic residues and framework residues of NA gene.Conclusions 17 strains virus were all sensitive to neuraminidase inhibitors,however,continuous resistance surveillance is necessary for control and prevention of influenza.
Pandemic influenza H1N1 virus;NA gene;Resistance
R373.1+3,Q939.92
A
1674-1129(2016)04-0458-03
10.3969/j.issn.1674-1129.2016.04.015
熊衍峰,女,1975年1月出生,主管技师,主要从事病原微生物检测工作
李如,女,1972年1月出生,副主任技师,主要从事传染病防制工作
(2016-04-25;
2016-07-29)
——赣州市审计局这样让困难群众感受温暖

