分娩方式影响婴儿皮肤真菌群落组成
应 时,谭 渊,全哲学
(1. 复旦大学 生命科学学院 微生物学与微生物工程系,上海200438; 2. 强生(中国)有限公司 研发部,上海 200245)
分娩方式影响婴儿皮肤真菌群落组成
应时1,谭渊2,全哲学1
(1. 复旦大学 生命科学学院 微生物学与微生物工程系,上海200438; 2. 强生(中国)有限公司 研发部,上海 200245)
婴儿出生之前生活在无菌的子宫里,出生以后婴儿皮肤第一次暴露在有大量微生物的环境中,形成了初始的皮肤微生物群落.因此分娩方式的选择,决定了婴儿最初接触的微生物源是母亲的阴道还是周围的环境.本研究对6对出生2~4个月的婴儿和他们母亲的皮肤表面6个部位的样品进行3次重复采样(共计189个达标样品)并通过高通量测序来探究不同的分娩方式对婴儿皮肤表面真菌群落组成的影响.研究发现不同分娩方式会造成2~4个月婴儿皮肤真菌群落的显著差异.孕期女性阴道里常见的念珠菌属(Candida)在顺产婴儿皮肤表面的丰度显著高于剖腹产婴儿.而母亲皮肤上最主要的马拉色菌属(Malassezia)在剖腹产婴儿皮肤表面的丰度比顺产婴儿更高.同时还发现剖腹产的母亲与自己孩子的皮肤真菌群落更相似,而顺产的母婴却没有类似的特征.
分娩方式; 皮肤真菌; 高通量测序; 群落组成
一般认为健康人子宫是无菌状态,所以出生前的婴儿皮肤表面也是无菌状态.但出生时婴儿就会立即接触到大量的微生物,形成最初的皮肤微生物群落.对于通过顺产出生的婴儿来讲,在生产时经过母亲阴道时将可能获得很多类型的初始微生物.这些初始微生物只有很少一部分会在皮肤表面稳定生存,并在以后成为成年时不同部位特异的微生物群落[1-3].但现代人越来越多地选择剖腹产分娩方式,这就意味着许多婴儿在出生时不能获得阴道微生物.分娩方式是对初生儿肠道微生物的形成影响最大的因素[4-7].有研究发现不同分娩方式会影响到初生婴儿的皮肤和口腔微生物,顺产婴儿的微生物群落往往与母亲的阴道微生物群落有一定的相似性[8].这些都说明分娩方式对初生儿的微生物群落形成的重要性,但这些研究都主要是针对细菌组成的考察,而分娩方式对初生儿皮肤真菌群落影响的系统性研究至今还没有.
皮肤作为人体最大的器官,是部位差异很大的微生物栖息地,其上除了细菌还存在大量的真菌[9].皮肤表面的真菌与人体健康或疾病密切相关,常见的银屑病、痤疮和脂溢性皮炎都可能与皮肤表面的常驻微生物马拉色菌(Malassezia)相关[10-12].婴儿在刚出生的第一年,其皮肤表面的微生物多样性会随着时间不断增加[13].所以研究分娩方式对婴儿皮肤表面真菌组成的影响就是从源头探究人体微生物群落的发展,这既有助于对成年人更复杂稳定的真菌群落演变的研究,也有助于对不同生产方式的婴儿从刚出生时开始就能有针对性地进行的皮肤真菌疾病的预防.
基于纯培养的分析微生物群落的传统方法只能针对特定的几种微生物,即使是基于克隆文库的不依赖培养的微生物群落结构分析也因测序数量的限制而无法准确体现整体微生物群落结构,但第二代高通量测序技术的发展可以解决这些问题[14].本研究对上海市区6位母亲和她们刚出生2~4个月的婴儿皮肤表面6个部位的3次采样样品进行焦磷酸测序,以探究不同分娩方式对婴儿皮肤表面真菌群落组成的影响.
1 材料和方法
1.1材料
1.1.1主要试剂和仪器
聚酯纤维拭子,MOBiO PowerSoilTMDNA Isolation Kit,MOBIO UltraCleanTMPCR Clean-up DNA Purification Kit,Axygen AxyPrep DNA胶回收试剂盒.
MOBIO VOTEX-Genie 2振荡仪,TaKaRa PCR仪,TBS-380 微型荧光计,Roche 454 GS-FLX焦磷酸测序仪.
1.1.2采样对象
本研究在实施并正式采样前已通过复旦大学伦理委员会的审核.所有参与采样的受试者或其监护人都了解本课题的研究目的、采样流程以及可能出现的不适症状,并签署了知情同意书.所有受试者的数据信息都被严格保密并不用作本研究之外的用途.
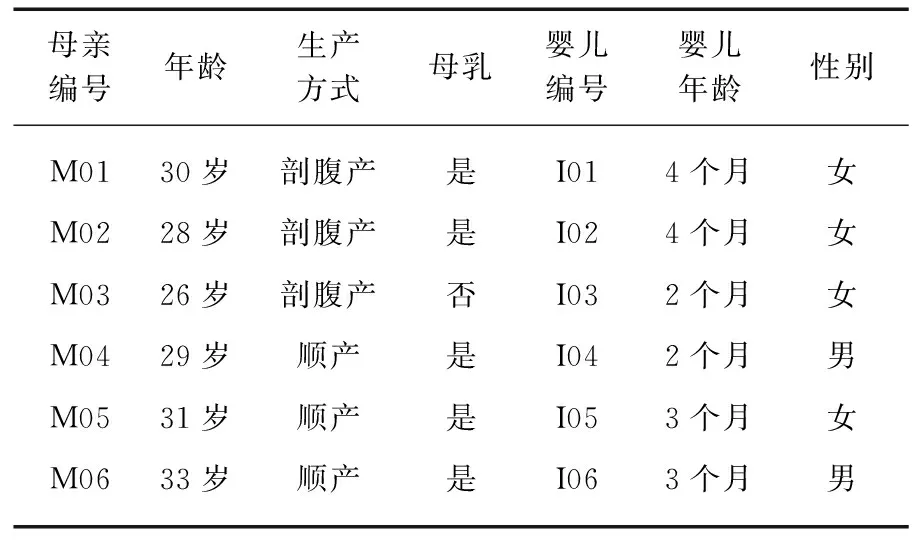
表1 母亲和婴儿的基本信息
本研究招募了6对母婴共12位受试者参与采样,母亲的年龄在26~33岁之间,婴儿年龄在2~4个月之间(表1).对所有受试者都进行了全套的皮肤科检查,通过问卷调查得到了他们的治疗和药物使用历史,并排除了在采样前6个月里患过皮肤病或使用过抗生素的受试者.通过筛选标准的受试者,都被要求在采样前12个小时内不对采样部位进行水洗(其中手上的部位只要求2个小时不水洗).本次研究采样了6个部位,依次为手背、指缝(采样位置在中指与无名指之间)、前臂、肘窝、额头和后背.对于对称的部位,随机选择一侧部位进行了采样.对每位受试者隔天进行3次重复采样.
1.2方法
1.2.1采样和DNA提取
采样时间集中在2011年7月份.具体的采样和DNA提取方法参见文献[15].其简要过程如下:先用蘸取过润湿液的无菌聚酯纤维拭子反复擦拭采样部位,然后取下拭子头放入DNA提取管中,最后按照改进过的MOBiO PowerSoilTMDNA Isolation Kit试剂盒说明书提取DNA.提取后的DNA样品将在-20℃的冰柜内储藏,以备后续实验.
1.2.2PCR扩增和高通量测序
采用巢式PCR对样品中的真菌基因组转录间隔区1区(Internal Transcribed Spacer 1, ITS1)进行扩增,该区域为高变区,但PCR引物对应的是相对保守的18S或5.8S rRNA基因末端[16-17].第一轮PCR使用通用引物对NSA3和NLC2,第二轮PCR使用带有焦磷酸测序用的接头(Adaptor)的长引物AdaB-NSI1和AdaA-58A2R.然后对PCR产物进行纯化、DNA浓度测定、样品混合和割胶回收.最后送至测序公司,使用Roche 454 GS-FLX焦磷酸测序仪进行测序.具体的引物序列、PCR条件和测序样品准备过程参见文献[15].
1.2.3序列分析
所有的序列分析过程都在QIIME软件集成平台上进行[18].首先对得到的原始序列进行质量筛选,去除Q值小于25、长度小于200bp、含有模糊碱基N或连续8个及以上相同碱基的序列.接着根据特异的标签序列对序列进行重命名,新的名字含有对应的样品信息.然后所有序列与UNITE数据库(https:∥unite.ut.ee/)比对进行嵌合体检测和去除.筛选后的序列使用USEARCH61的算法[19]根据95%的相似度进行可操作分类单位(OTU)划分,并挑选出每个OTU的代表序列,以80%的置信度使用RDP classifier方法[20]与UNITE数据库比对得到对应的分类信息.
1.2.4多样性分析和统计学分析
Alpha多样性是用来评估群落内的多样性程度,我们分析了2种Alpha多样性指数:Chao1值和Shannon指数.Chao1值主要反映物种丰富度,Shannon指数同时反映物种丰富度和均一度.Beta多样性是用来评估不同群落间的组成差异,我们的分析中根据两个样品的OTU种类和丰度(A)使用Bray-Curtis算法计算样品间的Bray-Curtis距离,距离数值越小说明两个样品的群落组成越相似.
我们使用t检验对不同分组样品的Alpha多样性指数值和Beta多样性的Bray-Curtis距离进行显著性检验,使用方差分析(ANOVA)检验真菌丰度在各分组间的差异.ANOSIM分析用于检验不同分组之间的群落组成差异,在P值显著(P<0.01)的结果中R值的大小反映了差异程度.R值越接近于1说明不同分组之间的群落组成差异大,也就表示这种分组所依据的因素对微生物群落的影响大;而R值接近于0说明不同分组之间的群落组成差异小,也就表示这种分组所依据的因素对微生物群落的影响小.
2 结果与分析
2.1测序结果
经过质量筛选得到长度在210~510bp之间的高质量序列100397条,这些序列聚类形成622个OTU(相似度阈值为95%).这些序列来自6对母婴的6个部位(3次重复采样),共计216个样品,其中27个样品因为没有达到每样品150条序列的最低标准而被舍去,最终用于分析的样品为189个.
2.2母亲皮肤的真菌群落特点
母亲皮肤表面的真菌主要来自两个门,子囊菌门(Ascomycota)和担子菌门(Basidiomycota).在属水平占主导的是担子菌门的马拉色菌属,它在不同部位样品中的平均丰度达到了64.0%.除此之外,丰度大于2.0%的属有子囊菌门的念珠菌属(Candida, 8.4%)、枝孢霉属(Cladosporium, 4.2%)和链格孢霉属(Alternaria, 2.9%).根据ANOSIM检验的结果,3次重复采样之间没有显著差异,对母亲皮肤表面的真菌群落组成影响最大的因素是不同的皮肤部位,而且其差异大于不同个体之间的差异(表2).
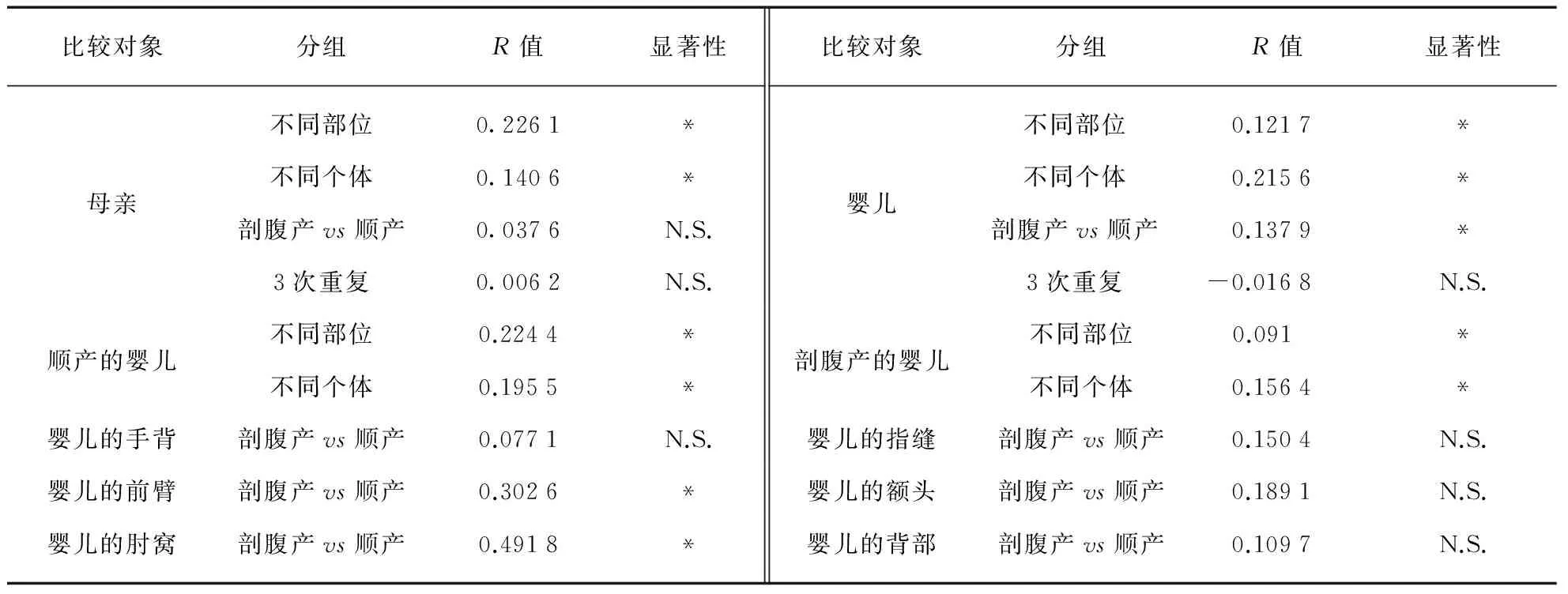
表2 不同分组的ANOSIM检验的结果
*:P<0.01, N.S.:P>0.01,无显著差异.
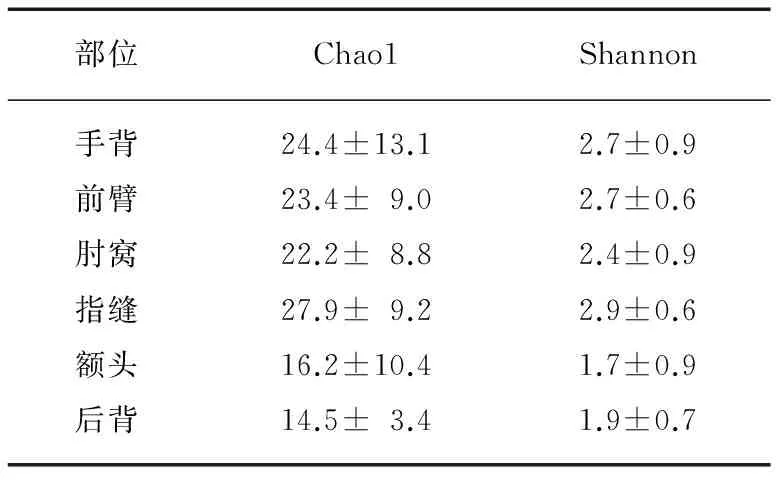
表3 母亲不同部位的Alpha多样性指数
不同皮肤部位的真菌群落多样性差异较大,油性部位(额头和背部)的Alpha多样性低于干性部位(手背和前臂)和湿性部位(指缝和肘窝)(表3),其中指缝部位的Shannon指数显著高于额头和背部(P=0.004和P=0.008).因此,不同属的真菌在各个部位中的丰度也存在着差异.丰度最高的马拉色菌属在油性部位额头和背部的丰度分别为82.4%和85.8%,显著高于手背(43.1%,P=0.006和P=0.002)和指缝(44.7%,P=0.008和P=0.003).其他几个主要属的真菌在不同部位的丰度虽然没有显著差异,但也有所不同(表4,图1,见第242页).念珠菌属在湿性和干性部位的丰度较高(8.3%~14.4%),而在油性部位的丰度较低(3.4%~5.0%).枝孢霉属则是在手背的丰度较高(8.9%),在其他部位较低(1.3%~5.4%).而链格孢霉属在手背丰度最高(7.6%),在后背丰度最低(0.5%).
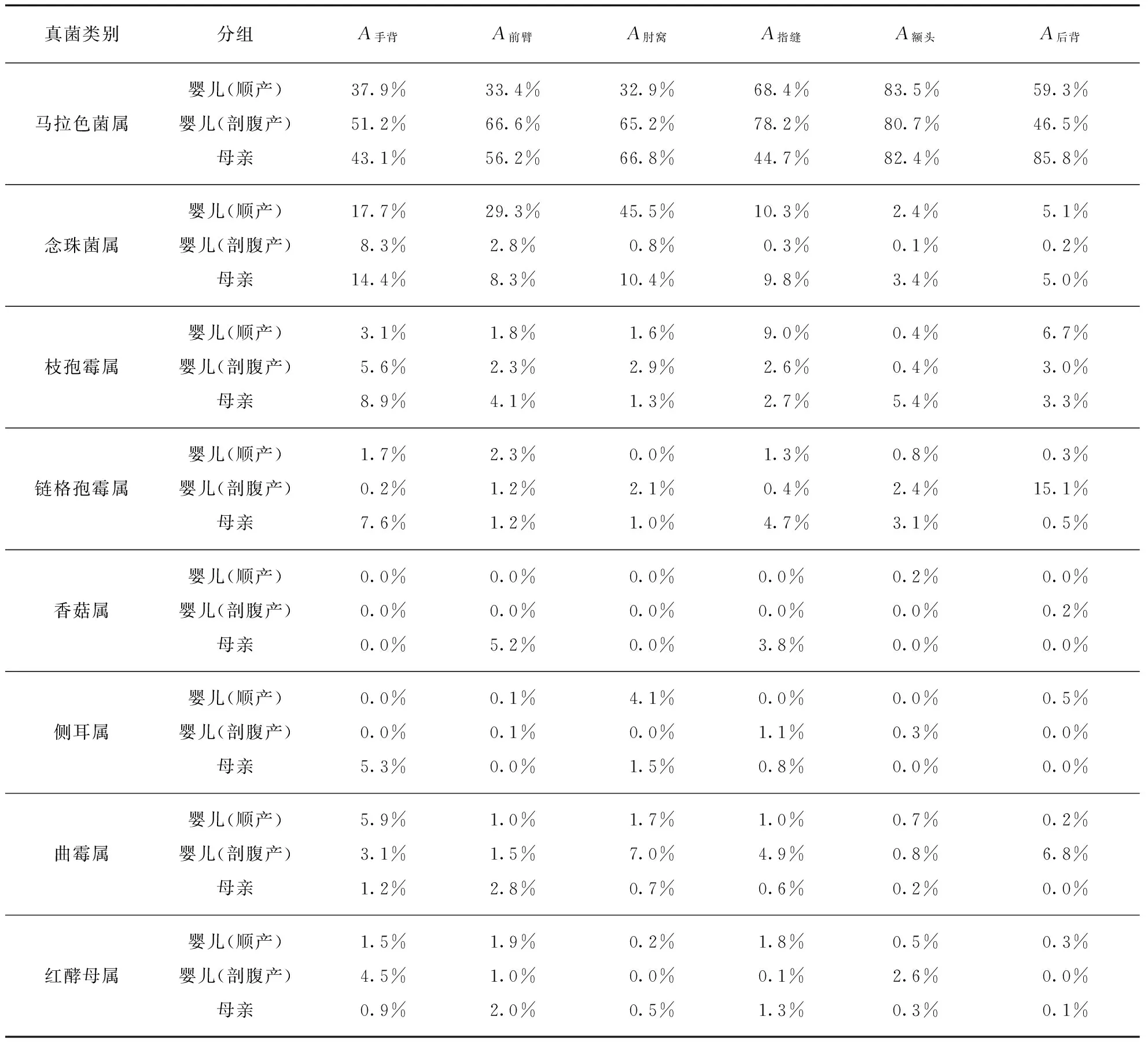
表4 母亲和不同分娩方式婴儿各部位的主要真菌组成
2.3婴儿皮肤的真菌群落特点
和母亲一样,婴儿皮肤表面的真菌也主要来自子囊菌门和担子菌门.各个部位中平均丰度最高的属也是担子菌门的马拉色菌属(59.4%).除此之外,丰度大于2.0%的属有来自子囊菌门的念珠菌属(8.9%)、枝孢霉属(3.2%)、曲霉属(Aspergillus,3.0%)和链格孢霉属(2.5%).比较母亲和婴儿相同部位的主要真菌的丰度差异,只发现指缝(母亲:44.7%,婴儿:73.9%,P=0.003)和后背(母亲:85.8%,婴儿:51.7%,P=0.003)的马拉色菌有显著差异.母婴相同部位的真菌群落的Alpha多样性没有显著差异.
ANOSIM检验结果与母亲相反,对婴儿皮肤表面真菌群落组成影响最大的因素是不同个体,其次是分娩方式以及部位,3次重复采样之间没有显著差异(表2).而且顺产婴儿的皮肤真菌群落组成受不同部位和个体因素的影响程度大于剖腹产婴儿受这些因素的影响;进一步比较婴儿不同部位所受分娩方式的影响,发现顺产婴儿和剖腹产婴儿真菌群落组成差异最大的部位是肘窝和前臂(表2).对于Alpha多样性,无论是Chao1值还是Shannon指数都表明顺产婴儿的皮肤真菌群落多样性显著高于剖腹产的婴儿(P=5.9×10-5和0.01).
顺产婴儿皮肤的马拉色菌属的丰度略小于剖腹产婴儿(52.5%,64.8%,P=0.03),而念珠菌属的丰度则显著高于剖腹产婴儿(17.9%,2.1%,P=1.6×10-6).具体到各个部位中,不同分娩方式的婴儿只在肘窝、前臂和指缝部位存在丰度差异显著的真菌(图2,表4).其中,顺产婴儿的念珠菌属的丰度在肘窝、前臂和指缝3个部位中都显著高于剖腹产婴儿(肘窝:45.5%,0.8%;前臂:29.3%,2.8%;指缝:10.3%,0.3%;P=0.001, 0.0007和0.001),而马拉色菌属的丰度只在前臂部位显著小于剖腹产婴儿(33.4%,66.6%,P=0.0004),另外,红酵母属(Rhodotorula)的丰度在指缝部位高于剖腹产婴儿(1.8%,0.05%,P=0.03).
2.4母亲和婴儿皮肤真菌群落的关联性
根据Beta多样性得到的样品间的Bray-Curtis距离,分别计算了每个母亲与1)自己的婴儿,2)相同生产方式的其他婴儿,以及3)不同生产方式婴儿的平均Bray-Curtis距离,以此来比较互相之间真菌群落差异的大小.在3个剖腹产的母亲中有两个母亲(M01和M03)的皮肤真菌群落,相比其他婴儿,都与自己的婴儿皮肤真菌群落更相似(P<0.05).如果与顺产婴儿相比,所有剖腹产母亲的真菌群落都与剖腹产婴儿的更接近(P<0.05,图3(a)).相反,顺产母亲的皮肤真菌群落并没有与自己的婴儿更相似(除了M05,图3(b)).
3 讨 论
本研究的测序深度相对来说比较低,平均每个样品只有531条序列,最少的样品只有150条.不过150条的测序深度也足够覆盖皮肤表面大部分丰度较高的微生物[21].有研究表明相似的测序深度并不会影响对主要的微生物群落特征的考察,并且还发现100~400条不等的测序深度反映出来的样品之间的相互关系是一致的[22].此外,有研究还发现测序深度较浅但样品数多的数据集,其优点明显大于样品数少而测序量大的数据集[23-24].本研究中所有部位的样品都间隔一天进行了3次重复采样,大大增加了样品数.而且3次重复采样的样品之间没有显著差异,证明了实验结果的可再现性,同时也说明人的皮肤真菌群落具有与细菌群落一致的随时间推移而相对稳定的特点[1, 25].
母亲皮肤表面的真菌群落中马拉色菌占主导,其他主要属包括念珠菌属、枝孢霉属和链格孢霉属,这与我们以前研究中的成年人女性皮肤真菌群落组成的结果一致[15].本研究中发现母亲不同皮肤部位的真菌群落有着显著差异,并且皮肤表面的微环境(油性、湿性和干性)是影响群落结构的重要因素.这与成年人皮肤表面微生物群落(不论是真菌还是细菌)具有部位特异性的研究结论一致[1,25-26].有研究进一步发现真菌群落多样性最高的是与脚相关的部位,如脚后跟、脚趾等;其次是与手相关的部位,如前臂、肘窝等;多样性最低的是躯干上的部位如额头、背部、腹股沟等[26].本研究虽然没有考察如此多的皮肤部位,但在分析的6个部位里也发现与手相关部位(手背、前臂、指缝和肘窝)的真菌群落多样性高于躯干上的皮肤部位(额头和背部).同时我们注意到多样性较低的两个部位均是油性部位,而马拉色菌作为嗜脂菌偏好油脂含量高的部位[27],所以这两个部位上的马拉色菌含量也远高于其他部位.可能也正是基于此,大量的马拉色菌挤占了其他真菌的生长空间,从而降低了油性部位的多样性.结果中也确实发现除了马拉色菌外的主要真菌在油性部位的丰度都要比在其他部位的丰度低.
而对于出生2~4个月的婴儿,他们不同皮肤部位间的真菌群落差异并没有作为成年人的母亲不同部位间的差异大.有研究采集了刚出生5分钟婴儿的皮肤、口腔、呼吸道分泌物和粪便的微生物,发现不同部位间的细菌群落没有显著性差异[8].但是婴儿的皮肤细菌群落多样性在出生的第一年会随着时间不断增加[13].皮肤的真菌群落可能也和细菌群落类似,有一个不断发展变化的过程.2~4个月婴儿的皮肤真菌群落虽然还没有体现出和成年人一样的部位特异性,但和刚出生的婴儿相比还是体现了一定的部位差异.而且母婴相同部位的主要真菌(除了马拉色菌)的丰度以及Alpha多样性都没有显著差异,说明2~4个月的婴儿的皮肤真菌群落组成已经与母亲的皮肤真菌群落比较接近.除了部位的差异性.本研究还发现分娩方式对婴儿的皮肤真菌群落的影响显著.顺产婴儿的皮肤真菌群落多样性显著高于剖腹产婴儿,并且顺产婴儿的个体差异和部位差异也都比剖腹产婴儿大.这可能是因为相比剖腹产婴儿,顺产婴儿由于分娩时经过阴道,多了真菌的一种来源.并且确实有研究发现刚出生婴儿皮肤表面的细菌群落与母亲阴道的细菌群落极为相似[8],说明顺产这种分娩方式确实会将阴道的微生物转移到婴儿的皮肤上.而且本研究中还发现顺产婴儿的念珠菌丰度显著高于剖腹产的婴儿.念珠菌是一种在大多数孕期女性阴道中比较常见的真菌[28],即使是健康的未孕女性中也有近20%的人阴道携带念珠菌[29].这意味着顺产婴儿皮肤上的念珠菌很有可能来自分娩时与母亲阴道的接触.而且念珠菌是一种常见的条件致病菌,会感染人体不同部位引起各种疾病[30].所以对于顺产婴儿皮肤含有较多的念珠菌的现象要引起重视,做好相关真菌疾病的早期防治.
本研究还考察了每一对母婴之间真菌群落的关联性,发现剖腹产的母亲与自己孩子的皮肤真菌群落更相似,而顺产的母亲却并不都与自己的小孩更相似.有研究发现婴儿脸颊的马拉色菌可能来自其母亲皮肤,并且从出生第一天起马拉色菌的丰度就不断增加,在第30天达到成人皮肤中马拉色菌的丰度[31].这说明婴儿在出生后由于与自己的母亲有更多的接触,使得母亲皮肤表面的真菌成为其真菌群落组成的重要来源.虽然与剖腹产的婴儿一样,顺产婴儿也都与自己的母亲生活在一起,但几个月之后也能保留顺产时所接触的母亲阴道的真菌,说明分娩方式对皮肤微生物组成的影响是较长期的.
本次的研究证实了婴儿皮肤的真菌群落如同细菌群落会受到分娩方式的影响,而且母亲阴道的真菌可能是顺产婴儿皮肤真菌群落的一个重要来源.这个结论提示我们对于青少年的皮肤真菌群落的演变与建立,需要从包括分娩方式等因素在内的更长时间轴去考量.虽然在本研究中对189个样本进行了分析,但因这些样本来源于6对母婴,所以个体差异可能引起一些干扰,因此需要对更多的母婴进行分析,提高所得结论的可信度.对于不同分娩方式婴儿皮肤真菌组成差异的考察,将有助于对婴幼儿皮肤清洁产品的开发以及早期真菌皮肤病的防治.
致谢: 本研究得到强生(中国)有限公司的资助.
[1]COSTELLO E K, LAUBER C L, HAMADY M,etal. Bacterial community variation in human body habitats across space and time [J].Science, 2009,326(5960):1694-1697.
[2]LEY R E, LOZUPONE C A, HAMADY M,etal. Worlds within worlds:Evolution of the vertebrate gut microbiota [J].NatRevMicrobiol, 2008,6(10):776-788.
[3]PALMER C, BIK E M, DIGIULIO D B,etal. Development of the human infant intestinal microbiota [J].PLoSBiol, 2007,5(7):e177.
[4]BIASUCCI G, BENENATI B, MORELLI L,etal. Cesarean delivery may affect the early biodiversity of intestinal bacteria [J].JNutr, 2008,138(9):1796S-1800S.
[5]PENDERS J, THIJS C, VINK C,etal. Factors influencing the composition of the intestinal microbiota in early infancy [J].Pediatrics, 2006,118(2):511-521.
[6]GRONLUND M M, LEHTONEN O P, EEROLA E,etal. Fecal microflora in healthy infants born by different methods of delivery:Permanent changes in intestinal flora after cesarean delivery [J].JPediatrGastrNutr, 1999,28(1):19-25.
[7]MACKIE R I, SGHIR A, GASKINS H R. Developmental microbial ecology of the neonatal gastrointestinal tract [J].AmJClinNutr, 1999,69(5):1035-1045.
[8]DOMINGUEZ-BELLO M G, COSTELLO E K, CONTRERAS M,etal. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns [J].ProcNatlAcadSciUSA, 2010,107(26):11971-11975.
[9]GRICE E A, SEGRE J A. The skin microbiome [J].NatRevMicrobiol, 2011,9(8):244-253.
[10]ZHANG E, TANAKA T, TAJIMA M,etal. Characterization of the skin fungal microbiota in patients with atopic dermatitis and in healthy subjects [J].MicrobiolImmunol, 2011,55(9):625-632.
[11]LEE Y W, BYUN H J, KIM B J,etal. Distribution ofMalasseziaspecies on the scalp in korean seborrheic dermatitis patients [J].AnnDermatol, 2011,23(2):156-161.
[12]PAULINO L C, TSENG C H, STROBER B E,etal. Molecular analysis of fungal microbiota in samples from healthy human skin and psoriatic lesions [J].JClinMicrobiol, 2006,44(8):2933-2941.
[13]CAPONE K A, DOWD S E, STAMATAS G N,etal. Diversity of the human skin microbiome early in life [J].JInvestDermatol, 2011,131(10):2026-2032.
[14]SHENDURE J, JI H. Next-generation DNA sequencing [J].NatBiotechnol, 2008,26(10):1135-1145.
[15]迟亮,谭渊,全哲学.上海健康人群夏冬两季皮肤真菌群落组成:基于高通量测序技术的研究 [J].复旦学报(自然科学版),2014,53(6):697-703.
[16]TOJU H, TANABE A S, YAMAMOTO S,etal. High-coverage ITS primers for the DNA-based identification of ascomycetes and basidiomycetes in environmental samples [J].PLoSOne, 2012,7(7):e40863.
[17]MARTIN K J, RYGIEWICZ P T. Fungal-specific PCR primers developed for analysis of the ITS region of environmental DNA extracts [J].BMCMicrobiol, 2005,5(1):28.
[18]CAPORASO J G, KUCZYNSKI J, STOMBAUGH J,etal. QIIME allows analysis of high-throughput community sequencing data [J].NatMethods, 2010,7(5):335-336.
[19]EDGAR R C. Search and clustering orders of magnitude faster than BLAST [J].Bioinformatics, 2010,26(19):2460-2461.
[20]COLE J R, WANG Q, CARDENAS E,etal. The Ribosomal Database Project:Improved alignments and new tools for rRNA analysis [J].NucleicAcidsRes, 2009,37(Database issue):141-145.
[21]LAX S, SMITH D P, HAMPTON-MARCELL J,etal. Longitudinal analysis of microbial interaction between humans and the indoor environment [J].Science, 2014,345(6200):1048-1052.
[22]BLASER M J, DOMINGUEZ-BELLO M G, CONTRERAS M,etal. Distinct cutaneous bacterial assemblages in a sampling of South American Amerindians and US residents [J].ISMEJ, 2012,7(1):85-95.
[23]SHADE A, GREGORY CAPORASO J, HANDELSMAN J,etal. A meta-analysis of changes in bacterial and archaeal communities with time [J].ISMEJ, 2013,7(8):1493-1506.
[24]KUCZYNSKI J, LIU Z Z, LOZUPONE C,etal. Microbial community resemblance methods differ in their ability to detect biologically relevant patterns [J].NatMethods, 2010,7(10):813-867.
[25]GRICE E A, KONG H H, CONLAN S,etal. Topographical and temporal diversity of the human skin microbiome [J].Science, 2009,324(5931):1190-1192.
[26]FINDLEY K, OH J, YANG J,etal. Topographic diversity of fungal and bacterial communities in human skin [J].Nature, 2013,498(7454):367-370.
[27]GUPTA A K, BATRA R, BLUHM R,etal. Skin diseases associated withMalasseziaspecies [J].JAmAcadDermatol, 2004,51(5):785-798.
[28]BONFANTE-GARRIDO R, BARROETA S, DE MONTILVA A. The vaginal fungi in pregnant women [J].Mycopathologia, 1969,37(1):39-44.
[29]GOLDACRE M J, WATT B, LOUDON N,etal. Vaginal microbial flora in normal young women [J].BritMedJ, 1979,1(6176):1450-1453.
[30]SCULLY C, EL-KABIR M, SAMARANAYAKE L P. Candida and oral candidosis:A review [J].CritRevOralBiolM, 1994,5(2):125-157.
[31]NAGATA R, NAGANO H, OGISHIMA D,etal. Transmission of the major skin microbiota,Malassezia, from mother to neonate [J].PediatrInt, 2012,54(3):350-355.
Delivery Modes Affected Skin Fungal Communities of Infants
YING Shi1, TAN Yuan2, QUAN Zhexue1
(1.Department of Microbiology and Microbial Engineering, School of Life Sciences, FudanUniversity,Shanghai200438,China; 2.ScientificAffairs,Johnson&JohnsonChinaLtd.,Shanghai200245,China)
Upon delivery, infants leave the microorganism-free uterus and are exposed for the first time to a wide variety of microbes which shape the initial microbiota’s establishment. Thus the delivery modes determine whether microbe source that an infant first contact is mother’s vagina or surrounding environments. To determine the influence of different delivery modes to the fungal community composition on infants’ skin, we analyzed fungal communities of six skin sites from 6 mothers and their corresponding 2—4 month old infants with three replications (189 qualified samples in total) through high-throughput pyrosequencing. The results show that delivery mode had significant influence on the skin fungal community compositions of 2-4 month old infants.Candida, the common fungi in vagina of pregnant women, was significantly more abundant on the skin of vaginally delivered infants than on the skin of infants born via C-section. In contrast,Malassezia, the dominant fungi on mothers’ skin, was more abundant on the skin of infants born via C-section. Meanwhile, C-section mother’s fungal community was significantly more similar to her own infant’s microbiota than to the microbiota of other infants. However, skin fungal communities of vaginally delivered mothers were no more similar to their own infants than to the other infants.
delivery modes; skin fungi; high-throughput pyrosequencing; community composition
0427-7104(2016)02-0239-08
2015-04-23
应时(1989—),男,硕士;全哲学,男,教授,通讯联系人,E-mail:quanzx@fudan.edu.cn.
Q 938.1+5
A

