利用SSR分子标记构建四倍体杂交冰草的遗传连锁图谱
姜志艳,于肖夏,于卓*,张志成,石悦,姜超
(1.内蒙古农业大学农学院,内蒙古 呼和浩特 010019;2.内蒙古科技大学数理与生物工程学院,内蒙古 包头 014010)
利用SSR分子标记构建四倍体杂交冰草的遗传连锁图谱
姜志艳1,2,于肖夏1,于卓1*,张志成1,石悦1,姜超1
(1.内蒙古农业大学农学院,内蒙古 呼和浩特 010019;2.内蒙古科技大学数理与生物工程学院,内蒙古 包头 014010)
为构建四倍体杂交冰草分子遗传连锁图谱,对深入开展冰草产量、抗性等重要性状的QTL定位及分子标记辅助育种提供依据,以四倍体杂种F2分离群体的347个单株及亲本蒙古冰草和航道冰草为材料,采用SSR分子标记技术和Joinmap 4.0软件进行了遗传作图研究。试验从256对SSR引物中筛选出条带清晰稳定、多态性丰富的适宜引物30对,PCR扩增得到224个SSR标记位点,平均每对引物扩增出7.47个位点,其中多态性标记位点185个,占82.6%。偏分离分析显示,在185个SSR多态性标记位点中有24个标记产生偏分离,占13.0%,符合植物遗传作图时通常偏分离标记比率<30%的要求,可用于遗传作图。构建了1张四倍体杂交冰草的分子遗传连锁框架图谱,该图谱包含14个连锁群、185个标记,其长度范围在123.0~202.6 cM之间,连锁群LG4最长、LG12最短,各连锁群的平均长度167.32 cM,覆盖基因组总长度2342.5 cM,标记间的平均距离12.66 cM。
杂交冰草;四倍体;SSR标记;遗传连锁图谱
冰草为多年生草本植物,隶属于禾本科小麦族(Triticeae)。其抗逆性强、适应性广,饲用和生态价值高,可用于退化的天然草地改良、人工草地建植及公路和铁路护坡等水土保持[1-2]。四倍体杂交冰草(Agropyronmongolicum×A.cristatumcv. Fairway,2n=4x=28)是课题组利用国产蒙古冰草(A.mongolicum,2n=2x=14)与产自美国的航道冰草(A.cristatumcv. Fairway,2n=2x=14)相组配,人工去雄授粉杂交获得的二倍体杂种F1,经秋水仙碱溶液加倍染色体而育成的优良牧草,其既有母本蒙古冰草耐寒抗旱、适应性强等优良特性,又有父本航道冰草再生性和分蘖性强、耐倒伏、叶量大、营养价值和产量高的特点[3-5]。
简单序列重复SSR(simple sequence repeat)是一项成熟的DNA标记技术,因其具有重复性好、多态性丰富、标记呈共显性等优点,在作物品种和杂种真实性鉴定、种质资源遗传差异分析、辅助选择育种、分子遗传连锁图谱建立等方面已得到应用[6-9]。由于大多数多年生禾本科或豆科牧草都具有多倍性和异花授粉等生物学特性[10],其复杂的遗传背景使得牧草的遗传作图、重要性状QTL定位及分子标记辅助育种研究工作相比作物进展较慢。1993年Brummer等[11]最早利用RFLP分子标记构建了四倍体紫花苜蓿(Medicagosativa)的遗传连锁图,之后在二倍体的羊茅(Festucaspp.)、黑麦草(Loliumspp.)、高丹草(Sorghum-Sudangrass)及二倍体杂交冰草等牧草上构建了分子遗传连锁图谱[12-15],而有关四倍体杂交冰草的遗传作图至今未见有研究报道。本试验以四倍体杂交冰草F2群体为作图材料,拟利用SSR分子标记技术构建四倍体杂交冰草分子遗传连锁图谱,为深入开展四倍体冰草重要农艺性状的QTL定位、分子标记辅助育种及基因图位克隆等奠定基础。
1 材料与方法
1.1供试材料
材料为四倍体杂交冰草F2群体的单株及其亲本蒙古冰草和航道冰草,种植于呼和浩特市赛罕区内蒙古农业大学农场的饲草材料圃内。试验于2014年5月—2015年5月在田间和室内进行。
1.2DNA的提取与纯度检测
从四倍体杂交冰草F2群体内随机取347个单株及其父母本各10个单株的嫩叶,用北京天根公司生产的植物基因组试剂盒提取DNA,1.6%琼脂糖凝胶电泳检测其DNA纯度后稀释至50 ng/μL,置于-20℃冰箱中备用[16]。
1.3SSR-PCR扩增反应体系和程序
SSR-PCR扩增体系:反应液总体积为20 μL,其中含50 μmol/L 的10×Buffer 2.0 μL、2.5 mmol/L的 dNTP 2.0 μL、25 mmol/L的MgCl21.8 μL、50 μmol/L的Forward primer和 Reverse primer各 1.0 μL、5 U/μL的Taq DNA聚合酶0.1 μL、模板DNA 1.0 μL、ddH2O 11.1 μL。SSR-PCR扩增程序:94℃ 5 min,94℃ 30 s,56℃ 30 s,72℃ 1 min,30个循环,72℃延伸10 min[17]。
1.4PCR扩增产物检测及SSR引物来源
PCR反应结束后,加入6.0 μL变性剂,于PCR仪上95℃变性5 min,用7%的变性聚丙烯酰胺凝胶进行电泳,功率70 W,预电泳30 min,电泳时间2 h。电泳胶板在乙醇-冰乙酸溶液中固定,AgNO3溶液中染色,NaOH-甲醛溶液中显影,风干后照相[18]。
试验所用的256对SSR引物,是从NCBI网站公布的禾本科作物小麦(Triticumaestivum)、玉米(Zeamays)和高粱(Sorghumbicolor)上开发的部分SSR引物中选取,送至上海生物工程有限公司进行合成。
1.5SSR多态性位点统计
按胶板点样顺序对每个位点上的多态性位点条带进行记录,有带记为“1”,无带记为“0”,带型模糊不清或缺失的记为“—”,生成“0”和“1”组成的原始矩阵进行统计分析。多态性标记位点百分率计算公式:P(%)=(K/N)×100,式中,K为多态性位点数目、N为观测位点总数[19]。同一SSR引物PCR扩增2~3次,统计电泳胶板条带清晰稳定后的多态性位点。
1.6SSR标记偏分离统计
依据孟德尔分离规律,四倍体杂交冰草F2群体基因型理论分离比为3∶1,将每个SSR标记的基因型数目进行卡方检验(χ2测验),凡P<0.05的SSR标记均列入偏分离标记之内。
1.7SSR分子遗传连锁图谱构建
将统计的SSR标记数据中的“0”和“1”原始矩阵分别转换成F2群体所要求的数据格式,应用计算机作图软件Joinmap 4.0,构建以SSR分子标记为基础的四倍体杂交冰草的分子遗传连锁图谱。具体步骤:开New project命令创建一个新的文件夹;选择Options,设置参数即选择作图群体模式、群体数量和标记位点数;导入数据,选择LOD Groupings (tree) 窗口,用Calculate命令进行标记连锁分组(LOD=3.0~10.0),选用LOD≥3.0标记连锁组群,运行Create Groups for Mapping命令得到用于作图的连锁群;选择Calculate map命令构建连锁图谱;用Map chart完成图谱的绘制。
2 结果与分析
2.1各材料基因组DNA的纯度检测
用DNA试剂盒提取的四倍体杂交冰草F2群体347个单株及其亲本的基因组DNA,其电泳条带清晰稳定、无降解、无蛋白和RNA污染(图1),表明各材料的DNA纯度高,符合SSR分子标记试验的要求。

图1 F2群体部分单株基因组DNA电泳纯度检测Fig.1 Genomic DNA electrophoresis purity test of partial plants of the F2 population P1:蒙古冰草;P2:航道冰草;1-31:F2群体部分单株。M:Marker DL 2000.P1:A. mongolicum; P2:A. cristatum cv. Fariway;1-31:Partial plants of the F2 population.
2.2SSR引物筛选及多态性分析
用亲本蒙古冰草和航道冰草及其四倍体杂种F2群体1个单株的基因组DNA为模板进行适宜引物筛选,从256对SSR引物中选出条带清晰、重复性和多态性好的SSR适宜引物30对(表1,图2)。
利用这30对引物对F2作图群体347个单株及亲本的DNA进行PCR扩增,共获得224个位点,平均每对引物扩增出7.47个位点,其中多态性标记位点185个,平均多态性标记位点百分率为82.6%,表明该四倍体杂交冰草F2群体的SSR多态性丰富(图3,表1)。
2.3SSR作图标记的分离及遗传连锁图谱特征分析
SSR标记分离及连锁图谱的基本特征如表2和图4所示。经卡方检验表明,当P<0.05时,在已获得的185个SSR多态性标记位点中,有161个标记符合孟德尔分离比例,其占标记总数的87.0%。另有24个SSR标记产生偏分离,偏分离比例仅为13.0%,符合植物遗传作图时通常偏分离标记比率<30%的要求[20],这185个SSR分子标记均可用于遗传作图。
利用Joinmap 4.0软件对185个SSR标记进行连锁分析,构建出1张四倍体杂交冰草的SSR分子遗传连锁框架图谱,该图谱包含14个连锁群、185个标记,14个连锁群的长度范围在123.0~202.6 cM之间,连锁群LG12最短、LG4最长,各连锁群的平均长度为167.32 cM,总长度为2342.5 cM,标记间的平均距离为12.66 cM。
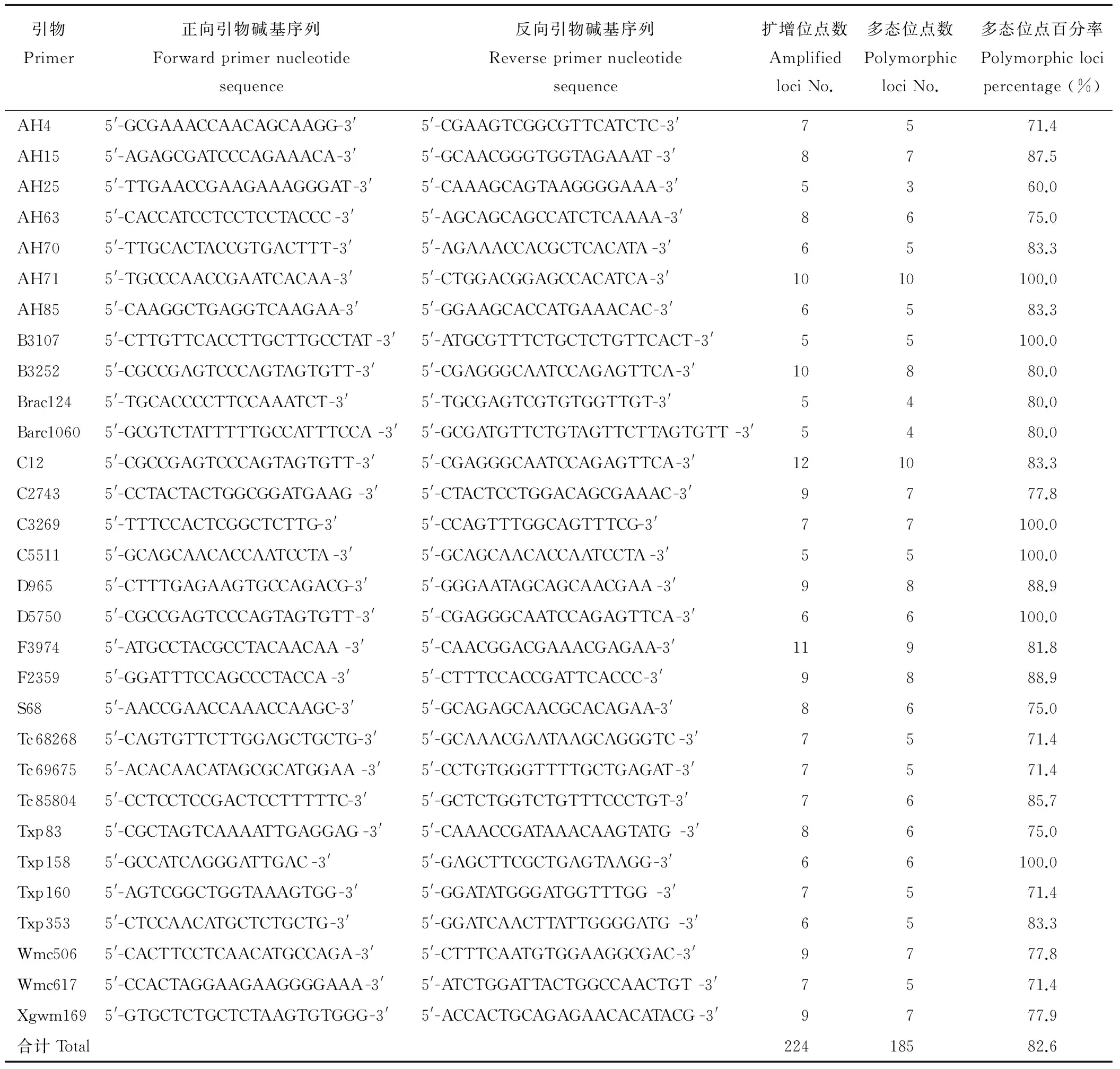
表1 适宜SSR引物碱基序列及其多态性标记位点Table 1 Nucleotide sequence of SSR primers and polymorphic markers loci

图2 用F2的1个单株及双亲的DNA对部分SSR引物筛选结果Fig.2 SSR primer screening result with one plant in F2 population and parents F2:F2 的1个单株;P1:蒙古冰草;P2:航道冰草。 F2:One plant of hybrid F2;P1:A. mongolicum;P2:A. cristatum cv. Fariway.

图3 引物D965(A)和F3974(B)对F2分离群体部分单株及其亲本DNA的扩增结果Fig.3 DNA amplified results of partial plants of F2 segregating population in hybrid wheatgrass and their parents using the primer D965(A)and F3974(B) P1:蒙古冰草;P2:航道冰草;1-55:F2群体部分单株。M:Marker DL 1000.P1:A. mongolicum;P2:A. cristatum cv. Fariway;1-55:F2 partial plants.

连锁群Linkagegroups连锁群长度Maplength(cM)连锁标记数No.oflinkedmarkers偏分离标记数(P<0.05)No.ofbiasedmarkers偏分离标记比率Ratioofdistortedmarkers(%)标记间平均距离Averagedistance(cM)LG1174.95120.034.98LG2175.410220.017.54LG3180.850036.16LG4202.629413.86.99LG5195.31827.111.49LG6147.541611.83.60LG7150.512216.712.54LG8141.430047.13LG9151.230050.40LG10178.014321.412.71LG11161.32328.77.01LG12123.07114.317.57LG13178.41010.117.84LG14182.250036.44合计Total2342.51852413.012.66

图4 四倍体杂交冰草SSR分子遗传连锁框架图谱Fig.4 SSR genetic linkage map of tetraploid hybrid wheatgrass
3 讨论
高密度的分子遗传连锁图谱是深入开展植物重要性状QTL定位及分子标记辅助育种等研究的基础[21-22]。与小麦、玉米、水稻(Oryzasativa)等农作物构建的遗传连锁图相比,目前国内外已构建的牧草遗传连锁图谱的图幅均偏小,且标记间空隙较大,使得遗传图谱在牧草分子标记辅助育种及重要性状QTL定位等研究方面较薄弱。本课题组曾利用蒙古冰草×航道冰草杂种F2分离群体构建了含7个连锁群、175个分子标记(AFLP标记152个、RAPD标记23个)的二倍体杂交冰草分子遗传图谱,其全长为416 cM,平均标记间密度为2.47 cM[15];本试验构建了含14个连锁群、185个SSR标记的四倍体杂交冰草分子遗传连锁图谱,其全长为2342.5 cM,远超过二倍体杂交冰草图谱的长度,表明四倍体的基因组明显大于二倍体;但标记间的密度(12.66 cM)小于二倍体,而且在各连锁群上标记的分布密度不均匀,如连锁群LG6上的标记数较多,密集了40个SSR标记,而连锁群LG8和LG9上SSR标记数均只有3个,这说明该图谱是一张SSR框架图谱,还有待于进一步扩大SSR标记数量或与其他分子标记如AFLP及SRAP等进行整合,来增加图谱中的分子标记数目、加大图谱密度,目前课题组正在开展四倍体杂交冰草遗传连锁图谱的加密研究。
遗传图谱精确度的高低很大程度取决于构建群体的大小,理论上作图群体越大,作图精度就越高,但若作图群体过大,就会使实验工作量和费用相应增加,故选择合适的群体数目很重要[23-24]。从作图效率看,作图群体的大小主要决定于3个方面:一是参照随机分离的结果计算出最大图距,二是两个标记间能够测算出重组的最小图距,三是所选用作图群体的类型。为了能够提高作图精度,在构建植物分子标记连锁图时需考虑作图群体的类型,一般认为作图所需的群体大小的顺序为F2>Rl>BC和DH[25]。目前在牧草上遗传图谱构建所用的作图群体在100~200个之间,本试验选用四倍体杂交冰草F2分离群体的347个单株进行遗传作图研究,主要考虑多年生牧草的多倍性,其数量性状分离复杂且表型受环境影响较大,因此控制试验误差显得尤为重要。
4 结论
试验筛选出条带清晰、重复性和多态性好的SSR适宜引物30对。对四倍体杂交冰草F2群体347个单株及亲本的基因组DNA进行PCR扩增得到185个SSR标记。构建了1张四倍体杂交冰草SSR分子遗传连锁框架图谱,其14个连锁群的长度变幅123.0~202.6 cM,覆盖基因组总长度2342.5 cM,标记间的平均距离12.66 cM。
References:
[1]Yun J F, Li R F, Mi F G. Interspecific hybridization and hybrid analysis of crested wheatgrass. Acta Agrestia Sinica, 1997, 5(4): 221-227.
[2]Wang F. Study on Genetic Diversity of Agropyron Gaertn Germolasm Resources[D].Lanzhou: Lanzhou University, 2009.
[3]Quan W, Yu Z, Wang Y H,etal. Isozyme analysis on lines of F4hybrid betweenAgropyronmongolicumandA.cristatumcv.Fairway. Acta Agrestia Sinica, 2006, 14(1): 14-19.
[4]Bao M L. Genetic Character Analysis on Chromosome Doubling Plants F2of Reciprocal Hybrids betweenA.mongolicumandA.cristatumcv. Fairway[D]. Huhhot: Inner Mongolia Agricultural University, 2012.
[5]Yu X X, Jiang Z Y, Yu Z,etal. The new variety breeding of tetraploid hybrid wheatgrass—Mengza No.1.Pratacultural Science, 2015, 32(5): 738-744.
[6]Cai H W, Inoue M, Yuyama N,etal. Isolation, characterization and mapping of simple sequence repeat markers in Zoysiagrass (Zoysiaspp.). Theoretical and Applied Genetics, 2005, 112(1): 158-166.
[7]Wang H M, Zhang Z Y, Chen Y L. SSR markers and their application in plant genetics. Journal of Northwest Normal University, 2003, 39(1):113-116.
[8]Ma Y H, Yu X X, Yu Z,etal. Cytological identification and SSR analysis of new strains of tetraploidy hybrid wheatgrass. Journal of Triticeae Crops, 2014, 34(2): 187-193.
[9]Hayward M D, Forster J W, Jones J G,etal. Genetic analysis ofLolium. I. Identification of linkage groups and the establishment of a genetic map. Plant Breeding, 1998, 117(5): 451-455.
[10]Jones E S, Natalia L, Mahoney M D,etal. An enhanced molecular marker based genetic map of perennial ryegrass (Loliumperenne) reveals comparative relationships with otherPoaceaegenomes. Genome, 2002, 45: 282-295.
[11]Brummer E C, Bouton J H, Kochert G. Development of an RFLP map in diploid alfalfa. Theoretical and Applied Genetics, 1993, 86(2): 329-332.
[12]Alm V, Fang C, Busso C S,etal. A linkage map of meadow fescue (FestucapratensisHuds.) and comparative mapping with other Poaceae species. Theoretical and Applied Genetics, 2003, 108(1): 25-40.
[13]Studer B, Boller B, Bauer,etal. Consistent detection of QTLs for crown rust resistance in Italian ryegrass (LoliummultiflorumLam. )across environments and phenotyping methods. Theoretical and Applied Genetics, 2007, 115(1): 9-17.
[14]Lu X P. Genetic Mapping and Gene Localization of Main Agronomic Characters ofSorghumbicolor×S.sudangrass[D]. Huhhot: Inner Mongolia Agricultural University, 2005.
[15]Li X L. Studies on Genetic Characters of Several Interspecific Hybrids F1inElymusand Construction of Molecular Genetic Map of Weatgrass[D]. Huhhot: Inner Mongolia Agricultural University, 2008.
[16]Li C Q, Yu X X, Ju T H,etal. Identification ofSolanumtuberosumhybrids by SSR molecular markers. Acta Botanica Boreali-occidentalia Sinica, 2012, 32(7): 1351-1360.
[17]Zhang Z Q, Yu X X, Ju T H,etal. Karyotype and SSR analysis of 3 potato hybrid lines. Acta Botanica Boreali-occidentalia Sinica, 2014, 34(7): 1318-1324.
[18]Yu Z, Xie R, Yu X X,etal. SSR analysis of Sorghum-Sudangrass new strains with low hydrocyanic acid content. Acta Prataculturae Sinica, 2014, 23(1): 223-228.
[19]Nei M. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences of the USA, 1973, 70(12): 3321-3323.
[20]Song X L, Sun X Z, Zhang T Z. Segregation distortion and its effect on genetic mapping in plants. Journal of Agricultural Biotechnology, 2006, 14(2): 286-292.
[21]Zhang Y H, Yu H F, Hou J H,etal. Construction of a genetic map of sunflower using a population of re-combinant inbred lines (RILs). Hereditas, 2014, 36(10): 1036-1042.
[22]Hu L L, Ye Y Q, Lv T T,etal. QTL mapping and genetic analysis for grain weight in wheat (Triticumaestivum) under different water environments. Acta Prataculturae Sinica, 2015, 24(8): 118-129.
[23]Hackett C A, Broadfoot L B. Effects of genotyping errors, missing values and segregation distortion in molecular marker data on the construction of linkage maps. Heredity, 1992, 69: 315-324.
[24]Zhang L Y, Wang S Q, Li H H,etal. Effects of missing marker and segregation distortion on QTL mapping in F2populations. Theoretical Applied Genetics, 2010, 121(6): 1071-1082.
[25]Yu X X. Development of a High-density Genetic Linkage Map and Mapping of QTLs for Dry-matter Content in Sweetpotato,Ipomoeabatatas(L.)Lam[D]. Beijing: China Agricultural University, 2013.
[1]云锦凤, 李瑞芬, 米福贵. 冰草的远缘杂交及杂种分析. 草地学报, 1997, 5(4): 221-227.
[2]王方. 冰草属植物种质资源遗传多样性研究[D]. 兰州: 兰州大学, 2009.
[3]权威, 于卓, 王月华, 等. 蒙古冰草×航道冰草杂种F4代株系同工酶分析. 草地学报, 2006, 14(1): 14-19.
[4]包美莲. 蒙古冰草与航道冰草正反交杂种染色体加倍植株F2遗传性状分析[D]. 呼和浩特: 内蒙古农业大学, 2012.
[5]于肖夏, 姜志艳, 于卓, 等. 四倍体杂交冰草新品种—蒙杂冰草1号的选育. 草业科学, 2015, 32(5): 738-744.
[7]王红梅, 张正英, 陈玉梁. SSR标记技术及其在植物遗传学中的应用. 西北师范大学学报, 2003, 39(1): 113-116.
[8]马艳红, 于肖夏, 于卓, 等. 四倍体杂交冰草新品系的细胞学鉴定及SSR分析. 麦类作物学报, 2014, 34(2): 187-193.
[14]逯晓萍. 高丹草遗传图谱构建及重要农艺性状的基因定位研究[D]. 呼和浩特: 内蒙古农业大学, 2005.
[15]李小雷. 几种披碱草种间杂种F1遗传特性及冰草分子图谱构建研究[D]. 呼和浩特: 内蒙古农业大学, 2008.
[16]李长青, 于肖夏, 鞠天华, 等. 马铃薯杂种F1的SSR鉴定. 西北植物学报, 2012, 32(7): 1351-1360.
[17]张自强, 于肖夏, 鞠天华, 等. 3个马铃薯杂种优良株系的核型及SSR分析. 西北植物学报, 2014, 34(7): 1318-1324.
[18]于卓, 谢锐, 于肖夏, 等. 低氢氰酸含量高丹草新品系及其亲本的SSR分析. 草业学报, 2014, 23(1): 223-228.
[20]宋宪亮, 孙学振, 张天真. 偏分离及对植物遗传作图的影响. 农业生物技术学报, 2006, 14(2): 286-292.
[21]张永虎, 于海峰, 侯建华, 等. 利用向日葵重组自交系构建遗传图谱. 遗传, 2014, 36(10): 1036-1042.
[22]胡亮亮, 叶亚琼, 吕婷婷, 等. 不同水分环境条件下小麦粒重QTL定位及遗传分析. 草业学报, 2015, 24(8): 118-129.
[25]于肖夏. 甘薯高密度分子连锁图谱的构建和干物质含量的QTL定位[D]. 北京: 中国农业大学, 2013.
*Construction of a genetic linkage map for tetraploid hybrid wheatgrass using a SSR molecular marker
JIANG Zhi-Yan1,2,YU Xiao-Xia1,YU Zhuo1*,ZHANG Zhi-Cheng1,SHI Yue1,JIANG Chao1
1.AgronomyCollege,InnerMongoliaAgriculturalUniversity,Huhhot010019,China;2.SchoolofMathematics,PhysicsandBiologicalEngineering,InnerMongoliaUniversityofScience&Technology,Baotou014010,China
To establish a genetic linkage map in tetraploid hybrid wheatgrass genetic mapping was conducted using a simple sequence repeats (SSR) molecular marker technique with ‘Joinmap’ 4.0 software. 347 individuals from the F2segregating population and their parents were utilized, this helped lay the foundation for further study of marker-assisted breeding, and quantitative trait locus (QTL) location of important traits in wheatgrass, such as disease resistance and yield. Thirty optimal primers with clear, stable and high polymorphic bands were screened from 256 tested SSR primers. A total of 224 SSR loci were obtained from polymerase chain reaction (PCR) amplification with an average of 7.47 loci per primer, of which 185 were polymorphic loci, accounting for 82.6% of all loci. Segregation distortion analysis showed that a total of 24 loci were distorted, accounted for 13.0% of all (185) polymorphic loci, which met the requirement of <30% segregation distortion for plant genetic mapping. These polymorphic loci would be useful for genetic mapping. In this study, a molecular genetic linkage map of tetraploid hybrid wheatgrass was constructed which contained 185 loci and 14 linkage groups with length ranging from 123.0 to 202.6 cM. The longest and the shortest linkage group were LG4 and LG12, respectively. The average length of each linkage group was 167.32 cM, total length of the genome was 2342.5 cM and the average marker distance was 12.66 cM.
hybrid wheatgrass; tetraploid; SSR marker; genetic linkage map
10.11686/cyxb2015410
http://cyxb.lzu.edu.cn
2015-09-06;改回日期:2015-10-21
国家自然科学基金面上项目(31172254)资助。作者简介:姜志艳(1976-),女,内蒙古包头人,讲师,博士。E-mail: L2359916@sina.com
Corresponding author. E-mail:yuzhuo58@sina.com
姜志艳,于肖夏,于卓,张志成,石悦,姜超. 利用SSR分子标记构建四倍体杂交冰草的遗传连锁图谱. 草业学报, 2016, 25(6): 94-101.
JIANG Zhi-Yan,YU Xiao-Xia,YU Zhuo,ZHANG Zhi-Cheng,SHI Yue,JIANG Chao. Construction of a genetic linkage map for tetraploid hybrid wheatgrass using a SSR molecular marker. Acta Prataculturae Sinica, 2016, 25(6): 94-101.

