Shewanella haliotis BP-1海藻酸裂解酶基因的克隆表达*
黄桂媛,温顺华,李 锋,卢明倩,王巧贞,廖 威,黄庶识**
(1.广西科学院生物物理实验室,广西南宁 530007;2.厦门万泰凯瑞生物技术有限公司,福建厦门 361003;3.黔南州民族师范学院化学化工学院,贵州都匀 558000;4.广西职业技术学院,广西南宁 530226)
Shewanella haliotis BP-1海藻酸裂解酶基因的克隆表达*
黄桂媛1,温顺华2,李锋3,卢明倩1,王巧贞1,廖威4,黄庶识1**
(1.广西科学院生物物理实验室,广西南宁530007;2.厦门万泰凯瑞生物技术有限公司,福建厦门361003;3.黔南州民族师范学院化学化工学院,贵州都匀558000;4.广西职业技术学院,广西南宁530226)
【目的】了解海洋细菌Shewanella haliotis BP-1中海藻酸裂解酶降解海藻酸钠的生物活性。【方法】应用基因克隆和大肠杆菌异源表达技术,过量表达海藻酸裂解酶,将粗酶液通过DEAE Sepharose FF柱分离纯化后检测其酶活性。【结果】从S.haliotis BP-1菌株的基因组DNA中克隆得到一个大小为2 157 bp的海藻酸裂解酶基因Alg17S,该基因编码的海藻酸裂解酶Alg17S属于PL17家族的蛋白,大小为79 726 Da,其中包括N端26个氨基酸的信号肽,与Saccharophagus degradans 2-40菌株产生的海藻酸裂解酶Alg17C具有高度同源性,相似性为52%。经纯化后获得的重组酶Alg17S和△snAlg17S(N端不含26个氨基酸的信号肽)均具有降解海藻酸钠的活性,但△snAlg17S对海藻酸钠的催化活性比Alg17S高,其酶比活力高达9 635 U/mg。【结论】重组海藻酸裂解酶△snAlg17S兼具高表达水平及高酶活性,是进一步研究海藻酸盐糖化和生物燃料生产的潜在的优势酶。
海藻酸裂解酶克隆表达酶活性测定Shewanella haliotis BP-1
0 引言
【研究意义】海藻酸具有在水介质中形成粘性溶液和凝胶的能力以及对生物无毒性的特点,在医药、化妆品、食品和生物技术等行业得到广泛应用[1]。同时海藻酸的降解产物褐藻寡糖具有广泛的生物活性,如促进植物生长、缓解植物非生物胁迫、抗肿瘤、抑菌、抗凝血、降血糖血脂、抗自由基氧化、抗炎症、免疫调节活性等作用,在绿色农业、医药保健、食品、日用化工等领域具有广阔的前景[2]。【前人研究进展】目前已有很多关于产海藻酸裂解酶细菌的报道,例如Pseudomonas sp.,Azotobacter sp.,Alteromonas sp.,Vibrio sp.,Sphingomonas sp.,Flavobacterum sp.等[3]。大多数细菌海藻酸裂解酶通常是分泌到细胞周质间隙或培养基即细胞外降解海藻酸的,但也有部分如Sphingomonas sp.A1菌株对海藻酸的降解作用发生在细胞质中,菌株A1在ABC转运蛋白的协作下将高分子海藻酸摄入细胞质中,然后进一步分解[3-7]。另外,在Carbohydrate-Active enZYmes(CAZY)数据库中登记的细菌海藻酸裂解酶基因已经超过500个[8]。1993年,Boyd等[9]首次克隆了假单胞菌(Pseudomonas alginovora)海藻酸裂解酶编码基因algL,并在大肠杆菌中成功表达,粗酶比活力为146 U/mg,是原始野生菌株的2.5倍;Uchimura等[10]从Vibrio sp.JAM-Aam菌株中克隆得到海藻酸裂解酶编码基因A9mT,并使其在大肠杆菌DH5α中分泌表达,从发酵液中分离纯化得到A9mT蛋白酶的生物活性高达1 401 U/mg;Li等[11]从Agarivorans sp.L11菌株中克隆到一个新的编码海藻酸裂解酶基因AlyL1,该基因在大肠杆菌中的重组表达蛋白AlyL1具有高的生物催化活性,比活力为1 370 U/mg。目前,已经有20多种海藻酸分解菌的海藻酸裂解酶基因被克隆,其中大部分基因已成功地进行了异源表达[12]。【本研究切入点】本实验室从北部湾海域分离筛选出一株Shewanella haliotis BP-1菌株(菌株BP-1),在该菌株的发酵液中分离纯化得到1种酶,该酶经基本的酶学性质分析,确定是外分泌的海藻酸裂解酶,分子量约为70 kDa,同时具有降解poly(M)和poly(G)的活性,酶的比活力高达40 420 U/mg(文献[13])。本研究在菌株BP-1的基因组注释结果中找出一个可能编码海藻酸裂解酶的基因Alg17S,并对其进行克隆表达以及重组酶的活性检测。【拟解决的关键问题】验证重组酶Alg17S具有降解海藻酸的生物活性,为阐明BP-1菌株利用海藻酸钠的代谢途径提供依据。
1 材料与方法
1.1菌株和质粒
海洋细菌菌株Shewanella haliotis BP-1从广西北海涠州岛采集到的腐烂马尾藻中筛选获得;实验所使用的细菌表达载体pET-11d、pET-27b购自上海捷瑞生物工程有限公司;pMD18-T载体购自TaKaRa公司;大肠杆菌Trans5α、BL21(DE3)购自北京全式金生物技术有限公司。
1.2酶与试剂
DNA聚合酶,DNA连接试剂盒购自TaKaRa公司;限制性内切酶购自NEB公司;琼脂糖凝胶回收试剂盒和质粒提取试剂盒均购自博日科技(Bioflux)公司;海藻酸钠为阿拉丁®(Aladdin®)品牌试剂,其它试剂均为国产或进口分析纯试剂;所得样品送往TaKaRa公司测序。
1.3培养基和抗生素
LB液体培养基(W/V):1%Tryptone、0.5% Yeast Extract和1% NaCl。抗生素氨苄青霉素(Ampicillin,Amp),贮液浓度100 mg/mL,终浓度50 μg/mL;抗生素卡那霉素(Kanamycin,Ka),贮液浓度50 mg/mL,终浓度25 μg/mL;异丙基-β-D-硫代吡喃半乳糖苷(IPTG)贮液浓度100 mmol/L,终浓度0.4 mmol/L。配制LB固体培养基时加入1.5%(W/V)的琼脂,抗生素则为液体培养基使用浓度的两倍。
1.4引物设计
根据菌株BP-1全基因组注释结果中可能编码海藻酸裂解酶基因的核酸序列,利用PrimerPremier 5.0软件设计两对PCR引物,Alg17S-F和Alg17S-R用于扩增海藻酸裂解酶完整的基因编码序列,△snAlg17S-F和△snAlg17S-R用于扩增不含信号肽的海藻酸裂解酶的基因编码序列。设计引物时分别在上游引物的5′端添加BspHⅠ酶切位点,下游引物的5′端添加BamHⅠ酶切位点,引物序列如下,酶切位点用下划线标出。
Alg17S-F:5′-GCGACTCATGAAAATACGATCCT-3′
Alg17S-R:5′-CGGGATCCGAATGTTCAGTCTT-3′
△snAlg17S-F:5′-GTTCATCATGAGCCCGTCA-
CTG-3′
△snAlg17S-R:5′-CGGGATCCGAATGTTCAGT-
CTT-3′
1.5基因组DNA的制备
菌株BP-1全基因组DNA的提取参照BioSpin Bacteria Genomic DNA Extraction Kit 说明书。用0.8%(W/V)的琼脂糖凝胶检测基因组DNA的大小。基因组DNA的浓度和纯度用Nanodrop法检测。
1.6海藻酸裂解酶基因的克隆与分析
以菌株BP-1基因组DNA为模板,利用Ex Taq DNA聚合酶PCR 扩增获得目的基因片段。PCR扩增程序:94℃预变性5 min;94℃变性1 min,54℃退火1 min,72℃延伸2 min 30 s,30个循环;72℃延伸10 min。海藻酸裂解酶基因的PCR产物经琼脂糖凝胶回收试剂盒回收纯化后与TA克隆质粒pMD18-T连接,转化大肠杆菌Trans 5α感受态细胞,提取阳性克隆的质粒送测序。利用NCBI(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)数据库在线分析基因的开放阅读框;使用软件DNASIS MAX Trial V3.0推导海藻酸裂解酶基因编码蛋白的氨基酸序列;通过Signalp-4.1(http://www.cbs.dtu.dk/services/SignalP/)预测分析海藻酸裂解酶的信号肽;使用ProtParam(http://web.expasy.org/protparam/)工具计算蛋白质的理论分子质量和理论等电点;依据蛋白质多序列比对的结果,采用MEGA 5.1构建Alg17S蛋白的系统进化树。
1.7海藻酸裂解酶基因的表达与纯化
将测序正确的重组质粒pMD18-T-Alg17S,pMD18-T-△snAlg17S和细菌表达载体pET-11d,pET-27b分别进行BspHⅠ和BamHⅠ酶切,酶切产物回收、连接构建得到相应的重组质粒pET-11d-Alg17S,pET-27b-△snAlg17S。阳性克隆子送TaKaRa公司测序。
将测序正确的重组质粒pET-11d-Alg17S,pET-27b-△snAlg17S转化大肠杆菌BL21(DE3),挑取阳性转化子接种至10 mL含相应抗生素的LB液体培养基中,37℃、200 r/min摇床震荡培养至OD600为0.6~0.8。按接种量为1%(V/V)将菌液接种至100 mL
加有相应抗生素的LB液体培养基中,37℃、200 r/min摇床震荡培养至OD600为0.8,向菌液中加入0.4 mL 100 mmol/L的IPTG使其终浓度为0.4 mmol/L,并将菌液转入20℃,200 r/min摇床诱导培养24 h。
蛋白分离纯化实验均在4℃下进行。将100 mL诱导菌液6 000 r/min离心10 min收集菌体,使用20 mmol/L,pH值7.5的磷酸缓冲液重悬后超声波细胞粉碎机裂解菌体;裂解的菌液经8 000 r/min,离心30 min后收集的上清液,即海藻酶裂解酶的粗酶液。蛋白粗酶液依次经过DEAE Sepharose FF柱(上海楷洋)和蛋白质截留分子量为10 kDa的超滤离心管(Millpore)进行分离纯化与浓缩。12% SDS-PAGE电泳分析蛋白纯化结果,Bradford方法[14]检测纯化的蛋白质浓度。
1.8海藻酸裂解酶活力定性测定
将获得的海藻酸裂解酶粗酶液点种于含0.2%(W/V)海藻酸钠的LB固体培养基上,37℃放置30 min,最后使用10%(W/V)氯代十六烷吡啶染色,观察重组酶Alg17S,△snAlg17S水解海藻酸钠透明圈产生的情况,同时以含有空载体的pET-11d/BL21(DE3)和pET-27b/BL21(DE3)重组菌作为阴性对照。
1.9海藻酸裂解酶活力定量测定
酶活性的测定参照Preiss方法[15]。取 0.1%(W/V)的海藻酸钠溶液(0.1 g海藻酸钠溶于100 mL ,pH 值7.5,0.1 mol/L的Tris-HCl缓冲液)2 mL,加入0.5 mL酶液,37℃水浴30 min后,沸水浴5 min终止反应,冷却至室温后测定反应体系在235 nm处的光吸收值。在此条件下,每分钟光吸收值增加0.001定义为一个酶活力单位(U)。
2 结果与分析
2.1编码海藻酸裂解酶基因序列分析与克隆
菌株BP-1全基因组中仅存在一个可能编码海藻酸裂解酶的基因Alg17S, 核酸序列见图1。 分析结果显示,该基因包含一个大小为2 157 bp的开放阅读框,编码含718个氨基酸残基的蛋白质海藻酸裂解酶Alg17S, 其理论分子量和理论等电点(pI)分别为79 726 Da和5.72,其中N端信号肽大小为26个氨基酸(图1下划线标出)。在CAZY数据库中,海藻酸裂解酶被归入多糖裂解酶(polysaccharide lyase,PL)蛋白家族[16]。NCBI在线分析蛋白质保守结构域发现,Alg17S蛋白与其他PL-17家族的蛋白一样存在两个保守的蛋白结构域:一个是蛋白质N端的海藻酸裂解酶超级家族结构域;一个是蛋白质C端的肝素酶(heparinas)Ⅱ/Ⅲ家族蛋白域[17]。同时,蛋白质多序列比对分析表明Alg17S蛋白存在PL17家族蛋白保守的2个可能的催化位点Y235和Y445(络氨酸 Try,Y)和3个底物相互作用位点D420(天冬氨酸Asp,D)、H410和H459(组氨酸His,H)[17]。如图2所示,Alg17S与S.degradans 2-40的海藻酸裂解酶Alg17C的进化距离最近,氨基酸序列的相似性为52%。

Y为Alg17S假定的催化位点;H、D为假定的底物相互作用位点
Y indicates the proposed catalytic sites;H and D substrate interacting sites for Alg17S
图1S.haliotis BP-1海藻酸裂解酶基因的核酸序列和推导的氨基酸序列
Fig.1Nucleotide and deduced amino acid sequence of the alginate lyase gene from S.haliotis BP-1
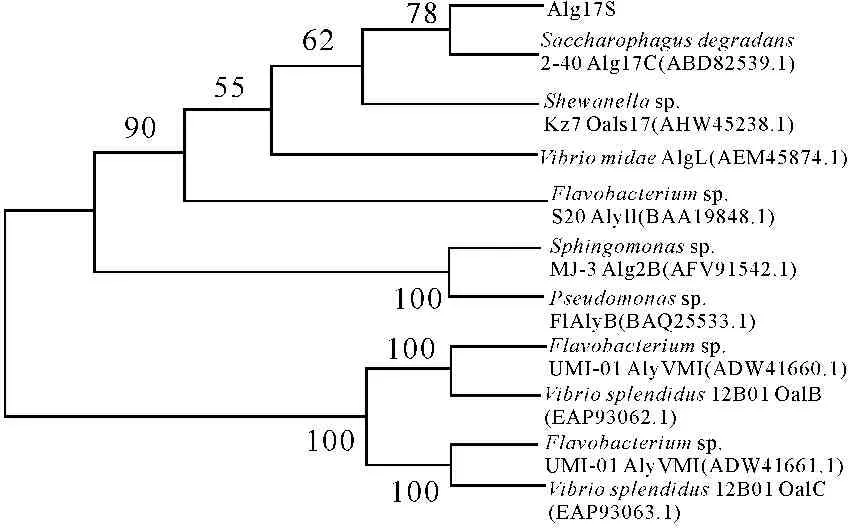
图2Alg17S与其它PL17家族蛋白的系统进化树
Fig.2Phylogenetic tree of Alg17S and other PL17 family
以菌株BP-1的基因组DNA为模板,使用引物Alg17S-F/Alg17S-R和△snAlg17S-F/△snAlg17S-R进行PCR扩增,分别得到大小为2 157 bp和2 085 bp的DNA片段(图3)。TA克隆、测序结果表明,Alg17S核酸序列和△snAlg17S核酸序列分别与全基因组中可能编码海藻酸裂解酶的核酸序列和去掉信号肽的海藻酸裂解酶的序列相似性均为100%。
2.2海藻酸裂解酶基因表达及酶活力的定性测定
成功构建的重组质粒pET-11d-Alg17S和pET-27b-△snAlg17S在大肠杆菌BL21(DE3)中表达并获得海藻酸裂解酶粗酶,该酶的大小约为70 kDa,与温顺华[13]从原始菌株BP-1中分离纯化得到的海藻酸裂解酶的大小相近。如图4a~b所示,重组菌pET-11d-Alg17S/BL21(DE3)和pET-27b-△snAlg17S/BL21(DE3)的粗酶液在含海藻酸钠的LB固体平板上都可以产生透明圈,说明BP-1菌株的海藻酸裂解酶无论有无信号肽都可以成功表达具有活性的蛋白酶;但是△snAlg17S蛋白水解海藻酸钠形成的透明圈亮度明显要比Alg17S的亮,这可能是前者重组酶的比活力要比后者高得多。同时pET-11d/BL21(DE3)(图4c)和pET-27b/BL21(DE3)(图4d)阴性对照组均没有透明圈生成。
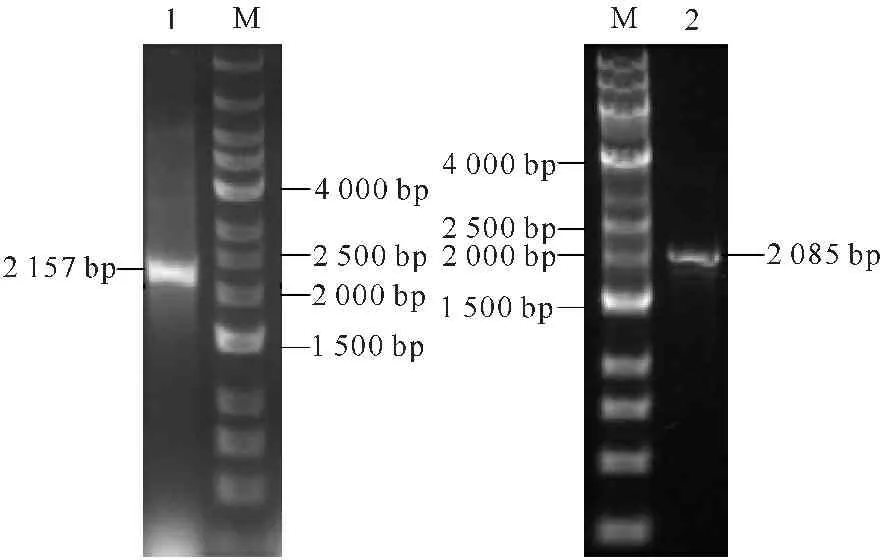
1:含信号肽的海藻酸裂解酶基因的PCR产物;2:不含信号肽的海藻酸裂解酶基因的PCR产物;M:1 kb DNA ladder
1:The PCR products of alginate lyase gene containing signal peptide;2:Alginate lyase gene PCR products without the signal peptide;M:1 kb DNA ladder
图3菌株BP-1海藻酸裂解酶基因PCR产物
Fig.3The alginate lyase gene PCR products from BP-1 strain

(a)pET-11d-Alg17S/BL21(DE3);(b)pET-27b-△snAlg17S/BL21(DE3);(c)pET-11d/BL21(DE3);(d)pET-27b/BL21(DE3)
图4重组菌产海藻酸裂解酶活性的定性测定
Fig.4Detection of alginate lyase activities of recombinant strain
2.3海藻酸裂解酶的纯化及酶活力的定量测定
如图5所示,纯化的蛋白酶分子量与从S.haliotis BP-1菌株中分离纯化得到的酶蛋白一样约为70 kDa。由表1可见,不含信号肽的海藻酸裂解酶△snAlg17S活性要比含信号肽的Alg17S高,或许信号肽的存在影响了蛋白酶的高级结构从而影响了酶对底物的结合催化活性。△snAlg17S纯蛋白比初始粗酶液纯化了30.3倍,酶的比活力高达9 635 U/mg,约为Alg17S的4.7倍,但是无论是△snAlg17S还是Alg17S,它们对海藻酸钠降解的最大酶比活力都远远高于Agrobacterium tumefaciens C58的AtU3025(20.5 U/mg)、Sphingomonas sp.A1的A1-IV(17.2 U/mg)和Pseudomonas sp.QD3的海藻酸裂解酶(173.4 U/mg)[18-19],这些结果表明菌株BP-1的海藻酸裂解酶对海藻酸钠具有更高的亲和力与更有效的水解能力。
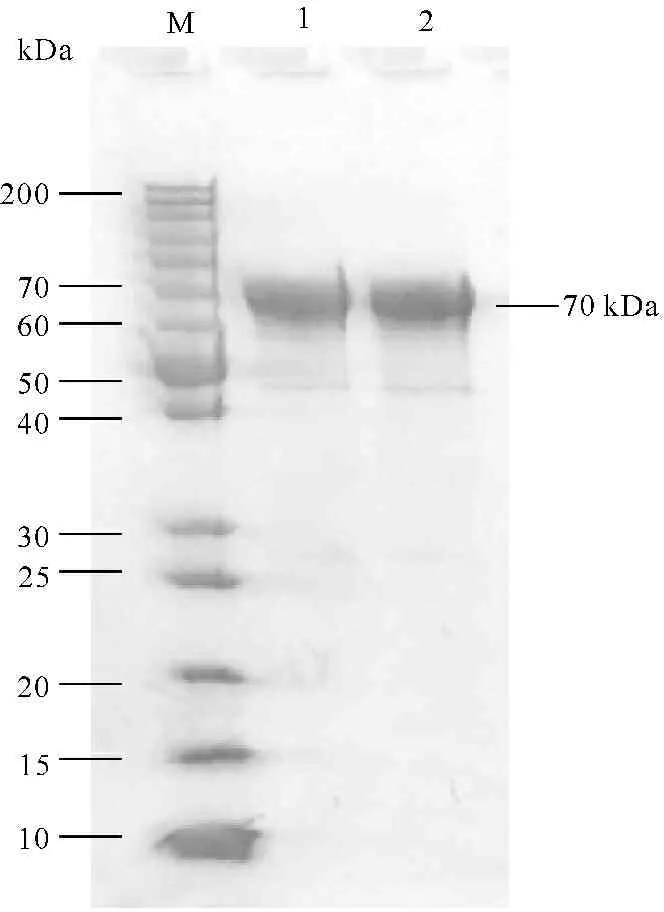
M:molecular weight markers;1:purified Alg17S;2:purified △snAlg17S
图5重组酶Alg17S、△snAlg17S的SDS-PAGE电泳
Fig.5SDS-PAGE of Alg17S and △snAlg17S
表1纯化的重组酶Alg17S和△snAlg17S的活性定量测定
Table 1The activity quantitative determination of purified recombinant enzyme Alg17S and △ snAlg17S

项目Item总蛋白Totalprotein(mg)总活力Totalactivity(U)比活力Specificactivity(U/mg)产量Yield(%)纯化倍数Purification(Fold)Alg17S粗酶液CrudeenzymesofAlg17S36.910670289100.01.0纯化的Alg17SPurificationofAlg17S0.241220603.97.1△snAlg17S粗酶液Crudeenzymesof△snAlg17S57.718331318100.01.0纯化的△snAlg17SPurificationof△snAlg17S0.21921963510.530.3
3 结论
从海洋细菌菌株S.haliotis BP-1中成功克隆的目的基因Alg17S、△snAlg17S的核酸序列和从其全基因组注释结果中找出的可能编码海藻酸裂解酶的核酸序列相似性为100%。海藻酸裂解酶归属于多糖裂解酶家族的PL17家族,分子量为79 726 Da。表达的海藻酸裂解酶重组蛋白Alg17S和△snAlg17S可以有效的水解海藻酸钠,且后者的水解能力明显高于前者。△snAlg17S具有较高的海藻酸降解酶活性,酶的比活力高达9 635 U/mg,约为Alg17S的4.7倍,为进一步研究海藻酸盐糖化和生物燃料的生产奠定了基础。
[1]HERNANDEZ-CARMONA G,MCHUGH D J,LOP-EZ-GUTIERREZ F.Pilot plant scale extraction of laginates from Macrocystis pyrifera[J].Apppl Phycol,2000,11:493-502.
[2]刘航,尹恒,张运红,等.褐藻胶寡糖生物活性研究进展[J].天然产物研究与开发,2012(S1):201-204.
LIU H,YIN H,ZHANG Y H,et al.Research progress on biological activities of alginate oligosaccharides[J].Nat Prod Res Dev,2012(S1):201-204.
[3]WONG T Y,PRESTON L A,SCHILLER N L.Alginate lyase:Review of major sources and enzyme characteristics,structure-function analysis,biological roles,and applications[J].Annual Review of Microbiology,2000,54:289-340.
[4]MURATA K,INOSE T,HISANO T,et al.Bacterial alginate lyase:Enzymology,genetics and application[J].Journal of Fermentation & Bioengineering,1993,76(93):427-437.
[5]TOMOO S,MIWA O,YOSHIO E.Novel alginate lyases from marine bacterium Alteromonas sp.strain H-4[J].Carbohydrate Research,1997,304(1):69-76.
[6]ZHEN Q Z,GUANG L Y,HUA S G,et al.Preparation and structure elucidation of alginate oligosaccharides degraded by alginate lyase from Vibro sp.510[J].Carbohydrate Research,2004,339(8):1475-1481.
[7]HUANG L,ZHOU J,XIAO L,et al.Characterization of a new alginate lyase from newly isolated Flavobacterium sp.S20[J].Journal of Industrial Microbiology & Biotechnology,2013,40(1):113-122.
[8]HENRISSAT B,COUTINHO P M,LEVASSEUR A,et al.CAZY Carbohydrate-Active Enzymes Database[EB/OL].[2014-04-15].http://www.cazy.org.
[9]SCHILLER N L,MONDAY S R,BOYD C M,et al.
Characterization of the Pseudomonas aeruginosa alginate lyase gene (algL):Cloning,sequencing,and expression in Escherichia coli[J].Journal of Bacteriology,1993,175(15):4780-4789.
[10]UCHIMURA K,MIYAZAKI M,NOGI Y,et al.Cloning and sequencing of alginate lyase genes from deep-sea strains of Vibrio and Agarivorans and characterization of a new Vibrio enzyme[J].Marine Biotechnology,2010,12(5):526-533.
[11]LI S,YANG X,ZHANG L,et al.Cloning,expression,and characterization of a cold-adapted and surfactant-stable alginate lyase from marine bacterium Agarivorans sp.L11[J].Journal of Microbiology & Biotechnology,2015,25(5):681-686.
[12]李锋,温顺华,黄庶冰,等.响应面法优化海带酸水解预处理工艺[J].可再生能源,2012,30(11):93-103.
LI F,WEN S H,HUANG S B,et al.Optimized pretreatmet process of acid hydrolysis Laminaria japonica by response surface methodology[J].Renewable Energy Resources,2012,30(11):93-103.
[13]温顺华.褐藻糖化预处理过程中褐藻胶裂解酶的作用研究[D].昆明:昆明理工大学,2013.
WEN S H.Study on Saccharification of Brown Algae by Alginate Lyase[D].Kunming: Kunming University of Science and Technology,2013.
[14]BRADFORD M M.A rapid method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].Analytical Biochemistry,1976,72(S1/S2):248-254.
[15]PREISS J,ASHWELL G.Alginic acid metabolism in bacteria.I.Enzymatic formation of unsaturated oligosaccharides and 4-deoxy-L-erythro-5-hexoseulose uronic acid[J].Journal of Biological Chemistry,1962,237(2):309-316.
[16]CANTAREL B L,COUTINHO P M,RANCUREE C,et al.The carbohydrate-active enzymes database (CAZY):An expert resource for glycogenomics[J].Nucleic Acids Research,2009,37(suppl 1):233-238.
[17]WANG L,LI S,YU W,et al.Cloning,overexpression and characterization of a new oligoalginate lyase from a marine bacterium,Shewanella sp.[J].Biotechnology Letters,2014,37(3):665-671.
[18]PARK H H,KAM N,LEE E Y,et al.Cloning and characterization of a novel oligoalginate lyase from a newly isolated bacterium Sphingomonas sp.MJ-3[J].Marine Biotechnology,2012,14(2):189-202.
[19]SAKATOKU A,TANAKA D,NAKAMURA S.Purification and characterization of an alkaliphilic alginate lyase AlgMytC from Saccharophagus sp.Myt-1[J].Journal of Microbiology & Biotechnology,2013,23(6):872-877.
(责任编辑:竺利波)
Gene Cloning and Expression of Alginate Lyase from Shewanella haliotis BP-1
HUANG Guiyuan1,WEN Shunhua2,LI Feng3,LU Mingqian1,WANG Qiaozhen1,LIAO Wei4,HUANG Shushi1
(1.Lab of Biophysics,Guangxi Academy of Sciences,Nanning,Guangxi,530007,China;2.Xiamen Innodx Biotech Co.Ltd.,Xiamen,Fujian,361003,China;3.School of Chemistry and Chemical Engineering,Qiannan Normal College for Nationalities,Duyun,Guizhou,558000,China;4.Guangxi Vocational and Technical College,Nanning,Guangxi,530226,China)
【Objective】Alginate lyase in Shewanella haliotis BP-1 strains was studied illustrate its biological activity of degrading alginate.【Methods】The gene cloning technology and the Escherichia coli heterologous expression technology were applied to overexpress the alginate lyase;And the enzyme activity was analyzed after the crude enzyme was separated and purified by DEAE Sepharose FF chromatography.【Results】The alginate lyase gene Alg17S,with a size of 2 157 bp,was cloned from S.haliotis BP-1 strain genomic DNA and encoded an alginate lyase Alg17S,which belonged to polysaccharide lyase(PL)17 family and had a size of 79 726 Da protein(including an N-terminal signal peptide of 26 amino acid signal peptide).Alg17S showed high sequence identity of 52% with PL-17 protein sequence Alg17C from Saccharophagus degradans 2-40.Both the purified recombinase Alg17S and the △snAlg17S(without the N-terminal signal peptide of 26 amino acids)can degrade alginate,but the enzymatic activity of △snAlg17S revealed a specific activity of 9 635 U/mg,which was more efficient than Alg17S.【Conclusion】The recombinant alginate lyase △snAlg17S that has both high-level expression and high enzymatic activity could be a potential enzyme for further researching on the alginate saccharification and the biofuels production.
alginate lyase,cloning and expression,enzyme activity determination,Shewanella haliotis BP-1
2016-04-28
2016-06-17
黄桂媛(1986-),女,研究实习员,主要从事分子生物学技术研究。
Q74
A
1005-9164(2016)03-0228-06
*国家自然科学基金项目(31560017),广西自然科学重点基金项目(2014GXNSFDA118012)和贵州省教育厅自然科学研究重点项目(黔教合KY[2014]286)资助。
**通讯作者:黄庶识(1964-),男,研究员,主要从事应用微生物学分子光谱研究,E-mail:hshushi@gxas.cn。
广西科学Guangxi Sciences 2016,23(3):228~233
网络优先数字出版时间:2016-07-13【DOI】10.13656/j.cnki.gxkx.20160713.003
网络优先数字出版地址:http://www.cnki.net/kcms/detail/45.1206.G3.20160713.0857.006.html

