南移池塘养殖仿刺参肠道菌群结构分析
杨求华,陆 振,林 琪,黄瑞芳,何丽斌,葛 辉,周 宸*,杜 虹*
(1.福建省水产研究所,福建省海洋生物增养殖与高值化利用重点实验室,福建 厦门 361013;2.汕头大学生物系,广东 汕头 515063)
南移池塘养殖仿刺参肠道菌群结构分析
杨求华1,陆振2,林琪1,黄瑞芳1,何丽斌1,葛辉1,周宸1*,杜虹2*
(1.福建省水产研究所,福建省海洋生物增养殖与高值化利用重点实验室,福建 厦门 361013;2.汕头大学生物系,广东 汕头 515063)
本文基于MiSeq高通量测序技术对南移福建池塘养殖仿刺参(Apostichopusjaponicus)肠道菌落结构进行分析。结果表明,仿刺参前肠(1C_1)、中肠(1C_2)和后肠(1C_3)测得的分类操作单元(OTUs)分别为424、444和414;三组样品的菌群物种丰度没有较大差别,但优势菌种存在较大差异;优势菌群方面,后肠的优势菌群为Haliea属和乳球菌属(Lactococcus)(分别占后肠总菌数的11.97%和10.37%),而前肠和中肠的优势菌群均是乳球菌属和芽孢杆菌属(Bacillus),两者在前肠菌落中的占比分别为27.91%和6.08%,在中肠中的占比分别为33.24%和6.97%。在重复性方面,三组样品的菌落组成都有重叠,重叠率为74.35%,其中前肠与中肠相似度较高,菌株种类有90%以上重叠。本文研究为仿刺参肠道益生菌的开发利用提供基础资料。
MiSeq高通量测序;仿刺参;池塘养殖;肠道菌群
仿刺参(Apostichopusjaponicus),隶属于棘皮动物门、海参纲、刺参科、刺参属,主要分布在西北太平洋沿岸、日本海、朝鲜半岛南部以及中国的黄海、渤海等地[1-3]。我国海参共有140余种,其中以仿刺参的品质最好,位于连云港海州湾的前三岛是仿刺参自然分布的最南界[4]。不过,自2003年起仿刺参的养殖就逐步扩展到福建等南方省份,“北参南养”的成功试验带动了福建省仿刺参养殖产业的蓬勃发展[5],每年的十一月到来年的三月是福建省的仿刺参养殖期[6-7]。仿刺参的养殖不仅带来了较好的经济效益,同时也存在一定的养殖风险。随着养殖密度和养殖规模的扩大,仿刺参的病害发生日益严重,研究结果表明,塔式弧菌(Vibriotubiashii)、灿烂弧菌(V.splendidus)等病原会引起仿刺参患上“腐皮综合征”等疾病[5-6]。
肠道菌群不仅是肠道的重要组成部分,还影响着动物的免疫调节、代谢吸收等生命机理[8-10],对肠道菌群结构的研究将为提高养殖产量、减少疾病发生提供基础参考数据。现有的肠道菌群结构分析方法包括传统分离培养法、变性梯度凝胶电泳(Denatured gradient gel electrophoresis,DGGE)和随机扩增引物多态性分析(Random amplified polymorphim DNA,RAPD)等[11],但这些方法存在一定的局限性。近年来,随着测序手段的不断发展,为了克服多数细菌不可培养和基因组多样性的问题,高通量测序技术应运而生,现已被广泛应用于水产动物肠道菌群结构分析中。基于MiSeq高通量测序技术克服了传统方法的局限性,应用该技术对环境微生物的菌群结构分析均获得了较好的实验结果[12-19]。本文基于微生物16S rRNA的MiSeq高通量测序技术对福建省池塘养殖仿刺参的肠道菌群结构进行分析,为南移养殖仿刺参肠道益生菌的开发提供基础资料。
1 材料和方法
1.1材料、仪器
实验用仿刺参采自福建省莆田市城关镇某池塘养殖场,选择体质健壮体、体表颜色正常、个头大小一致、体表完好的个体用于实验研究,采样时间为2015年1月29日。
超微量分光光度计(Nano drop 2000;美国Thermo Scientific公司)用于DNA浓度测定,细菌DNA提取试剂盒(北京艾德莱生物科技有限公司)用于肠道菌群DNA提取。
1.2样品采集方法
选取大小一致的仿刺参10头,在无菌环境下,用75%酒精冲洗仿刺参体表2~3遍,用剪刀剪开仿刺参体腔,取出肠道,用无菌生理盐水(0.9%)冲洗肠道外壁,取出内含物;将采集的前肠(1C_1)、中肠(1C_2)、后肠(1C_3)内含物混合分别置于无菌EP管中称重;在无菌研钵内充分研磨,冲洗研钵用5 mL无菌生理盐水从而得到仿刺参肠道内含物匀浆[18]。
1.3MiSeq高通量测序流程
正向引物338F 5′-ACTCCTACGGGAGGCAGCA-3′;
反向引物806R 5′-GGACTACHVGGGTWTCTAAT-3′。
先PCR扩增,检测方法采用琼脂糖凝胶电泳进行,切胶回收产物按照Illumina公司标准流程测序。
2 结果
2.1池塘养殖仿刺参肠道提取DNA质量
提取仿刺参前肠(1C_1)、中肠(1C_2)和后肠(1C_3)的肠道内含物细菌基因组DNA,琼脂糖凝胶电泳检测结果显示,提取的DNA产物浓度符合MiSeq高通量测序的要求(表1)。

表1 仿刺参肠道内容物基因组DNA质量检测结果
2.2样品序列数目
三组样品测得的序列条数分别为39 620(1C_1)、32 700(1C_2)和38 895(1C_3),序列的平均长度为450 bp左右,符合高通量测序要求(表2)。
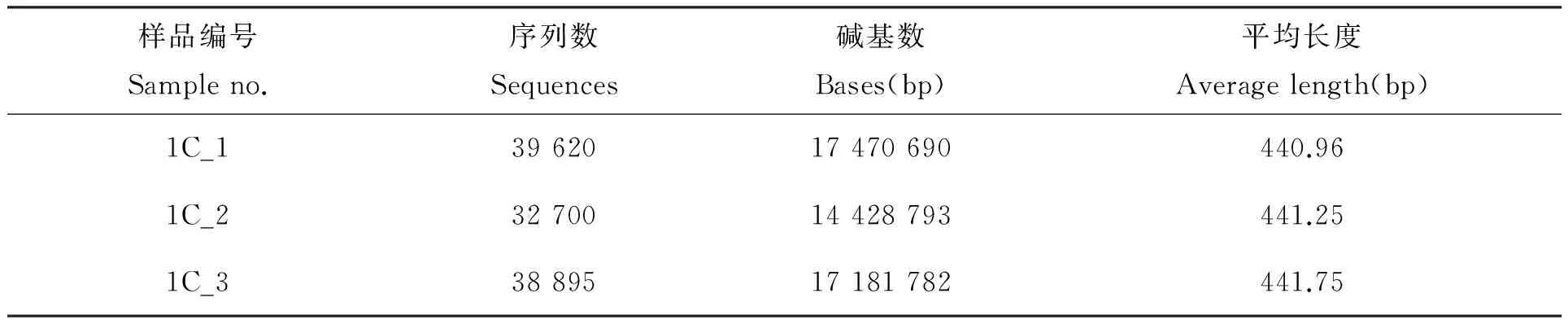
表2 样品序列数统计表
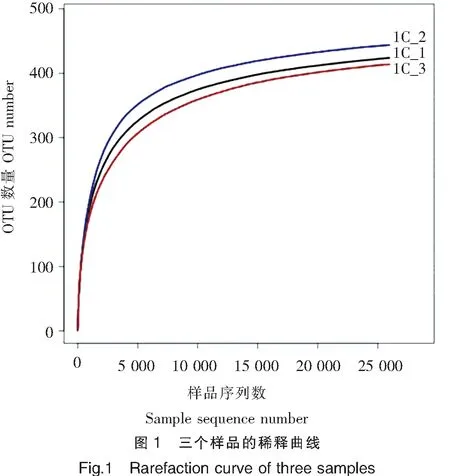
2.3稀释性曲线(Rarefaction curve)
样品稀释性曲线的总体趋势为:在序列数小于10 000条时,操作分类单元[OTU:Operational Taxonomic Units是在系统发生学或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系、属、种、分组等)设置的同一标志]的数量迅速增加;随序列数的增加,OTU数量增加速度减缓;当序列数达到10 000~25 000时趋于平台期。本文的样品在最后都趋于平台期(图1),说明三组样品的数据可靠,测序数量接近饱和,通过对样品数据进行抽平分析,三组样品的取样条数为25 943。
2.4细菌群落结构分析
2.4.1南移池塘养殖仿刺参前、中、后肠肠道菌群结构多样性指数分析
如表3中所示,三组样品的覆盖率均在99%以上,表明数据测序完整;各组样品的OTU数量都在430左右;三组样品(1C_1、1C_2、1C_3)的丰富度指数和多样性指数差异不大,其中丰富度指数(Chao/Ace)分别为457/445、474/466、438/436,多样性指数(Simpson/Shannon)分别为0.065/4.28、0.087 7/4.14和0.027/1.24。

表3 三个样品多样性指数
注:Chao 是用Chao1算法估计样品中所含OTU数目的指数,Chao1在生态学中常用来估计物种总数,由Chao(1984)最早提出。Ace:用来估计群落中OTU数目的指数,由Chao提出,是生态学中估计物种总数的常用指数之一,与Chao 1的算法不同。Simpson:用来估算样品中微生物多样性指数之一,由Edward Hugh Simpson(1949)提出,在生态学中常用来定量描述一个区域的生物多样性;Simpson指数值越大,说明群落多样性越低。Shannon:用来估算样品中微生物多样性指数之一,它与Simpson多样性指数常用于反映alpha多样性指数;Shannon值越大,说明群落多样性越高。
2.4.2池塘养殖仿刺参前、中、后肠肠道群落结构分析
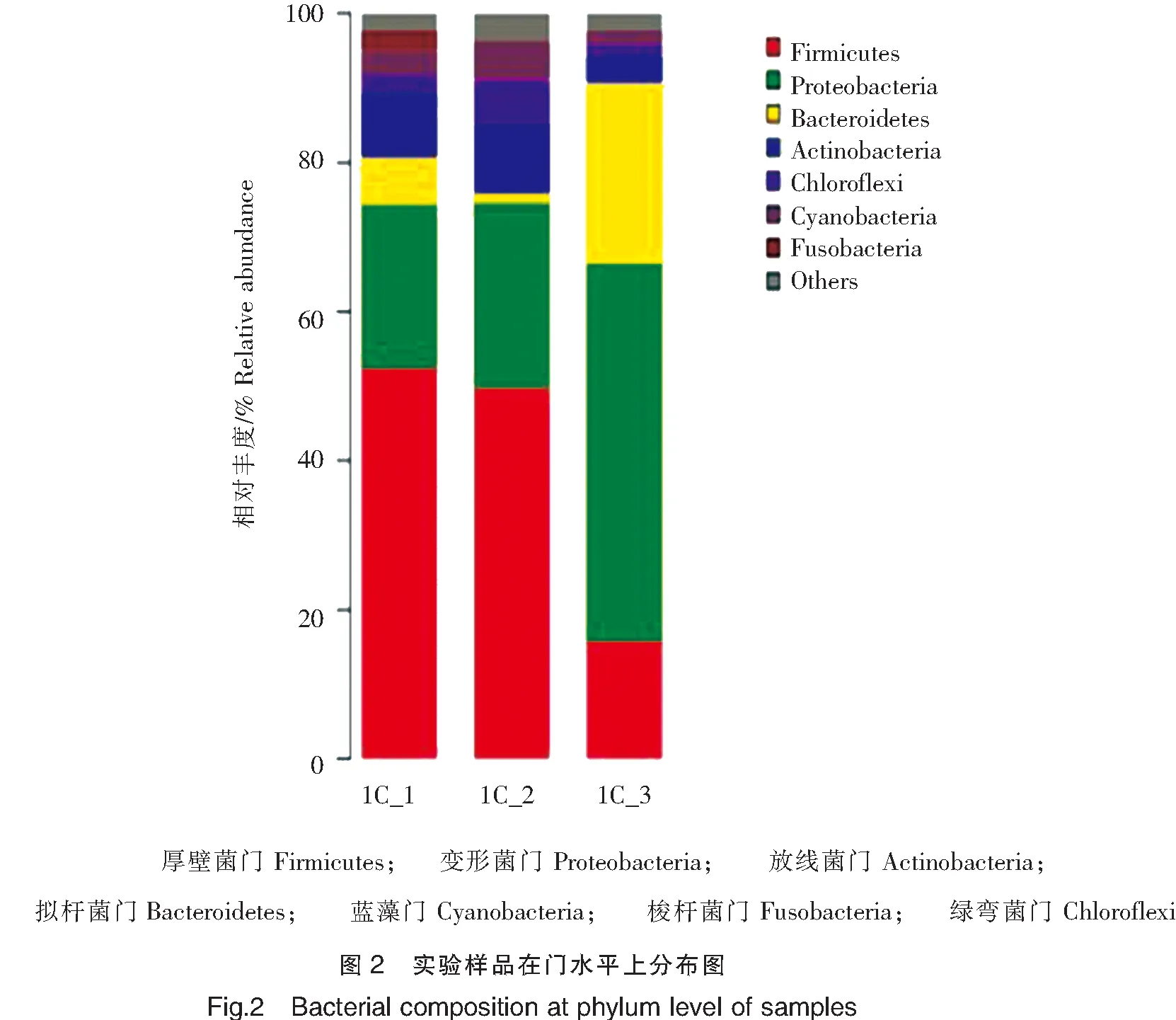
表4 实验样品在门水平上的丰度

分类Taxonomy丰度/% Abundance1C_11C_21C_3厚壁菌门Firmicutes52.5149.9816.24变形菌门Proteobacteria22.0324.4550.16放线菌门Actinobacteria8.469.343.48拟杆菌门Bacteroidetes6.291.5324.28蓝藻门Cyanobacteria3.284.941.72梭杆菌门Fusobacteria2.630.200.00绿弯菌门Chloroflexi2.465.891.69疣微菌门Verrucomicrobia0.590.930.51酸杆菌门Acidobacteria0.160.660.20衣原体门Chlamydiae0.120.580.05硝化螺旋菌门Nitrospirae0.120.010.00浮霉菌门Planctomycetes0.040.050.84软壁菌门Tenericutes0.030.010.00芽单胞菌门Gemmatimonadetes0.020.150.14螺旋菌门Spirochaetae0.000.000.00TM70.770.660.43SR10.290.010.00OD10.040.090.07TM60.110.470.17SHA-1090.040.020.00CKC40.000.040.02
在门的水平上(图2、表4),南移池塘养殖仿刺参的前肠、中肠、后肠肠道菌群主要包括21个细菌门,分别为厚壁菌门、软壁菌门、变形菌门、绿弯菌门、放线菌门、拟杆菌门、酸杆菌门、蓝藻门、疣微菌门、梭杆菌门、酸杆菌门、衣原体门、硝化螺旋菌门、浮霉菌门、软壁菌门、芽单胞菌门等。其中厚壁菌门和变形菌门在前肠、中肠中的占比较高,二者在前肠中占细菌总数的百分比分别为52.51%和22.03%,在中肠中占的百分比分别为49.98%和24.45%;而后肠则以变形菌门、拟杆菌门和厚壁菌门含量最高,分别占细菌总数的50.16%、24.28%和16.24%。此外,在中肠内没有发现螺旋菌门,后肠中没有发现梭杆菌门、软壁菌门和硝化螺旋菌门。

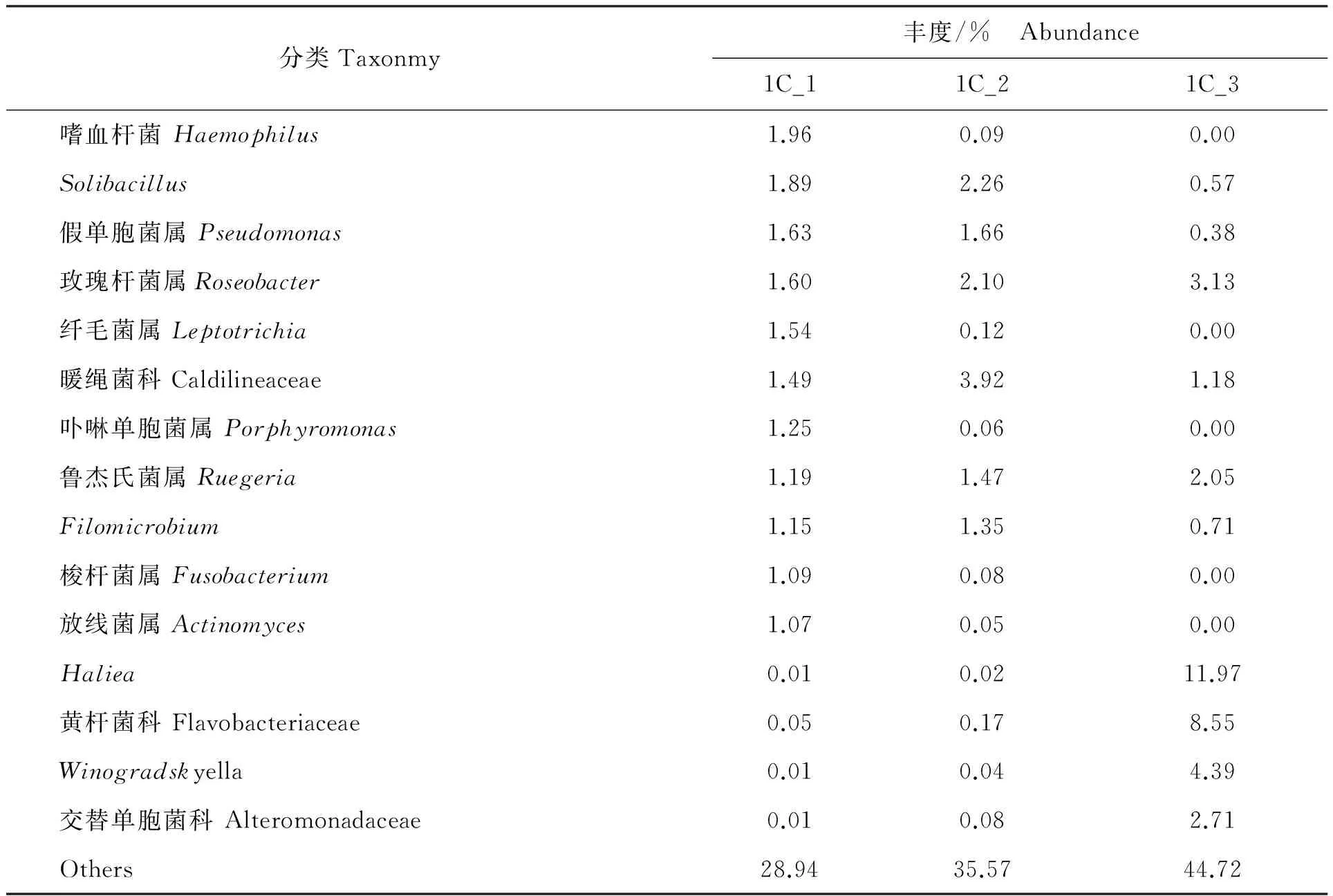
表5 实验样品在科、属水平上的丰度

分类Taxonmy丰度/% Abundance1C_11C_21C_3乳球菌属Lactococcus27.9133.2410.37芽孢杆菌属Bacillus6.086.973.20链球菌属Streptococcus4.051.190.24普氏菌属Prevotella3.890.280.00节杆菌属Arthrobacter2.332.700.27罗氏菌属Rothia2.260.080.00奈瑟氏菌属Neisseria2.230.120.00红杆菌科Rhodobacteraceae2.202.153.93韦永氏球菌属Veillonella2.090.120.00蓝细菌Cyanobacteria2.084.091.61
续表5
在属的水平上(图3,表5),南移池塘养殖仿刺参前肠、中肠、后肠中共检测到268个细菌种属,主要包括乳球菌属、Haliea、链球菌属、芽孢杆菌属、黄杆菌科等。其中乳球菌属在前肠和中肠中所占比例最高,分别为27.91%和33.24%;而后肠中以Haliea属和乳球菌属的含量最高,占后肠细菌总数的比例分别为11.97%和10.37%。
2.4.3Venn图和样本聚类树


将所得结果用作图软件和层类聚类分析做Venn图(图4)和样品聚类树(图5)。结果表明,在重复性方面,三组样品的菌群组成都有重叠,重叠率为74.35%,其中前肠和中肠的菌群相似度较高,菌群种类上有90%以上的重叠。而特异性菌株方面,在前肠、中肠和后肠中都有发现特有菌株,数量分别为14、3、55,以后肠中发现的特有菌株数量最多,其功能有待进一步研究。从样品聚类树可以看出前肠和中肠的菌群结构具有较高的相似度,而后肠与前、中肠的菌群结构间存在一定的差异性。
3 讨论
本研究参考高菲[20]等人的方法选取多个仿刺参的前肠、中肠和后肠,分别混合装在灭菌的冻存管内混合均匀。高通量结果显示,福建南移池塘养殖仿刺参的肠道菌群总体以厚壁菌门为主(主要为芽孢杆菌属和乳球菌属),其次为拟杆菌门和变形菌门。乳球菌属、芽孢杆菌属和链球菌属等厚壁菌门菌株主要存在于仿刺参的前肠和中肠;拟杆菌门中的黄杆菌属和Winogradskyella属在后肠中的占比(所占比例分别为8.55%和4.39%)明显高于前肠和中肠,而同一门中的普氏菌属则在前肠中占比较高(占比为3.85%);变形菌门的红杆菌科和Haliea属在后肠中的占比分别为3.39%和11.97%,尤其是Haliea属在后肠中占比较高。有研究结果表明,厚壁菌门的细菌含有多种与淀粉降解酶有关基因,当厚壁菌门在肠道内占优势时,有助于宿主分解营养物质为宿主提供能量[21-23]。本研究中,前肠和中肠中的厚壁菌门占有较高优势,分析此类菌株有助于宿主对营养物质的分解和吸收;而后肠中多为仿刺参的消化物,是营养物质经消化分解后的堆积场所,其优势菌株变为变形菌门和拟杆菌门为主。
在本研究中,仿刺参前肠、中肠和后肠的多样性差异不大,仅在菌群结构的组成方面存在差异。而高菲等[20]采用PCR-DGGE技术对北方养殖仿刺参肠道内含物的群落组成研究结果显示,仿刺参前肠内含物的微生物多样性最低,后肠最高,中肠其次;造成差异的原因也许是由于不同的研究手段及不同地域生长环境。有资料指出,海参的前肠是用酶对事物进行消化的场所,后肠的主要作用为对营养物质的吸收[24-25]。因此,前、中、后肠中种类繁多的细菌群落组成是与仿刺参肠道的具体功能密切相关。
本文采用高通量测序技术与微生物16S rDNA V3-V4可变区序列分析技术相结合[26-27],分析福建省南移的池塘养殖仿刺参肠道菌群结构组成。结果表明,MiSeq高通量测序技术能快速、准确地分析肠道中的细菌群落结构,克服了细菌常规分离培养的限制。肠道的微生物菌群是肠道的重要组成部分,仿刺参的生理状态不同能够影响肠道菌群的变化,相对应的肠道菌群结构变化又会反过来影响仿刺参的生理状态;仿刺参的肠道菌群组成会受外部环境的影响,从而影响仿刺参的吸收、消化等功能。本研究首次利用高通量测序技术揭示了南移养殖仿刺参肠道菌群结构的多样性和丰度,且检测到的细菌多数是传统方法未检测到的,对于这些细菌的分离培养方法以及其与仿刺参的生理功能之间的关系还有待进一步研究。本研究将为仿刺参的健康养殖和疾病防控等方面的研究提供参考资料。
[1]廖玉麟.中国动物志:棘皮动物门.海参纲[M].北京:科学出版社,1997:48-152.
[2]张春云,王印庚,荣小军,等.国内外海参自然资源、养殖状况及存在问题[J].海洋水产研究,2004,25(3):89-97.
[3]Bordbar S,Anwar F,Saari N.High-value components and bioactives from sea cucumbers for functional foods:a review[J].Mar Drugs,2011,9(10):1761-805.
[4]Kanno M,Li Q,Kijima A.Isolation and characterization of twenty microsatellite loci in Japanese sea cucumber(Stichopusjaponicus)[J].Mar Biotechnol,2005,7(3):179-183.
[5]杨求华,葛辉,方旅平,等.池塘养殖刺参病原菌塔式弧菌的分离与鉴定[J].南方水产科学,2014,10(4):45-51.
[6]葛辉,何丽斌,林琪,等.福建海区网箱养殖刺参“腐皮综合征”[J].福建水产,2012,34(2):105-110.
[7]李正良.刺参南方池塘养殖技术[J].福建水产,2011,33(5):77-80.
[8]Mackie R I,White B A.Recent advances in rumen microbial ecology and metabolism:potential impact on nutrient output[J].J Dairy Sci,1990,73(10):2971-2995.
[9]Hegarty R S,Goopy J P,Herd R M,et al.Cattle selected for lower residual feed intake have reduced daily methane production[J].Journal of Animal Science,2007,85(6):1479-1486.
[10]Zhou M,Hernandez-sanabria E,Guan L L.Assessment of the microbial ecology of ruminal methanogens in cattle with different feed efficiencies[J].Appl Environ Microbiol.,2009,75(20):6524-6533.
[11]李祎,郑伟,郑天凌.海洋微生物多样性及其分子生态学研究进展[J].微生物学通报,2013,40(4):655-668.
[12]Quail M A,Kozarewa I,Smith F,et al.A large genome center’s improvements to the Illumina sequencing system[J].Nature Methods,2008,5(12):1005-1010.
[13]Meyer M,Stenzel U,Hofreiter M.Parallel tagged sequencing on the 454 platform[J].Nature Protocols,2008,3(2):267-278.
[14]Mardis E R.The impact of next-generation sequencing technology on genetics[J].Trends Genetics,2008,24(3):133-141.
[15]秦楠,栗东芳,杨瑞馥.高通量测序技术及其在微生物学研究中的应用[J].微生物学报,2011,51(4):445-457.
[16]Zwolinski M D.DNA sequencing:Strategies for soil microbiology[J].Soil Science Society of America Journal,2007,71(2):592-600.
[17]He J Z H,Hughes J.Molecular Bacterial diversity of a forest soil under residue management regimes in subtropical Australia[J].FEMS Microbiology Ecology,2006,55(1):38-47.
[18]黄诗婷,陈荣元,刘克韶,等.四个拟穴青蟹养殖池水环境中细菌群落结构的比较研究[J].应用海洋学学报,2012,31(3):387-395
[19]杨章武,杨铿,葛辉,等.基于宏基因组测序技术分析凡纳滨对虾育苗中生物絮团细菌群落结构[J].福建水产,2015,37(2):91-97.
[20]高菲,孙慧玲,许强,等.刺参消化道内含物细菌群落组成的PCR-DGGE分析[J].中国水产科学,2010,17(4):671-680.
[21]Bates S T,Clemente J C,Flores G E,et al.Global biogeography of highly diverse protistan communities in soil[J].The ISME Journal,2013,7(3):652-659.
[22]Fouts D E,Szpakowski S,Purushe J,et al.Next Generation Sequencing to Define Prokaryotic and Fungal Diversity in the Bovine Rumen[J].PLoS ONE,2012,7(11):e48289.
[23]Justin L S,Jian X,Douglas D L,et al.Glycan Foraging in Vivo by an Intestine- Adapted Bacterial Symbiont[J].Science,2005,307(5717):1955-1959.
[24]Filiminova G F,Tokin I B.Structural and functional peculiarities of the digestive system ofCucumariafrondosa(Echinodermata:Holothuroidea)[J].Mar Biol,1980,60(3):9-16.
[25]Khripounoff A,Sibuet M.The nutrition of abyssal echinoderms.I.Feeding of holothuroids[J].Mar Biol,1980,60(2):17-26.
[26]Shi P,JIA S,Zhang T,et al.Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water[J].Water Reasearch,2013,47(1):111-120.
[27]He J Z,Xu Z H,Hughes J.Analyses of soil fungal communities in adjacent natural forest and hoop pine plantation ecosystems of subtropical Australia using molecular approaches based on 18S rRNA genes[J].FEMS Microbiology Letters,2005,247(1):91-100.
Bacterial component analysis of the gut of South Apostichopus japonicus cultured in pond
YANG Qiuhua1,LU Zhen2,LIN Qi1,HUANG Ruifang1,HE Libin1,GE Hui1,ZHOU Chen1*,DU Hong2*
(1.Key Laboratory of Cultivation and High-value Utilization of Marine Organisms in Fujian Province,Fisheries Research Institute of Fujian,Xiamen 361013,China;2.Department of Biology,Shantou University,Shantou 515063,China)
The gut microbiota is essential in shaping many of its host’s functional attributes.The gut microbiota is necessary for the proper physiological development of the gut and for the animal’s ability to digest and convert plant mass into food products.Thus based on high throughput sequencing technology MiSeq,the research aims at analyzing the structure of intestinal bacterial colonies ofApostichopusjaponicuscultured in pond polyculture of Fujian.The results showed that the units of classification operation(OTUs)of the three samples were 424,444 and 414,respectively.The dominant strains were quite different among these samples.The hindgut dominant bacteria wereHaliea(11.97%)andLactococcus(10.37%),while both foregut and midgut’s dominance wereLactococcus andBacillus.In addition, the percentage ofLactococcus in foregut and midgut were 27.91% and 6.08%,respectively,as forBacilluswere 33.24% and 6.97%.In repeatability,colonies consisting of three samples had overlapping(74.35%),which foregut and midgut had high similarity,the strain types more than 90% overlap.Therefore,the study aimed at providing basic information for the development and utilization ofApostichopusjaponicuintestinal flora.
MiSeq high-throughput sequencing;Apostichopusjaponicus;pond culture;gut microbiota
2016-03-17
福建省科技计划项目(2014R1003-1);福建省海洋高新产业发展专项项目(闽海洋高新[2015]29号);闽台重要海洋生物资源高值化开发技术公共服务平台(2014FJPT01);福建重要海洋经济生物种质库与资源高效开发技术公共服务平台(14PZY017NF17).
杨求华(1988-),男,硕士,工程师,从事水产生物疾病研究.E-mail:qhyang1314@163.com
周宸(1961-),男,教授级高工,从事水产病害防治技术研究.E-mail: ccdafj@fjscs.ac.cn
杜虹(1976-),女,教授,从事海洋生态学等方面的研究.E-mail:hdu@stu.edu.cn
S 917.4
A
1006-5601(2016)03-0192-10
杨求华,陆振,林琪,等.南移池塘养殖仿刺参肠道菌群结构分析[J].渔业研究,2016,38(3):192-201.

