乙型肝炎基因分型的研究进展*
张桂前,高建梅,孙 鹥 综述,段 勇 审校
(1.云南省第一人民医院检验科,昆明 650032;2.昆明医科大学第一附属医院检验科,昆明 650032)
·综述·
乙型肝炎基因分型的研究进展*
张桂前1,高建梅1,孙鹥1综述,段勇2△审校
(1.云南省第一人民医院检验科,昆明 650032;2.昆明医科大学第一附属医院检验科,昆明 650032)
乙型肝炎;基因分型;乙型肝炎病毒
乙型肝炎(简称乙肝)病毒(HBV)感染是严重的公共卫生问题,全球约20亿人感染过HBV,其中3.5亿人成为了慢性HBV感染者[1],约15%~40%的HBV感染者最终发展成为了肝硬化或肝癌[2],全球每年约100万人死于乙肝所致的肝衰竭、肝硬化和原发性肝癌。HBV的感染呈地方性流行,超过75%的HBV感染者分布于西太平洋地区和东南亚国家,乙肝表面抗原(HBsAg)携带率在西欧、北美仅0.1%~0.9%,而在亚洲国家高达5%~10%。中国累计有7亿人曾感染过HBV,有1.2亿人成为了病毒携带者,每年约60万人死于HBV感染引起的相关疾病。HBV感染的临床转归不仅与患者年龄和免疫力有关,而且也与病毒株的基因型密切相关,所以研究HBV基因型有重要的临床意义。通过HBV基因分型,可以了解基因型的地区分布特点以及在人群中的变异和进化趋势。
1 概 述
HBV属于嗜肝病毒的一种,核酸为部分共价闭合环状双链DNA,其双链长度不同,与病毒mRNA互补全长的一条链为负链,而另一条为正链,长度不定,约为负链的50%~100%。病毒基因组长度在3 182~3 221个核苷酸, 含4个部分重叠的开放读码框(ORF),即前S/S区、前C/C区、P区和X区。S-ORF分为前S1区、前S2区和S区,各有其起始密码子ATG,编码大、中、小3种包膜蛋白;C-ORF分为前C区和C基因区,各自有起始密码ATG,编码HBeAg及HBcAg,这一区段最保守,是免疫攻击的靶表位所在;P-ORF 是最长的阅读框,与C-ORF、S-ORF、X-ORF重叠,编码病毒聚合酶;X-ORF编码HBx蛋白,HBx是一种多功能的反式调节因子,可反式激活增强子和启动子的转录功能。在病毒基因组上,不仅有编码蛋白的结构基因,还有调节元件,包括4个启动子、2个增强子、糖皮质激素应答元件、负调节元件、CCAAT元件等。由于HBV复制过程中需经过RNA中间体的逆转录,而该逆转录酶又缺乏校正功能,所以HBV具有高变异性,其突变率约为(1.4~3.2)×10-5位点/年。因此,在生活环境和免疫压力下,HBV不断变异从而形成了不同的准种、基因型和基因亚型。
2 乙型肝炎的基因分型和流行分布
由于全基因序列分析比较复杂,HBV旧的分型原则可根据部分基因特别是S基因序列进行分型,因为S基因的序列相对其他序列而言异质性最小,也更加保守,从而根据S区基因序列的异质性≥4%的标准代替全基因序列分型。但新近提出的分型原则认为必须要以全基因组序列作为分型依据,并纠正了一些错误分型。例如,Khedive等[3]对伊朗的HBV携带者681 bp的分离株进行分析,报告了5株亚型(D5和D8),这与已知的地理分布不一致;以及Shi等[4]对B3亚型的错误分型等。目前HBV有8个证实基因型(A至H)和2个暂定基因型(I和J)[5]。其中,基因型A又进一步分为A1~A7亚型;B型分为B1~B9亚型;C型有C1~C16亚型;D型有D1~D8亚型;基因型F只有4种亚型F1~F4;其他基因型目前还未发现亚型存在。
基因型分布在不同的地区和不同的人种中。有关HBV基因型研究表明,B和C基因型主要分布在亚洲,而欧洲最常见的基因型是A和D型[6]。地中海、中东、中亚地区以D型为主,北美地区C型为主,其次是A和B型,H型少量分布在美国中部。印度的基因型以D型为主,D型是全球分布最广的基因型,E型是西非的优势亚型,F型是在南美洲和中美洲发现的。G型是目前HBV基因分型中最少见的一个型,它是2000年时由来自美国和法国的标本中分离的。G型导致感染是通常要有其他型的HBV存在,最常见的就是G型合并A2型,因为A2型能够增强G型病毒的复制[7]。我国以B和C两种基因型为主,也有少量的A和D基因型和B/C基因型混合感染,其中北方以C型为多,南方以B型占优势,各省之间并不完全相同[8]。越来越多的研究表明在不同的国家、地区和人群中流行的HBV存在基因型上的差异。见表1。
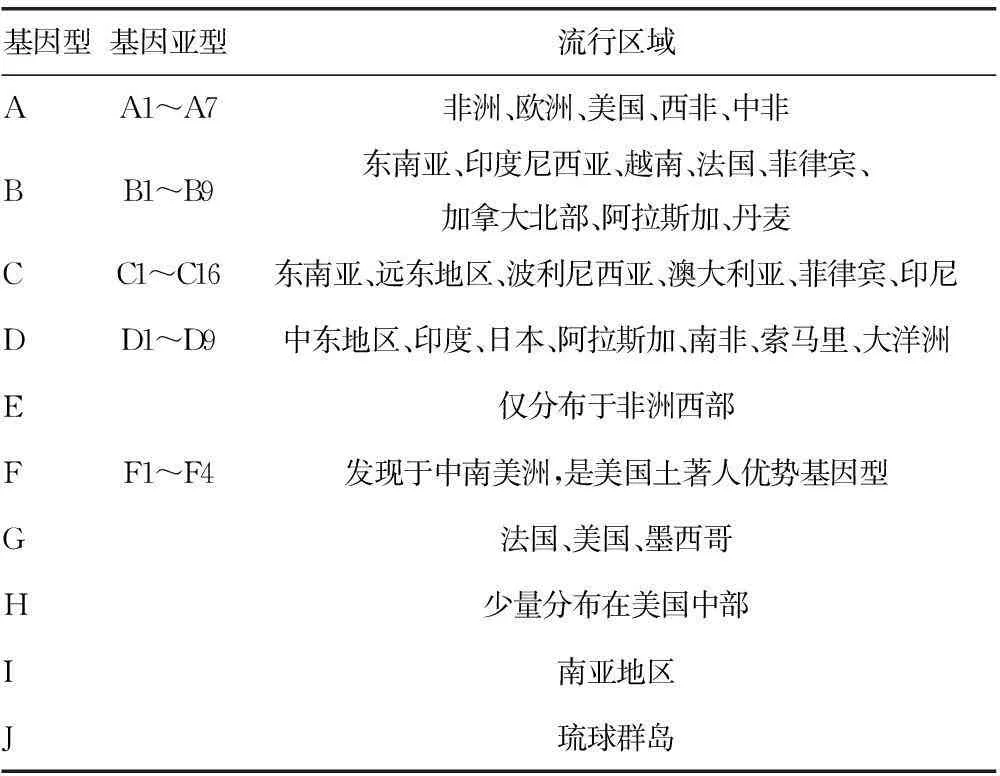
表1 HBV基因型的地理分布表[9-14]
3 HBV基因分型的方法
至今HBV基因分型的检测方法主要包括以下 10 种[15-17]:
3.1全基因测序法全基因测序法是PCR扩增HBV全基因DNA,扩增产物经胶回收后进行测序并做种系进化统计,分析不同病毒株之间的亲缘关系,得出各病毒株的型别,该方法是分析和确定基因型的最准确方法,也是 HBV 基因分型的金标准。但耗时、费用高,不适用于大规模流行基因病学调查。
3.2聚合酶链反应-限制性片段长度多态性(PCR-RFLP) 基因分型法PCR-RFLP基因分型法是先将待测的靶DNA片段进行扩增,再用限制性核酸内切酶对扩增产物进行酶切,不同基因型的序列产生的限制性片段数目和长度不同,最后经电泳分析靶DNA片段而分型。此法不需杂交和测序,是一种有效、简便的分型方法,适用于流行病学调查研究。缺点是:酶切位点易受基因变异的影响,且混合感染或酶切不完全时,会出现复杂条带,影响结果判断。
3.3单克隆抗体酶联免疫吸附试验(ELISA)分型法单克隆抗体ELISA分型法是根据HBV前S2区的特异性表位设计特异的单克隆抗体,并用辣根过氧化物酶进行标志,一般采用双抗体夹心法分析联合检测,可以得到各待测标本的表位组合,这些表位与基因型存在对应关系,根据单克隆抗体表位鉴定出不同的基因型。该方法操作简便,可用于大样本的检测。但对一些混合型感染和HBsAg低水平表达的标本可能无法鉴别。
3.4型特异性引物-PCR分型法型特异性引物-PCR分型法是对HBV各型别全序列或某目标序列(S区)进行比对分析,找出每种基因型的独特序列,设计相应的引物进行两次或一次PCR扩增,经过扩增后得到不同长度的相应基因产物,通过琼脂糖凝胶电泳分析确定不同的基因型。该方法简化了操作步骤,特异性较高,是目前应用比较多的一种适合大样本研究的方法,不足是存在非特异性扩增。
3.5PCR 微板核酸杂交-ELISA法PCR微板核酸杂交-ELISA法是将基因扩增、核酸分子杂交和酶联免疫显色3种技术融为一体,发挥了核酸分子杂交技术特异性高、PCR检测技术灵敏度高和酶联显色检测方便的优点。首先将待测核酸进行PCR扩增,将扩增产物加入预先包被HBV通用探针的微孔板,再加入HBV各基因型显色探针,进行微板核酸杂交,然后通过酶联免疫显色判断结果。该方法耗时短、特异性强、且准确可靠,可用于大规模的HBV分型检测。
3.6线性探针分析(LiPA)基因分型法LiPA基因分型法遵循反向杂交的原理,根据待测序列设计各型特异性线性探针并固化在支持物上,对待测标本特定序列进行PCR扩增,然后将扩增产物与固相探针反向杂交,经显色反应得出结果。该法最大优点是可鉴别用直接测序不能区分的混合型感染。
3.7实时定量 PCR-溶解曲线分析实时定量PCR-溶解曲线分析是将传统PCR检测模式中的PCR扩增和检测相结合,并用溶解曲线分析确定基因型,不同的基因型其目标序列与探针结合时GC含量间的互补性差异导致不同的熔解温度(TM)。该方法快速准确,灵敏度高,可鉴别混合型感染。
3.8限制性片段质谱多态性(RFMP)RFMP以限制性内切酶切割PCR产物,产生型特异性寡核苷酸片段,产生片段的分子量用质谱进行分析。该方法检测限100 copy/mL,灵敏度较高,可以检测突变型和野生型标本,但仪器体积庞大,价格昂贵。
3.9多重PCR多重PCR是在同一PCR反应体系里加入2对以上引物,同时扩增出多个核酸片段,扩增产物经琼脂糖凝胶电泳分析确定其基因型。其经济、快速,可用于大规模研究,可检测混合基因型,但单核苷酸多态性(SNP)酶切位点可影响到方法的灵敏度。
3.10PCR-侵入法PCR-侵入法能检测不同的基因型和亚型,先用多重PCR扩增S序列,然后经过2个侵入反应,第1个侵入反应是将扩增产物与型特异的寡核苷酸初级探针(P1或P2)结合,根据基因型对P1或P2的5′端进行切割,导致5′翼片的释放,在第2个反应中,切割下来的5′翼片能和其通用的FRET盒结合,导致其切割并释放特异性荧光,产生的荧光信号与HBV基因型相对应。该方法有极高的灵敏度,可检测混合基因型,同样SNP酶切位点可影响到方法的灵敏度。
4 乙肝基因型的致病性和治疗反应
研究资料显示,不同基因型/亚型其临床表现不同。日本一项包括585例慢性乙肝患者的研究表明,基因型B较C型进展至肝硬化更缓慢,更早发生HBeAg血清转换和有更少的炎症活动[18];基因型C和D引起的肝脏疾病比基因型A和B更严重,转化成肝癌的可能性更大。与基因型B相比,基因型C患者HBeAg阳性和HBV DNA、丙氨酸氨基转移酶(ALT)水平、肝脏病理的炎症和纤维化程度均较高;基因型C患者HBeAg更易在年龄偏大时发生血清转换延迟,对干扰素的应答率也较低。顾锡炳等[19]的研究认为C基因型感染者非特异性细胞毒性T淋巴细胞(CTL)比B基因型感染者高,非特异性细胞免疫清除HBV的作用相对较弱,在清除HBV的过程中可起肝细胞损伤。这可能是导致C基因型HBV DNA水平、HBeAg阳性率、肝功能损害高于B基因型感染者的原因。美国的研究报道了CHB患者中D基因型更容易导致肝炎的发作,而瑞士的研究显示A基因型较D型更容易慢性化。35岁前B基因型HBV易形成肝细胞癌(HCC),而50岁以后C型HBV感染者比B基因型更易形成HCC。在所有年龄段内,混合型的病毒载量和HBeAg阳性率最高,提示混合型的HBV感染者较单个基因型感染者更容易形成HCC[20]。一般患者都会合并感染多于2个不同基因型的HBV,例如在亚洲的B和C基因型的合并感染,G基因型引发的慢性感染需要A基因型或H基因型的存在等。
不同基因型的HBV感染者对抗病毒治疗的反应也存在差异。一项台湾的研究报道指出,干扰素治疗后,B基因型的HBeAg血清转换率为41%,而同组中C基因型的转换率仅为15%[21],且基因型A和B对干扰素为基础的治疗比基因型C和D有更好的反应。B基因型感染的预后一般较好,B基因型HBeAg阳性的CHB患者对IFN-α治疗的应答率高于C基因型,A基因型患者高于D基因型。研究显示B型CHB患者经拉米夫定治疗后HBV DNA阴转率、HBeAg血清转换率及ALT复发率均显著高于B+C及C基因型CHB患者,这可能与C基因型预后不良有关[22]。B1基因亚型的患者比B2基因亚型具有更高的HBeAg阳性率及拉米夫定耐药的发生率。也有研究表明:C2基因亚型比B2基因亚型更易发生拉米夫定耐药,在CHB患者中更明显。目前有感染B2基因亚型的HCC患者术后易复发的报道[23],提示基因型不仅与HCC的发生有密切关系,同时也影响着肿瘤的复发和转移。当然,有些基因型的临床结果和治疗反应等仍存在争议,如基因型D[24]等,为此还需要做进一步的研究。
5 小 结
全球化和移民人口的增大加快了病毒株之间的分布和重组,如基因型A/D、A/E、C/D、G/C、D/F、A/F等的重组,由于重组导致基因片段在不同的病毒株之间的移动是导致HBV多样性的原因[25],而移民是全球性HBV分布的混杂因素,大量的移民改变了HBV基因型的分布,导致了越来越多异国株的出现[26-27]。目前HBV基因分类方法尚不完善,本领域的专家开始关心那些新发现的亚型的分类是否准确。Pourkarim等[28]认为分型时应该系统地对病毒基因组进行分析,而不是仅仅对部分序列进行分析。Mahmoud等提出了新术语“recombino-亚型”和“immigro-亚型”;Pourkarim也同时认为,对于重组株没必要给予特定的分型,只需归于重组亚型就好。HBV基因型的分类存在很多复杂因素,对此还需要做更多的研究。
[1]Liaw YF,Chu CM.Hepatitis B virus infection[J].Lancet,2009,373(9663):582-592.
[2]Fattovich G,Bortolotti F,Donato F.Natural history of chronic hepatitis B:special emphasis on disease progression and prognostic factors[J].J Hepatol,2008,48(2):335-352.
[3]Khedive A,Norouzi M,Ramezani F,et al.Hepatitis B virus surface protein mutations clustered mainly in CTL immune epitopes in chronic carriers:results of an Iranian nationwide study[J].J Viral Hepat,2013,20(7):494-501.
[4]Shi W,Zhu C,Zheng W,et al.Subgenotype reclassification of genotype B hepatitis B virus[J].BMC Gastroenterol,2012,12:116-119.
[5]Yousif M,Kramvis A.Genotype D of hepatitis B virus and its subgenotypes:An update[J].Hepatol Res,2013,43(4):355-364.
[6]Lazarevic I,Cupic M,Delic D,et al.Distribution of HBV genotypes,subgenotypes and HBsAg subtypes among chronically infected patients in Serbia[J].Arch Virol,2007,152(11):2017-2025.
[7]Zaaijer HL,Boot HJ,Van Swieten P,et al.HBsAg-negative mono-infection with hepatitis B virus genotype G[J].J Viral Hepat,2011,18(11):815-819.
[8]Zeng G,Wang Z,Wen S,et al.Geographic distribution,virologic and clinical characteristics of hepatitis B virus genotypes in China[J].J Viral Hepat,2005,12(6):609-617.
[9]Kurbanov F,Tanaka Y,Mizokami M.Geographical and genetic diversity of the human hepatitis B virus[J].Hepatol Res,2010,40(1):14-30.
[10]Sharma S,Sharma B,Singla B,et al.Clinical significance of genotypes and precore/basal core promoter mutations in HBV related chronic liver disease patients in North India[J].Dig Dis Sci,2010,55(3):794-802.
[11]Kim BJ.Hepatitis B virus mutations related to liver disease progression of Korean patients[J].World J Gastroenterol,2014,20(2):460-467.
[12]Thedja MD,Muljono DH,Nurainy N,et al.Ethnogeographical structure of hepatitis B virus genotype distribution in Indonesia and discovery of a new subgenotype,B9[J].Arch Virol,2011,156(5):855-868.
[13]Shi W,Zhang Z,Ling C,et al.Hepatitis B virus subgenotyping:history,effects of recombination,misclassifications,and corrections[J].Infect Genet Evol,2013,16:355-361.
[14]Alvarado-Mora MV,Pinho JR.Distribution of HBV genotypes in Latin America[J].Antivir Ther,2013,18(3 Pt B):459-465.
[15]Malmström S,Berglin-Enquist I,Lindh M.Novel method for genotyping hepatitis B virus on the basis of TaqMan real-time PCR[J].J Clin Microbiol,2010,48(4):1105-1111.
[16]邹淑慧,应颖,周艳,等.基于基因序列的乙型肝炎病毒基因分型方法的比较[J].南昌大学学报(理科版),2013,37(2):179-184.
[17]Guirgis BS,Abbas RO,Azzazy HM.Hepatitis B virus genotyping:current methods and clinical implications[J].Int J Infect Dis,2010,14(11):e941-e953.
[18]Sumi H,Yokosuka O,Seki N,et al.Influence of hepatitis B virus genotypes on the progression of chronic type B liver disease[J].Hepatology,2003,37(1):19-26.
[19]顾锡炳,杨小娟,王栋,等.乙型肝炎病毒B,C基因型引起的慢性乙型肝炎患者特异性、非特异性细胞免疫比较[J].中国科学(生命科学),2010,40(5):428-432.
[20]Toan NL,Song le H,Kremsner PG,et al.Impact of the hepatitis B virus genotype and genotype mixtures on the course of liver disease in Vietnam[J].Hepatology,2006,43(6):1375-1384.
[21]Kao JH,Wu NH,Chen PJ,et al.Hepatitis B genotypes and the response to interferon therapy[J].J Hepatol,2000,33(6):998-1002.
[22]方军伟,顾美芬.拉米夫定治疗B、B+C、C基因型慢性病毒性乙型肝炎患者的临床疗效观察[J].中国预防医学杂志,2009,10(6):532-533.
[23]Yin J,Zhang H,Li C,et al.Role of hepatitis B virus genotype mixture,subgenotypes C2 and B2 on hepatocellular carcinoma:compared with chronic hepatitis B and asymptomatic carrier state in the same area[J].Carcinogenesis,2008,29(9):1685-1691.
[24]Karimi A,Moezzi M,Imani R.Sequence-based genotyping of hepatitis B virus in general population[J].Med J Islam Repub Iran,2015,29:165-168.
[25]Croagh CM,Desmond PV,Bell SJ.Genotypes and viral variants in chronic hepatitis B:A review of epidemiology and clinical relevance[J].World J Hepatol,2015,7(3):289-303.
[26]Bissinger AL,Fehrle C,Werner CR,et al.Epidemiology and genotyping of patients with chronic hepatitis B:genotype shifting observed in patients from central Europe[J].Pol J Microbiol,2015,64(1):15-21.
[27]Coppola N,Alessio L,Pisaturo M,et al.Hepatitis B virus infection in immigrant populations[J].World J Hepatol,2015,7(30):2955-2961.
[28]Pourkarim MR,Amini-Bavil-Olyaee S,Kurbanov F,et al.Molecular identification of hepatitis B virus genotypes/subgenotypes:revised classification hurdles and updated resolutions[J].World J Gastroenterol,2014,20(23):7152-7168.
2016-01-29修回日期:2016-05-06)
云南省应用基础研究计划-昆医联合专项资助项目(2014FB094)。
10.3969/j.issn.1673-4130.2016.15.033
A
1673-4130(2016)15-2136-04

