葡萄牙牡蛎外套膜转录组测序及壳色基因挖掘
严璐琪,郭香,巫旗生,祁剑飞,宁岳,王晓清*,曾志南*
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.水产高效健康生产湖南省协同创新中心,湖南 常德415000;3.福建省水产研究所,福建 厦门 361000)
葡萄牙牡蛎外套膜转录组测序及壳色基因挖掘
严璐琪1,2,郭香3,巫旗生3,祁剑飞3,宁岳3,王晓清1,2*,曾志南3*
(1.湖南农业大学动物科学技术学院,湖南 长沙 410128;2.水产高效健康生产湖南省协同创新中心,湖南 常德415000;3.福建省水产研究所,福建 厦门 361000)
采用Illumina高通量测序平台对黑壳与金壳葡萄牙牡蛎(Crassostrea angulata)外套膜组织开展转录组测序研究,其中黑壳组总计产出5 857 718 092 nt数据,筛选后的干净阅读子58 004 560个;金壳组产出6 727 447 326 nt数据,干净阅读子66 614 854个。将发掘的新基因与COG、GO、KEGG、Swiss-Prot、NR数据库进行序列比对,最终得到各数据库注释的新基因数量分别为147、397、203、567、1 347个。通过对差异表达基因的序列进行生物学功能注释和聚类分析以及利用已知贝壳基质蛋白序列进行搜索,鉴定出 14条可能与壳色表达有关的贝壳基质蛋白基因,其中5条下调基因可能与金壳基因表达相关。
葡萄牙牡蛎;壳色;转录组
投稿网址:http://xb.ijournal.cn
葡萄牙牡蛎(Crassostrea angulata)属软体动物门(Mollusca),双壳纲(Bivalvia),珍珠贝目(Pterioida),牡蛎科(Ostreidae),是广泛分布于世界各地的重要经济贝类。2013年中国牡蛎的总产量为4 218 644 t,占全国贝类养殖总产量的33.1%[1]。为进一步发展牡蛎生产,目前已有关于葡萄牙牡蛎选育研究的报道[2]。牡蛎的视觉性状越来越受到消费者和育种人员的关注[3]。贝类的壳色是受遗传控制的。海产经济贝类壳色研究主要是针对滨螺、皱纹盘鲍、海湾扇贝、牡蛎和蛤类[4]等。通过壳色选育,已培育出“中科红”海湾扇贝新品种[5]。太平洋牡蛎壳色在大多数情况下呈正态分布,具有数量性状遗传特征,但在个别家系中,其壳色仅仅受少数几个主效基因的控制[6-7]。
目前,葡萄牙牡蛎研究主要集中在育种、养殖方面[8-10],关于分子生物学研究方面的尚少[11]。转录组测序(RNA-seq)是对某一物种的mRNA进行高通量测序,具有高通量、低成本、高效率等特点,有助于快速获得大量分子生物学信息。笔者应用RNA-seq技术对金壳和黑壳葡萄牙牡蛎进行高通量转录组测序分析,描绘葡萄牙牡蛎的转录组图谱,筛选与葡萄牙牡蛎壳色相关的基因,旨在为葡萄牙牡蛎基因结构信息的完善及潜在新基因的挖掘提供参考数据。
1 材料与方法
1.1样品的采集
用黑壳和金黄壳色葡萄牙牡蛎亲本各1个构建全同胞家系,分别选取黑壳与金壳子代个体各3个,采集外套膜组织样品,置于液氮中速冻,于-70 ℃保存,备用。
1.2RNA的提取
使用Trizol试剂分别提取黑壳与金壳葡萄牙牡蛎外套膜组织的总RNA[12],分别采用Nanodrop、Qubit 2.0、Aglient 2100方法检测RNA样品的纯度、浓度和完整性等,样品RIN≥9。
1.3RNA文库的构建及测序
取5 μg RNA,用带有Oligo(dT)的磁珠富集其中的mRNA,加入Fragmentation Buffer将mRNA进行随机打断。以mRNA为模板,用六碱基随机引物(random hexamers)合成第1条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA polymerase I合成第2条cDNA链,利用AMPure XP beads纯化cDNA。将纯化的双链cDNA进行末端修复,加A尾并连接测序接头,然后用AMPure XP beads进行片段大小选择,最后通过PCR富集,得到cDNA文库。
文库构建完成后,分别使用Qubit 2.0和Agilent 2100对文库的浓度和插入片段大小进行检测,待检测合格后,用Illumina HiSeq 2500进行高通量测序,测序读长为PE100。
1.4转录组数据分析
由Illumina HiSeq 2500测序所得的原始数据称为原始测序序列(Raw Data)。对Raw Data进行数据过滤,去除其中的接头序列及低质量 Reads,获得高质量的Clean Data。将Clean Data与太平洋牡蛎的参考基因组进行序列比对,获得Mapped Data。基于Mapped Data,进行插入片段长度检验、随机性检验等测序文库质量评估和表达量分析、可变剪接分析、新基因发掘和基因结构优化等。根据基因在不同样品或不同样品组中的表达量进行差异表达分析、差异表达基因功能注释和功能富集等。
将FPKM[13](fragments per kilobase of transcript per million fragments mapped)作为衡量转录本或基因表达水平的指标。
基于太平洋牡蛎基因组序列,使用Cufflinks[13]软件对Mapped Reads进行拼接,并与原有的基因组注释信息进行比较,寻找原来未被注释的转录区,发掘葡萄牙牡蛎的新转录本和新基因,从而补充和完善原有的基因组注释信息。
2 结果与分析
2.1测序数据分析及注释
经Illumina HiSeq 2500深度测序,得到原始序列数据,其中黑壳组总计产出数据5 857 718 092 nt,去除接头序列、空读序列以及低质量序列后,得到干净阅读子58 004 560个;金壳组产出数据6 727 447 326 nt,干净阅读子66 614 854个,总计12.59 Gb Clean Data。碱基组成和质量分析结果表明,原始测序数据的碱基组成情况良好(图 1),低质量碱基测序序列(<30)的比例均低于10%(图2)。此结果说明测序质量良好。与太平洋牡蛎基因组的比对分析结果表明,黑壳色和金壳色葡萄牙牡蛎分别有38 090 056条(65.76%)和43 081 54条(64.67%)比对到参考基因组上。

图1 黑壳色葡萄牙牡蛎(左)和金壳色葡萄牙牡蛎(右)原始数据的碱基组成Fig.1 Bases content distribution of black shell and golden shell Crassostrea angulata
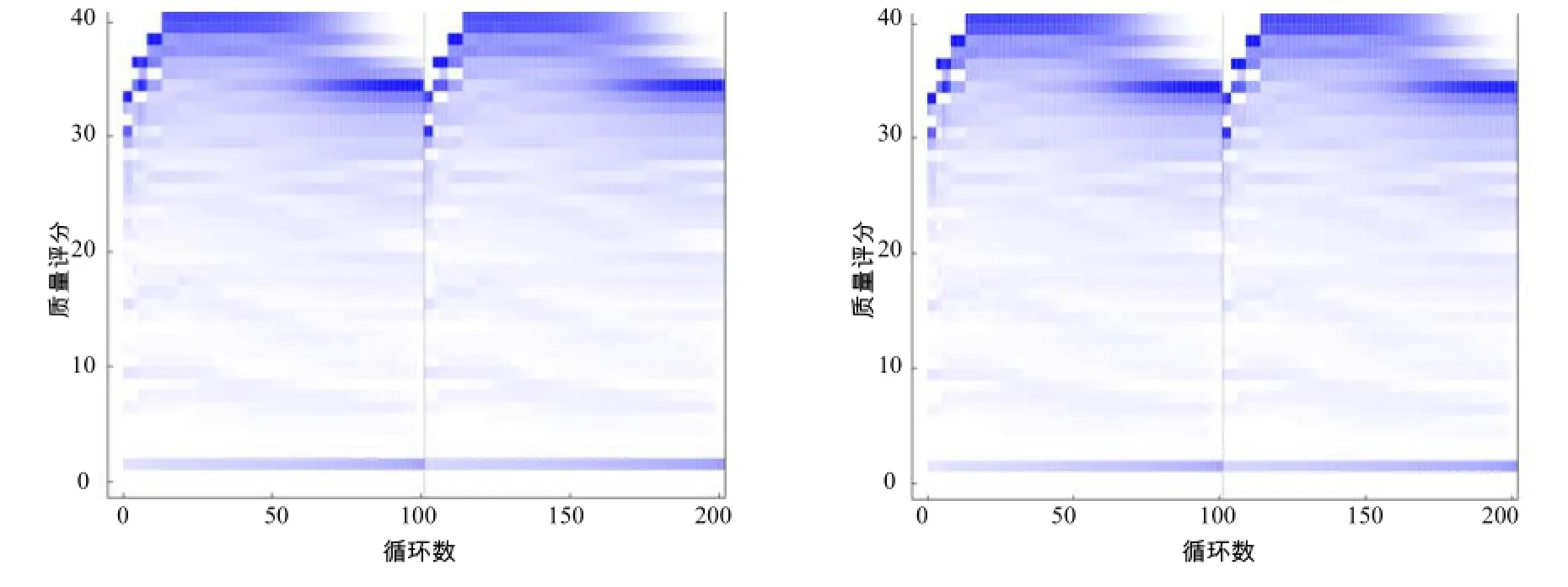
图2 黑壳色葡萄牙牡蛎(左)和金壳色葡萄牙牡蛎(右)原始数据碱基的质量评分Fig.2 Base quality distribution of black shell and golden shell Crassostrea angulata
过滤掉编码肽链过短(少于 50个氨基酸残基)或只包含单个外显子的序列,在葡萄牙牡蛎转录组中共发掘1 856个新基因。使用BLAST[14]软件将发掘的新基因与COG、GO、KEGG、Swiss-Prot、NR数据库进行序列比对,获得新基因的注释信息,最终得到各数据库注释的新基因数量分别是 147、397、203、567、1 347个。
2.2基因差异表达分析
在差异表达基因检测过程中,将Fold Change≥2 且FDR<0.01作为筛选标准,结果表明,黑壳色葡萄牙牡蛎与金壳色葡萄牙牡蛎之间有 1 082个差异表达基因,分别有707个上调基因,375个下调基因。
将上述差异表达基因进行功能注释到 COG、GO、KEGG、Swiss-Prot、NR库,成功获得注释的基因数分别为193、370、156、553、1 025个。葡萄牙牡蛎的差异基因在功能注释过程中所匹配的基因序列主要来自有基因组数据的太平洋牡蛎(Crassostrea gigas),占太平洋牡蛎基因组数据的96.39%。
与COG数据库进行比对后的差异表达基因可归属于21个大类(图3),数量最多的功能类为一般功能预测类(R),达56条,其余依次为未知功能类(S)、氨基酸转运和代谢类(E)、次生代谢物生物合成、转运和代谢类(Q)、信号转导机制类(T)。

图3 差异表达基因经COG注释后各功能分类基因的数目Fig.3 Functional classification of DEGs by COG
将葡萄牙牡蛎差异表达基因注释到GO库,共获得370个注释结果,包含3个主要分支,即细胞组分、分子功能和生物学过程。在细胞组分方面,包含差异表达基因最多的二级功能是细胞部分类、细胞类、细胞器类;在分子功能方面,包含差异表达基因最多的二级功能是结合活性、催化活性;在生物学过程方面,所包含差异表达基因最多的二级功能是单组织过程和细胞过程(图4)。
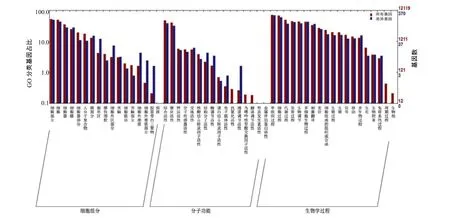
图4 差异表达基因GO二级节点的注释统计结果Fig.4 Statistical map of DEGs on two level node annotation by GO
葡萄牙牡蛎外套膜转录组共 203个差异表达基因在KEGG数据库获得注释,共涉及86种通路,其中,差异基因数量最多的通路分别是细胞外基质受体相互作用通路、神经信号传递通路、吞噬体通路、黏着斑通路等。按照KEGG中通路类型进行分类的差异表达基因的注释结果见图5。
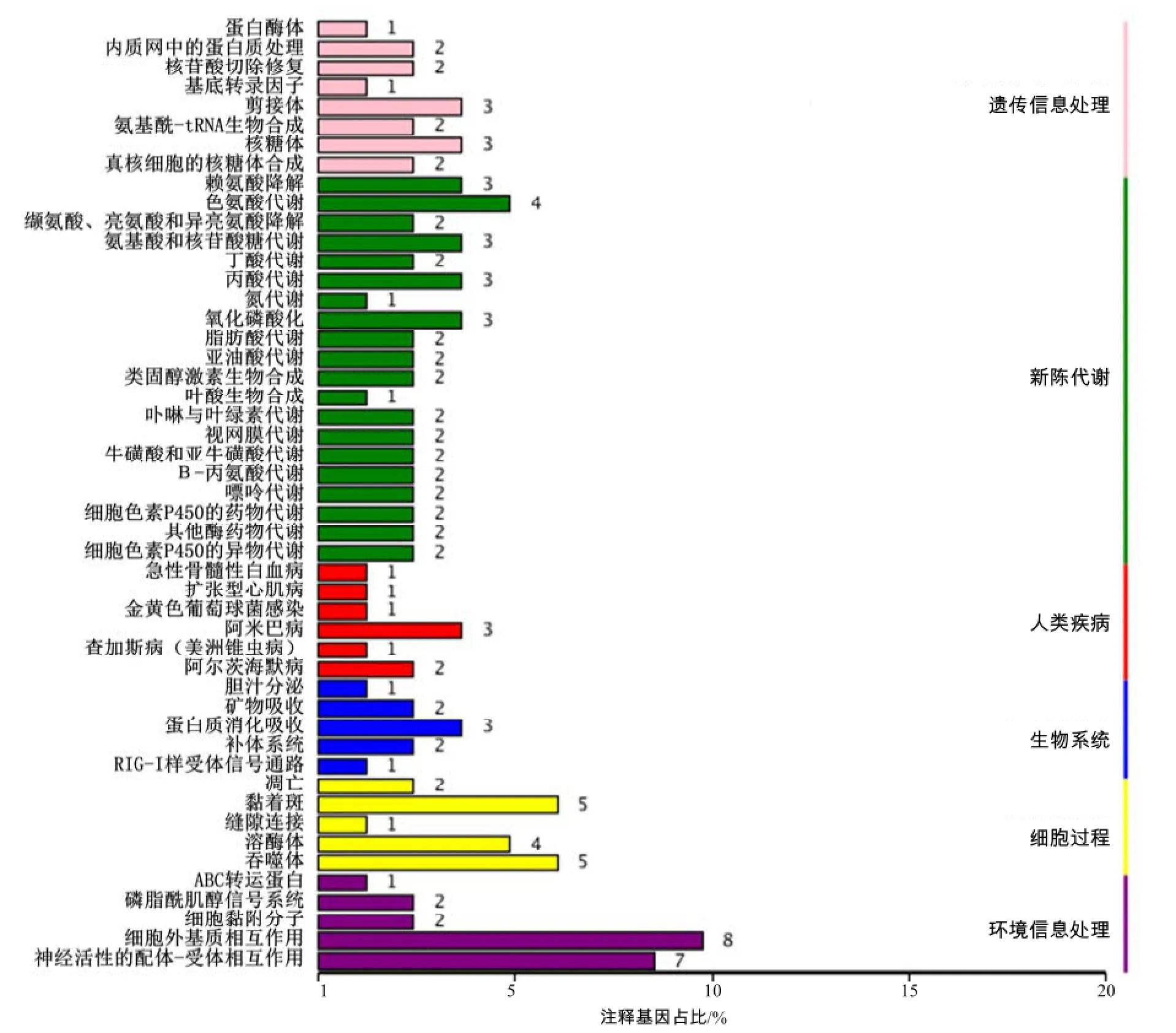
图5 差异表达基因的KEGG分类结果Fig.5 Classification map of DEGs by KEGG
2.3壳色基因的挖掘
根据葡萄牙牡蛎外套膜转录组1 025个差异表达基因功能注释的结果以及数据库中已经公布的代表性贝壳基质蛋白的序列,筛选出与已知贝壳基质蛋白具有高同源性的基因14条,其中5条下调基因可能与金壳基因表达相关(表1)。这5条基因分别为CGI_10014905、CGI_10005327、CGI_10024093、CGI_10013406和CGI_10016016。

表1 可能与金壳基因表达相关的基因Table 1 Genes related to the expression of the golden shell
3 结论与讨论
根据数据库中已公布的代表性贝壳基质蛋白序列,在1 025个注释到的差异表达基因中进行筛选,共选出5条下调基因可能与金壳基因的表达相关。CGI_10014905、CGI_10005327、CGI_10024093注释到Perlucin蛋白[15-16]。Perlucin蛋白同时存在于鲍属和贻贝属中,对鲍壳的生物结构起着重要的作用,特别是对鲍壳矿物相的晶核生成、定性生长、表面形貌具有调控作用。CGI_10013406注释到Prisilkin-39蛋白[17],Prisilkin-39富含甘氨酸、酪氨酸和丝氨酸,被认为是一种双功能贝壳基质蛋白,即参与棱柱层的蛋白质框架构建,又可以抑制文石型碳酸钙晶体的形成。CGI_10016016注释到Protein Pif80。Pif蛋白[18]被认为与贝壳内的几丁质成分和文石型碳酸钙晶体一起组成复合体,它参与了贝壳珍珠质层的形成,并赋予了珍珠质层优异的力学性能。笔者将对这5条基因进行分子实验验证,以便进一步分析葡萄牙牡蛎壳色相关基因的表达模式,探讨壳色基因与生长的调控关系,建立良好的分子基础研究平台。
本研究中采用Illumina高通量测序平台,对黑壳与金壳葡萄牙牡蛎外套膜组织开展转录组测序研究,其中黑壳色组总计产出5 857 718 092 nt数据,筛选后的干净阅读子共58 004 560个;金壳色组总计产出6 727 447 326 nt数据,筛选后的干净阅读子共66 614 854个。碱基质量值Q30均不小于90.64%,测序数据与太平洋牡蛎基因组的比对效率分别为65.67%和64.67%,表明数据正常,能够满足后续分析需求。通过差异表达基因分析,得到差异表达基因1 082个,其中94.73%成功获得注释。本研究中得到的大量数据可以为后续金壳基因挖掘提供参考依据。
[1] 农业部渔业渔政管理局.2014中国渔业统计年鉴[M].北京:中国农业出版社,2014.
[2] 郑怀平,王迪文,林清,等.太平洋牡蛎与葡萄牙牡蛎两近缘种间杂交及其早期阶段生长与存活的杂种优势[J].水产学报,2012(2):210-215.
[3] Clydesdale F M.Color as a factor in food choice[J]. Critical Reviews in Food Science and Nutrition,1993,33 (1):83-101.
[4] 管云雁,何毛贤.海产经济贝类壳色多态性的研究进展[J].海洋通报,2009(1):108-114.
[5] 李国江,盛红禄,宋京辉,等.海洋贝类养殖新品种——‘中科红’海湾扇贝[J].科学养鱼,2005(2):22.
[6] Hedgecock D, Perry G M L, Voigt M L.Mapping heterosis QTL in the Pacific oyster Crassostrea gigas[J]. Aquaculture,2006,272:267-268.
[7] Jianlong Ge,Qi Li,Hong Yu,et al.Identification and mapping of a SCAR marker linked to a locus involved in shell pigmentation of the Pacific oyster (Crassostrea gigas)[J].Aquaculture,2014,434:249-253.
[8] 巫旗生,曾志南,宁岳,等.葡萄牙牡蛎工厂化人工育苗技术[J].福建水产,2015,37(5):399-405.
[9] 於俊琦,陈琛,冀德伟,等.高效生态循环养殖系统中的葡萄牙牡蛎单体养殖试验[J].科学养鱼,2013(10):41-42.
[10] 王华青,张志杨,梁和平.单体葡萄牙牡蛎筏式笼养试验[J].中国水产,2014(2):70-71.
[11] 巫旗生.福建沿海主要养殖牡蛎种类及其遗传多样性研究[D].长沙:湖南农业大学,2012.
[12] 王桂玲,李家乐.淡水育珠蚌外套膜提取总RNA的改良方法[J].生物技术通报,2008(S1):356-361.
[13] Trapnell C,Williams B A,Pertea G,et al.Transcript assembly and quantification by RNA Seq reveals unannotated transcripts and isoform switching during cell differentiation[J].Nature Biotechnology,2010,28(5):511-515.
[14] Altschul S F,Madden T L,Schaffer A A,et al.Gapped BLAST and PSI BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[15] Weiss I M,Kaufmann S,Mann K,et al.Purification and characterization of perlucin and perlustrin,two new proteins from the shell of the mollusk Haliotis laevigata[J].Biochemical and Biophysical Research Communications,2000,267(1):17-21.
[16] Mann K,Weiss I M,André S,et al.The amino-acid sequence of the abalone(Haliotis laevigata) nacre protein perlucin:Detection of s functional C-type lectin domain with galactose/mannose sepecificity[J].The Federation of European Biochemical Societies Journal,2000,267(16):5257-5264.
[17] Kong Y M,Jing G,Yan Z G,et al.Cloning and characterization of Prisilkin-39,a novel matrix protein serving a dual role in the prismatic layer formation from the oyster Pinctada fucata[J].Journal of Biological Chemistry,2009,284(16):10841-10854.
[18] Suzuki M,Saruwatari K,Kogure T,et al.An acidic matrix protein,Pif,is a key macromolecule for nacre formation[J].Science,2009,325(5946):1388-1390.
责任编辑:王赛群
英文编辑:王 库
Transcriptome analysis and shell gene mining on Crassostrea angulata
Yan Luqi1,2, Guo Xiang3, Wu Qisheng3, Qi Jianfei3, Ning Yue3, Wang Xiaoqing1,2*, Zeng Zhinan3*
(1.College of Animal Science and Technology, Hunan Agricultural University, Changsha 410128, China; 2.Collaborative Innovation Center for Efficient and Health Production of Fisheries in Hunan Province, Changde, Hunan 415000, China;3.Fisheries Research Institute of Fujian, Xiamen, Fujian 361000, China)
Shell coloration is one of important traits for genetic breeding, for improving genome structure and explore new potential gene and further determine the genetic structure of golden shell gene of oyster, the transcriptome sequencing of Crassostrea angulata with black shell and golden shell mantle were performed by Illumina platform. Data were obtained from black shell and golden shell transcriptome were 5 857 718 092 nt and 6 727 447 326 nt respectively,out of which 58 004 560 and 66 614 854 clean reads were remained after screening, respectively. To compare with the database from COG, GO, KEGG, Swiss-Prot and NR, new gene numbers corresponding to each database were 147, 397,203, 567, and 1 347 respectively. To further explore the shell color genes of oyster, functional annotation, clustering analysis and screening from the reported shell matrix proteins on the differential expression gene (DEGs), 14 genes related to shell color expression were identified out and 5 down-regulated genes out of them might be related to the expression of golden shell.
Crassostrea angulata; shell pigmentation; transcriptome
严璐琪(1991—),女,江苏无锡人,硕士研究生,主要从事水产经济动物遗传育种研究,825954514@qq.com;*通信作者,王晓清,博士,教授,主要从事水产动物育种研究,wangxiao8258@126.com;*通信作者,曾志南,博士,研究员,主要从事海水养殖和贝类遗传育种研究,xmzzn@sina.com
Q915.817;S917.4
A
1007-1032(2016)04-0409-06
2015-12-21 修回日期:2016-01-20
现代农业产业技术体系建设专项(CARS-48);福建省种业工程项目(201451477-9);福建省属公益类科研院所基本科研专项(2015R1003-8)

