诺如病毒河北卢龙株NHBGR59全基因组序列分析
敖元云 虞结梅 李莉莉 靳淼 段招军
·论著·
诺如病毒河北卢龙株NHBGR59全基因组序列分析
敖元云虞结梅李莉莉靳淼段招军
100052 北京,中国疾病预防控制中心病毒病预防控制所腹泻室
【摘要】目的分析河北卢龙县腹泻儿童粪便标本中存在的诺如病毒株NHBGR59的全基因组序列,了解其基因结构特点和进化特征。方法根据454高通量测序获得的两个重叠群(contig)和诺如病毒参考株序列(GenBank登录号KM198534)设计NHBGR59特异性引物,用重叠反转录聚合酶链反应(Reverse transcription PCR,RT-PCR)和cDNA末端快速扩增的技术(rapid-amplification of cDNA ends,RACE)的方法扩增其全长基因,经过分子克隆、测序获得全基因组序列,然后进行三个开放阅读框(Open Reading Frames,ORFs)序列和系统进化树的分析。结果诺如病毒河北卢龙珠HBGR59病毒全基因组全长为7 563 bp(除了PolyA尾),病毒基因组分为三个ORFs:ORF1(5~5 098 nt)、ORF2(5 079~6 731 nt)和ORF3(6 731~7 510 nt),其中ORF1和ORF2之间有20 bp重叠,ORF2和ORF3之间有1 bp重叠。经过全基因组序列分析发现其和基因组II基因型6(genogroup II GenotypeVI, GII.6)诺如病毒株30443同源性最高(同源性为98.0%)。ORF2以及ORF3核苷酸和氨基酸序列与株30443同源性最高(分别为97.0%,98.0%;99.0%,98.0%)。ORF1的基因和氨基酸序列与台湾暴发的急性胃肠炎GII.6株11-FJ-5(GenBank号KM461694)同源性最高(99.0%,99.3%),只在高突变P2区发生一个氨基酸突变位点:aa308D→N;其中衣壳区的氨基酸序列与其他GII.6型诺如病毒相似度为73.0%~99.3%。进化树分析发现河北卢龙株HBGR59与GII.6型株30443和11-FJ-5亲缘关系最近。结论河北卢龙株HBGR59为诺如病毒GII.6型,进化关系上与已报道的GII.6型株30443和11-FJ-5最近。
【主题词】诺如病毒;GII.6型;进化树;基因组
Fund program: National Natural Science Foundation of China (81290345)
诺如病毒(Norovirus,NoVs)是一种单股、无包膜的RNA病毒,属于杯状病毒属,是引起儿童和成人非细菌性胃肠炎的最主要病原体[1-2]。诺如病毒基因组包括三个开放阅读框(Open readding frames,ORFs)。ORF1编码非结构蛋白,包括RNA聚合酶(RNA-dependent RNA polymerase,RdRp);ORF2和ORF3分别编码主要(VP1)和次要(VP2)衣壳蛋白。根据诺如病毒RNA多聚酶区和衣壳区的核苷酸序列,将诺如病毒分为5个基因组(genogroup,G),GI~GV,其中G1、GII和GIV主要感染人类,而GIII和GV分别感染牛和鼠[3-4]。根据ORF2全基因序列,分别进一步分为GI型8个和GII型23个,其中GII.4基因型一直是全球胃肠炎暴发的优势流行株,由GII.6型引起的暴发很少有报道[5-7]。但是,最近发现了两起由GII.6型诺如病毒引起的儿童急性胃肠炎暴发,其中一起来自上海的一所小学[8],另一起来自中国台湾(未发表),而且前期来自日本的研究发现GII.6型诺如病毒已成为第二大优势株,仅次于GII.4型[9]。因此,对于我国GII.6型诺如病毒的基因结构特点和进化特征的研究是非常重要。本研究对河北卢龙检测到的诺如病毒GII.6型毒株进行全基因组的测序和序列分析,以了解其遗传特征。
1材料与方法
1.1标本来源和处理本实验中NHBGR59阳性毒株来源于2013年河北省卢龙县10个月男性腹泻患儿(急性胃肠炎)粪便标本,是通过454高通量测序发现的。取豌豆大小(约1 g)的粪便样品加入1 ml处理液PBS,制成10%便悬液,8000 rmp/min离心5 min,取上清液,保存于-20 ℃。
1.2所用试剂核酸提取试剂盒Viral Nucleic Acid Extraction Kit II购自Geneaid公司;Superscript II反转录酶,RNA酶抑制剂,dNTP购于Invitrogen;Ex-TaqDNA和LA-TaqDNA聚合酶购自日本TaKaRa公司;3′RACE/5′RACE试剂盒购自美国Clontech公司;随机引物和PGEM T-easy连接载体购于Promega公司;快速T4DNA连接酶购于北京擎科有限公司;DH5a感受态购自北京博迈德公司;引物合成和DNA测序来自北京天一辉远公司。
1.3所用软件Primer Premier 5用于引物设计、Clustalx用于多序列比对、DNAMAN6.0用于序列相似度分析、DNAStar software package用于序列拼接和MEGA 5.0用于序列进化树分析。
1.4病毒cDNA的合成取上述从粪便标本悬液离心上清中提取的RNA作为模板,用随机引物进行反转录,得到病毒的cDNA。反应体系为15 μl,组成为:5×First Buffer 2.05 μl,Random Primers 0.5 μl,dNTP(10Mm)0.5 μl,DTT 0.5 μl,Superscript II反转录 0.5 μl,RNase Inhibitor 0.5 μl,H2O 2.2 μl,RNA 7.5 μl。反应条件为:42 ℃ 60 min延伸,99 ℃ 5 min灭活。
1.5病毒全基因组扩增由高通量测序平台产生的53条序列(reads),利用DNAStar软件进行重叠区(overlap)的拼接,拼接后获得的两段重叠群(Contig),长度分别为2 350个核苷酸(Nucleotide, nt)和1 173 nt,设计两对特异性引物进行确认,然后根据确证后的Contig和参考诺如病毒株30443(GenBank登录号KM198498)设计特异性引物进行重叠PCR扩增,两末端序列采用3′RACE/5′RACE试剂盒进行验证,扩增产物经过1.5%琼脂糖凝胶电泳鉴定,对阳性产物切胶纯化,连接至PGEM T-easy载体,挑选阳性克隆送北京天一辉远公司测序。所使用的引物见表1。
2结果
2.1病毒全基因组扩增和拼接本次重叠PCR扩增共产生5个特异性片段,长度分别为1 590 nt, 2 296 nt, 1 960 nt, 1 069 nt和1 579 nt,经DNAstar软件包中SeqMan软件拼接后得到接近全长序列(7 495 nt)。两末端序列经过3′RACE/5′RACE方法扩增,分别得到274 nt和271 nt(表1),再次拼接和确证后得到河北卢龙株NHBGR59病毒全长序列为7 563 nt(除了PolyA尾),经过与其他NoVs GII参考序列比对,NHBGR59分为三个ORFs和5′/3′UTR区:ORF1(5~5 098 nt),ORF2(5 079~6 731 nt),ORF3(6 731~7 510 nt)以及3′UTR(7 511~7 563 nt),其中ORF1和ORF2之间有20 bp重叠,ORF2和ORF3之间有1 bp重叠。此序列已收录于GenBank (序列号为AU870455)。
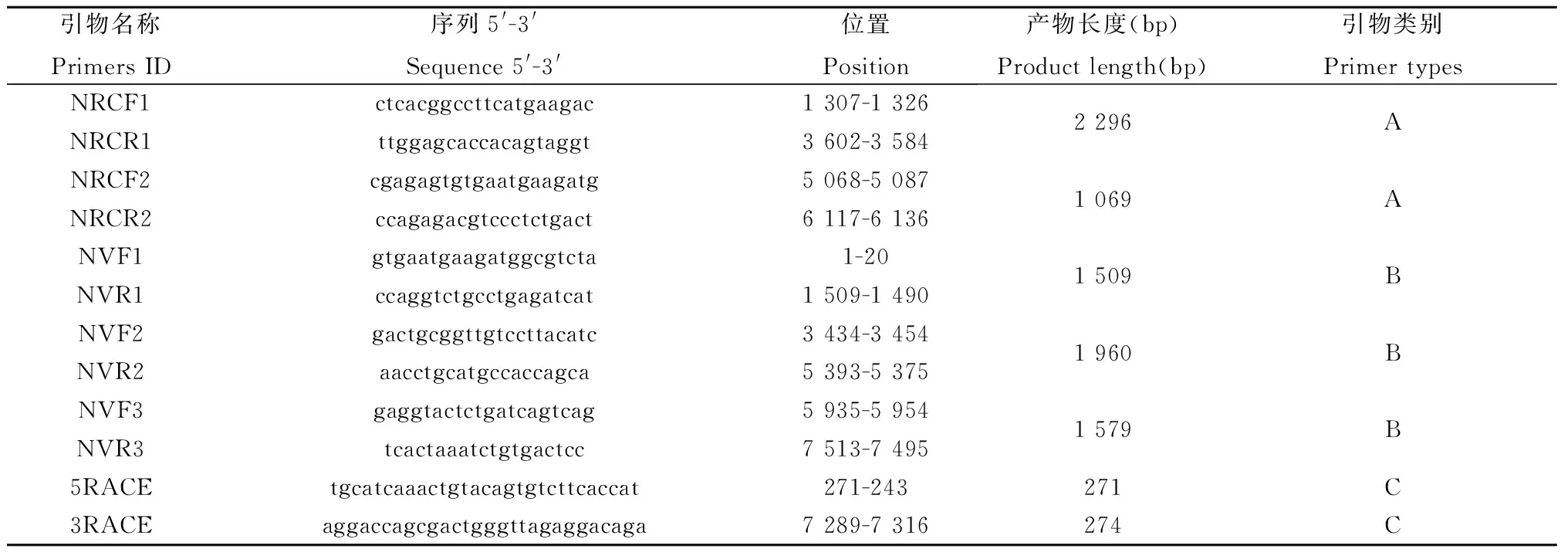
表1 诺如病毒全基因组扩增所用引物
注:A 确证NHBGR59两个Contigo所用的引物;B 用重叠PCR扩增NHBGR59未知片段所用的引物;C扩增NHBGR59两末端序列所用的引物
Note: A Primers used for confirmation of two contigs of NHBGR59;B Primers used for amplification of unknown sequence of NHBGR59;C Primers used for amplification of 3′/5′ end sequences of NHBGR59
2.2病毒序列同源性比较将NHBGR59全基因组核苷酸序列进行Blast比对发现其和GII.6型诺如病毒株30443(GenBank登录号KM198534)同源性最高,为98%,将ORF1、ORF2、ORF3核苷酸和氨基酸分别进行Blast分析发现ORF1的核苷酸和氨基酸序列与引起2014年中国台湾暴发急性胃肠炎的GII.6株11-FJ-5(GenBank号KM461694)同源性最高(99.0%,99.3%)(文章未发表),只在高突变P2区发生一个氨基酸突变位点:aa308D→N(存在P2亚区);而ORF2以及ORF3核苷酸和氨基酸序列与 30443株同源性最高(分别为97.0%,98.0%;99.0%;98.0%)。将NHBGR59 VP1核苷酸和氨基酸和GenBank中所有的GII.6型诺如病毒VP1核苷酸和氨基酸序列比对发现其相似度分别约为80.0%~99.0%,73.0%~99.3%。
2.3病毒进化树分析将NHBGR59分别与已报道的19条NoVs全基因组和15条NoVs VP1区氨基酸参考序列用MEGA 5.0软件(Neighbor-joining的方法)绘制系统进化树。从全基因组进化树(图1A)可以发现其和GII.6型诺如病毒株30443在一个亚枝上,亲缘关系较近。衣壳区进化树(图1B)显示HBGR59与株11-FJ-5亲缘关系最近,而且显示GII.6型诺如病毒株可进一步分为三个亚群(Ⅰ~Ⅲ)。其中NHBGR59 衣壳区的氨基酸和此三个亚群的病毒相似度分别约为73.0%,90.0%和99.0%左右,而且以NHBGR59为代表的Ⅲ群病毒的VP1比Ⅰ和Ⅱ群病毒长三个氨基酸。
3讨论
诺如病毒是人急性胃肠炎暴发流行的重要病原,可引起各年龄段人群急性胃肠炎。从20世纪90年代至今,GⅡ.4基因型一直是该病原的主要的流行株[5-7]。但最近我国发生的两起GII.6型诺如病毒的暴发,提示此型诺如病毒在我国扩散和再次暴发的可能性较大,应该引起我国对GII.6型诺如病毒的高度重视。本研究利用测序的技术对一名由诺如病毒引起的急性胃肠炎的儿童的粪便标本中该型病毒基因组进行扩增、测序、拼接和分析,为我国GII.6型诺如病研究提供了科学基础数据。

A NHBGR59全基因组进化树; B NHBGR59衣壳蛋白区进化树图1 诺如病毒NHBGR59系统进化树 A Phylogenetic tree of complete genome; B Phylogenetic tree of VP1 aa Fig.1 Phylogenetic tree of norovirus strain NHBGR59
诺如病毒尚不能在体外培养,而且也没有合适的动物模型,因此分析其全基因组序列是研究诺如病毒进化和发现新变异株的有效方法。本研究对NHBGR59进行全基因组序列和进化树的分析,表明NHBGR59株属于诺如病毒GII.6型毒株, 同时提示诺如病毒GII.6型毒株具有较大的差异性。主要结构蛋白VP1中P2亚区为诺如病毒的高突变区,含有特异性的抗原位点,且为受体和潜在中和抗体的结合位点[10],因此P2亚区氨基酸抗原位点的分析对预示新的病毒变异株出现具有重要意义。本研究发现NHBGR59的VP1和P2区与2014年中国台湾暴发的11-FJ-5毒株同源性最为相近(99.3%),仅存在一个氨基酸位点的差异(存在于P2区),结合发生时间的先后顺序,提示11-FJ-5可能是来自NHBGR59的变异株,但需要更多的证据。由于缺乏足够有效长度的序列以及其它按时间和地区分布的序列数据,使我们难以分析NHBGR59、上海和台湾暴发的GII.6诺如病毒株彼此间的进化和传播关系。
近期中国发生两起GII.6型诺如病毒暴发,其全基因组序列均未报道,因此河北卢龙株NHBGR59全基因序列可以作为我国乃至全球GII.6型NoVs比对的重要参考序列,对丰富我国NoVs毒株序列具有重要意义。同时,本研究也提示加强我国诺如病毒监测的必要性和重要性,以及时发现和预警新毒株可能引起的大流行。
4参考文献
[1]Patel MM, Hall AJ, Vinje J, et al. Noroviruses: a comprehensive review [J]. J Clin Virol, 2009, 44(1): 1-8. doi: 10.1128/CMR.00011-14.
[2]Bok K, Green KY. Norovirus gastroenteritis in immunocompromised patients [J]. N Engl J Med, 2012, 367(22):2126-2132. doi: 10.1056/NEJMc1301022.
[3]Zheng DP, Andoa T, Fankhauser RL, et al. Norovirus classification and proposed strain nomenclature [J]. Virology, 2006, 346(2): 312-323. doi:10.1016/j.virol.2005.11.015.
[4]Hoa Tran TN, Trainor E, Nakagomi T, et al. Molecular epidemiology of noroviruses associated with acute sporadic gastroenteritis in children: global distribution of genogroups, genotypes and GII.4 variants [J]. J Clin Virol, 2013, 56(3):185-193. doi: 10.1016/j.jcv.2012.11.011.
[5]Zheng DP, Widdowson MA, Glass RI, et al. Molecular epidemiology of genogroup II-genotype 4 noroviruses in the United States between 1994 and 2006 [J]. J Clin Microbiol, 2010, 48(1): 168-177. doi: 10.1128/JCM.01622-09.
[6]田毅, 裴银辉, 靳淼, 等. 诺如病毒广州株GZ20133135全基因组序列测定及分析 [J]. 中华实验和临床病毒学杂志, 2014,28(3):184-186. doi: 10.3760/cma.j.issn.1003-9279.2014.03.009.
[7]Siebenga JJ, Vennema H, Zheng DP, et al. Norovirus illness is a global problem: emergence and spread of norovirus GII.4 variants, 2001-2007 [J]. J Infect Dis, 2009, 200(5): 802-812. doi: 10.1086/605127.
[8]Luo LF, Qiao K, Wang XG. et al. Acute gastroenteritis outbreak caused by a GII.6 norovirus [J]. World J Gastroenterol, 2015, 21(17):5295-5302. doi: 10.3748/wjg.v21.i17.5295.
[9]Chan-It W, Thongprachum A, Khamrin P, et al. Emergence of a new norovirus GII.6 variant in Japan, 2008-2009 [J]. J Med Virol, 2012, 84(7):1089-1096. doi: 10.1002/jmv.23309.
[10]Roth AN, Karst SM. Norovirus mechanisms of immune antagonism[J]. Curr Opin Virol, 2015, 16:24-30. doi: 10.1016/j.coviro.
通信作者:段招军,Email: zhaojund@126.com
DOI:10.3760/cma.j.issn.1003-9279.2016.03.003
基金项目:国家自然科学基金( 81290345)
(收稿日期:2016-03-06)
Complete sequence analysis of norovirus strain NHBGR59 of Hebei Lulong
AoYuanyun,YuJiemei,LiLili,JinMiao,DuanZhaojun
DiarrheaDepartment,NationalInstituteforViralDiseaseControlandPrevention,ChineseCentreforDiseaseControlandPrevention,Beijing100052,ChinaCorrespondingauthor:DuanZhaojun,Email:zhaojund@126.com
【Abstract】ObjectiveTo study the gene and evolution of the complete sequence of NHBGR59 strain isolated from children with diarrhea in Hebei Lulong. MethodsThe full-length genome was amplified based on the two contigs and reference sequence of strain of 30443 (KM198534) by methods of reverse transcription PCR and rapid-amplification of cDNA ends. The PCR products was cloned and sequenced. HBGR59 was performed on sequence analysis and phylogenetic tree. ResultsThe complete sequence of NHBGR59 was 7 563 bp (excluding PolyA tail), including three open reading frames (ORFs): ORF1 (5-5 098 nt), ORF2 (5 079-6 731 nt) and ORF3(6 731-7 510 nt), with 20 nucleotide (nt) overlapping in ORF1 and ORF2, and 1 nt overlapping in ORF2 and ORF3. By sequence analysis, NHBGR59 showed the highest homology in complete sequence with norovirus strain 30443 (98% identity), and the ORF2 and ORF3 also showed the highest identity to strain 30443 (97.0% and 99.0% in nt; 98.0% and 98.0% in amino acid (aa)), while the ORF2 displayed the highest homology (99.0% in nt, 99.3% in aa) with the strain 11-FJ-5 (KM461694), which was related to GII.6 norovirus gastroenteritis outbreaks in Taiwan in 2014, and only one amino acid was different (aa308D→N in P2 region) between them. The phylogenetic analysis showed NHBGR59 clustered tightly with strains of 30443 and 11-FJ-5 in norovirus genogroup II genotype 6 (GII.6). ConclusionsNHBGR59 belongs to norovirus GII.6, which is the mostly close to the recently reported strain 30443 and 11-FJ-5.
【Key words】Norovirus; Genogroup II genotype 6; Phylogenetic tree; Genome

