葡萄与咖啡基因组的多倍化过程及共线性分析
李育先,夏瑞燕,王金朋,王希胤
(华北理工大学生命科学学院基因组学与计算生物学研究中心,河北唐山063000)
葡萄与咖啡基因组的多倍化过程及共线性分析
李育先,夏瑞燕,王金朋,王希胤
(华北理工大学生命科学学院基因组学与计算生物学研究中心,河北唐山063000)
葡萄;咖啡;全基因加倍;重复基因;同源片段
以葡萄和咖啡的基因组为研究对象,基于比较基因组学和生物信息学的研究方法,通过比较基因组结构上的同源关系,确定物种进化中基因组加倍的性质,获取由全基因组加倍产生的重复基因。研究表明:葡萄和咖啡都发生过一次古老的全基因三倍化事件,产生了大量的同源重复基因;研究获得了种内、种间的同源基因,对物种间的同源基因进行了区分,并构建了葡萄和咖啡基因组的联合比对图谱,对这一结果进行形象地展示;在葡萄、咖啡基因组内分别获取了1 427(5.4%)个、1 543(6.0%)个由多倍化产生的重复基因对,在2个物种基因组间获取了7 462个重复基因对,为深入研究葡萄和咖啡基因组的进化提供了参考。
全基因组加倍(Whole-genome polyploidization)是被子植物基因组进化过程中的重要特征[1],被子植物在约200百万年前的进化过程中经历了持续的全基因组加倍事件。如双子叶植物的祖先在约1亿3 000万年前共同经历了一次古老的全基因组三倍化事件[1],此后大豆独立发生了一次全基因组二倍化事件[2],棉花则独立发生了一次全基因的五倍化事件[3]。在单子叶植物中,禾本科的共同祖先物种在约9 600万年前发生了一次全基因组二倍化事件[4,5],此后,玉米又发生了一次全基因组的二倍化事件[6]。持续的多倍化事件使得现有的被子植物常常形成多倍体[7],在全基因组加倍事件发生时,还常常伴随着染色体片段的融合、丢失和断裂等事件,使整个基因组实现重排,来继续保持基因组二倍性的特点,这使得祖先基因分配到多条染色体中去,它们的子基因组在基因的排列和基因组的大小上表现出明显的差异,也使得要了解被子植物的基因组进化变得十分困难[8]。丰富的研究还表明,多倍化作为被子植物物种分化和进化的主要动力,可能有助于被子植物的起源,一个明显的事实是多倍化之后,禾本科在地球上迅速扩增,并使它们成为地球上的主要植物类群之一[13]。随着越来越多物种的全基因组完成测序,多倍化事件在植物进化过程中的广泛性已逐渐为人所知。全基因组加倍产生了成千上万的重复基因,研究表明重复基因为遗传创新提供了重要的材料来源。这些重复基因通过基因功能分化、同源基因的置换、基因表达的变化、拷贝基因的剂量效应、调控路径和功能网络的调节等实现了基因组的遗传创新,使得加倍后的物种能成功地生存下来[11。
葡萄隶属于葡萄科,是重要的水果和饮料原料,是第4个测序的开花植物,第1个测序的水果作物[19]。咖啡隶属于茜草科,是世界上种植最广泛的热带经济作物,全球的种植面积超过1 100万公顷,每天的消费量超过了22.5亿杯[20]。目前2个物种的全基因组均以测序并注释完成[19-20],这为2个物种基因组的比较分析提供了重要的研究原料。研究对葡萄和咖啡的全基因组进行深入地比较基因组学分析,通过分析基因组间和基因组内的结构同源关系,并利用这种同源信息,确定各物种全基因加倍事件发生的规模,并获取这些事件产生的重复基因片段,区分物种间的直系同源基因和种外旁系同源基因,为2个物种基因组的其他相关研究提供了参考。
1 材料和方法
1.1 全基因组序列数据
葡萄(Vitis vinifera)的全基因组序列来自于植物比较基因组学平台Phytozome v11:https://phytozome.jgi.doe.gov/pz/portal.html#;咖啡(Coffea canephora)的全基因组序列来自于咖啡基因组数据库Coffee Genome Hub[21]:http://coffee-genome.org/。
1.2 全基因组的同源比对与结构解析
为确定基因组内以及基因组间的片段同源关系,研究中去除了原始基因组数据中没有锚定染色体的基因组信息,并使用序列相似性比对软件BLAST,对葡萄和咖啡基因组内、间的全基因组序列进行比对,获得任意基因对在基因组内和基因组间的相似性信息。为保证基因序列间相似性信息的可靠性,研究对结果进行了严格的阈值限定与筛选(E-value<10-5,Score≥100)。
在基因组序列同源比对结果的基础上,依据每个基因在染色体上的物理位置或相对位置,构建葡萄和咖啡基因组内、间的结构同源性点阵图。分析同源性点阵图展示的基因组结构特征,推测2个物种在基因组层面发生的全基因组加倍事件。这一过程主要基于Perl语言编程,利用编写的内部程序,读取BLAST的格式化输出文件,生成结构同源性点阵图。
1.3 推断并获取基因组片段的同源共线关系
利用多重序列共线搜索工具McScan[22]和ColinearScan[23],搜索基因组内和基因组间的同源共线片段,结合基因组结构的同源分析结果,区分开不同加倍事件产生的基因组内和基因组间的重复基因,整合所得结果,生成两物种同源基因对的联合比对图谱。
2 结果与分析
将物种内的同源性点阵图和物种间的同源性点阵图相结合,可以推断2个物种在基因组层面发生过的全基因组加倍事件,区分不同全基因组加倍事件产生的重复基因。研究利用结构同源性点阵图对葡萄、咖啡基因组的内部结构进行了解析,印证了咖啡和葡萄共有的古老全基因加倍事件的存在[19,21]。
2.1 基因组内的同源比对与结构解析
从葡萄和咖啡的基因组内同源性点阵图,可以清楚看到葡萄和咖啡共同发生过古老的全基因组三倍化事件,这次事件即是在双子叶祖先中发生的,如图1所示。
图1展示了葡萄和咖啡各自基因组内的结构同源性点阵图,以图1(a)为例:二维平面图中的横轴从左向右依次排列的矩形框代表葡萄的19条染色体,纵轴从上到下依次排列的矩形框也代表葡萄的19条染色体,基因按在染色体上的实际位置顺序在各条染色体上依次排列;图中每个矩形框内的有色点代表对应染色体上序列相似的同源基因对,其中红色的点代表序列相似性最好的同源基因基因对,蓝色的点代表序列相似性次好的同源基因对,灰色的点代表其他序列相似性较差的同源基因对。
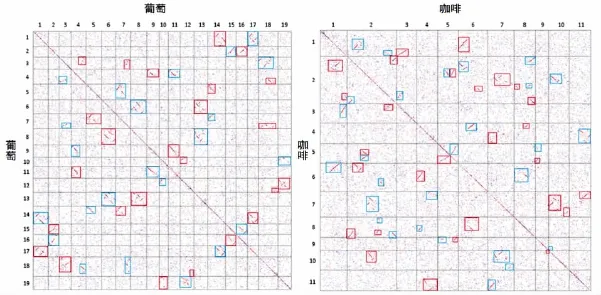
图1 基因组内同源性点阵图
从图1中能够观察到大量的同源基因对构成了一系列的同源线(同源片段),这些线上的基因对即是由基因加倍产生的重复基因。从图1中可以发现,2个物种在基因组内都保留了大量的同源共线片段。葡萄(缩写Vv)基因组内较大的同源共线片段主要存在于染色体Vv1-Vv14,Vv1-Vv17,Vv2-Vv15,Vv2-Vv16,Vv3-Vv4,Vv3-Vv7,Vv3-Vv18,Vv4-Vv9,Vv4-Vv10,Vv4-Vv18,Vv5-Vv7,Vv5-Vv14,Vv6-Vv8,Vv6-Vv13,Vv7-Vv14,Vv7-Vv18,Vv8-Vv13,Vv9-Vv11,Vv10-Vv12,Vv10-Vv19,Vv12-Vv18,Vv12-Vv19,Vv14-Vv17,Vv15-Vv16;咖啡(缩写Cc)基因组内较大的同源共线片段主要存在于染色体Cc1-Cc2,Cc1-Cc3,Cc1-Cc6,Cc2-Cc2,Cc2-Cc3,Cc2-Cc5,Cc2-Cc6,Cc2-Cc7,Cc2-Cc8,Cc2-Cc10,Cc4-Cc6,Cc4-Cc7,Cc4-Cc8,Cc4-Cc11,Cc5-Cc5,Cc5-Cc9,Cc6-Cc8,Cc7-Cc10,Cc7-Cc11,Cc9-Cc10。
图1(a)显示,这些由重复基因构成的同源共线片段可以明显的分为2组,即每个葡萄的染色体同源片段在基因组内呈现1:2的对应关系,如葡萄的1号染色体与葡萄的14和17号染色体分别对应。为了区分2组同源共线片段,分别使用红色和蓝色的矩形将2次同源拷贝进行了标记。这是因为双子叶植物的共同祖先在约1.3亿年前发生了一次全基因组的三倍化事件(Triplication)[19],这次事件使物种的每个基因生成了另外2个同源拷贝,之后在经历一系列的染色体片段的断裂、融合等进化过程形成了现在的19条葡萄染色体。咖啡与葡萄有相类似的情况,它的祖先基因组也发生了这次全基因组的三倍化事件[21],因此反映在同源性点阵图上,如图1(b)所示,基因组内部的同源共线片段也表现出1:2的对应关系。
2.2 基因组间的同源比对与结构解析
图2展示了葡萄-咖啡基因组间的结构同源性点阵图:二维平面图中的横轴从左向右依次排列的矩形框代表葡萄的19条染色体,纵轴从上到下依次排列的矩形框代表咖啡的11条染色体,基因按实际位置的排列顺序在各条染色体上依次排列;图中每个矩形框内的有色点代表对应染色体上序列相似的同源基因对,分别使用红色、蓝色和灰色的点代表序列相似性最好、次好和较差的同源基因对。
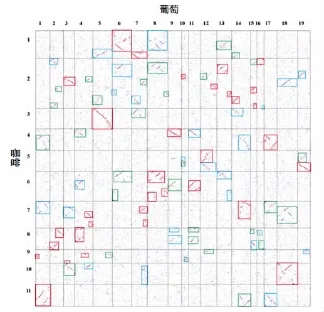
图2 葡萄-咖啡基因组间同源性点阵图
从葡萄和咖啡基因组间的同源性点阵图,如图2所示,葡萄和咖啡基因组间呈现出明显的1:3对应关系,即每个葡萄的染色体对应3组咖啡的同源共线片段,每个咖啡的染色体也对应3组葡萄的同源共线片段。并且这3组同源片段都可以清楚的分为1组最好的同源片段(红色矩形框)和2组较差的同源片段(蓝色和绿色矩形框)。如葡萄的17号染色体与咖啡的4、7和11号染色体分别对应,其中,葡萄的17号染色体与咖啡的4号染色体是真正的直系同源片段,在图中呈现红色的线,葡萄的17号染色体与咖啡的7号、11号染色体是种外的旁系同源片段,在图中呈现蓝色的线。这说明2个物种在分化形成后都没有独立发生全基因层面的加倍事件,也即表现为1:1的最好直系同源关系;2个物种共同发生过一次全基因组的三倍化事件,也即表现为1:2的次好种外旁系同源关系。这一结论与前面二者各自基因组内的同源比对结果保持了一致,也与此前2个物种基因组的相关研究保持了一致[19,21]。即是说双子叶的祖先在分化为葡萄和咖啡2个物种后,没有发生独立的全基因加倍事件。
2.3 基因组的同源共线性分析
利用前面提到的共线搜索软件,结合基因组结构分析的结果,对葡萄和咖啡基因内、间的同源共线基因进行了严格的搜索。在葡萄基因组内,共得到86个旁系同源共线片段,共包含了1 427个旁系同源基因对,平均每个片段包含约16.6个旁系同源基因对,其中长度大于4、10、20、50的旁系同源共线片段的个数分别是312、126、47和0,最长的旁系同源共线片段分布在葡萄的4号和18号染色体之间,包含了61个旁系同源共线基因对;在咖啡基因内,共得到92个旁系同源共线片段,共包含了1 543个旁系同源共线基因对,平均每个片段包含约16.8个旁系同源基因对,其中长度大于4、10、20、50的旁系同源共线片段的个数分别是263、46、26和5,最长的旁系同源共线片段分布在咖啡的1号和6号染色体之间,包含了91个旁系同源基因对。在葡萄和咖啡的基因组间共得到了149个直系同源片段和153个种外旁系同源片段,其中直系同源片段包含了7 462个基因对,平均每个片段包含约50.0个基因对,旁系同源片段包含了2 924个种外旁系同源基因对,平均每个片段包含约19.1个基因对,物种间最长的同源片段主要存在于葡萄的5号和咖啡的3号染色体之间,包含了327个同源基因对。
图3展示了以葡萄和咖啡基因组的同源关系为基础的,两个物种同源基因的共线联合比对关系。图中共包括6个圈,第1、3、5圈和第2、4、6圈分别代表了葡萄(V)和咖啡(C)的3组旁系同源基因。利用19条葡萄的染色体为参考(图例中的19种颜色),依据葡萄和咖啡基因组内保留的旁系同源基因、葡萄与咖啡之间保留的直系同源基因和种外旁系同源基因,绘制了图3所示的2个物种的同源联合比对图。
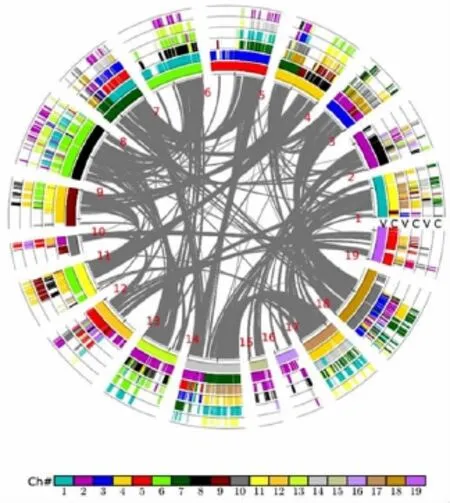
图3 葡萄-咖啡基因组的同源联合比对图
利用前面基因组间结构解析的结果,即物种间1:1的直系同源关系,1:2的旁系同源基因关系,将搜索得到的同源共线基因,依据物种间的同源关系进行整合排列。研究中将2个基因组的比对信息,生成了一张2个物种的同源联合比对图,如图3所示,以更好的展示物种内、间的片段同源关系。上面的结果显示,咖啡基因组内的同源共线片段和同源基因对的数量要高于葡萄中的相应数量,但是2个物种内同源共线基因对的平均数量基本保持了一致,分别是16.6和16.8。由于2个物种在分化后都没有发生独立的全基因组加倍事件,因此表现为同源共线片段的平均基因对数量十分接近。全基因组加倍常伴随着不一致的基因丢失,这很可能是物种分化的一个驱动力[5,22]。葡萄和咖啡内部同源共线片段和同源基因对的数量不同,很可能是因为葡萄在进化过程中发生了更严重的染色体片段的丢失事件,导致其比咖啡丢失了更多的重复基因,表现为基因组内同源共线片段和同源基因对的数量要低于咖啡中的这一数量。
3 结论
(1)物种基因组层面的多倍化事件增加了其基因组内容的复杂性,也为物种的遗传进化提供了原材料[1]。该项研究通过比较葡萄和咖啡2个基因组的结构特征,清晰地印证了咖啡和葡萄基因组中曾经发生的古老全基因组三倍化事件;通过严格的共线搜索,确定了2个物种内、物种间的同源共线片段,并依据直系同源的相似性高于种外旁系同源的原则,对不同物种间的同源共线片段进行了区分。研究在葡萄、咖啡基因组内分别得到了由多倍化产生的1 427个、1 543个重复基因对,分别占2个物种基因组大小的5.4%和6.0%,在2个物种基因组间得到了7 462对重复基因对,这为进一步研究葡萄和咖啡基因组的进化提供了参考。
(2)重复基因的分析还发现,咖啡比葡萄保留了更多的重复基因数量,这是因为2个物种在全基因加倍后的进化过程中,基因的丢失比率并不一致,并且葡萄中重复基因的丢失比率更高。基因组加倍产生的重复基因为物种的进化提供了原料和基础,在伴随着差异性的丢失频率,加倍产生的重复基因很可能间接促进了物种的分化。
参考文献:
[1] TANG HB,BOWERS JE,WANG XY,et al.Synteny and collinearity in plant genomes[J].Science.2008,320(5875):486-488.
[2] SCHMUTZ J,CANNON SB,SCHLUETER J,et al.Genome sequence of the palaeopolyploid soybean[J].Nature.2010,463(7278):178-183.
[3] WANG XY,GUO H,WANG JP,et al.Comparative genomic de‐convolution of the cotton genome revealed a decaploid ancestor and widespread chromosomal fractionation[J].New Phytologist.2015.
[4] FUJISAWA M,BABA T,NAGAMURA Y,et al.The map-based sequence of the rice genome[J].Nature,2005,436(436):793-800.
[5] WANG XY,WANG JP,JIN D,et al.Genome Alignment Spanning Major Poaceae Lineages Reveals Heterogeneous Evolutionary Rates and Alters Inferred Dates for Key Evolutionary Events[J].Mol Plant.2015,8(6):885-898.
[6] SCHNABLE PS,WARE D,FULTON RS,et al.The B73maize genome:complexity,diversity,and dynamics[J].Science.2009,326(5956):1112-1115.
[7] BOWERS JE,CHAPMAN BA,RONG J,et al.Unravelling angiosperm genome evolution by phylogenetic analysis of chromosomal duplication events[J].Nature.2003,422(6930):433-438.
[8] WANG XY,SHI XL,HAO BL,et al.Duplication and DNA segmental loss in the rice genome:implications for diploidization[J].New Phytologist.2005,165(3):937-946.
[9] LYSAK MA,CHEUNG K,KITSCHKLE M,et al.Ancestral chromosomal blocks are triplicated in Brassiceae species with varying chromosome number and genome size[J].Plant Physiol.2007,145(2):402-410.
[10] GORDON JL,BYRNE KP,WOLFE KH.Additions,losses,and rearrangements on the evolutionary route from a reconstructed ancestor to the modern Saccharomyces cerevisiae genome[J].PLoS Genet.2009,5(5):e1000485.
[11] WANG XY,TANG HB,BOWERS JE,et al.Comparative inference of illegitimate recombination between rice and sorghum duplicated genes produced by polyploidization[J].Genome Res.2009,19(6):1026-1032.
[12] GAETA RT,CHRIS PJ.Homoeologous recombination in allopolyploids:the polyploid ratchet[J].The New phytologist.2010,186(1):18-28.
[13] JIAO Y,WICKETT NJ,AYYAMPALAYAM S,et al.Ancestral polyploidy in seed plants and angiosperms[J].Nature.2011,473(7345):97-100.
[14] QIAN W,ZHANG J.Gene dosage and gene duplicability[J].Genetics.2008,179(4):2319-2324.
[15] COATE JE,DOYLE JJ.Quantifying whole transcriptome size,aprerequisite for understanding transcriptome evolution across species:an example from a plant allopolyploid[J].Genome biology and evolution.2010,2:534-546.
[16] RATNAPARKHE MB,WANG XY,LI JP,et al.Comparative analysis of peanut NBS-LRR gene clusters suggests evolutionary innovation among duplicated domains and erosion of gene microsynteny[J].New Phytologist.2011,192(1):164-178.
[17] DAVIDSON RM,GOWDA M,MOGHE G,et al.Comparative transcriptomics of three Poaceae species reveals patterns of gene expression evolution[J].The Plant journal:for cell and molecular biology.2012,71(3):492-502.
[18] SOLTIS DE,BELL CD,KIM S,et al.Origin and early evolution of angiosperms[J].Annals of the New York Academy of Sciences.2008,1133:3-25.
[19] JAILLON O,AURY JM,NOEL B,et al.The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla[J].Nature.2007,449(7161):463-467.
[20] DENOEUD F,CARRETERO-PAULET L,DEREEPER A,et al.The coffee genome provides insight into the convergent evolution of caffeine biosynthesis[J].Science.2014,345(6201):1181-1184.
[21] DEREEPER A,BOCS S,ROUARD M,et al.The coffee genome hub:a resource for coffee genomes[J].Nucleic Acids Res.2015,43(Database issue):D1028-1035.
[22] TANG HB,WANG XY,BOWERS JE,et al.Unraveling ancient hexaploidy through multiply-aligned angiosperm gene maps[J].Genome Research.2008,18(12):1944-1954.
[23] WANG XY,SHI XL,LI Z,et al.Statistical inference of chromosomal homology based on gene colinearity and applications to Arabidopsis and rice[J].BMC bioinformatics.2006,7.
Analysis of Collinearity and Polyploidization Process of Grape and Coffee Genome
LI Yu-xian,XIA Rui-yan,WANG Jin-peng,WANG Xi-yin
(College of Life Sciences,North China University of Science and Technology,Tangshan Hebei 063000,China)
grape;coffee;whole-genome polyploidization;duplication gene;homologous fragment
Based on the research methods of comparative genomics and bioinformatics,and the homologous relationship of the genome structure of grape and tomato,the properties of the genome duplication in the evolution of the two species and the duplicate genes produced by whole genome duplication were determined through this analysis.The research results showed that grape and coffee had experienced an ancient whole genome triplication events,and a large number of homologous duplication genes were produced;The homologous genes in this two species were obtained,and the homologous genes of the species were distinguished,and a united alignment of grape and coffee genome was constructed.1 427(5.4%)and 1 543(6.0%)duplicated gene pairs which produced by polyploidization was obtained in the genomes of grapes and coffee,respectively.7 462duplication gene pairs were identified in the grapes and coffee genome,which provides a reference for the further study of the evolution of the grape and coffee genomes.
Q343.1
A
2095-2716(2016)04-0038-07
2016-05-23
2016-09-27
华北理工大学研究创新项目(2015S24);华北理工大学大学生创新创业训练计划项目(X2015165)。
——紫 苏

