不同温度下大鼠脑组织RNA降解与早期PMI的相关性
吕叶辉,李志宏,托 娅,刘 丽,李 堃,卞 杰,马剑龙,陈 龙(.上海健康医学院基础医学院,上海 038;.复旦大学基础医学院法医学系,上海 0003)
不同温度下大鼠脑组织RNA降解与早期PMI的相关性
吕叶辉1,李志宏1,托娅1,刘丽1,李堃1,卞杰1,马剑龙2,陈龙2
(1.上海健康医学院基础医学院,上海 201318;2.复旦大学基础医学院法医学系,上海 200032)
摘要:目的 探讨大鼠脑组织8种RNA指标,在不同温度下的表达水平与早期死亡时间(PMI)的相关性。方法 将222只SD大鼠随机分为对照组(死后0h)和4个实验组,实验组断颈处死后分别置于5℃、15℃、25℃和35℃的环境中,于死后1~24 h内9个时间点提取脑组织RNA,利用实时荧光定量PCR技术检测8种RNA指标(β-actin、GAPDH、RPS29、18S rRNA、5S rRNA、U6 snRNA、miRNA-9及miRNA-125b)的表达水平,geNorm软件选取合适内参,SPSS软件对内参标准化RNA指标进行回归分析,R软件构建推断PMI的数学模型,另选6只已知PMI的SD大鼠予以验证。结果 5S rRNA、miR-9和miR-125b表达稳定,可作为内参指标。β-actin和GAPDH具有良好的时序性降解规律,在24h内随PMI延长不断降解。R软件拟合得ΔCt值随PMI和温度变化的数学模型可用以推断PMI。运用β-actin和GAPDH验证模型的平均误差率分别为14.1%和22.2%。结论 β-actin和GAPDH表达水平与PMI和环境温度相关性良好。本研究建立的数学模型可为温度变化条件下的早期PMI推断提供参考。
关键词:法医病理学;RNA;死亡时间;温度;降解;大鼠
死亡时间(PMI)是指发现、检查尸体时距死亡发生时的时间间隔,PMI的精确推断有助于迅速确定涉案罪犯以及排除嫌疑人,为侦查部门提供重要线索,并为司法实践提供重要科学依据。利用传统的方法进行PMI推断,其结果易受主观因素和外界环境因素的影响,导致推断的精确性较低。
随着分子生物学技术在法医学领域的广泛应用,利用RNA的时间依赖性降解规律推断PMI正逐渐成为研究热点[1],但针对RNA在不同温度下早期PMI降解规律的研究较少。本研究利用实时荧光定量
1 材料与方法
1.1仪器与试剂
仪器:9700型PCR扩增仪、Prism 7500型荧光定量PCR仪(美国AB公司),NanoDrop 1000分光光度计(美国Thermo公司),Agilent 2100生物分析仪(美国Agilient公司)。
试剂:TRIzol试剂盒(美国 Invitrogen公司),RNAlater保护液、PrimeScriptTMRT试剂盒、One Step PrimeScriptTMmiRNA cDNA Synthesis试剂盒、SYBR® Premix Ex TaqTM试剂盒(日本TaKaRa公司),DEPC水(焦碳酸二乙酯处理并经高温高压灭菌的MiliQ纯水)。
1.2实验动物分组及处理
清洁级成年雄性SD大鼠228只,体质量(220± 20)g,由复旦大学上海医学院实验动物部提供,随机选取其中6只备用,其余222只大鼠断颈处死后随机分为对照组(死后即刻0h,6只)和4个实验组(每组54只)。实验组大鼠尸体分别置于(5±1)℃、(15±1)℃、(25±1)℃和(35±1)℃环境中,并分别在死后1、3、6、9、12、15、18、21、24 h(每个时间点6只)提取大鼠额叶脑组织50~100mg。另取6只备用的大鼠,断颈处死后随机分为3组,尸体分别置于(10±1)℃、(20±1)℃和(30±1)℃环境中,并分别在死后10、20h(每个时间点1只)提取大鼠额叶脑组织50~100 mg,用以验证数学模型。所取脑组织均用无菌眼科剪剪碎后分装于RNAlater保护液中,置于-80℃冻存。
1.3总RNA提取
准确称取脑组织质量,加入液氮研碎组织后转入匀浆器中,按照TRIzol试剂盒总RNA提取说明书操作步骤提取总RNA,加入30μL DEPC水溶解后置于-80℃冻存。NanoDrop 1000分光光度计测定RNA的纯度及浓度,用光密度(D)表示,并检测D260/D280值。Agilent 2100生物分析仪测定RNA完整性,用RNA完整性指数(RNA integrity number,RIN)表示。
1.4指标选择
本研究选择8种RNA指标,分别为β-肌动蛋白(β-actin)、甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、核糖体蛋白S29 (ribosomal protein S29,RPS29)、18S核糖体RNA(18S ribosomal RNA,18S rRNA)、5S核糖体RNA(5S ribosomal RNA,5S rRNA)、U6微核RNA(U6 small nuclear RNA,U6 snRNA)、微小RNA-9(miRNA-9,miR-9)及微小RNA-125b(miRNA-125b,miR-125b)。引物设计至少跨一个外显子或外显子区域以保证cDNA正确的合成与扩增,扩增产物片段长度均在200bp以下。引物由上海生工生物技术有限公司合成,引物序列见表1。
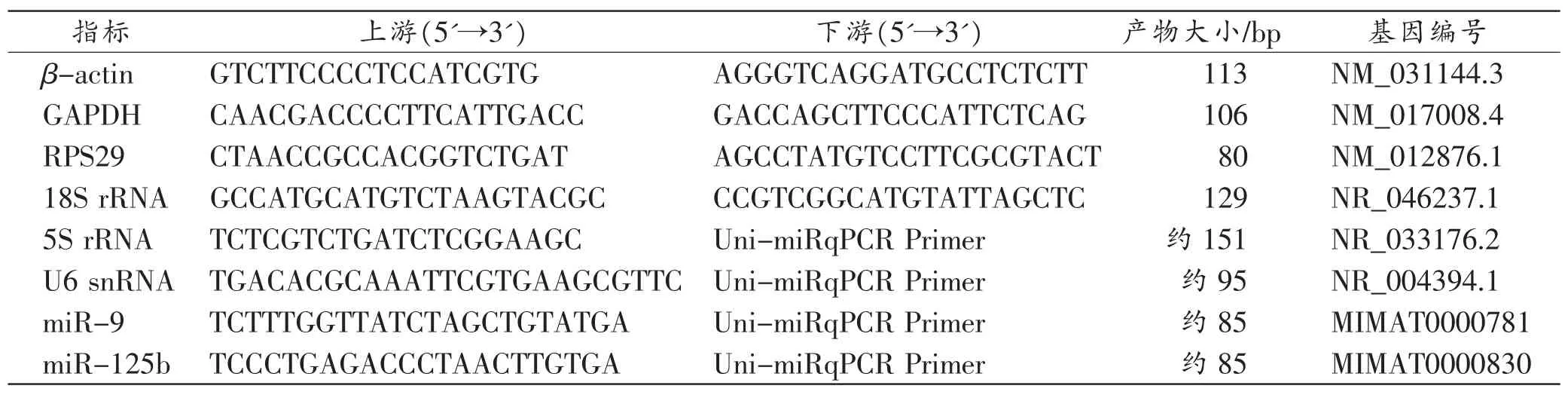
表1 实验选取的RNA指标及引物序列
1.5总RNA反转录
应用PrimeScriptTMRT试剂盒对总RNA进行反转录,以用于β-actin、GAPDH、RPS29、18S rRNA的实时荧光定量PCR。取RNA样本500 ng进行反转录,按照试剂盒说明书配置10μL反应体系。反应条件:37℃ 15 min,85℃ 5 s,1个循环。应用One Step PrimeScriptTMmiRNA cDNA Synthesis试剂盒对总RNA进行反转录,以用于5S rRNA、U6 snRNA、miR-9、miR-125b的实时荧光定量PCR。取RNA样本500ng进行反转录,按照试剂盒说明书配置10μL反应体系,反应条件:37℃ 60 min,85℃ 5 s,1个循环。将反转录产物cDNA以1∶10稀释,置于-20℃冻存。
1.6实时荧光定量PCR
应用SYBR®Premix Ex TaqTM试剂盒进行实时荧光定量PCR反应。按照试剂盒说明书配制20 μL反应体系,并应用Prism 7500型荧光定量PCR仪进行扩增。扩增条件:95℃ 30s,1个循环;95℃ 5s,60℃34 s,40个循环;95℃ 15 s,60℃ 1 min,95℃ 15 s,1个循环。每个样本重复三次,结果由系统自动生成,从而得到各个RNA指标的循环阈值(cycle threshold,Ct值)。
1.7统计学分析
1.7.1RNA浓度及完整性检测
运用SPSS v22.0(美国SPSS公司)对RNA单位质量浓度及RIN进行统计分析,多组间运用单因素方差分析,两组间运用t检验进行差异性分析。检验水准α=0.05。
1.7.2内参选择
运用geNorm v3.5软件(比利时Biogazelle公司)[2]对RNA指标的Ct值进行统计学处理。根据平均表达稳定度M值确定最稳定的指标,M值越小,指标的稳定性越高。根据平均表达稳定度的配对变异V值判断引入新的内参指标是否会对标准化因子产生显著影响,默认V值为0.15,如果Vn/Vn+1(n>1)小于0.15(当多个V值符合时选取最小值),说明n或n+1个RNA指标可以作为内参指标使用。除内参指标外,其余RNA指标作为候选指标。
1.7.3模型建立
运用SPSS v22.0、Excel 2010(美国Microsoft公司)软件对候选RNA指标 Ct值进行内参标准化(ΔCt=Ct候选指标-Ct内参均值),内参均值为内参指标的几何平均数,并对不同温度组下候选RNA指标ΔCt值及PMI进行三次方回归分析并计算相关系数r,平均相关系数较高的候选指标将被用于构建模型。运用R v3.2.3软件(美国RStudio公司)构建以温度(x)、PMI(y)为自变量,ΔCt值(z)为因变量的三元二次方程,即z=k1x2+k2x+k3xy+k4y+k5y2+k0。使用MATLAB v7.0软件(美国MathWorks公司)将方程转为三维可视化数学模型。检验水准α=0.01。
1.7.4模型验证
将已知PMI的备用大鼠样本数据代入模型进行验证,并计算推断PMI和实际PMI之间的差异程度,用推断误差及误差率表示,推断误差=|PMI推断-PMI实际|,误差率=|PMI推断-PMI实际|/PMI实际×100%。
2 结果
2.1RNA纯度、浓度、完整性及实时荧光定量PCR结果
测得所有RNA样本的D260/D280均在1.8~2.0,证明RNA纯度较高。各组间RNA单位质量浓度差异无统计学意义(P>0.05)。对照组RIN值为9.45±0.08 (9.2~9.7),5℃组为9.20±0.11(8.6~9.6),15℃组为8.40±0.25(7.2~9.6),25℃组为7.42±0.55(4.8~9.2),35℃组为7.06±0.73(3.5~9.4)。与对照组相比,5℃组RIN值差异无统计学意义(P>0.05),而15℃、25℃和35℃组RIN值降低(P<0.05),表明在较高温度下RNA逐渐降解,其完整性随PMI延长逐渐降低。
所有样品均成功反转,得到Ct值。扩增曲线和溶解曲线完好,表明样本均成功特异性扩增。
2.2内参选择结果
4个不同温度组的RNA指标的平均表达稳定度M值及标准化因子的配对变异Vn/Vn+1值,如表2所示。以5℃组为例,RNA指标平均表达稳定度M值由高到低排序依次为β-actin>GAPDH>18S rRNA>RPS29>U6 snRNA>miR-9>5S rRNA=miR-125b,表明5S rRNA和miR-125b稳定性最好,而β-actin和GAPDH较不稳定;相比其他Vn/Vn+1值,V3/V4小于0.15且最小,表明选择3个或4个RNA指标作为5℃组内参最为合适,即miR-9、5S rRNA、miR-125b或U6 snRNA、miR-9、5S rRNA、miR-125b。

表2 不同温度组RNA指标的稳定性对比
以此类推,15℃组内参为miR-9、5S rRNA、miR-125b或 U6 snRNA、miR-9、5S rRNA、miR-125b;25℃组内参为miR-9、miR-125b或5S rRNA、miR-9、miR-125b;35℃组内参为miR-9、miR-125b或5S rRNA、miR-9、miR-125b。
综合4个温度组对RNA指标进行分析,5S rRNA、miR-9和miR-125b在各温度组均有较好的稳定性;U6 snRNA在5℃、15℃组相对稳定,但在25℃、35℃组稳定性下降;β-actin和GAPDH在4个温度组中均不稳定。因此,4个温度组均选择5S rRNA、miR-9和miR-125b作为内参指标,其余RNA指标作为候选指标。
2.3PMI推断的模型建立
对候选RNA指标的Ct值进行内参标准化,即Ct内参均值=(Ct5S rRNA×CtmiR-9×CtmiR-125b)1/3。利用统计软件对4个温度组内5种候选RNA指标的ΔCt值和PMI进行三次方回归分析并计算相关系数r,如表3所示。
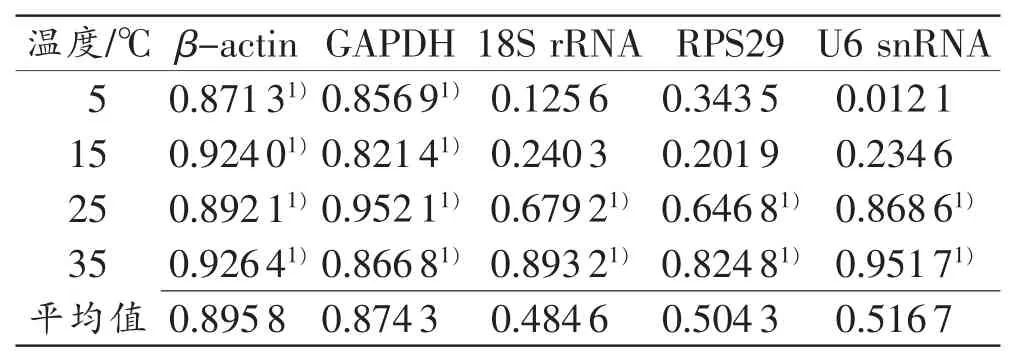
表3 不同温度组内参标准化的RNA指标与PMI的相关性分析(r值)
结果表明,18S rRNA、RPS29和U6 snRNA在5℃、15℃时与PMI相关性较低,但在25℃、35℃时与PMI相关性提高;β-actin和GAPDH在4个温度组内都与PMI有着良好的相关性,且β-actin和GAPDH的平均r值均高于0.87;因此选择β-actin和GAPDH用于构建推断PMI的数学模型。
运用R v3.2.3软件构建三元二次方程,统计结果表明x、xy、y、y2、k0项具有统计学意义(P<0.01),重新计算后得到β-actin的方程为z=0.0048x+0.0023xy+ 0.033 1 y-0.001 5 y2+3.567 7,GAPDH的方程为z= -0.002 7 x+0.003 0 xy+0.028 3 y-0.000 5 y2+2.650 8;决定系数R2分别为0.9375和0.9017,表明拟合优度较高。
使用MATLAB v7.0软件将方程转为三维可视化数学模型,β-actin和GAPDH的模型如图1~2所示。结果显示,早期PMI内,β-actin和GAPDH的ΔCt值随着PMI延长不断增加,而且ΔCt值增加的速率不仅与PMI相关,也与环境温度相关。
2.4PMI推断的模型验证
运用R软件将6只备用大鼠样本的ΔCt值及温度分别代入β-actin、GAPDH模型进行验证,分别得到两者的PMI推断值,并计算推断PMI与实际PMI的误差率,如表4所示。运用β-actin模型进行PMI推断,平均推断误差为1.9h,平均误差率为14.1%;运用GAPDH模型进行PMI推断,平均推断误差为3.0h,平均误差率为22.2%。对于环境温度较高(30℃)、PMI较长(20 h)的样本,运用该模型进行PMI推断时,推断误差和误差率相对较低。
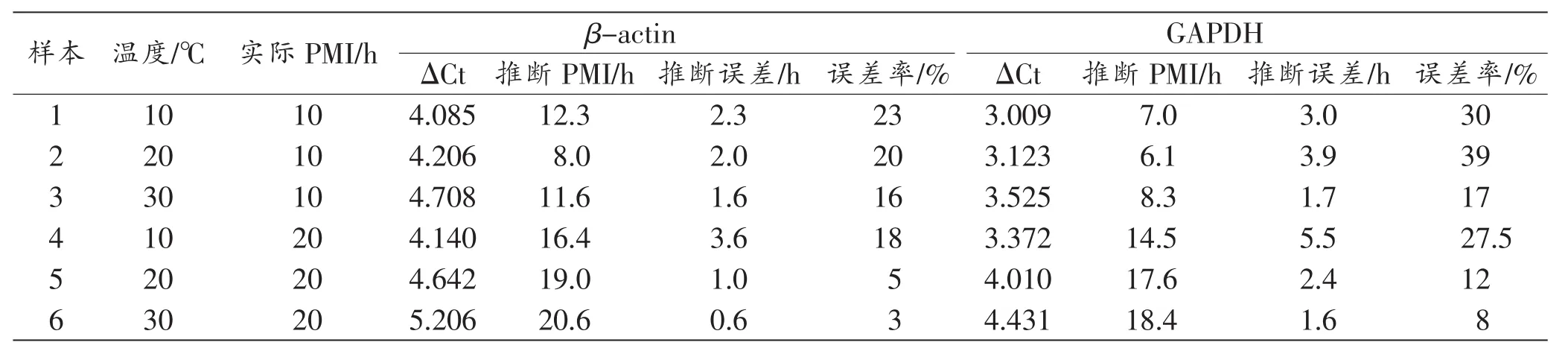
表4 β-actin和GAPDH数学模型验证结果
3 讨论
早期PMI(24 h内)是指尸体未出现明显腐败现象的时期,对于法医鉴定来说更常用也更重要,因此早期PMI的精确推断一直是法医学研究的重点和难点。近年来随着分子生物学的发展,利用生物体内大分子来推断PMI成为当前研究的热点,尤其是依据RNA的时间依赖性降解规律推断PMI已经取得了一些进展[3]。由于RNA的降解不仅与PMI有关,在很大程度上也受到环境温度的影响,因此相关研究也会考虑此影响因素,但多数研究仅将实验温度设定在室温或单个环境温度下[4-5],而在实际检案中尸体处于不断变化的环境温度中,因此单一的实验温度设定会限制其实用性。本研究设定4个连续温度的实验组,即5℃、15℃、25℃和35℃,选择受其他因素影响较小的脑组织作为检材,8种RNA作为指标,利用实时荧光定量PCR检测RNA降解规律以推断PMI。
作为快速高效的RNA定量技术,实时荧光定量PCR技术在PMI推断的研究中得到广泛应用。为了保证结果的可重复性和客观性,在应用该技术时应对各项操作进行统一规范。Bustin等[6]提出的标准化实时荧光定量PCR指南(the minimum information for publication of qRT-PCR experiments,MIQE)正逐渐被广泛认可。本实验流程均按照MIQE指南进行,包括样本的采集、处理和准备,RNA的质量控制、反转录效率、引物设计、扩增曲线、溶解曲线检测灵敏性、特异性、内参指标的选择、数据处理等。其中,定量PCR结果很大程度上依赖于内参指标的标准化处理[6-8]。因此,筛选出稳定适用、较少受PMI和温度影响的内参指标就显得尤为必要。
经geNorm软件统计后,5S rRNA、miR-9和miR-125b在4个温度组中都比较稳定,因此将这3种指标作为本研究的内参指标,其余指标作为候选指标。5S rRNA在miRNA研究中常被选作内参基因[9-10],其存在于核糖体蛋白复合物中,可适度隔绝核酶和其他因素的影响,使之保持稳定。相对于18S rRNA,5S rRNA片段较短,受PMI及温度因素的影响较小,更适合作为PMI推断的内参指标。miRNA属于非编码小RNA家族,其长度只有21~25 bp,以往研究[11-13]证实,miRNA在死后一段时间内表达稳定。在不同温度组中,miR-9 和miR-125b在死后24h内均保持稳定,表明miRNA适合作为PMI推断的内参指标。此外miR-9和miR-125b均是脑组织特异性miRNA[14]。β-actin和GAPDH是常用的RNA指标,性质相对稳定保守[15]。本课题组在前期研究中也常选用β-actin和GAPDH作为候选指标进行PMI推断[11-12,16]。U6 snRNA在PMI相关研究中也常被选作内参指标[7,11],张萍等[17]证实死亡早期人体心肌组织中U6 snRNA表达稳定,其稳定性可能与其本身的发卡结构及存在的细胞核中没有核酶有关。本研究脑组织中U6 snRNA在温度较低时表达稳定,但在温度较高(尤其是35℃组)时稳定性降低,即U6 snRNA稳定性与温度有关,不适合作为本研究内参指标,这可能是由于通常状态下与snRNA结合成复合物的小核核糖核蛋白(small nuclear ribonucleoprotein,snRNP)在较高温度时降解所致[18]。RPS29和18S rRNA也是检测PMI的常用指标,降解规律在不同实验条件下呈现出不同的模式[5,11-12],还需进一步研究。
对上述5种候选RNA指标进行内参标准化处理,在各温度组分别对ΔCt值和PMI进行回归分析,并依据相关系数筛选出与PMI相关性最好的RNA指标。结果表明,β-actin、GAPDH在不同条件下均具有较高的相关系数。内参选择时也印证了这一点,即β-actin和GAPDH在4个温度组中都具有较高的M值。相较其他候选RNA指标,β-actin和GAPDH更容易受到PMI的影响,在24h内随PMI延长不断降解,拥有良好的时序性降解规律,与一些前期研究[11-12,19]一致。考虑到环境温度的影响,为了使PMI推断结果更加精确,本研究使用R软件分别对β-actin和GAPDH进行多参数拟合,得到ΔCt值、温度和PMI三个变量的三元二次方程,决定系数R2分别为0.9375和0.9017,表明拟合优度较高。说明在不同温度下,β-actin和GAPDH有着相似的降解规律,ΔCt值与PMI的关系近似符合三元二次方程分布。在24 h内,β-actin和GAPDH的ΔCt值随着PMI延长不断增加,表明RNA 随PMI不断降解。温度较低时ΔCt值增加速率较慢,而温度较高时ΔCt值增加速率较快,表明ΔCt值升高速率与环境温度也呈正相关关系,证实随着环境温度的增高,RNA的降解速率加快。
为确保PMI推断的准确性,本研究还利用已知PMI的备用大鼠样本对数学模型进行验证。运用βactin模型进行验证时,实际PMI为10h的样本,推断误差在2.3 h内;实际PMI为20 h的样本,推断误差在3.6h内。运用GAPDH模型进行验证时,实际PMI 为10h的样本,推断误差在3.9h内;实际PMI为20h的样本,推断误差在5.5h内。相较GAPDH,利用βactin模型进行PMI推断更为精确,这可能是由于βactin模型拥有更高的决定系数,拟合优度较好。环境温度越高、PMI越长,利用该模型推断PMI的精确性越高,推测是由于随PMI的延长和环境温度增高,RNA降解速率加快以致ΔCt增加明显所致。
本研究利用实时荧光定量PCR技术观察大鼠脑组织8种RNA指标在5℃、15℃、25℃和35℃环境中的早期降解规律。实验结果显示,在4个温度组中,5S rRNA、miR-9和miR-125b表达稳定,适合作为内参指标。经内参标准化处理后β-actin和GAPDH与PMI和环境温度有着较好的相关性,表明RNA降解不仅与PMI有关,也与环境温度有关。经R软件拟合ΔCt值、温度和PMI三个变量,分别得到β-actin和GAPDH的数学模型推断PMI。用已知PMI的大鼠样本进行验证,结果证实运用该数学模型推断早期PMI相对准确有效,有望成为早期PMI推断的新方法。在今后的研究中,本课题组将对以上实验结果进一步使用人体资料进行验证。
参考文献:
[1]Bauer M.RNA in forensic science[J].Forensic Sci Int Genet,2007,1(1):69-74.
[2]Vandesompele J,De Preter K,Pattyn F,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biol,2002,3(7):H34.
[3]张恒,李文灿,张萍,等.组织RNA检测用于死亡时间推断的研究进展[J].中国法医学杂志,2012,27(4):292-295.
[4]Birdsill AC,Walker DG,Lue L,et al.Postmortem interval effect on RNA and gene expression in human brain tissue[J].Cell Tissue Bank,2011,12(4):311-318.
[5]Sampaio-Silva F,Magalhaes T,Carvalho F,et al. Profiling of RNA degradation for estimation of post morterm interval[J].PLoS One,2013,8(2):e56507.
[6]Bustin SA,Benes V,Garson JA,et al.The MIQE guidelines:minimum information for publication of quantitative real-time PCR experiments[J].Clin Chem,2009,55(4):611-622.
[7]Zhang H,Zhang P,Ma KJ,et al.The selection of endogenous genes in human postmortem tissues[J].Sci Justice,2013,53(2):115-120.
[8]Lardizabal MN,Nocito AL,Daniele SM,et al. Reference genes for real-time PCR quantification of microRNAs and messenger RNAs in rat models of hepatotoxicity[J].PLoS One,2012,7(5):e36323.
[9]Tea M,Michael MZ,Brereton HM,et al.Stability of small non-coding RNA reference gene expression in the rat retina during exposure to cyclic hyperoxia[J]. Mol Vis,2013,19:501-508.
[10]Gharbi S,Shamsara M,Khateri S,et al.Identification of Reliable Reference Genes for Quantification of MicroRNAs in Serum Samples of Sulfur Mustard-Exposed Veterans[J].Cell J,2015,17(3):494-501.
[11]Lü YH,Ma KJ,Zhang H,et al.A time course study demonstrating mRNA,microRNA,18S rRNA,and U6 snRNA changes to estimate PMI in deceased rat’s spleen[J].J Forensic Sci,2014,59(5):1286-1294.
[12]Ma J,Pan H,Zeng Y,et al.Exploration of the R code-based mathematical model for PMI estimation using profiling of RNA degradation in rat brain tissue at different temperatures[J].Forensic Sci Med Pathol,2015,11(4):530-537.
[13]Odriozola A,Riancho JA,de la Vega R,et al. miRNA analysis in vitreous humor to determine the time of death:a proof-of-concept pilot study[J].Int J Legal Med,2013,127(3):573-578.
[14]Griffiths-Jones S,Grocock RJ,van Dongen S,et al. miRBase:microRNA sequences,targets and gene nomenclature[J].Nucleic Acids Res,2006,34(Database issue):D140-D144.
[15]Thellin O,Zorzi W,Lakaye B,et al.Housekeeping genes as internal standards:use and limits[J]. J Biotechnol,1999,75(2-3):291-295.
[16]吕叶辉,张恒,潘晖,等.不同死因大鼠心肌内多种RNA相对表达量变化与PMI的关系[J].法医学杂志,2014,30(1):7-12.
[17]张萍,马开军,张恒,等.实时RT-PCR常用内对照在死亡早期人心肌内的稳定性[J].法医学杂志,2012,28(2):81-84.
[18]Yong J,Golembe TJ,Battle DJ,et al.snRNAs contain specific SMN-binding domains that are essential for snRNP assembly[J].Mol Cell Biol,2004,24(7):2747-2756.
[19]朱怡,董迎春,梁伟波,等.小鼠死后脑RNA降解与死亡时间的相关性[J].法医学杂志,2011,27(3):161-163.
(本文编辑:邹冬华)
中图分类号:DF795.1
文献标志码:A
doi:10.3969/j.issn.1004-5619.2016.03.002
文章编号:1004-5619(2016)03-0165-06
基金项目:上海健康医学院种子基金项目;上海高校青年教师培养资助计划项目
作者简介:吕叶辉(1989—),男,硕士,主要从事基础医学教学、科研工作;E-mail:yeyeliushu@163.com
通信作者:陈龙,男,博士,主任法医师,副教授,主要从事法医病理学及法医临床学研究;E-mail:chenlong@shmu.edu.cnPCR技术,检测死后24h内大鼠脑组织8种RNA在5℃、15℃、25℃和35℃环境下的变化规律,建立各指标与PMI之间的关系模型,以期为不同温度下早期PMI的推断提供新思路。
收稿日期:(2016-01-30)
Correlation between RNA Degradation Patterns of Rat’s Brain and Early PMI at Different Temperatures
LÜ Ye-hui1,LI Zhi-hong1,TUO Ya1,LIU Li1,LI Kun1,BIAN Jie1,MA Jian-long2,CHEN Long2
(1.School of Basic Medical Science,Shanghai University of Medicine&Health Science,Shanghai 201318,China;2.Department of Forensic Medicine,School of Basic Medical Science,Fudan University,Shanghai 200032,China)
Abstract:Objective To explore the correlation between early postmortem interval(PMI)and eight RNA markers of rat’s brain at different temperatures.Methods Total 222 SD rats were randomly divided into control group(PMI=0 h)and four experimental groups.And the rats in the experimental groups were sacrificed by cervical dislocation and respectively kept at 5℃,15℃,25℃ and 35℃ in a controlled environment chamber.The RNA was extracted from brain tissues,which was taken at 9 time points from 1 h to 24 h postmortem.The expression levels of eight markers,β-actin,GAPDH,RPS29,18S rRNA,5S rRNA,U6 snRNA,miRNA-9 and miRNA-125b,were detected using real-time fluorescent quantitative PCR,respectively.Proper internal reference was selected by geNorm software.Regression analysis of normalized RNA markers was performed by SPSS software.Mathematical model for PMI estimation was established using R software.Another 6 SD rats with known PMI were used to verify the mathematical model.Results 5S rRNA,miR-9 and miR-125b were suitable as internal reference markers for their stable expression.Both β-actin and GAPDH had well time-dependent degradation patterns and degraded continually with prolongation of PMI in 24 h postmortem.The mathematical model of the variation of ΔCt values with PMI and temperature was set up by R software and the model could be used for PMI estimation.The average error rates of model validation using β-actin and GAPDH were 14.1%and 22.2%,respectively.Conclusion The expression levels of β-actin and GAPDH are well correlated with PMI and environmental temperature.The mathematical model established in present study can provide references for estimating early PMI under various temperature conditions.
Key words:forensic pathology;RNA;postmortem intervals;temperature;degradation;rats

