不同年份和产地美味牛肝菌的红外光谱鉴别研究
杨天伟,李 涛,李杰庆,张 雪,王元忠,刘鸿高*
1. 云南农业大学农学与生物技术学院,云南 昆明 650201
2. 云南省农业科学院药用植物研究所,云南 昆明 650200
3. 玉溪师范学院资源环境学院,云南 玉溪 653100
4. 云南技师学院,云南 安宁 650300
不同年份和产地美味牛肝菌的红外光谱鉴别研究
杨天伟1, 2,李 涛3,李杰庆1,张 雪4,王元忠2*,刘鸿高1*
1. 云南农业大学农学与生物技术学院,云南 昆明 650201
2. 云南省农业科学院药用植物研究所,云南 昆明 650200
3. 玉溪师范学院资源环境学院,云南 玉溪 653100
4. 云南技师学院,云南 安宁 650300
采用傅里叶变换红外光谱技术结合多元统计分析建立快速鉴别不同年份、不同产地美味牛肝菌的方法。采集2011年—2014年云南26个不同地区152个美味牛肝菌样品的红外光谱,使用正交信号校正(orthogonal signal correction,OSC)、微波压缩(wavelet compression)方法对原始光谱进行优化处理,OSCW校正前后的光谱数据进行偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA),比较光谱预处理前后PLS-DA的分类效果。将152个美味牛肝菌随机分为训练集(120个)和验证集(32个),建立OSCW校正前后的PLS分类预测模型。结果显示,经OSCW处理后的PLS-DA分类效果明显优于处理前的结果,主成分得分图能准确区分不同年份、不同产地美味牛肝菌样品,表明OSCW处理能有效滤除光谱中的噪音及与因变量无关的干扰信息,提高光谱分析的准确性和计算速率。OSCW处理前PLS模型训练集的R2和RMSEE分别为0.790 1和21.246 5,验证集的R2和RMSEP分别为0.922 5和14.429 2; OSCW预处理后训练集的R2和RMSEE分别为0.852 3和17.238 1,验证集的R2和RMSEP分别为0.845 4和20.87,表明OSCW预处理提高了训练集的预测效果,但OSCW-PLS出现了过拟合现象降低验证集的预测能力,因此,OSCW不适宜与PLS结合建立模型。OSCW结合PLS-DA能滤除光谱中大量的干扰信息,准确区分不同年份、不同产地美味牛肝菌样品,为野生食用菌的鉴别分类提供可靠依据。
红外光谱; 正交信号校正-微波压缩; 偏最小二乘判别分析; 美味牛肝菌; 鉴别
引 言
牛肝菌是世界性著名食用菌,味道鲜美,营养丰富,日益受到消费者亲睐,具有重要的经济价值、食药用价值和生态价值[1, 2]。美味牛肝菌又名白牛肝菌、大脚菇,富含蛋白质、氨基酸、多糖、矿质元素、维生素等,经常食用可以增强人体免疫力,具有清热除烦,养血补虚,抗氧化,抗病毒等药用功效,是极具开发应用价值的野生食用菌之一[3-6]。云南丰富的森林资源和多样的气候条件,孕育了丰富的野生食用菌,其中美味牛肝菌产量高、分布广,受气候条件和生长环境影响,不同产地美味牛肝菌的营养成分、化学成分的积累和药效成分不尽相同[7]。
传统食用牛肝菌鉴别主要根据外表特征,生长特性,孢子的显微结构,菌肉、菌管的颜色和变色反应等进行分类[8]。然而不同种类间形态相似性大,如茶褐牛肝菌(Boletusbrunneissimus)和深褐牛肝菌(Boletusobscureumbrinus); 同种牛肝菌在不同生长环境及不同生长阶段存在大量的表形差异,如美味牛肝菌菌盖颜色有灰白色、淡褐色、黄褐色、红褐色或深褐色,菌管有乳白色、淡褐色,偶尔成黄绿色,其颜色变化较大,不易准确鉴别[9]; 此外新鲜牛肝菌子实体水分含量高,多数加工成干片保存和出售,导致以假充真,以次充好的现象时有发生[10],难以进行质量控制和市场管理。目前对食用菌的鉴别研究主要有光谱技术和分子生物学方法; 周在进等[8, 11]应用红外光谱结合分层抽样、聚类分析等方法研究了不同产地、种类食用菌的鉴别方法; 孙素琴等[12]用红外光谱法鉴别了不同厂家生产的36种灵芝产品; 本课题组采用紫外光谱结合化学计量学方法对牛肝菌不同部位和产地进行了鉴别研究[13],但紫外光谱法受提起溶剂、提起时间等影响较大,单一溶剂提取难以全面反映样品的整体信息。魏海龙等[14]设计5对ITS引物并对ITS区段进行PCR扩增,实现牛肝菌属卷边组Boletussect.Appendiculati物种的识别; Wu等[2]测定了牛肝菌属4个基因片段的DNA系列,结合牛肝菌形态特征及孢子的显微结构,建立了牛肝菌科分子发育系统框架; Mello等[15]根据ITS片段设计引物,对美味牛肝菌、铜色牛肝菌(BoletusaereusBull.)等进行了分子鉴别; Lian等[16]应用ITS设计引物辨别了美味牛肝菌和其他蘑菇; 分子生物学法仪器操作复杂,价格昂贵,推广应用具有局限性。
傅里叶变换红外光谱具有快速、简便、样品用量少、非破坏性等优点; 能有效反映出成分复杂物质体系的组分特征,实现对样品多组分、多指标分析,是一种综合的质量控制和物种鉴别方法[17-18],已被广泛应用于农业[19]、食品[20]、中药[21]等行业的定性、定量分析。本文采集了2011年—2014年云南26个不同地区152个美味牛肝菌子实体的红外光谱信息,采用正交信号校正(orthogonal signal correction)、微波压缩(wavelet compression)法对光谱进行预处理,预处理前后的光谱数据进行PLS-DA分析; 结果显示OSCW处理后能够很好的区分不同年份及不同产地美味牛肝菌样品,表明OSC能滤除大量与分类变量无关的干扰信息,同时对光谱进行微波压缩处理可以在极少量信息丢失的前提下提高光谱分析速率,为快速鉴别不同年份、不同产地美味牛肝菌提供辅助方法。
1 实验部分
1.1 仪器
Frontier型傅里叶变换红外光谱仪(美国Perkin Elmer公司,采用DTGS检测器,光谱分辨率为4 cm-1); YP-2型压片机(上海市山岳科学仪器有限公司); FW-100型高速粉碎机(天津市华鑫仪器厂); 80目标准筛盘(浙江上虞市道墟五四仪器厂)。
1.2 材料
2011年—2014年采自云南26个不同产地的152个美味牛肝菌子实体,均由云南农业大学刘鸿高教授鉴定为美味牛肝菌BoletusedulisBull,来源见表1。
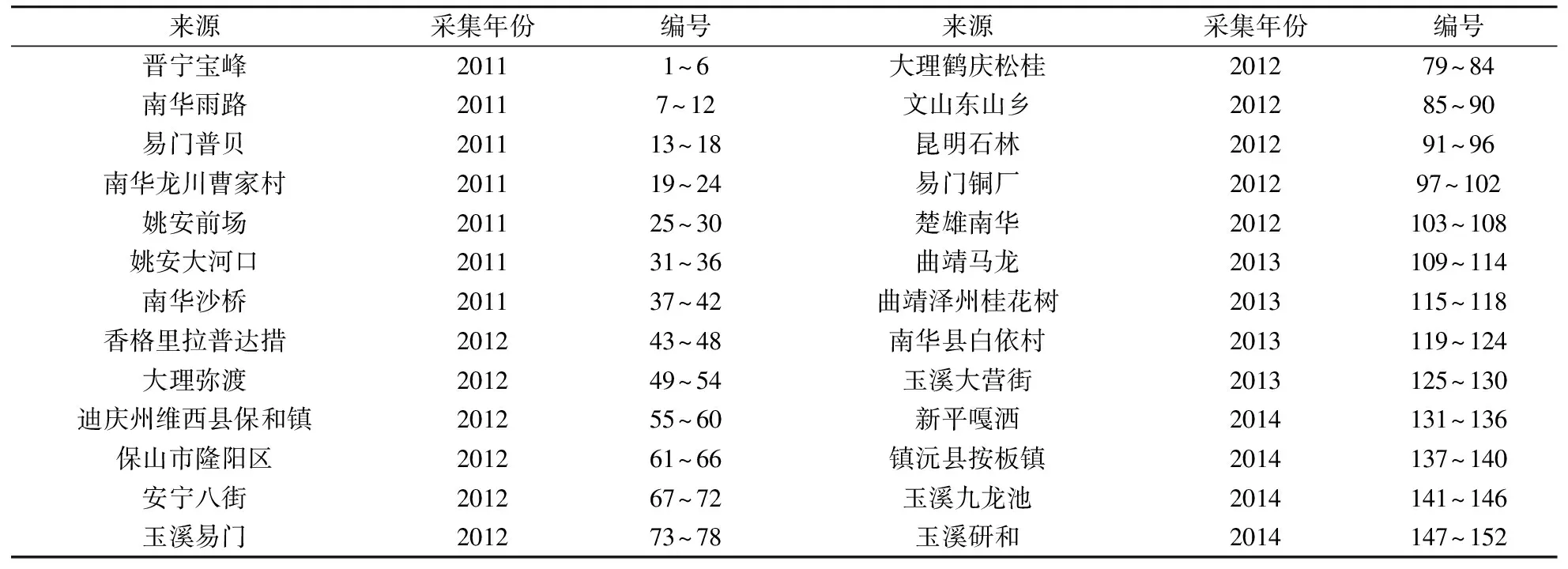
表1 样品信息
1.3 样品处理及光谱采集
样品采集后清洗干净,50 ℃烘干,粉碎,过80目筛,备用。称取样品粉末(1.5±0.2)mg,KBr粉末(100±0.2)mg,放入玛瑙研钵充分混合磨成细粉,压片; 仪器预热1 h后测定光谱,设定累加扫描次数为16次,光谱扫描范围4 000~400 cm-1。重复称取7份样品1,分别压片、测定,考察方法重现性; 取一片样品1重复测定7次考察方法精密度; 取一片样品1,分别在0,10,20,30,40,50,60 min(每次测完立即放到红外线灯下以免吸潮)时测定红外光谱,考察稳定性; 其余样品重复测定3次,取平均光谱; 扫描时均已扣除CO2和水的干扰。
1.4 光谱预处理
常见的光谱预处理方法主要有平滑,求导,标准正态变量(standard normal variate,SNV),多元散射校正(multiplicative scatter correction,MSC),纯净分析信号/处理(net analyte signal/processing,NAS/P),正交信号校正(orthogonal signal correction,OSC)等方法。其中正交信号校正(OSC)运用数学上正交的方法有效滤除自变量X中与因变量Y无关的信息,增强自变量、因变量间的相关性,提高主成分解释能力和光谱分析的针对性,对OSC处理后的X变量进行微波压缩以提高运算速度和效率[22-24]。152个美味牛肝菌子实体样品红外光谱见图1; 原始光谱经过正交信号校正-微波压缩(OSCW)处理后结果见图2。
1.5 光谱数据处理
采用偏最小二乘判别分析法(PLS-DA),对OSCW处理前后的光谱数据进行分析比较。152个美味牛肝菌样品随机抽取32个作为验证集,其余120个作为训练集,结合PLS建立模型。根据相关系数(R2)、校正均方根误差(RMSEE)和预测均方根误差(RMSEP)评价模型可行性,其计算见式(1)—(式3); 当R2越接近1,RMSEP和RMSEE越小且RMSEP≤RMSEE时认为所建模型预测效果最佳。
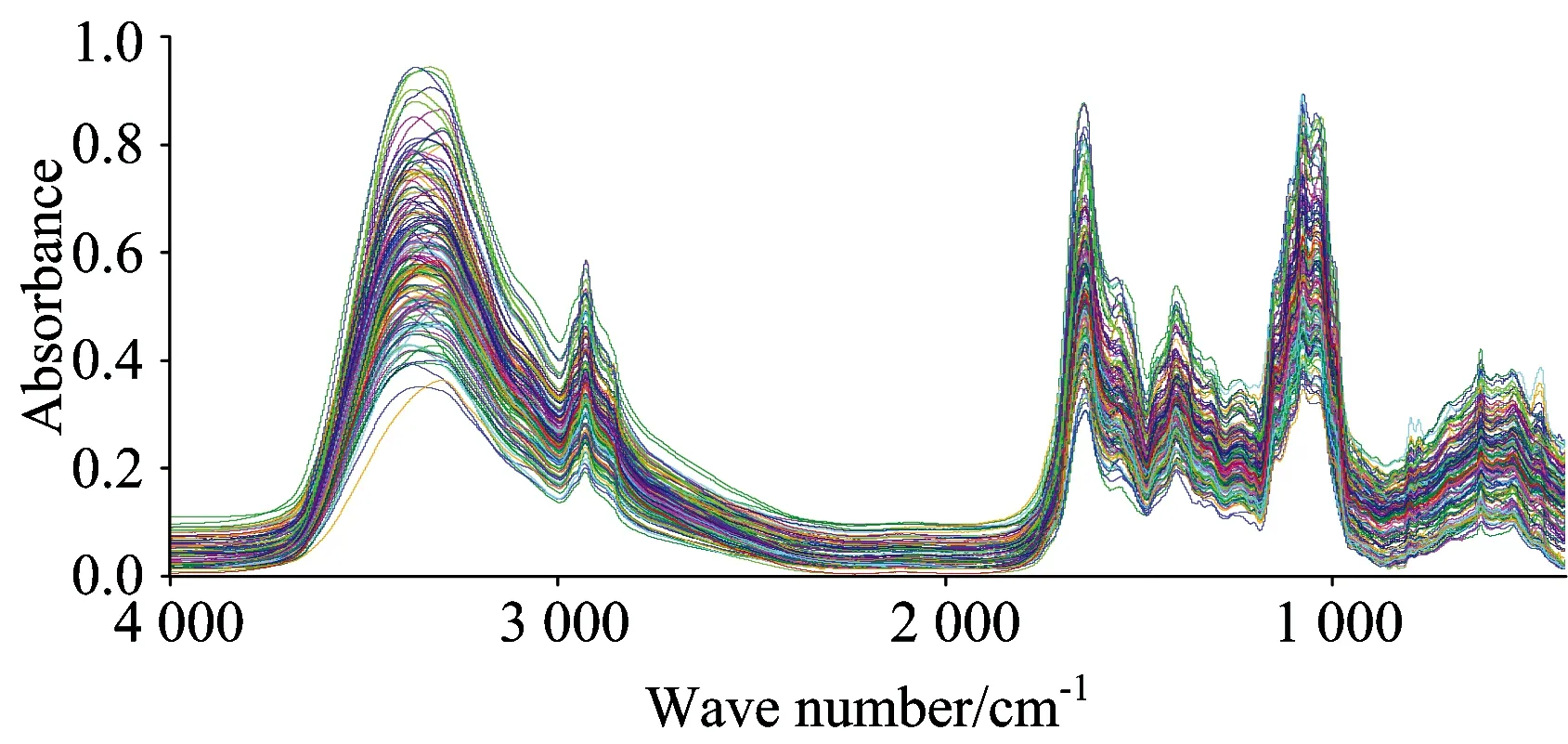
图1 美味牛肝菌的红外光谱
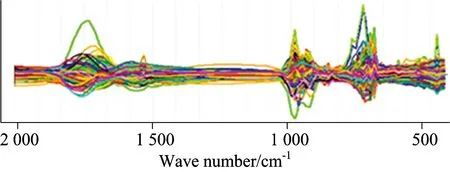
图2 正交信号校正-微波压缩预处理后美味牛肝菌的红外光谱
(1)
(2)
(3)
2 结果与讨论
2.1 方法学考察
1.3中用于重现性、精密度、稳定性考察的光谱信息分
别计算平均光谱,通过Omnic8.2软件的光谱检索功能,以三个平均光谱分别建立重现性、精密度、稳定性的光谱数据库:C1,J1和W1。 将方法学考察的原始光谱作为未知样品,分别在相应的光谱数据库进行检索,系统自动计算出能反映样品亲密程度的匹配分值,分值越大匹配程度越高,方法越可靠[19]。结果显示重现性的匹配值在99.32~99.99之间,RSD为0.26%; 精密度的匹配值在99.93~99.99之间,RSD为0.25%; 稳定性的匹配值介于99.14~99.85之间,RSD为0.366%; 表明该方法稳定、可行。
2.2 美味牛肝菌红外光谱特征分析
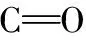
2.3 OSCW预处理
为了增强光谱分析的准确性和针对性,消除与采集年份、产地无关的信息,对原始光谱进行正交信号校正-微波压缩(OSCW)处理,结果见图2和表2。由图2可以看出经过OSCW处理后光谱曲线变得紧凑,表明已消除大量干扰信息,保留了有用信息。表2为OSCW的参数和结果,由表2可知OSC计算出的两个新组分之间角度为90°具有正交性,第二个组分计算后的剩余平方和(remaining sum of squares,RSS)为13.92%,表明与年份、产地无关的86.08%信息已从X变量母集中移除。通过正交微波(orthogonal Wavelet)对X变量母集进行压缩以提高运算速率,选用多贝西(daubechies)小波函数和离散小波变换法(discrete wavelet transform)进行压缩,压缩水平为8,此时能解释98%的原始光谱信息。
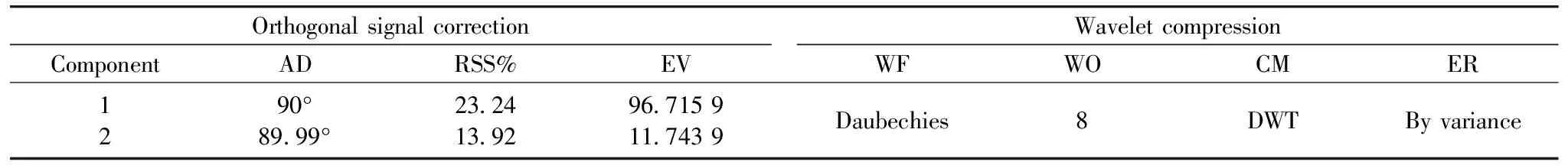
表2 正交信号校正-微波压缩总结
Note: AD: angle in degrees; RSS: remaining sum of squares; EV: EigenValue: WF: wavelet function; WO: wavelet order; CM: compression method; ER: energy retained; DWT: discrete wavelet transform
2.4 PLS-DA分析
2.4.1 不同年份美味牛肝菌PLS-DA分析
设光谱数据为自变量X,不同采集年份的分类为因变量Y,对OSCW处理前后的光谱信号进行PLS-DA分析,结果见图3和图4。图3为决定主成分数的R2Y和Q2值。R2Y表示所提取主成分能解释Y变量的比例,Q2表示主成分能预测Y变量的比例,一般认为R2Y和Q2应大于0.5且越接近1越好[24]。图4为OSCW处理前后主成分t1和t2的得分图。
由图3(a)可知OSCW处理前一共提取了8个主成分,其R2Y和Q2分别为0.764和0.528表明这8个主成分能够解释和预测76.4%和52.8%的Y变量,R2Y和Q2均远小于1; 图4(a)可以看出采于不同年份美味牛肝菌样品不能区分开,表明OSCW处理前PLS-DA分析效果较差。由图3(b)可知OSCW处理后提取了6个主成分,R2Y和Q2分别为0.893和0.752,主成分的累积解释水平和预测水平都大幅度提高。由前2个主成分得分图4(b)可以看出,不同年份美味牛肝菌样品能够很好的区分开,表明不同年份美味牛肝菌对化学成分的积累情况不同,这可能与气候变化有关,如2014年云南的降水量较其他年份更充沛。结果显示红外光谱经OSCW预处理后滤除了大量的干扰信息,PLS-DA分类效果变好。OSCW结合PLS-DA适用于不同年份美味牛肝菌的鉴别分析。
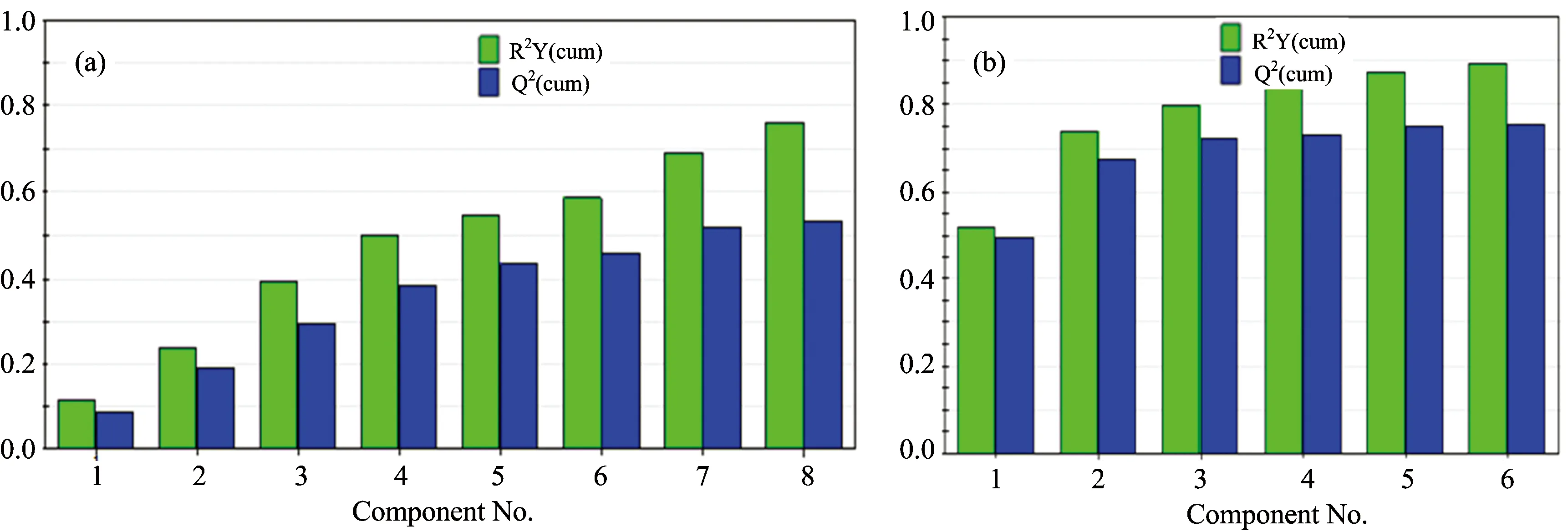
图3 OSCW处理前后的主成分数及R2Y,Q2值
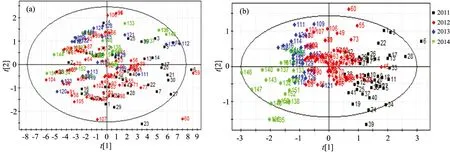
图4 不同年份美味牛肝菌OSCW处理前后主成分得分图
2.4.2 不同产地美味牛肝菌PLS-DA分析
以2012年采集的不同产地美味牛肝菌光谱数据为自变量,产地分类为因变量,比较原始光谱与正交信号-微波压缩(OSCW)处理后的PLS-DA分析结果。图5为11个不同产地美味牛肝菌PLS-DA的前3个主成分三维得分图。由原始光谱PLS-DA的前3个主成分得分图5(a)可知不同产地美味牛肝菌样品不能够很好地区分开。图5(b)显示了OSCW预处理后PLS-DA的前3个主成分得分图,由图可知2012年采自11个不同产地的样品除了2个样品聚类有误外,其余样品均能按不同产地正确分类,表明正交信号-微波压缩滤除了大量干扰信息,保留与美味牛肝菌产地有关的信息,产地分类效果提高,结果令人满意。
2.5 PLS建模分析
基于美味牛肝菌红外光谱数据建立PLS预测模型,结果见图6和图7。由图6可知OSCW预处理前(a)和OSCW处理后(b)训练集样本的观测值与预测值之间的相关系数r分别为0.790 1和0.852 3,校正均方根误差RMSEE分别为21.246 5和17.238 1; 表明OSCW处理可以明显提高模型训练集的预测率。
图7可知OSCW预处理前(a)和OSCW处理后(b)验证集样本的观测值与预测值之间的相关系数R2分别为0.922 5和0.845 4,预测均方根误差RMSEP分别为14.429 2和20.87。结合图6可知OSCW处理前模型的RMSEP
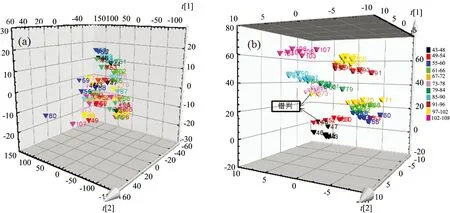
图5 不同产地美味牛肝菌OSCW处理前后主成分得分图
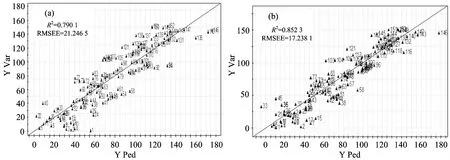
图6 OSCW预处理前后训练集的PLS模型预测结果
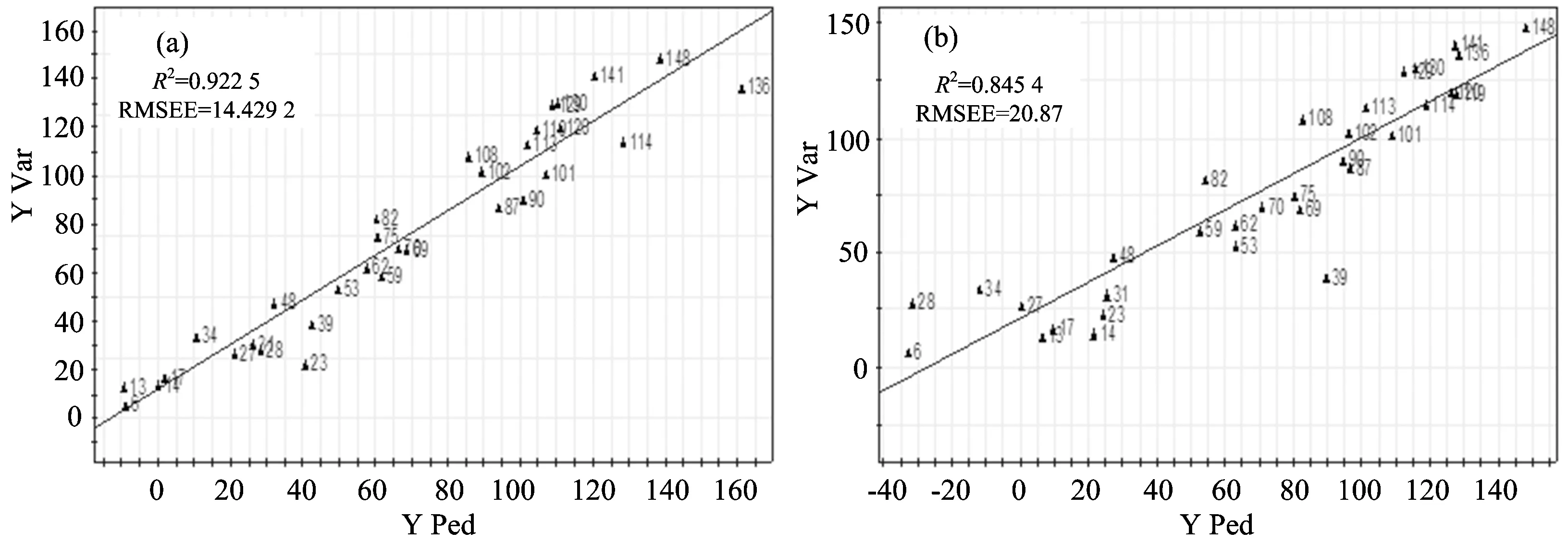
图7 OSCW预处理前后验证集的PLS模型预测结果
3 结 论
采集不同年份、不同产地美味牛肝菌样品的红外光谱,用Omnic8.2软件的光谱检索功能考察方法的重现性、精密度和稳定性,显示该方法稳定、可靠。应用正交信号校正-微波压缩(OSCW)方法处理光谱,对OSCW处理前后的美味牛肝菌光谱数据进行PLS-DA分析,结果显示OSCW处理后PLS-DA对美味牛肝菌不同年份及不同产地的分类效果明显优于光谱预处理前的效果,表明OSC能够滤除大量与分类变量无关的干扰信息,在确保极少量信息丢失的前提下对数据进行微波压缩明显提高了光谱分析的计算速率。
152个美味牛肝菌光谱数据分为训练集和验证集,分别以OSCW处理前后的光谱数据,建立PLS分类预测模型,以相关系数r、校正均方根误差RMSEE和预测均方根误差RMSEP评价模型预测效果。结果显示OSCW处理前训练集的r小于预处理后,而RMSEE大于预处理后,表明OSCW处理能够提高模型训练集的预测效果; OSCW预处理后模型的RMSEP>RMSEE,r小于光谱预处理前的数值,表明OSCW处理后光谱出现过拟合现象,降低了模型验证集的预测能力。
红外光谱数据通过OSCW处理能够滤除光谱矩阵中的噪音信号及与因变量无关的干扰信息,提高光谱分析的针对性和计算速率,但OSCW-PLS会出现过拟合现象,不适宜建立分类预测模型。OSCW与PLS-DA结合区分不同年份、不同产地美味牛肝菌样品,结果令人满意,为食用菌的快速鉴别分类和质量控制提供行之有效的方法,具有实际推广价值。
[1] Wang X M, Zhang J, Wu L H, et al. Food Chemistry, 2014, 151: 279.
[2] Wu G, Feng B, Xu J P, et al. Fungal Diversity, 2014, 69: 93.
[3] DENG Bai-wan, CHEN Wen-qiang, LI Xin-sheng(邓百万, 陈文强, 李新生). Food Science(食品科学), 2005, 25(11): 255.
[4] Kalac P. Food Chemistry, 2009, 113(1): 9.
[5] ZHOU Ling-xian, YIN Jian-zhong(周玲仙, 殷建忠). Edible Fungus(食用菌), 2008, (4): 61.
[6] Luo Aoxue, Luo Aoshuang, Huang Jiandong, et al. Molecules, 2012, 17(7): 8079.
[7] LI Shu-hong, ZHAO Yong-chang, YU Fu-qiang, et al(李树红, 赵永昌, 于富强, 等). Edible Fungi of China(中国食用菌), 2011, 30(5): 34.
[8] ZHOU Zai-jin, LIU Gang, REN Xian-pei(周在进, 刘 刚, 任先培). Laser and Infrared(激光与红外). 2010, 40(9): 970.
[9] DAI Yu-cheng, TOLGOR Bau, CUI Bao-kai, et al(戴玉成, 图力古尔, 催宝凯, 等). Illustrations of Medicinal Fungi in China(中国药用真菌图志). Harbin: Northeast Forestry University Press(哈尔滨: 东北林业大学出版社), 2013. 74.
[10] WANG Xiang-hua, LIU Pei-gui, YU Fu-qiang(王向华, 刘培贵, 于富强). Color atlas of wild commercial mushrooms in Yunnan(云南野生商品蘑菇图鉴). Kunming: Yunnan Science and Technology Press(昆明: 云南科技出版社), 2004. 125.
[11] ZHOU Zai-jin, LIU Gang, REN Xian-pei(周在进, 刘 刚, 任先培). Laser and Infrared(激光与红外), 2009, 39(11): 1158.
[12] SUN Su-qin, DU De-guo, LIANG Xi-yun, et al(孙素琴, 杜德国, 梁曦云, 等). Chinese Journal of Analytical Chemistry(分析化学), 2001, 29(3): 309.
[13] YANG Tian-wei, CUI Bao-kai, ZHANG Ji, et al(杨天伟, 崔宝凯, 张 霁, 等). Mycosystema(菌物学报), 2014, 33(2): 262.
[14] WEI Hai-long, LI Hai-bo, WANG Li-ling, et al(魏海龙, 李海波, 王丽玲, 等). Mycosystema(菌物学报), 2014, 33(2): 242.
[15] Mello A, Ghignone S, Vizzini A, et al. Journal of Biotechnology, 2006, 121(3): 318.
[16] Lian B, Zang J P, Hou W G, et al. Electronic Journal of Biotechnology, 2008, 11(3): 102.
[17] ZHOU Ye, ZHANG Qing-wei, LI Pei-fu, et al(周 晔, 张庆伟, 李佩孚, 等). Chinese Journal of Traditional Chinese Medicine(中国中药杂志), 2013, 38(019): 3309.
[18] Alexandrakis D, Downey G, Scannell A G M. Food and Bioprocess Technology, 2012, 5(1): 338.
[19] REN Xian-pei, LIU Gang, ZHOU Zai-jin, et al(任先培, 刘 刚, 周在进, 等). Laser and Infrared(激光与红外), 2009, 39(9): 944.
[20] Tarantilis P A, Troianou V E, Pappas C S, et al. Food Chemistry, 2008, 111(1): 192.
[21] Sun S Q, Chen J B, Zhou Q, et al. Planta Medica, 2010, 76(17): 1987.
[22] Wold S, Antti H, Lindgren F, et al. Chemometrics and Intelligent Laboratory Systems, 1998, 44: 175.
[23] Bylesjö M, Rantalainen M, Cloarec O, et al. Journal of Chemometrics, 2006, 20(8-10): 341.
[24] LI Si-hai, PAN Xin-bo, REN Zhen, et al(李四海, 潘新波, 任 真, 等). Chinese Journal of Experimental Traditional Medical Formulae(中国实验方剂学杂志), 2013, 19(12): 132.
(Received Sep. 13, 2014; accepted Dec. 25, 2014)
*Corresponding author
Study on the Discrimination ofBoletusEdulisfrom Different Years and Origins with FTIR
YANG Tian-wei1, 2, LI Tao3, LI Jie-qing1, ZHANG Xue4, WANG Yuan-zhong2*,LIU Hong-gao1*
1. College of Agronomy and Biotechnology, Yunnan Agricultural University, Kunming 650201, China
2. Institute of Medicinal Plants, Yunnan Academy of Agricultural Sciences, Kunming 650200, China
3. College of Resources and Environment, Yuxi Normal University, Yuxi 653100, China
4. Yunnan Technician College, Anning 650300, China
In order to establish a rapid method for discriminatingBoletusedulismushroom, Fourier transform infrared spectroscopy combined with multivariate statistical analysis were used to studyB.eduliswhich were collected from different origins and different years. The original infrared spectra of all the 152B.edulissamples collected from 2011 to 2014 and 26 different areas of Yunnan Province were optimized with orthogonal signal correction and wavelet compression (OSCW) method. The spectral data that before and after being preprocessed with OSCW were analyzed with partial least squares discriminant analysis (PLS-DA). The classification results of PLS-DA were compared. Then the 152B.edulissamples were randomly divided into a training set (120) and a validation set (32) to establish the PLS classification prediction model. The results showed that, after OSCW processing, the classification result of PLS-DA was significantly better than the other one which was not processed by OSCW. Principal component score plot can accurately distinguishB.edulissamples collected from different years and different origins. It indicated that OSCW can effectively eliminate the noise of spectra and reduce the unrelated interference information about the dependent variables to improve the accuracy and calculation speed of spectral analysis. Before OSCW preprocessed, theR2and RMSEE of PLS model of the training set were 0.790 1 and 21.246 5 respectively whileR2and RMSEP of the model of validation set were 0.922 5 and 14.429 2. After OSCW pretreatment,R2and RMSEE of the training set were 0.852 3 and 17.238 1 whileR2and RMSEP of validation set were 0.845 4 and 20.87. It suggested that OSCW could improve the predictive effect of the training set, but the over-fitting of OSCW-PLS may reduce the predictive ability of validation set. Therefore, it was unsuitable to establish a model with OSCW combined with PLS. In a conclusion, OSCW combined with PLS-DA can eliminate a large amount of spectrum interference information. This method could accurately distinguishB.edulissamples collected from different years and different origins. It could provide a reliable basis for the discrimination and classification of wild edible fungi.
Infrared spectroscopy; OSCW; PLS-DA;Boletusedulis; Discrimination
2014-09-13,
2014-12-25
国家自然科学基金项目(31260496,31160409,31460538)和云南省自然科学基金项目(2011FB053,2011FZ195)资助
杨天伟,1989年生,云南农业大学农学与生物技术学院硕士研究生 e-mail: yangtianweizj@126.com *通讯联系人 e-mail: boletus@126.com; honggaoliu@126.com
TS201.2
A
10.3964/j.issn.1000-0593(2016)07-2117-07

