浸入式可见/近红外光谱技术的藻种鉴别研究
朱红艳,邵咏妮,蒋璐璐,郭安鹊,潘 健,何 勇*
1. 浙江大学生物系统工程与食品科学学院,浙江 杭州 310058 2. 浙江经济职业技术学院,浙江 杭州 310018 3. 西北农林科技大学葡萄酒学院,陕西 杨凌 712100
浸入式可见/近红外光谱技术的藻种鉴别研究
朱红艳1,邵咏妮1,蒋璐璐2,郭安鹊3,潘 健1,何 勇1*
1. 浙江大学生物系统工程与食品科学学院,浙江 杭州 310058 2. 浙江经济职业技术学院,浙江 杭州 310018 3. 西北农林科技大学葡萄酒学院,陕西 杨凌 712100
对藻类的识别分类及其生化分析已成为海洋生物学的研究热点之一。以普通小球藻、蛋白核小球藻、微绿球藻、莱茵衣藻为样品,通过便携式USB4000微型光纤光谱仪、Y形光纤和探针,卤素光源构建的光谱采集系统对不同浓度梯度的120个微藻样本进行浸入式可见/近红外透射光谱的原位采集,比较去基线、卷积平滑等光谱预处理方法的效果,并基于连续投影算法(SPA)筛选特征波长,通过偏最小二乘法(PLS)、最小二乘支持向量机(LS-SVM)和极限学习机(ELM)进行建模,探讨采用透射光谱原位快速鉴别四种不同藻种的可行性。结果表明:卷积平滑的处理效果较为理想,有效波长可用于代替原始光谱建立微藻种类判别分析模型。SPA-LV-SVM和SPA-ELM的预测效果显著高于SPA-PLS,三者的平均预测正确率分别是80%,85%,65%。浸入式可见/近红外光谱技术和便携式光纤探针结合的藻种鉴别方法,有效实现了对四种微藻的鉴别,为藻种鉴别和藻种分类研究领域提供了一种新思路。
微藻;可见/近红外透射光谱;藻种鉴别;极限学习机
引 言
微藻(microalgae)是一类可以将二氧化碳转化为潜在的生物燃料、食品、饲料和高价值的生物活性分子并能进行光合作用的真核微生物[1]。微藻具有生态和生物学价值,是一种重要的生物质资源。微藻成为国际上很多研究项目的焦点[2-4],这种兴趣来自于微藻中含有丰富的营养及生理活性成分,具有预防心血管、癌症等疾病的功效。目前,对藻类的识别分类及其生化分析已经成为海洋生物学的研究热点之一。
传统的微藻鉴定方法主要依赖于显微镜对微藻形态结构的观察和一些藻体所固有的生理生化特性。以形态学的细微特征对藻进行鉴定存在许多困难和争议,于是人们开始把目光投向各类分子生物学技术。但通过序列分析获得的结果和根据形态学分类的结果有时存在一些偏差,而且物种遗传多样性对特异性探针的设计也产生了不利影响[5]。通过具有特征性的生理生化物质的测定来鉴定微藻,是最近发展起来的一门科学,称为化学分类法(chemotaxonomy)[6]。目前能够用于化学分类的海洋浮游藻类“特征性化学成分”有脂肪酸、碳水化合物(糖类)、色素和氨基酸等。
可见/近红外光谱可充分利用全谱段或多波长下的光谱数据进行定性或定量分析。国内外利用此技术进行苹果[7]、茶叶[8]、酸奶等品种的鉴别。而对藻种鉴别的研究还很少;据于该方法的快速、简单和活体生物无损检测的特点,探讨通过便携式USB4000微型光纤光谱仪、Y形光纤和探针,卤素光源构建的光谱采集系统对不同浓度梯度的微藻样本进行浸入式可见/近红外透射光谱的原位采集,通过不同算法的建模,研究浸入式可见/近红外光谱在藻种分类领域应用的可行性,为以后更深入的研究提供参考。
1 实验部分
1.1 微藻及其培养
选用普通小球藻(Chlorellavulgaris)、蛋白核小球藻(Chlorellapyrenoidosa)、莱茵衣藻(Chlamydomonasreinhardtii)(购于中国科学院野生生物种质库——淡水藻种库)和微绿球藻(Nannochloropsisoculata)(购于中国科学院海洋研究所)作为研究对象。四种微藻备受研究者青睐,其中小球藻是一类单细胞绿藻,属于绿藻门、小球藻属,广泛分布于自然界,淡水水域中种类最多[9]。已有研究表明,小球藻含丰富的蛋白质、脂质、多糖、食用纤维、维生素、微量元素和活性代谢产物[10]。莱茵衣藻属于绿藻门、衣藻属,是一类单细胞真核鞭毛藻类,广布于水沟、洼地和含微量有机质的小型水体中,早春晚秋最为繁盛。一些含蛋白质较丰富的种类,可培养作饲料或食用[11]。微绿球藻属于绿藻门、胶球藻科,具有易培养、繁殖迅速、营养丰富的特点,在水产养殖中应用较为广泛。微绿球藻中含有丰富的碳水化合物、蛋白质、脂肪和多种微量元素[12]。
四种微藻对生物柴油的生产都是很好的选择[2],作为高产油微藻在实验室条件下易培养,生长速度快,因形态和大小相似在显微镜下无法辨别开。微藻所用培养基根据藻种库提供的标准来配制,普通小球藻、蛋白核小球藻和莱茵衣藻均为SE培养基培养,微绿球藻为f/2培养基培养。将四种藻在相同的温度、光照(25 ℃,2500 Lux-3500 Lux,周期为12 h昼12 h夜)下培养。
1.2 样本制备
分别取培养的四种藻液10 mL于15 mL的离心管中,以相对浓度梯度为0.8的n次方来稀释藻液,共10个浓度梯度。所含藻液分别是:10,10×0.8,10×0.82,10×0.83,10×0.84,10×0.85,10×0.86,10×0.87,10×0.88和10×0.89mL,即10,8.0,6.4,5.1,4.1,3.3,2.6,2.1,1.7和1.3 mL。之后,用蒸馏水稀释,分别获得10 mL样品在10个浓度水平。每种微藻重新取样共测三次,可采集到120个样本(每种30个)的可见/近红外透射光谱,随机获取80个样本用作建模,其余40个样本用于预测。
1.3 可见/近红外光谱的采集
构建的光谱采集系统如图1所示,它由一个便携式USB4000微型光纤光谱仪、用于传输和接收的Y形光纤、卤素光源和一台电脑组成。其中光纤一端连接有光纤探针。研究提出的浸入式光谱采集方法是指通过把光纤探针浸泡于藻液中固定的深度来相继得到光谱数据。在环境温度为23~26 ℃范围内采集光谱,光谱范围是346~1 038 nm,每隔0.3 nm采集一次。平均测量次数为5次,积分时间为13 ms。采集光谱数据前应通过蒸馏水来校准,所得数据由电脑中的光谱采集软件快速处理并保存。所采用的化学计量学算法和数字图像处理技术主要由Unscrambler○R10.1(CAMO AS, Oslo, Norway),MATLAB7.11(The Mathworks, Natick, USA),OriginPro 8.5(OriginLab, Northampton, USA)软件实现。
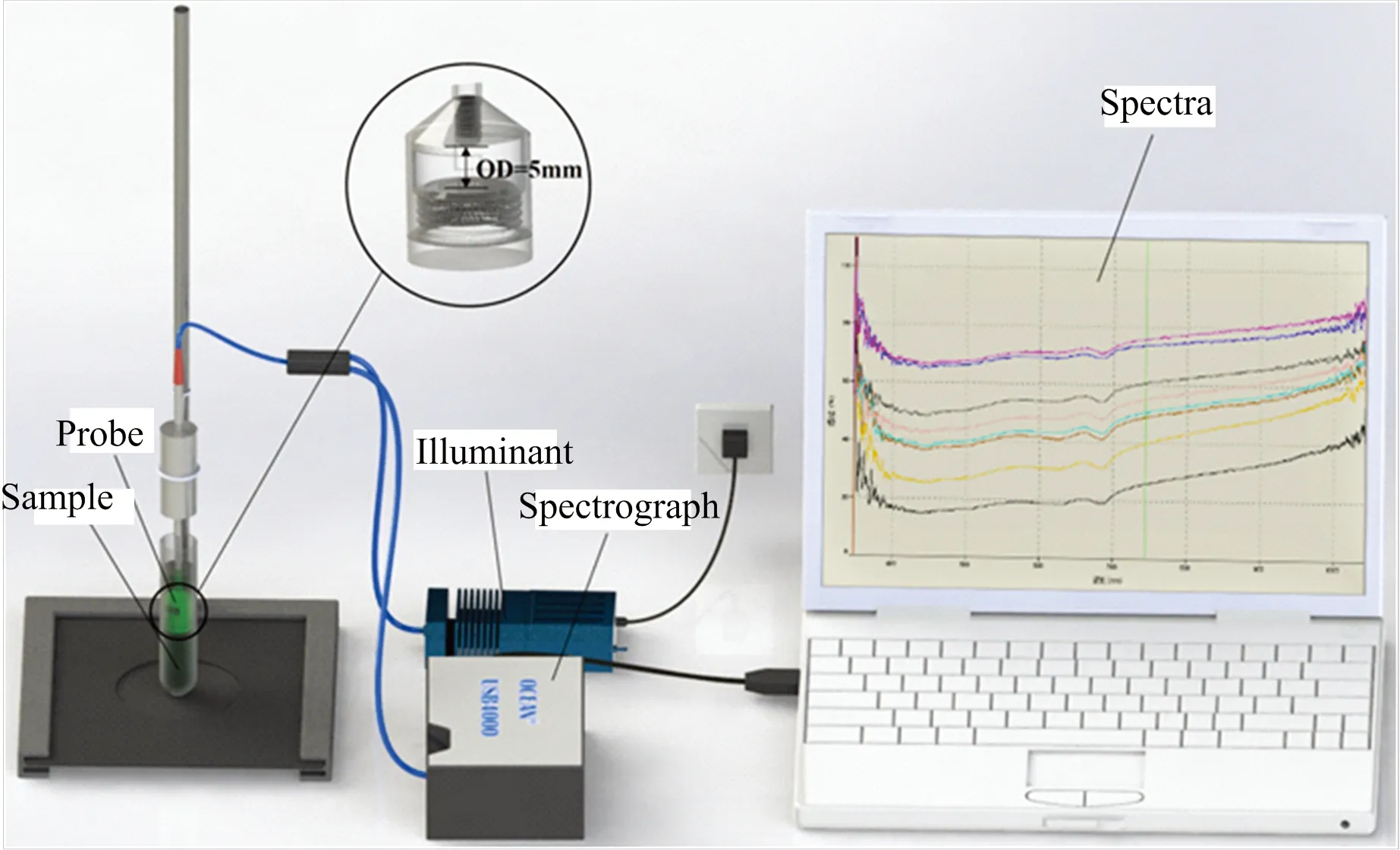
Fig.1 The spectral acquisition system for identification of microalgae species
1.4 化学计量学方法
通常完整的光谱可能包含数百个变量,因此减少不提供信息的变量是一个重要的方法和策略来获得更好的预测和简单而有效的模型。使用最佳波长比全波长在多变量分析中可能更有效。连续投影算法(SPA)是新发展的包含三个阶段的一种算法[13]。用SPA后,信息变量拥有最少共线性,减少了数据冗余度。偏最小二乘法(PLS)是最常用的一种光谱建模算法,PLS能够在矩阵分解过程中考虑光谱和物质目标特性,因此可以更好地选取光谱中有利于建模的信息,减少干扰组分因素。极限学习机(ELM)是黄广斌教授于2004年针对SLFNs(即含单个隐藏层的前馈型神经网络)的监督型学习算法提出来的一种简单实用的新型算法[14]。它能够有效克服传统神经网络训练参数选取复杂、易陷入局部最优等问题,并以其学习速度快、泛化能力强等优点被广泛关注[15]。Suykens提出的最小二乘支持向量机(LS-SVM)是对经典支持向量机(SVM)的一种改进。LS-SVM只需要对一个等式方程组求解对偶空间中的二次规划问题,从而降低了计算的复杂性,并且加快了计算的速度[16]。
2 结果与讨论
2.1 不同藻种的可见/近红外光谱特征
四种微藻的可见/近红外光谱图平均曲线见图2。图中横坐标为光谱波长,采用藻种光谱波长范围是346~1 038 nm,纵坐标为透射率。由图可知,4条曲线没有明显区别,且峰值均出现在450,650和690 nm附近,采用原始光谱很难将各个微藻种类分开。为消除光谱数据在采集时首端与末端产生的部分噪音,对图2截取430~930 nm波段的光谱数据进行分析。参与鉴别的光谱数据阵从120×3 649减少到120×2 582。
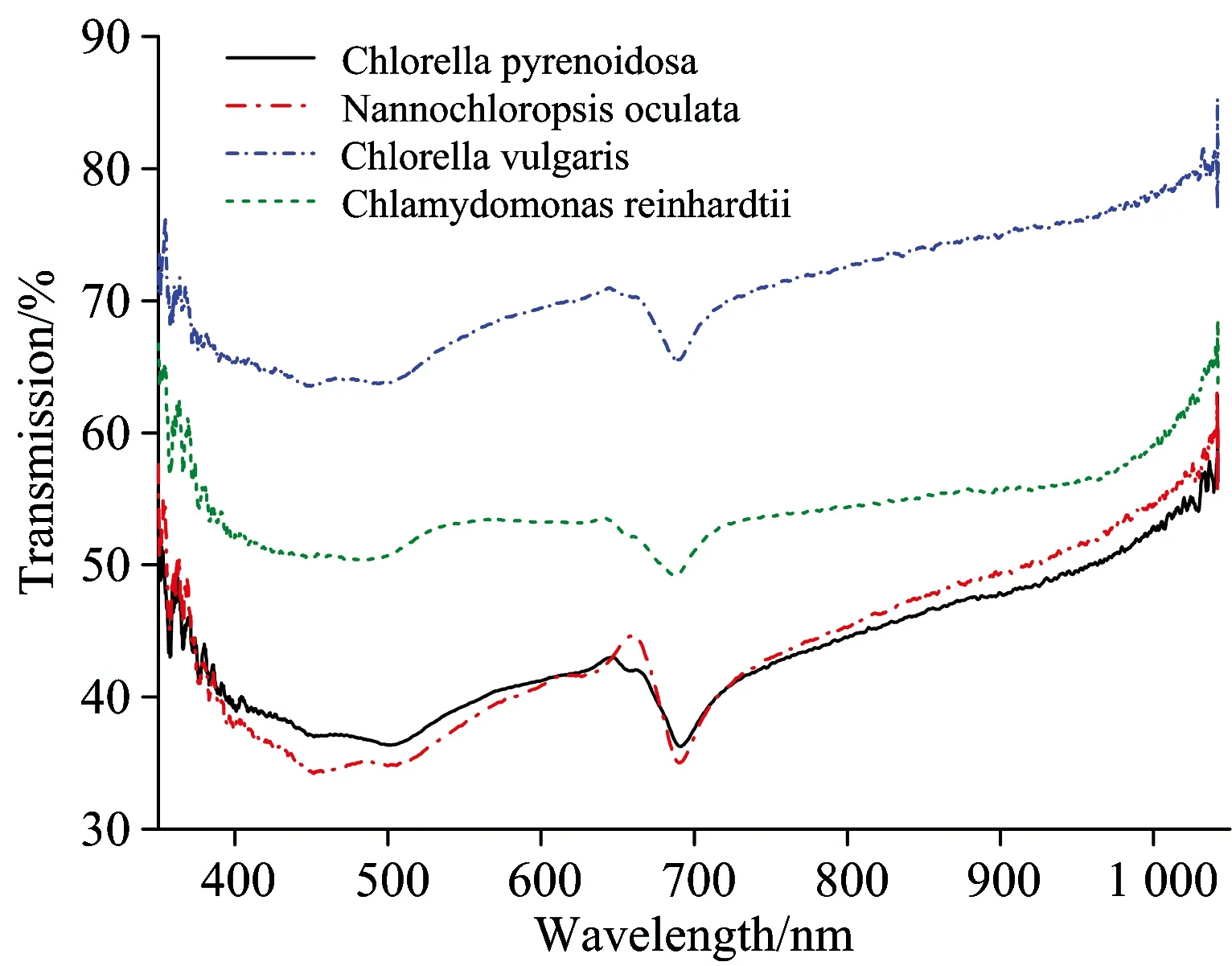
Fig.2 Visible/Near infrared transmission spectroscopy of four different microalgae species
2.2 光谱预处理方法的比较
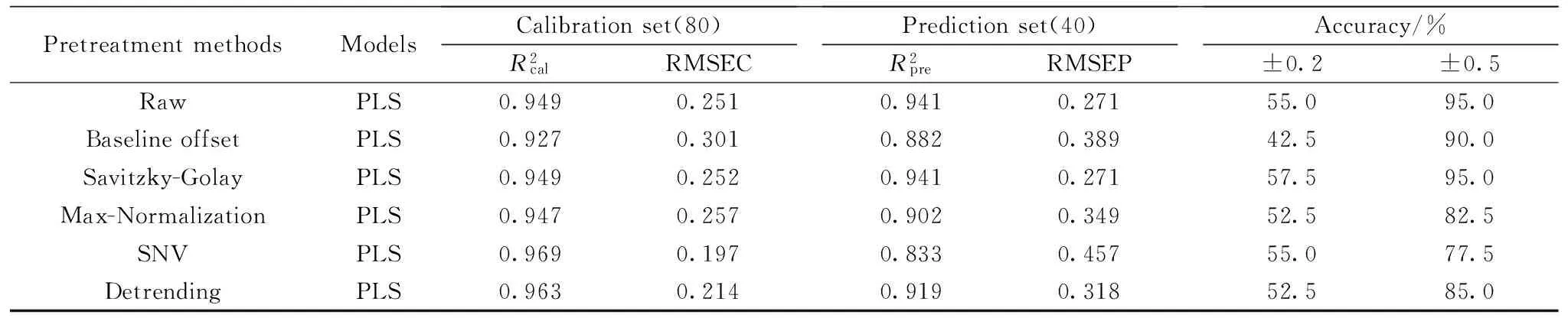
Table 1 Prediction results of identification of microalgae species with pretreatment methods
2.3 SPA选取有效波长
用SPA对光谱数据进行处理,被SPA选择的特征变量会根据重要性进行排列。用SPA选择的最相关的变量被称为有效波长(EWs)。从光谱中选择出6个有效波长(512.8,688.48,442.23,432.27,713.46和929.4 nm)。所选择的波长被认为是模拟波长,可能和四种微藻中色素含量有关。选择的有效波长(442.23,432.27和512.8 nm)可能和类胡萝卜素的吸收有关,因为四种微藻中所含色素主要包括β-胡萝卜素、海胆酮、玉米黄质,这些色素已被证实在450和510 nm的范围内有很强的吸收谱带[17]。出现稍微的偏移可能是因为:(1)不同藻种所含的色素含量不同;(2)同藻种不同个体的色素含量不同;(3)同一个体不同采集时间的色素含量变化影响采集的光谱变化。
另外,Workman等[18]已经证实,在玉米黄质分子中有两个R—C—OH在513 nm处有特征峰,这在某种程度上可以解释所选择的有效波长512.8 nm。688.48 nm的有效波长可能是因为叶绿素a在675 nm处有特征峰有关,也可能是其颜色在可见光区。而713.46和929.4 nm可能对微藻中的某些物质具有敏感性。因此,以上的波长分配表明SPA对四种微藻中相关变量的选择是很有帮助的。
2.4 全波段和有效波长PLS结果比较
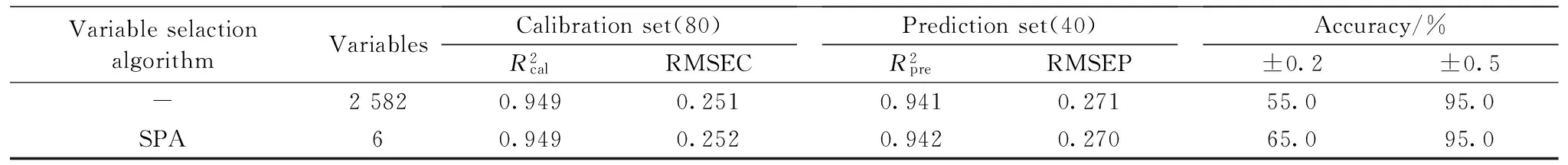
Table 2 PLS modeling results of full spectral and effective wavelengths
2.5 SPA-PLS,SPA-LV-SVM,SPA-ELM建模结果比较
通过用SPA选择出的6个有效波长作为模型输入值,输出值为微藻的不同品种(将蛋白核小球藻、微绿球藻、普通小球藻、莱茵衣藻品种值分别设为1,2,3,4)。进行的SPA-PLS模型是用化学计量学软件Unscrambler进行的分析,且计算结果是阈值为±0.2时的正确率。利用Matlab采用自编程序LV-SVM和ELM进行比较。由表3可知,三者的平均预测正确率分别是65%,80%,85%,SPA-LV-SVM和SPA-ELM的预测效果显著高于SPA-PLS。表中蛋白核小球藻和莱茵衣藻的预测正确率不如微绿球藻和普通小球藻。但用ELM的方法,4者的预测正确率均在80%以上。说明LV-SVM,ELM和SPA结合的方法能将四种微藻有效地辨别开。
3 结 论
以普通小球藻、蛋白核小球藻、微绿球藻和莱茵衣藻作为研究对象,通过自构建的光谱采集系统获取四种微藻的浸入式可见/近红外透射光谱信息。用Unscrambler软件处理相关的光谱数据,比较了不同预处理方法下PLS建模的藻种鉴别效果,卷积平滑的处理效果最好。用SPA选出的6个特征波长与微藻所含的色素有关。同时,SPA-PLS与Full-Spectral-PLS相比,不仅正确率有所提高,光谱变量也有99.78%的显著降低。说明利用SPA来提取有效波长能够极大地提高计算效率和简化运算过程。采用Matlab自编程序LV-SVM,ELM和PLS进行比较,SPA-LV-SVM和SPA-ELM的预测效果显著高于SPA-PLS,能够有效鉴别四种微藻。因此提出的SPA-LV-SVM和SPA-ELM组合算法为复杂体系的光谱非线性建模提供了一种有力的工具。
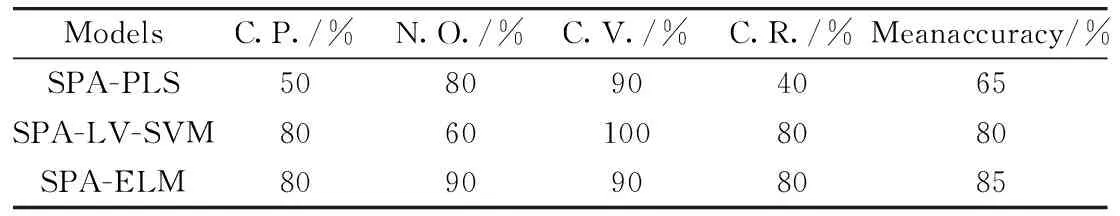
Table 3 Results of classification models using effective wavelengths for classifying microalgae
Note:C.P.:Chlorellapyrenoidosa, N.O.:Nannochloropsisoculata, C.V.:Chlorellavulgaris, C.R.:Chlamydomonasreinhardtii
研究所建立的浸入式可见/近红外光谱技术藻种鉴别方法,有效实现了对四种微藻的鉴别,为藻种鉴别和藻种分类研究领域提供了一种新思路。与化学分类法相比,该法具有无需配制任何溶液以及化学测定、简化的操作步骤、较短的检测时间等优点,同时也避免了由于操作人员操作不熟练或者主观因素带来的测量误差,分类预测结果更为准确。同时,可见/近红外光谱和便携式光纤探针结合的方法在微藻种类原位鉴别方面提供了一种新型、快速、无损的方式,应用前景广阔。
[1] Wei X, Jie D, Cuello J J, et al. Trac Trends in Analytical Chemistry, 2014, 53: 33.
[2] Chisti Y. Biotechnology Advances, 2007, 25(3): 294.
[3] Spolaore P, Joannis-Cassan C, Duran E, et al. Journal of Bioscience and Bioengineering, 2006, 101(2): 87.
[4] Hu Q, Sommerfeld M, Jarvis E, et al. The Plant Journal, 2008, 54(4): 621.
[5] GAO Yang, LIANG Jun-rong, GAO Ya-hui, et al(高 杨,梁君荣,高亚辉,等). Marine Sciences(海洋科学), 2005, 29(1): 67.
[6] YAO Peng, YU Zhi-gang(姚 鹏,于志刚). Marine Environmental Science(海洋环境科学), 2003, 22(1): 75.
[7] Reid L M, Woodcock T, O’Donnell C P, et al. Food Research International, 2005, 38(10): 1109.
[8] He Y, Li X, Deng X. Journal of Food Engineering, 2007, 79(4): 1238.
[9] Ahmad A L, Yasin N H M, Derek C J C, et al. Environmental Technology, 2014, 35(17): 2244.
[10] HU Kai-hui, WANG Shi-hua(胡开辉,汪世华). Journal of Wuhan Polytechnic University(武汉工业学院学报), 2006, 24(3): 27.
[11] ZHANG Chi, HU Hong-jun, LI Zhong-kui, et al(张 弛,胡鸿钧,李中奎,等). Journal of Wuhan Botanical Research(武汉植物学研究), 2000, 18(3): 189.
[12] Umdu E S, Tuncer M, Seker E. Bioresource Technology, 2009, 100(11): 2828.
[13] Araújo M C U, Saldanha T C B, Galvo R K H, et al. Chemometrics and Intelligent Laboratory Systems, 2001, 57(2): 65.
[14] Huang G B, Zhu Q Y, Siew C K. Neural Networks, Proc. IEEE International Joint Conference on, 2004, 2: 985.
[15] GUO Wen-chuan, WANG Ming-hai, GU Jing-si, et al(郭文川, 王铭海, 谷静思, 等). Optics and Precision Engineering(光学精密工程), 2013, 21(10): 2720.
[16] Langeron Y, Doussot M, Hewson D J, et al. Engineering Applications of Artificial Intelligence, 2007, 20(3): 415.
[17] HUI Bo-di(惠伯棣). Carotenoid Chemistry and Biochemistry(类胡萝卜素化学及生物化学). Beijing: China Light Industry Press(北京: 中国轻工业出版社), 2005. 152.
[18] Workman Jr J, Weyer L. Practical Guide to Interpretive Near-Infrared Spectroscopy. CRC Press, 2007.
*Corresponding author
Identification of Microalgae Species Using Visible/Near Infrared Transmission Spectroscopy
ZHU Hong-yan1, SHAO Yong-ni1, JIANG Lu-lu2, GUO An-que3, PAN Jian1, HE Yong1*
1. College of Biosystems Engineering and Food Science, Zhejiang University, Hangzhou 310058, China
2. Zhejiang Technology Institute of Economy, Hangzhou 310018, China 3. College of Enology, Northwest A&F University, Yangling 712100, China
At present, the identification and classification of the microalgae and its biochemical analysis have become one of the hot spots on marine biology research. Four microalgae species, includingChlorellavulgaris,Chlorellapyrenoidosa,Nannochloropsisoculata,Chlamydomonasreinhardtii, were chosen as the experimental materials. Using an established spectral acquisition system,which consists of a portable USB 4000 spectrometer having transmitting and receiving fiber bundles connected by a fiber optic probe, a halogen light source, and a computer, the Vis/NIR transmission spectral data of 120 different samples of the microalgae with different concentration gradients were collected, and the spectral curves of fourmicroalgae species were pre-processed by different pre-treatment methods (baseline filtering, convolution smoothing, etc.). Based on the pre-treated effects, SPA was applied to select effective wavelengths (EWs), and the selected EWs were introduced as inputs to develop and compare PLS, Least Square Support Vector Machines (LS-SVM), Extreme Learning Machine (ELM)models, so as to explore the feasibility of using Vis/NIR transmission spectroscopy technology for the rapid identification of four microalgae species in situ. The results showed that: the effect of Savitzky-Golay smoothing was much better than the other pre-treatment methods. Six EWs selected in the spectraby SPA were possibly relevant to the content of carotenoids, chlorophyll in the microalgae. Moreover, the SPA-PLS model obtained better performance than the Full-Spectral-PLS model. The average prediction accuracy of three methods including SPA-LV-SVM, SPA-ELM, and SPA-PLS were 80%, 85% and 65%. The established method in this study may identify four microalgae species effectively, which provides a new way for the identification and classification of the microalgae species. The methodology using Vis/NIR spectroscopy with a portable optic probe would be applicable to a diverse range of microalgae species and proves to be a rapid, real-time, non-destructive, precise method for the physiological and biochemical detection for microalgae.
Microalgae; Visible/Near infrared (Vis/NIR) transmission spectroscopy; Species identification; Extreme Learning Machine(ELM)
Aug. 29, 2014; accepted Dec. 15, 2014)
2014-08-29,
2014-12-15
浙江省自然科学基金项目(LY14C130008),教育部博士点基金项目(20130101120149),国家自然科学基金项目(31072247)和浙江省教育厅科研项目(Y201327409)资助
朱红艳,女,1992年生,浙江大学生物系统工程与食品科学学院博士研究生 e-mail: hyzhu-zju@foxmail.com *通讯联系人 e-mail:yhe@zju.edu.cn
S931.1
A
10.3964/j.issn.1000-0593(2016)01-0075-05
- 光谱学与光谱分析的其它文章
- Determination of Component Contents of Blend Oil Based on Characteristics Peak Value Integration
- Identification of Haploid Maize Kernel Using NIR Spectroscopy in Reflectance and Transmittance Modes: A Comparative Study
- 基于光谱吸收法和荧光法的甲烷和二氧化硫检测系统的研究
- 基于TDLAS-WMS的痕量甲烷气体检测仪
- 推扫误差对计算光谱成像数据重构的影响分析
- ICP-MS用于云南南部四种特色蜂蜜的植物源鉴别分析

