5株PCV2四川株全基因组的克隆与遗传进化分析
杨泽晓,马玉峰,汪开毓,王 印,赵希仑,姚学萍,任冉阳,陈 平,胡 凌
(1 四川农业大学 动物医学院,四川 温江 611130;2 动物疫病与人类健康四川省重点实验室,四川 温江 611130;3 金宇保灵生物药品有限公司,内蒙古 呼和浩特 010030)
5株PCV2四川株全基因组的克隆与遗传进化分析
杨泽晓1,2,马玉峰1,3,汪开毓1,王印1,2,赵希仑1,姚学萍1,任冉阳1,陈平1,胡凌1
(1 四川农业大学 动物医学院,四川 温江 611130;2 动物疫病与人类健康四川省重点实验室,四川 温江 611130;3 金宇保灵生物药品有限公司,内蒙古 呼和浩特 010030)
[摘要]【目的】 了解目前四川省猪圆环病毒2型(PCV2)的分子流行病学特征和遗传变异情况,探讨PCV2感染的控制对策。【方法】 根据GenBank中发表的PCV2基因组序列设计2对特异性引物,对2012-2014年临床送检的PCV2感染病例材料中的5株PCV2全基因组序列进行扩增测序,并利用DNAStar等生物信息分析软件对获得的PCV2基因组序列进行分子特性与遗传演变分析。【结果】 5株PCV2四川株全基因组序列长度都为1 767 bp,均具有滚环复制起点(ORC)典型结构。序列比对和进化分析显示,5株PCV2四川株的基因序列同源性很高,基因组核苷酸序列及ORF1、ORF2和ORF3基因序列同源性分别为98.4%~99.9%,97.5%~99.6%,99.4%~100%和96.2%~100%,ORF1、ORF2和ORF3基因推导氨基酸序列同源性分别为98.4%~99%,99.6%和91.5%~100%;与47条参比PCV2毒株核苷酸序列的同源性比较发现,5株PCV2与国内一些新出现的PCV2毒株亲缘关系较近,且均属于PCV2d基因型毒株。【结论】 5株PCV2四川株之间尽管存在着不同程度的基因变异,但遗传进化相对较稳定。
[关键词]PCV2;基因克隆;遗传进化分析
猪圆环病毒(Porcine circovirus,PCV)是一种单股环状DNA病毒,病毒子呈球形,无囊膜,直径17~20 nm,属于圆环病毒科(Circoviridae)圆环病毒属(Circovirus)成员[1]。目前常见的PCV包括PCV1和PCV2 2种血清型,其基因组长度分别为 1 759 和1 768 bp(或1 767 bp)[2],其中PCV1发现于传代细胞PK-15中,可能广泛存在于猪体内以及猪的原代和传代细胞系中,几乎无致病性,也被称为非致病PCV(npPCV)[3];而PCV2具有致病性,被认为是导致断奶仔猪多系统衰竭综合征(PMWS)的主要病原[1],此外PCV2还与怀孕母猪繁殖障碍(SAMS)[4]、幼龄仔猪A2型先天性震颤(CT-A2)[5]、断奶猪和育肥猪的呼吸道疾病(PRDS)、猪皮炎和肾炎综合征(PDNS)[6]、产房仔猪拉稀[7]、间质性肺炎(IP)、增生性坏死性肺炎(PNP)等疾病密切相关,因此又将PCV2感染称为猪圆环病毒相关疾病(Porcine circovirus-associated disease,PCVAD)或者猪圆环病毒病(PCVD)[7-8]。由于PCV2致病机理尚不清楚,这增加了其控制难度,但一般认为猪只感染PCV2后免疫功能会受到损害,机体抵抗力降低,更易遭受其他病原的并发、继发感染或者在应激条件下发病甚至加重病情[7]。20世纪90年代,PCV2感染作为PMWS病因在加拿大被报道后,世界很多国家证实有PCV2的存在和流行[2],成为目前全球养猪生产中被广泛关注的一种病原。我国自2000年发现猪群中存在PCV2感染以来,由PCV2感染所致疾病不断见诸报道,已经给养猪业造成重大经济损失,严重威胁着养猪业的健康发展[7]。近些年来随着PCV2灭活疫苗的使用,其流行情况得到了一定程度的控制,但是在临床检测诊断中仍有PCV2感染病例不断出现[9],加之与其他疾病的混合感染,PCV2感染控制的形势仍然十分严峻。研究表明,PCV2基因序列的遗传变异和演变可能与PCV2毒力变化、致病机理及PCVAD发生等相关,2004年de Boisséson等[10]研究发现,来源于PMWS猪群与无PMWS猪群的PCV2序列同源性为94.6%~99.9%,且非PMWS猪群来源的PCV2序列比PMWS猪群来源的PCV2序列变异大;还有研究分析认为,PCV2的变异演化与时间有一定的相关性,其变异周期约为3年[11];且2009年郭龙军等[12]报道在我国已经发现2株基因组大小为1 766 bp的PCV2毒株。因此,研究PCV2近年来在我国的遗传变异情况和演变规律,对目前PCVAD等疾病发生和控制的相关研究具有重要意义。为了解目前四川省PCV2流行株的遗传变异情况,本研究设计并合成了2对特异性引物,克隆了2012-2104年临床送检的5株PCV2病毒全基因组,并进行序列分析,以期为进一步控制PCV2感染发生的相关研究提供科学材料和依据。
1材料与方法
1.1毒株、菌种及试剂
DL2000 DNA Marker和pMD19-T载体购自成都飞克(天泰)科技有限公司;蛋白酶K、2×Taq PCR Mastermix、E.coliDH5α感受态细胞、琼脂粉、TIANpre Mini Plasmid Kit(DP103)和TIANgel Midi Purification Kit(DP209)等购自成都安雅生物试剂有限公司;PCV2感染病料样品5份(对应毒株分别标记为SCh201201、SCh201202、SCh201301、SCh201401和SCh201402)由四川农业大学动物检疫研究室收集保存。
1.2引物设计与合成
根据GenBank(http://www.ncbi.nlm.nih.gov)中发表的PCV2基因组序列,用DNAStar和Premier5.0软件分析后将PCV2基因组分为分别包含ORF1和ORF2基因的2个大片段(命名为Fa、Fb片段)设计2对特异性引物,同时合成pMD19-T 载体测序通用引物MR13-47和RV-M,3对引物的信息见表1。引物由上海英潍捷基贸易有限公司合成,按照合成说明书将其稀释至10 μmol/L,-20 ℃冻存备用。
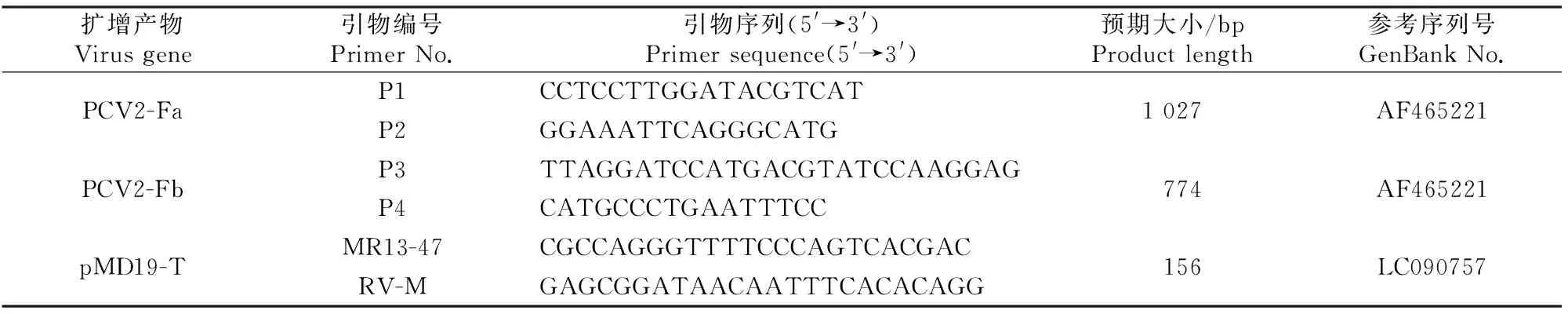
表 1 PCV2基因组PCR扩增相关引物
1.3PCV2四川株基因的提取
取5份受检PCV2感染阳性样品并标记,以正常组织样品作阴性对照,按照参考文献[2]的方法抽提病毒基因组DNA,-20 ℃冻存备用。
1.4PCV2四川株基因组片段的PCR扩增
分别以1.3节抽提的5份受检PCV2病料总DNA和正常组织样品DNA作为检测和阴性对照模板,以P1/P2、P3/P4为引物对,按下面反应体系和反应程序进行PCR扩增。反应体系为:2×PCR PreMixture 25 μL,上、下游引物(10 μmol/L)各1.0 μL,DNA模板2 μL,ddH2O 21 μL。反应程序为:95 ℃预变性5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 1 min,35个循环; 72 ℃延伸10 min。反应结束后取5 μL产物进行10 g/L琼脂糖凝胶电泳分析。
1.5目的基因的克隆鉴定
按TIANgel Midi Purification Kit(DP209)说明书纯化回收5株PCV2的PCR产物;按照pMD19-T Vector kit 说明书进行连接,并将连接产物转化DH5α感受态细胞,接种含氨苄青霉素(Amp)的LB平板,挑取阳性克隆扩增培养;按TIANpre Mini Plasmid Kit(DP103)说明书抽提重组质粒,分别用特异性引物(P1/P2、P3/P4)和pMD19-T载体通用引物MR13-47/RV-M(表1)进行抽提质粒的PCR鉴定,反应体系和程序同1.4节。将2种PCR鉴定均为阳性的克隆送往上海英潍捷基贸易有限公司进行序列测定。
1.6PCV2四川株基因组的序列分析
用DNAStar等软件对克隆的5株PCV2基因组序列进行分析后,收集GenBank中已经发表的国内外47条不同基因型的PCV2毒株序列[12-13]和2条PCV1毒株全序列(表2),进行PCV2基因组及其主要基因(ORF1、ORF2和ORF3)的核苷酸和氨基酸同源性分析;并使用MEGA4.1软件绘制PCV2四川株基因组序列和47条参考毒株基因组序列的系统进化树,进行遗传变异分析。
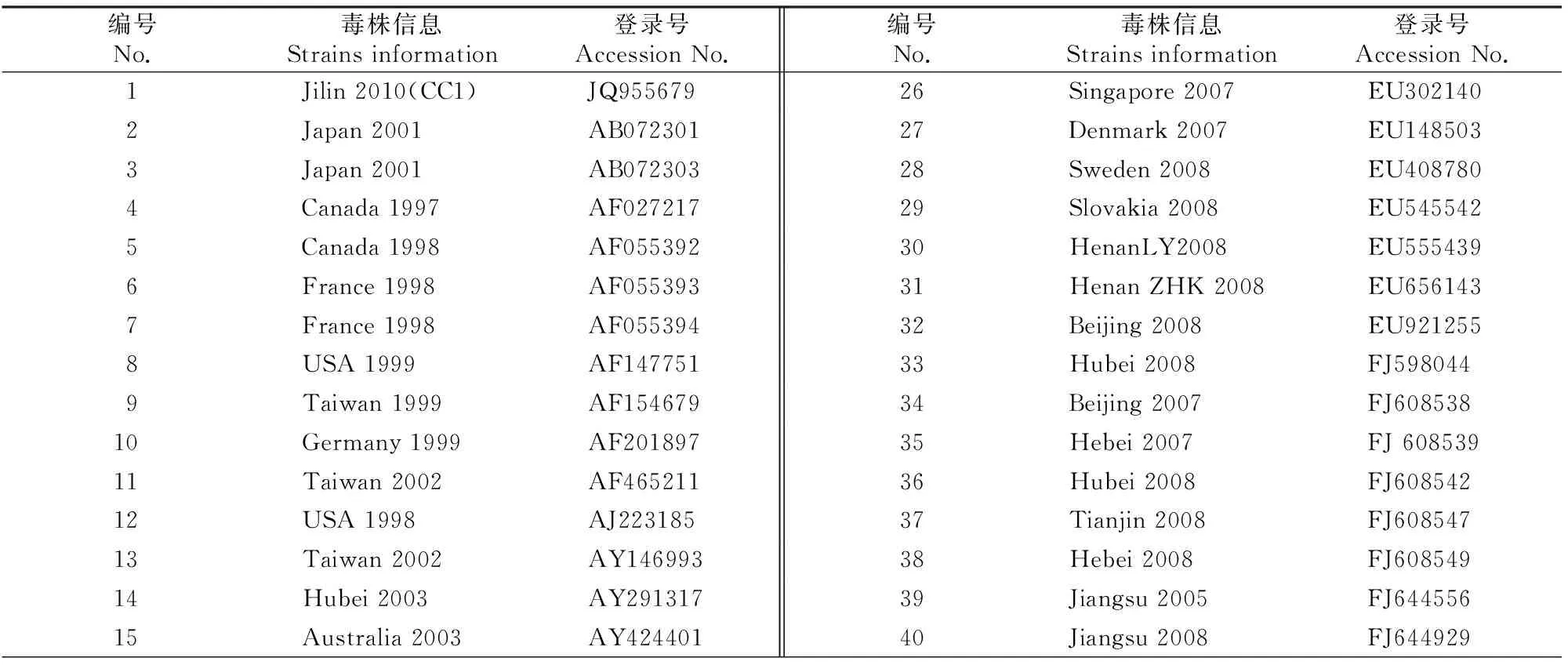
表 2 PCV2 全基因组序列分析的参考序列
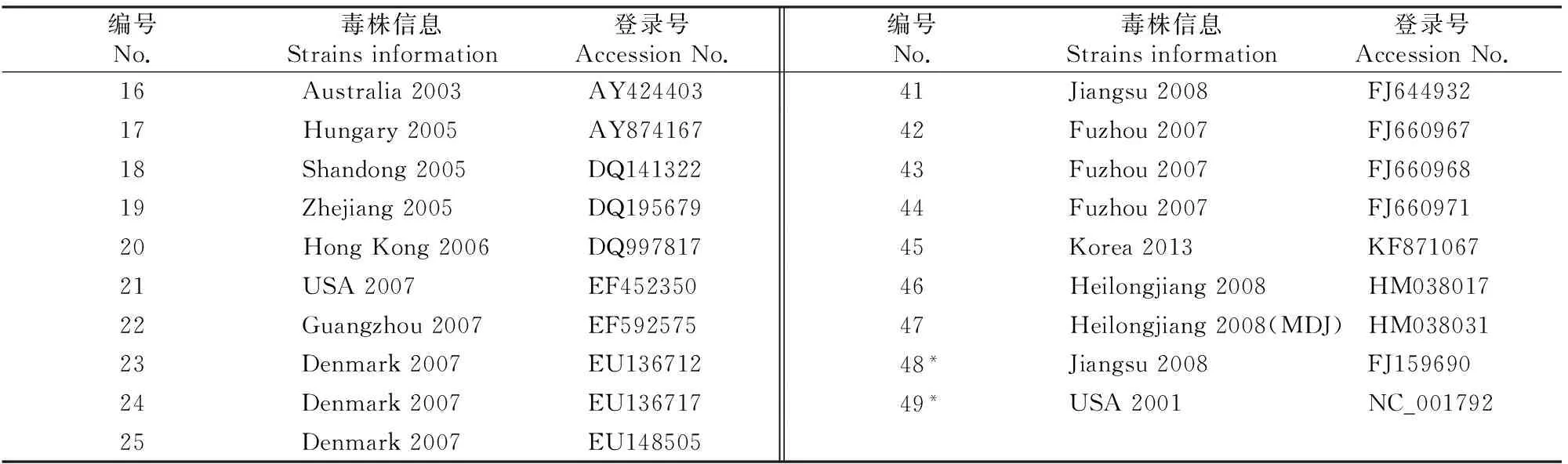
续表 2 Continued table 2
注:*为PCV1毒株。
Note:* means PCV1 isolate.
2结果与分析
2.1PCV2四川株基因组的PCR扩增
分别以P1/P2、P3/P4为引物,按照1.4节中的反应体系和反应程序,对1.3节抽提的受检PCV2阳性病料中5株PCV2的基因组进行PCR扩增,结果每株PCV2都扩增出约1 027 bp和774 bp与预期长度一致的2条特异性DNA条带(图1), 即Fa片段和Fb片段,阴性对照样品扩增结果为阴性。

图 1 5株PCV2四川株基因组的PCR扩增
2.2PCV2四川株基因组片段的克隆鉴定
按照1.5节操作分别纯化回收5株PCV2基因组扩增的10个预期大小DNA片段,连接转化和抽提重组质粒,然后分别用相应特异性引物和pMD19-T 载体通用引物进行PCR鉴定,结果如图2所示。由图2可知,5株PCV2基因Fb片段重组质粒均扩增出约774 bp的特异性片段,而用pMD19-T载体通用引物(RV-M/MR13-47)PCR均扩增出约930 bp的特异性片段;Fa片段重组质粒均扩增出约1 027 bp的特异性片段,而用pMD19-T载体通用引物PCR则均扩增出约1 183 bp的DNA片段,都与预期长度相一致。故将2种PCR鉴定均为阳性的重组质粒分别命名为SCh201201-Fa、SCh201201-Fb、SCh201202-Fa、SCh201202-Fb、SCh201301-Fa、SCh201301-Fb、SCh201401-Fa、SCh201401-Fb、SCh201402-Fa和SCh201402-Fb,并进行测序。测序结果通过NCBI网站Blast分析显示,重组质粒连接基因片段与GenBank中提交的PCV2序列(GU325763.1、FJ644929.1、HQ113117.1等)的同源性均高达99%,表明成功克隆了5株PCV2基因组序列并构建了其重组质粒。5株PCV2的基因组测序结果经拼接后已提交GenBank,PCV2毒株SCh201201、SCh201202、SCh201301、SCh201401和SCh201402的登录号分别为KF530836、KF530837、KJ596438、KM272211和KM272212。
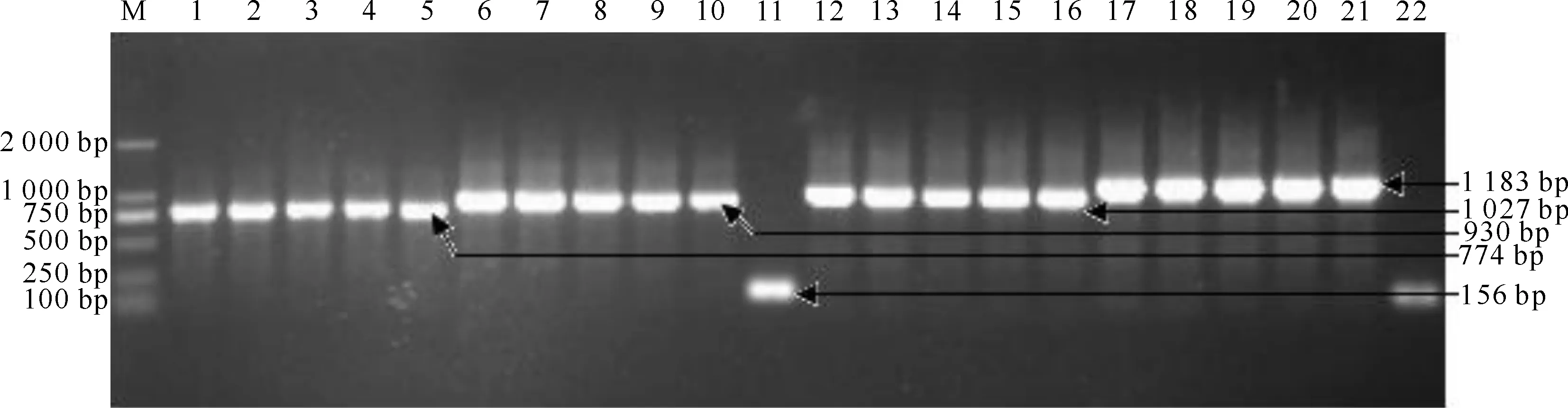
图 2 PCV2四川株基因组重组质粒的PCR鉴定
2.3PCV2四川株基因组的序列特征
采用DNAStar软件的MegAlign程序对5株PCV2基因组序列进行拼接和特征分析,结果表明,受检5株PCV2四川株的基因组序列全长均为 1 767 bp,其滚环复制起点(ORC)都属于典型的结构序列:包括环内9碱基保守序列(-AAGTATTAC-)、茎环结构下游4个(H1、H2、H3和H4)6碱基保守区(-CG/AGCAG-),H1与H2无间隔,H2、H3、H4相隔5个核苷酸(CACCT),仅H4碱基序列为-CAGCAG-。5株PCV2基因组的2个最大开放阅读框(ORFs)ORF1(Rep蛋白编码基因)和ORF2(Cap蛋白编码基因)长度不变,ORF1长945 bp,编码314个氨基酸残基;ORF2长705 bp,编码234个氨基酸残基;除SCh201402株(KM272212)ORF3基因(在ORF1的大阅读框内)第103位密码子点突变为终止密码子外,其他4株PCV2的ORF3基因长度均为315 bp,编码104个氨基酸残基。 5株受试PCV2基因组与基因组大小为1 768 bp的PCV2毒株(Canada 1998、Japan 2001等)相比,均在第1 042位置处缺失1个碱基T,与我国新出现的基因组大小为1 766 bp的PCV2毒株相比,Jilin 2010(CC1)株则在1 054位置处多缺失1个C碱基,而Heilongjiang 2008(MDJ)株是在39位置处多缺失1个C碱基。
2.4PCV2四川株全基因组同源性比较与遗传进化分析
采用DNAStar软件对供试的5株PCV2四川株基因组序列与GenBank中发表的国内外49条毒株参考序列(表2)进行核苷酸同源性分析,结果表明,5株供试PCV2四川株的核苷酸同源性为98.4%~99.9%,其中SCh201401(KM272211)与SCh201301(KJ596438)同源性最低,为98.4%,5株供试PCV2四川株的核苷酸同源性处于参比的47株PCV2毒株序列的核苷酸同源性93%( Denmark 2007与Sweden 2008)~99.9%(Australia 2003与Australia 2003)内;与2株PCV1全序列(Jiangsu 2008、USA 2001)的同源性为69.1%~69.6%,也在参比PCV2毒株与PCV1的序列同源性68.7%(Australia 2003与USA 2001)~70.5%(Denmark 2007与Jiangsu 2008)范围内,表明供试5株PCV2四川株基因组核苷酸序列遗传演化相对稳定。
使用MEGA4.1软件Boot-strapped Neighbor-Joining方法,对PCV2四川株基因序列和49条国内外PCV的毒株序列绘制系统发生进化树并进行遗传演化分析,结果见图3。由图3可知,PCV1和PCV2形成PCV的2个大分支。PCV2毒株又可分为较独立的4个分支(PCV2a,PCV2b,PCV2c,PCV2d),其中参比的12株基因组大小为1 768 bp的不同年份PCV2毒株形成PCV2a分支;参比的4株国内PCV2毒株(Heilongjiang 2008、Heilongjiang 2008(MDJ)、Hubei 2003、Jiangsu 2008)和1株PCV2新加坡株(Singapore 2007)形成PCV2d分支,另外本试验的5株PCV2四川株全都处于PCV2d基因型分支;2株PCV2丹麦株构成PCV2c分支;其他参比的28株年份不同的国内外PCV2毒株构成了数量庞大的PCV2b基因型分支,该分支包括了大多数国内毒株并呈现复杂的遗传变异趋势;基因型PCV2c和PCV2d毒株的出现时间相对较晚。
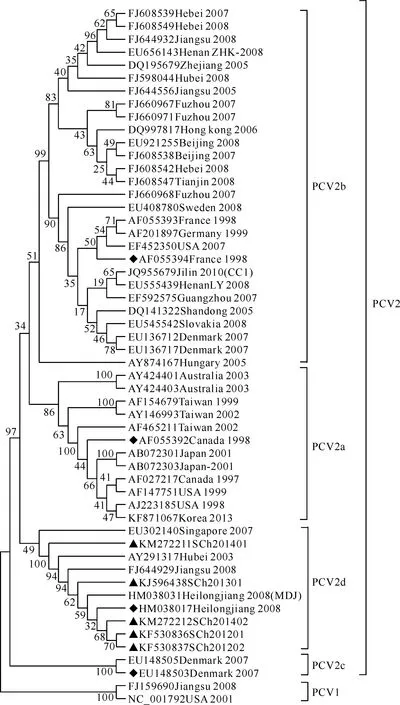
图 3 PCV2四川株基因组核苷酸序列的系统发育分析
2.5PCV2四川株重要蛋白基因及其推导氨基酸的序列分析
通过DNAStar软件,对5株PCV2四川株与国内外47条PCV2毒株参考序列的ORF1、ORF2和ORF3进行核苷酸和推导氨基酸序列的同源性比较发现,PCV2四川株间ORF1的核苷酸和氨基酸序列同源性分别为97.5%~99.6%和98.4%~99%,与参比毒株ORF1核苷酸和氨基酸序列的最低同源性分别是96.4%和97.1%(SCh201402与Taiwan 2002),而47条参考毒株ORF1间核苷酸和氨基酸序列的同源性分别为96.3%(Taiwan 2002与Heilongjiang 2008(MDJ)等)~100%(France 1998与Germany 1999等)和97.1%(Fuzhou 2007与HenanLY 2008等)~99.7%(Canada 1998与Korea 2013等)。PCV2四川株ORF2间核苷酸和推导氨基酸序列的同源性分别为99.4%~100%和99.6%,与参比毒株ORF2核苷酸与氨基酸序列的最低同源性分别是88.1%和87.2%(SCh201301与Denmark 2007),而47条参考毒株ORF2间核苷酸和推导氨基酸序列的同源性分别为84.3%(Taiwan 2002与Denmark 2007等)~100%(2株Denmark 2007间)和84.2%(Denmark 2007与Japan 2001、Australia 2003等)~99.6%(2株Denmark 2007间)。PCV2四川株ORF3间核苷酸和推导氨基酸序列的同源性分别为96.2%~100%和91.5%~100%,与参比毒株ORF3核苷酸与氨基酸序列的最低同源性分别是96.2%和91.5%(SCh201401与Denmark 2007等),而47条参考毒株ORF3核苷酸和推导氨基酸序列的同源性分别为95.9%(Jiangsu 2005与Denmark 2007)~100%(Hebei 2007与Hebei 2008等)和 92.5%(Hubei 2003与Denmark 2007)~100%(Hebei 2007与Hebei 2008等)。由同源性分析结果可知,参比PCV2毒株的3个主要ORFs中,其核苷酸和氨基酸序列的遗传稳定性表现为ORF1>ORF3>ORF2; PCV2四川株ORF1和ORF2核苷酸及其推导氨基酸序列与参比毒株最低同源性高于参比序列间的最低同源性,仅ORF3氨基酸序列与参比毒株最低同源性(91.5%)低于参比序列间的最低同源性(92.5%),但PCV2四川株间的ORF1、ORF2和ORF3基因序列同源性都在参比毒株间的序列同源性范围内,这表明PCV2四川株基因遗传仍相对稳定。
选取PCV2四川株与4种PCV2基因型参比株(每基因型选2株)的ORF2基因推导的氨基酸序列,通过DNAStar软件对其在5个抗原表位区A(57-91)、B(121-137)、C(183-191)、D(206-215)、E(230-233)[14]和核内定位信号区(N端的41个氨基酸)[15]的变异情况进行比对,结果见图4。
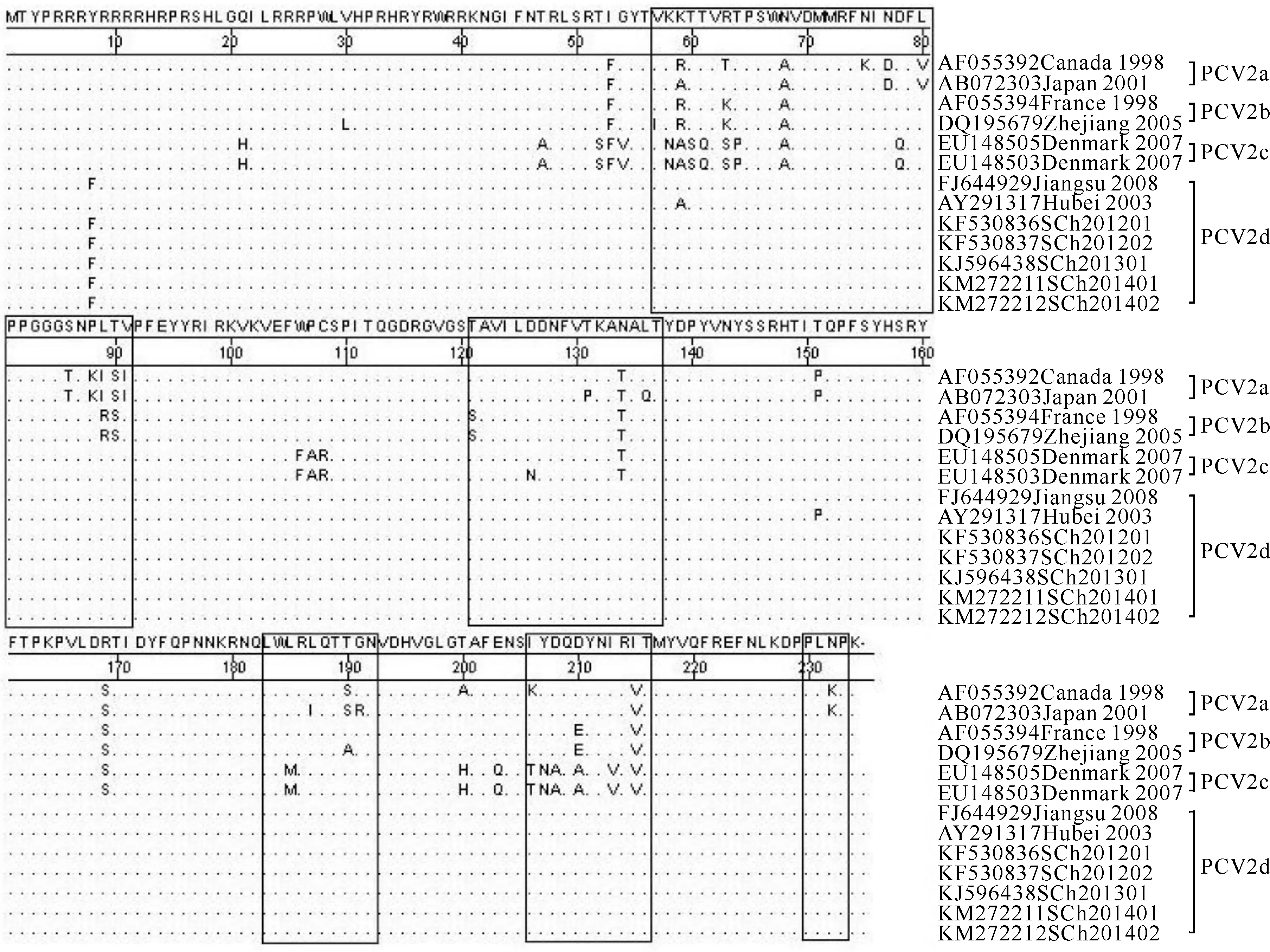
图 4PCV2 ORF2推导的氨基酸序列框内为参比的5个抗原表位区
Fig.4Induced amino acid sequences based on PCV2 ORF2 Antigentic epitope region of 5 reference in box
由图4可知,5株PCV2四川株ORF2编码的Cap蛋白都包括234个核苷酸残基,它们在5个抗原表位区和核内定位信号区的氨基酸高度一致,与其他2株d型PCV2相比,第8位氨基酸由R变为F,第59位氨基酸由A变为K,第151位氨基酸由P变为T;与其他基因型PCV2相比,在第8、21、30、47、52、53、54、57等74处氨基酸残基存在差异,其中第8、53、59、63、68、134和215位等7处氨基酸残基与其他基因型参比毒株均不同,包括A区3处、B区1处、D区1处和抗原表位以外区域2处。
3讨论
随着对PCV2感染控制研究和临床生产实践的深入,人们已经逐步认识到PCV2虽不像猪瘟病毒、高致病性蓝耳病毒和伪狂犬病毒那样毒力强大,但由于PCVAD对养猪生产危害的多系统性、多阶段性、多方位性、广泛性及其病程的长久性等特点,其对养猪业的危害可能比其他重要疫病的危害还要大[7]。因此控制和消灭PCVAD将具有重要而特殊的意义。本研究对最近3年分离的5株PCV2的基因组进行了克隆与分析,结果显示,受检5株PCV2四川株基因组全长1 767 bp,基因组核苷酸同源性在98.4%~99.9%,与参比的基因组大小为1 767 bp的PCV2毒株序列一样都有ORC典型的茎环保守结构,均在第1 042位比基因组大小为 1 768 bp的PCV2毒株缺失1个碱基T。对PCV2 的ORF1、ORF2和ORF3核苷酸和氨基酸序列同源性的比较发现,PCV2四川株3个主要开放阅读框的核苷酸和氨基酸的序列同源性(除SCh201402株ORF3蛋白氨基酸序列同源性仅为91.5%外)都分布在47个PCV2参比毒株间序列同源性范围内,表明5株PCV2四川株遗传变异相对稳定。考虑到ORF3蛋白参与PCV2感染细胞的凋亡,与PCV2致病性有关[14,16],加之SCh201402株ORF3蛋白氨基酸序列同源性低于参比毒株序列同源性,笔者对其ORF3基因序列进行了分析,结果显示,其ORF3基因第103个密码子突变为终止密码子,由于ORF3基因与Rep蛋白第103-207位氨基酸序列基因(ORF1)是反向互补序列,因此导致 Rep蛋白第105位氨基酸出现了非极性氨基酸错义突变,由异亮氨酸(I)突变为亮氨酸(L)。PCV2 SCh201402株ORF3在103位密码子的无意义突变能否被宿主内校正tRNA校正读译及其对Rep蛋白功能、病毒毒力的影响有待进一步研究。
对54株PCV的进化分析显示,PCV2a、PCV2b 2种基因型的毒株数量较多,且与一些欧洲分离株亲缘关系较近,包括不同年份的PCV2毒株,如早期出现的USA 1998株和France 1998株,出现较晚的HenanLY 2008株、Korea 2013株以及2013年卢权威等[13]分离于河南的2株PCV2;PCV2c基因型仅出现在国外,由2007年丹麦出现的2株PCV2毒株组成;PCV2d基因型主要由出现较晚的毒株组成,包括供试的5株PCV2四川株、国内的4株PCV2毒株与1株新加坡PCV2株。由此不难看出,虽然供试的5株PCV2四川株亲缘关系较近,同属于PCV2d基因型,但世界上PCV2的演变是多基因型化的,而不是基因型间的简单演替。此外,通过与2株基因组大小为1 766 bp的PCV2毒株[12](Jilin 2010(CC1)和Heilongjiang 2008(MDJ)株)比较分析发现,它们并没有像PCV2a基因型(1 768 bp)毒株一样单独形成一个新的基因亚型,而是分别处于PCV2b和PCV2d两个基因型分支上,序列比对显示它们分别在1 054处和39处不同位置出现核苷酸缺失,推测是来自于不同基因亚型PCV2毒株的突变,这也提示不同基因亚型PCV2毒株都存在缺失突变的潜力。总体来看,近年来的研究虽然显示目前PCV2遗传演变仍然相对稳定,但是PCV2a、PCV2b、PCV2c和PCV2d 4个基因型以及基因组大小为1 766 bp毒株的出现也暗示PCV2的遗传演变不断加剧且趋势复杂, PCV2毒株间的遗传距离不断扩大,变异方式不断复杂化——碱基转换、颠换和缺失等。目前人们在PCV2起源方面的研究认为: PCV1和PCV2基因组结构组成相似,同源性在68%以上,推测PCV1和PCV2间存在一定的亲缘关系,二者之间的差异可能是发生较大突变而形成的[17]。因此有理由相信PCV2在今后发生较大突变的可能性是存在的,国内外PCV2流行的复杂化趋势应该引起足够的重视。
PCV2 ORF2编码的主要结构蛋白(Cap) 具有良好的免疫原性,一般由233~234个氨基酸残基组成。本研究表明,5株PCV2四川株ORF2长度都为705 bp,氨基酸序列同源性为99.6%,在参比毒株间最低氨基酸序列同源性(87.2%)以上,表明PCV2四川株虽然属于新的PCV2d基因型,免疫原性蛋白仍然比较保守,现用的PCV2灭活疫苗应该可以针对PCV2四川流行株产生保护性抗体。然而不同基因型PCV2 毒株ORF2蛋白氨基酸序列比较结果显示,与其他参比PCV2相比,PCV2四川株Cap蛋白的氨基酸残基有74处发生了突变,其中有3处(第8、21和30位)发生在N端41个氨基酸以内,由于这些突变均不在核内对蛋白定位起重要作用的12-18和34-41两个区域[16]中,对PCV2蛋白的细胞核内定位不会产生显著影响,但是PCV2四川株有7处氨基酸残基与其他基因型参比毒株相比都发生了突变,并且5处发生在抗原表位区(A、B、D区),其毒株抗原性是否改变还有待血清学试验来证实。贾怀杰等[18]曾对PCV2-GSLZ株Cap蛋白的氨基酸同源性进行了比较,发现其同源性有的不超过15%;还有研究发现多个PCV2d型毒株不能被mAb 1D2识别[19]。由此可见,PCV2的ORF2变异相对不稳定,并且可能会影响到PCV2抗原表位的变化,因此随着PCV2的遗传演变,其血清型和致病力是否会受到影响还有待今后的研究证实。
本研究通过PCR等常规方法对2012-2014年PCV2四川株全基因组进行了克隆测序和遗传演变分析,结果显示,受检5株PCV2四川株间基因序列和氨基酸序列都具有很高的同源性,属于由我国一些新出现毒株组成的PCV2d基因型,与参比毒株相比出现了一定变异但遗传演化相对稳定。本研究补充和明确了近期四川省PCV2流行株的分子流行性病学特征,为PCV2感染的防控研究提供了新的科学材料和参考依据。
[参考文献]
[1]蔡宝祥.家畜传染病学 [M].4版.北京:中国农业出版社,2001:221-222.
Cai B X.Lemology of domestic animals [M].4th ed.Beijing:Chinese Agriculture Press,2001:221-222.(in Chinese)
[2]杨泽晓,庞歌,王印,等.猪圆环病毒多重PCR方法的建立 [J].西北农业学报,2010,19(11):8-12.
Yang Z X,Pang G,Wang Y,et al.Development of the multiplex PCR for porcine circovirus detection [J].Acta Agriculturae Boreali-occidentalis Sinica,2010,19(11):8-12.(in Chinese)
[3]Dulac G C,Afshar A.Porcine circovirus antigens in PK-15 cell line and evidence of antibodies to circovirus in Candian pigs [J].Can J Vet Res,1989,53:431-433.
[4]West K H,Bystrom J M,Wojnarowicz C,et al.Myocarditis and abortion associated within trauterine infection of sows with porcine circovirus 2 [J].J Vet Diagn Invest,1999,11:530-532.
[5]Stevenson G W,Kiupel M,Mittal S K,et al.Tissue distribution and genetic typing of porcine circoviruses in pigs with naturally occurring congenital tremors [J].J Vet Diagn Invest,2001,13(1):57-62.
[6]Molnar T,Glavits R,Szeredi L,et al.Occurrence of porcine der-matitis and nephropathy syndrome in Hungary [J].Acta Vet Hung,2002,50(1):5-16.
[7]宋长绪,梁鹏帅.猪圆环病毒病(PCVD)研究进展 [J].广东农业科学,2010(9):34-37.
Song C X,Liang P S.Research and progress of porcine circovirus disease (PCVD) [J].Guangdong Agricultural Sciences,2010(9):34-37.(in Chinese)
[8]Mesu A P,Labqrque G G,Naumynck H J,et al.Seroprevalence of porcine circovirus types 1 and 2 in the Belgian pig population [J].Vet Q,2000,22:234-236.
[9]杨泽晓,侯义宏,曾辉,等.猪圆环病毒2型 LAMP检测方法的建立与初步应用 [J].中国兽医科学,2014,44(2):152-158.
Yang Z X,Hou Y H,Zeng H,et al.Development and application of LAMP method for porcine circovirus type2 detection [J].Chinese Veterinary Science,2014,44(2):152-158.(in Chinese)
[10]de Boisséson C,Béven V,Bigarré L,et al.Molecular characterization of porcine circovirus type 2 isolates from post-weaning multisystemic wasting syndrome-affected and nonaffected pigs [J].Journal of General Virology,2004,85(2):293-304.
[11]Dupont K.Genomic analysis of PCV2 isolates from Danish archives and a current PMWS case control study supports a shift in genotypes with time [J].Vet Microbiol,2008,128:56-64.
[12]郭龙军,陆月华,危艳武,等.我国部分地区猪圆环病毒2型分离株的遗传变异分析 [J].中国预防兽医学报,2009,31(11):856-859.
Guo L J,Lu Y H,Wei Y W,et al.Genetic variation of porcine circovirus type 2 isolates from different regions in China [J].Chinese Journal of Preventive Veterinary Medicine,2009,31(11):856-859.(in Chinese)
[13]卢权威,郭官鹏,崔保安,等.2株猪圆环病毒2型的分离鉴定及全基因组序列分析 [J].西北农林科技大学学报:自然科学版,2013,41(9):43-48.
Lu Q W,Guo G P,Cui B A,et al.Isolation,identification,and genomic sequence of two PCV2 isolates [J].Journal of Northwest A&F University:Nat Sci Ed,2013,41(9):43-48.(in Chinese)
[14]陈陆,彭志锋,杨霞,等.10株猪圆环病毒2型(PCV2)河南株全基因组的克隆与序列分析 [J].中国兽医学报,2010,30(2):166-174.
Chen L,Peng Z F,Yang X,et al.Sequence analysis of complete genome of ten porcine circovirus type 2 strains in Henan province [J].Chinese Journal of Veterinary Science,2010,30(2):166-174.(in Chinese)
[15]Liu Q,Tikoo S K,Babiuk L A.Nuclear location of the ORF2 protein encoded by porcine circovirus type 2 [J].Virol,2001,285(1):91-99.
[16]Liu J,Chen I,Kang J.Characterization of a previously unidentified viral protein in porcine circovirus type 2-infected cells and its role in virus-induced apoptosis [J].Journal of Virology,2005,79(13):8262-8274.
[17]Mankertz J,Buhk H J,Blaess G,et al.Transcription analysis of porcine cir-covirus (PCV) [J].Virus Genes,1998,16(3):267-276.
[18]贾怀杰,景志忠,房永祥,等.猪圆环病毒2型甘肃兰州株全基因组序列的克隆及序列分析 [J].中国畜牧兽医,2010,37(1):45-51.
Jia H J,Jing Z Z,Fang Y X,et al.Cloning and sequence analysis of the complete genome of porcine circovirus type 2 Gansu Lanzhou strain [J].China Animal Husbandry & Veterinary Medicine,2010,37(1):45-51.(in Chinese)
[19]Wang F,Guo X,Ge X,et al.Genetic variation analysis of Chinese strains of porcine circovirus type 2 [J].Virus Res,2009,145(1):151-156.
Cloning and phylogenetic analysis of complete genome of five PCV2 strains from Sichuan
YANG Ze-xiao1,2,MA Yu-feng1,3,WANG Kai-yu1,WANG Yin1,2,ZHAO Xi-lun1,YAO Xue-ping1,REN Ran-yang1,CHEN Ping1,HU Ling1
(1CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Wenjiang,Sichuan611130,China;2KeyLaboratoryofAnimalDiseaseandHumanHealthofSichuanProvince,Wenjiang,Sichuan611130,China;3TheJinyubaolingBiologicalPhamceuticalCo.,Ltd,Hohhot,InnerMongolia010030,China)
Abstract:【Objective】 This study investigated the molecular epidemiology characteristics and genetic heterogeneity of PCV2 in Sichuan to provide reference for control and prevention of PCV2 infection.【Method】 Two pairs of special primers were designed and synthesized according to the full genome sequences of PCV2 published in GenBank.Then they were used to amplify and sequence the full genome sequences of 5 PCV2 strains in clinical samples of PCV2 infection cases collected from Sichuan during 2012 to 2014 using PCR.Molecular epidemiology characteristics and genetic heterogeneity of 5 PCV2 were also analyzed using DNAStar and MEGA 4.1.【Result】 The genomic sequences of 5 PCV2 strains were all 1 767 bp in the length and had the classic ORC structure in sequence.Phylogenetic analysis showed that the genomes of 5 PCV2 Sichuan strains were closely related to each other.The nucleotide homology of PCV2 full length genome,ORF1,ORF2 and ORF3 were 98.4%-99.9%,97.5%-99.6%,99.4%-100% and 96.2%-100%,respectively.The amino acid sequence homologies of ORF1,ORF2 and ORF3 were 98.4%-99%,99.6% and 91.5%-100%,respectively.Phylogenetic analysis also showed that the 5 PCV2 Sichuan strains were close to some recent PCV2 isolates in China and belonged to genotype PCV2d. 【Conclusion】 The evolution of PCV2 was stable although there were nucleotide mutations at different degree among the 5 strains in Sichuan.
Key words:PCV2;gene clone;phylogenetic analysis
DOI:网络出版时间:2016-03-1408:4510.13207/j.cnki.jnwafu.2016.04.001
[收稿日期]2014-08-16
[基金项目]“十二五”国家科技支撑计划项目(2013BAD12B04);四川农业大学双支计划项目(00270401)
[作者简介]杨泽晓(1980-),男,河南南阳人,副教授,博士,硕士生导师,主要从事动物疫病防治与兽医公共卫生研究。
[中图分类号]S852.65+9.2
[文献标志码]A
[文章编号]1671-9387(2016)04-0001-09
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20160314.0845.002.html
E-mail:yzxyang2003@126.com

