基于线粒体COⅠ和COⅡ基因的沙果小食心虫与梨小食心虫的分子鉴定
王 康,李玉婷,郑 燕,段辛乐,张 蒙,彭 雄,陈茂华
(西北农林科技大学 植物保护学院,农业部西北黄土高原作物有害生物综合治理重点实验室,植保资源与病虫害治理教育部重点实验室,陕西 杨凌 712100)
基于线粒体COⅠ和COⅡ基因的沙果小食心虫与梨小食心虫的分子鉴定
王康,李玉婷,郑燕,段辛乐,张蒙,彭雄,陈茂华
(西北农林科技大学 植物保护学院,农业部西北黄土高原作物有害生物综合治理重点实验室,植保资源与病虫害治理教育部重点实验室,陕西 杨凌 712100)
[摘要]【目的】 建立基于线粒体COⅠ和COⅡ基因序列的沙果小食心虫、梨小食心虫分子鉴定方法。【方法】 田间采集试虫,利用PCR和基因测序技术,扩增2种食心虫的线粒体COⅠ和COⅡ基因,将获得的COⅠ和COⅡ基因序列在GenBank中进行BLAST比对,并通过GeneDoc软件分析基因序列相似性,利用Mega 5.05软件分别计算基于COⅠ和COⅡ基因序列的遗传距离,并分别构建COⅠ和COⅡ基因的系统发育树。【结果】 在分析的2种食心虫样本中,沙果小食心虫COⅠ基因的种内相似度在99.4%以上,梨小食心虫COⅠ基因的种内相似度在99.6%以上,2种食心虫COⅠ基因的种间相似度为95.0%~95.6%;沙果小食心虫COⅡ基因的种内相似度在 99.0% 以上,梨小食心虫COⅡ基因的种内相似度在99.3%以上, 2种食心虫COⅡ基因的种间相似度为94.6%~95.5%;2种食心虫COⅠ基因种间存在30个稳定变异位点,COⅡ基因种间有26个稳定变异位点。2种食心虫种内遗传距离为 0.001~0.006(COⅠ)和0.001~0.010(COⅡ),种间遗传距离为0.046~0.052(COⅠ)和0.047~0.057(COⅡ),基于COⅠ和COⅡ基因的种间遗传距离均显著大于种内遗传距离。分别构建COⅠ和COⅡ基因单倍型的系统发育树,结果显示,在2个基因的系统发育树上,2种食心虫的基因序列分别位于不同的进化支,且置信度均达到100%,同种食心虫的基因序列位于相同的进化枝。【结论】 可以根据本研究所用的COⅠ和COⅡ基因序列的差异性,进行沙果小食心虫和梨小食心虫的分子鉴定。
[关键词]沙果小食心虫;梨小食心虫;COⅠ基因;COⅡ基因;单倍型;分子鉴定
沙果小食心虫(GrapholitadimorphaKomai)和梨小食心虫(GrapholitamolestaBusck)是重要的蛀果害虫,二者均属于鳞翅目(Lepidoptera)卷蛾科(Tortricidae)。沙果小食心虫寄主植物为苹果、沙果、李等,在我国部分地区报道有发生[1-2]。梨小食心虫又名东方果蛀蛾,寄主植物主要为桃、苹果、梨、杏、李、山楂等,是世界性的主要蛀果害虫之一,在我国分布广泛[3]。沙果小食心虫和梨小食心虫经常混合发生,而且发生期部分重叠,二者幼虫和成虫外部形态都非常相似,而且性信息素主要成分亦相似[4-5],因此在田间调查和监测种群动态时,准确鉴定这2种食心虫十分困难[1,6-7]。
基于PCR技术的分子鉴定可以检测不同物种间及相同物种内不同个体间的细微差异,这为物种鉴定提供了新的手段[8-10]。线粒体DNA具有基因序列相对保守、无重组和单拷贝、进化速率适中、易于检测等优点,因此常被应用于昆虫的分子鉴定与系统进化研究之中,其中线粒体细胞色素氧化酶亚基Ⅰ(COⅠ)和线粒体细胞色素氧化酶亚基Ⅱ(COⅡ)等基因应用较为广泛[11-12]。常虹等[13]基于线粒体COⅠ基因构建了齿小蠹属(Ips)昆虫的快速鉴定方法,很好地解决了植物检疫中截获的齿小蠹幼虫或残缺成虫的鉴定难问题;游中华等[14]报道了基于线粒体COⅠ基因的9种蓟马的分子鉴定方法;通过COⅠ基因序列的分析发现,弄蝶(Astraptesfulgerator)是由多个物种组成的混合种[15];王戎疆等[16]利用线粒体COⅡ基因构建蛱蝶属(Polyura) 5种蝴蝶的系统发育树并进行聚类关系分析,结果和形态学结果相一致;杨飞龙等[17]通过比较大量中华按蚊(Anophelessinensis)标本的COⅠ、COⅡ基因及几种核糖体基因序列,建立了该虫基于COⅡ等基因的分子鉴定标准。
本研究从田间采集沙果小食心虫和梨小食心虫试虫,提取试虫的基因组DNA,PCR扩增试虫的线粒体COⅠ和COⅡ基因序列,并对扩增的序列进行测序,以此建立沙果小食心虫和梨小食心虫分子鉴定方法,为这2种害虫的准确鉴定及预测预报提供了重要依据。
1材料与方法
1.1样本采集
利用沙果小食心虫(GrapholitadimorphaKomai)和梨小食心虫(GrapholitamolestaBusck)信息素诱捕器(诱捕器为三角胶粘式,购自北京中捷四方生物科技公司),在2种害虫的发生期,于陕西省的5个苹果园,分别诱集2种食心虫成虫样本(表1)。诱芯的载体中空,由硅橡胶制成,每个质量0.3~0.5 g,每个诱芯的性信息素含量不低于0.001 g,纯度90%~97%。选择较为完整的试虫,在实验室利用体视显微镜进行准确的形态学鉴定,鉴定后的样本保存于无水乙醇中,置于-20 ℃冰箱中保存备用。
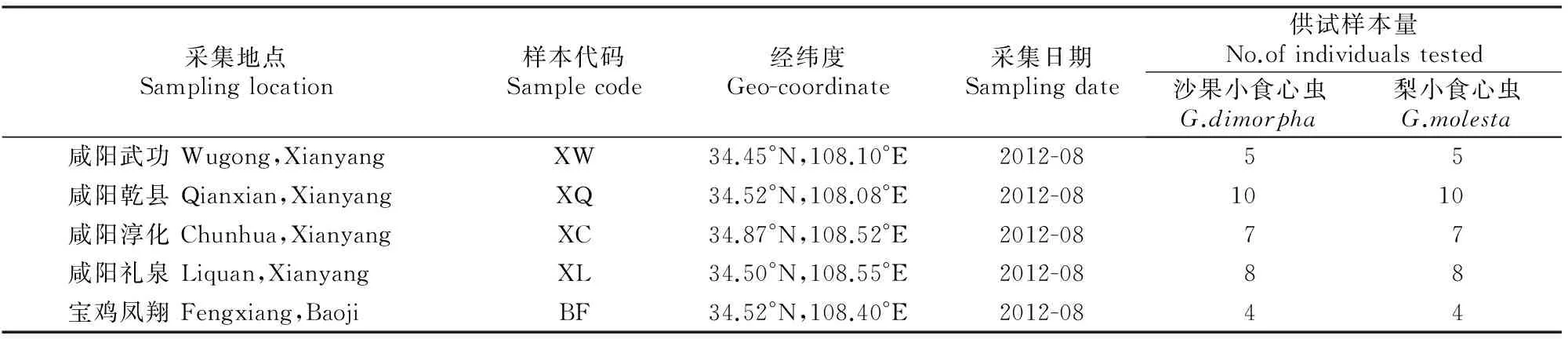
表 1 沙果小食心虫和梨小食心虫样本的采集信息
1.2基因组DNA提取
在采集的样本中,随机选取沙果小食心虫和梨小食心虫样本,利用EasyPureTMGenomic DNA Kit(北京全式金生物技术有限公司)提取单头试虫基因组DNA,基因组DNA的提取方法按照试剂盒说明书步骤进行,用1%琼脂糖凝胶电泳检测提取的DNA质量,提取的基因组DNA置于-20 ℃冰箱保存备用。
1.3PCR扩增及序列测定
参照已有的报道,选用扩增2种食心虫COⅠ和COⅡ基因的引物[18],稍作修改。扩增2种食心虫COⅠ基因的上、下游引物序列分别为:5′-CAA-CATTTATTTTGATTTTTTGG-3′,5′-TCCAATGCACTAATCTGCCATATTA-3′;扩增2种食心虫COⅡ基因的上、下游引物序列分别为:5′-ATG-GCAGATTATATGTAATGG-3′,5′-GTTTAAG-AGACCAGTACTTG-3′。
PCR扩增反应总体系为25 μL,其中含2×PCR master mix 12.5 μL,上、下游引物各3 μL,DNA模板2 μL(含20~50 ng DNA),加ddH2O至25 μL。PCR扩增程序为:94 ℃预变性2 min;94 ℃变性30 s,(CO Ⅰ 基因:52 ℃/CO Ⅱ 基因:57 ℃)退火延伸30 s,72 ℃延伸30 s,共35个循环;循环结束后72 ℃延伸5 min。利用1%琼脂糖凝胶电泳检测PCR扩增产物。
将扩增的PCR产物直接进行测序,测序由生工生物工程(上海)有限公司完成。
1.4序列分析
将所测2种食心虫COⅠ和COⅡ基因在GenBank中进行BLAST搜索,并将获得的COⅠ和COⅡ基因登录到GenBank。通过GeneDoc软件分析基因序列相似性,利用Mega 5.05软件中的Kimura2-parameter(K2P)双参数模型计算遗传距离,并利用邻接法(Neighbor-Joining,NJ)构建系统发育树[19]。
2结果与分析
2.12种试虫线粒体COⅠ和COⅡ基因序列分析
在所有沙果小食心虫和梨小食心虫试虫中都成功扩增到线粒体COⅠ和COⅡ基因序列,其中COⅠ基因序列长度为720 bp,COⅡ基因序列长度为682 bp,所有序列均无碱基插入和缺失。将所获得的序列在GenBank中进行BLAST分析,结果显示,本研究获得的沙果小食心虫COⅠ基因和COⅡ基因序列与GenBank中登录的沙果小食心虫COⅠ基因(登录号:AB603522)和COⅡ基因(登录号:JQ404438)对应序列的相似性分别为99.17%~99.31%和 99.19%~100%;测序获得的梨小食心虫COⅠ基因和COⅡ基因序列与GenBank中登录的梨小食心虫线粒体全基因组(登录号:HQ116416)上对应的COⅠ基因和COⅡ基因序列的相似性分别为 99.72%~100%和99.56%~100%。
沙果小食心虫和梨小食心虫COⅠ和COⅡ基因序列的比对结果分别见图1和图2。供试样本共获得3个沙果小食心虫COⅠ基因单倍型(GenBank登录号:KC923197~KC923199),这3个单倍型在4个碱基位点上具有多态性,3个单倍型之间的相似度在99.4%以上。供试样本共获得6个梨小食心虫COⅠ基因单倍型(GenBank登录号:KC923200~KC923205),6个单倍型在5个碱基位点上具有多态性,6个单倍型之间的相似度在99.6%以上。沙果小食心虫和梨小食心虫COⅠ基因种间的相似度在95.0%~95.6%,COⅠ基因在2种食心虫之间存在30个变异位点(图1,表2)。
供试样本共获得4个沙果小食心虫COⅡ基因单倍型(GenBank登录号:KC923206~KC923209),这4个单倍型在7个碱基位点上具有多态性,4个单倍型之间的相度在99.0%以上(表3)。

图 1沙果小食心虫和梨小食心虫COⅠ基因序列比对
黑色显示种间变异位点,下图同
Fig.1Alignment of COⅠ sequences ofGrapholitadimorphaandGrapholitamolesta
The interspecific variation loci are indicated by black color.the same below

图 2沙果小食心虫和梨小食心虫COⅡ基因序列比对
Fig.2Alignment of COⅡ sequences ofGrapholitadimorphaandGrapholitamolesta
供试样本共获得8个梨小食心虫COⅡ基因单倍型(GenBank登录号:KC923210~KC923217),这8个单倍型在8个碱基位点上具有多态性,8个单倍型之间的相似度在99.3%以上。沙果小食心虫和梨小食心虫COⅡ基因种间的相似度在94.6%~95.5%,种间存在26个变异位点(图2,表3)。
2.22种试虫遗传距离及系统发育树的构建
由表2可以看出,沙果小食心虫COⅠ基因序列间的遗传距离在0.001~0.006,梨小食心虫COⅠ基因序列间的遗传距离在0.001~0.004,2种食心虫COⅠ基因种间遗传距离为0.046~0.052,种间遗传距离显著高于种内遗传距离(df=29,t=2.05,P<0.001)。
由表3可以看出,COⅡ基因中,沙果小食心虫序列间遗传距离在0.001~0.010,梨小食心虫序列间遗传距离在0.001~0.007,2种食心虫COⅡ基因种间序列遗传距离在0.047~0.057,种间遗传距离显著高于种内遗传距离(df=58,t=2.00,P<0.001)。

表 2 沙果小食心虫和梨小食心虫COⅠ基因单倍型间的相似度和遗传距离
注:对角线上方为单倍型间相似度(%),对角线下方为单倍型间遗传距离。GD1~GD3为沙果小食心虫COⅠ基因单倍型,其GenBank登录号依次为KC923197~KC923199;GM1~GM6是梨小食心虫COⅠ基因单倍型,其GenBank登录号依次为KC923200~KC923205。
Note:The similarities between haplotypes (%) are above the diagonal,and the genetic distances between haplotypes are below the diagonal.GD1-GD3 are haplotypes ofG.dimorphaCOⅠ gene (GenBank accession No:KC923197-KC923199),and GM1-GM6 are the haplotypes ofG.molestaCOⅠ gene (GenBank accession No:KC923200-KC923205).

表 3 沙果小食心虫和梨小食心虫COⅡ基因单倍型间的相似度和遗传距离
注:对角线上方为单倍型间相似度(%),对角线下方为单倍型间遗传距离。GD1~GD4为沙果小食心虫COⅡ基因单倍型,其GenBank登录号依次为KC923206~KC923209;GM1~GM8是梨小食心虫COⅡ基因单倍型,GenBank登录号依次为KC923210~KC923217。
Note:The similarities between haplotypes (%) are above the diagonal,and the genetic distances between haplotypes are below the diagonal.GD1-GD4 are haplotypes ofG.dimorphaCOⅡ gene (GenBank accession No:KC923206-KC923209),and GM1-GM8 are the haplotypes ofG.molestaCOⅡ gene (GenBank accession No:KC923210-KC923217).
以苹果蠹蛾(Cydiapomonella)COⅠ基因序列(GenBank登录号:JQ928797)作外类群,将本研究获得的沙果小食心虫和梨小食心虫COⅠ基因单倍型序列和1个已登录的沙果小食心虫COⅠ基因序列(GenBank登录号:AB603522),以及3个已登录的梨小食心虫COⅠ基因序列(GenBank登录号:AB603521、FJ449660、HQ700338)构建系统发育树,结果(图3)显示,沙果小食心虫和梨小食心虫的COⅠ基因单倍型各自聚在一支。
以苹果蠹蛾(C.pomonella)线粒体全基因(GenBank登录号:JX407107)中COⅡ基因序列作外类群,将本研究获得的2种食心虫COⅡ基因单倍型序列和已登录的相应沙果小食心虫COⅡ基因序列(GenBank登录号:JQ404438)和梨小食心虫COⅡ基因序列(GenBank登录号:EU671060)构建系统发育树,结果(图4)表明,所有的沙果小食心虫COⅡ基因单倍型聚为一支,所有的梨小食心虫单倍型聚为另外一支。
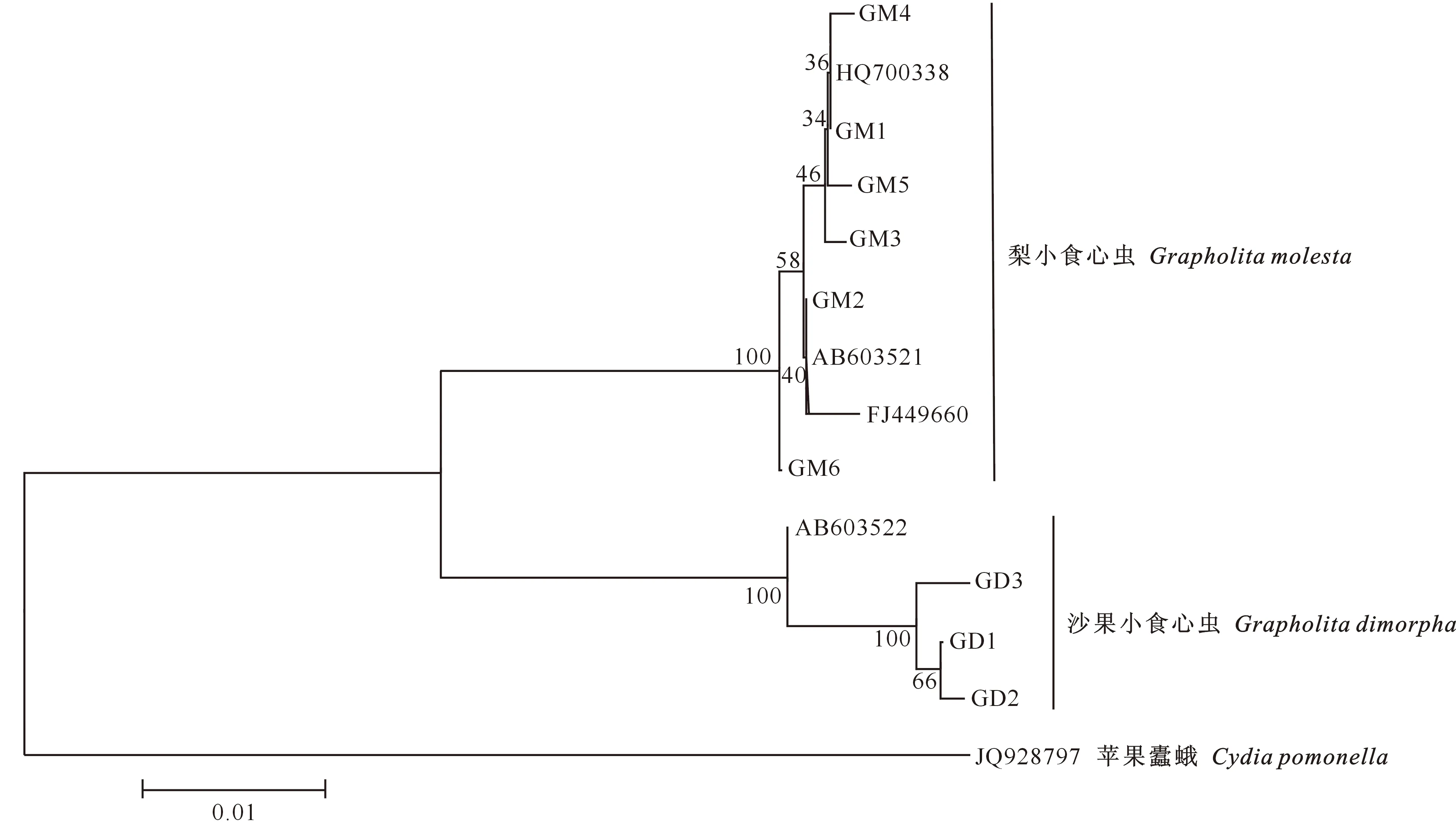
图 3 基于沙果小食心虫和梨小食心虫COⅠ基因单倍型的系统发育树GD1~GD3和GM1~GM6为本研究获得的沙果小食心虫和梨小食心虫COⅠ基因单倍型;AB603522为GenBank已登录的相应沙果小食心虫COⅠ基因序列;AB603521、FJ449660、HQ700338为GenBank已登录的相应梨小食心虫COⅠ序列;JQ928797为GenBank已登录的相应苹果蠹蛾COⅠ基因序列
3讨论
线粒体COⅠ和COⅡ基因序列目前已广泛地应用到昆虫近缘种的鉴定之中[20]。在利用线粒体基因进行分子鉴定时,不同物种间相同的线粒体基因核苷酸序列差异是最重要的标准[21-22]。Hebert等[23]通过分析比较200种鳞翅目昆虫COⅠ基因序列后发现,COⅠ基因核苷酸序列3%的差异可以作为鉴定不同物种的标准,即不同物种COⅠ基因差异大于3%,同一物种COⅠ基因序列差异小于3%,目前大量的相关研究证实了这一标准的准确性[24-25]。本研究中,2种食心虫COⅠ和COⅡ基因种内核苷酸差异小于1.0%,而种间核苷酸差异大于4.4%,因此本研究的COⅠ和COⅡ基因序列可以准确地应用于沙果小食心虫和梨小食心虫的分子鉴定中。
Aliabadian等[26]研究指出,当种间遗传距离和种内遗传距离不存在重叠区域,且分离程度较高时,目标片段能准确鉴定物种。本研究结果显示,基于COⅠ和COⅡ基因的沙果小食心虫和梨小食心虫的种内遗传距离均小于0.010,而种间遗传距离大于0.046,基于COⅠ和COⅡ基因的种内遗传距离和种间遗传距离不存在重叠区域,并且种内遗传距离与种间遗传距离差异显著(P<0.001)。另外,有分析认为,种间平均遗传距离一般为种内平均遗传距离的10倍以上[27],本研究基于COⅠ基因的种间平均遗传距离(0.050 0)为种内平均遗传距离(0.002 7)的18.2倍,基于COⅡ基因的种间平均遗传距离(0.050 6)为种内平均遗传距离(0.003 8)的13.2倍。因此,基于COⅠ和COⅡ基因的沙果小食心虫和的梨小食心虫种间和种内遗传距离分析结果表明,COⅠ和COⅡ基因可以用于这2种食心虫的分子鉴定。另外,稳定的种间差异位点是确定种的重要手段。朱振华等[28]通过比较6种果实蝇mtCytb基因序列,发现种间存在30个比较稳定的变异位点,并以此作为鉴定不同种的标准。本试验发现,2种食心虫在COⅠ基因序列上有30个稳定变异位点,COⅡ基因序列上有26个稳定差异位点,这些位点的碱基及其变化可以作为梨小食心虫和沙果小食心虫分子鉴定时的重要参考。
4结论
本研究结果表明,沙果小食心虫、梨小食心虫的COⅠ和COⅡ基因种内的相似度在99.0%以上,种间相似度在95.6%以下,且2个基因的种间遗传距离显著高于种内遗传距离,2种食心虫的COⅠ基因单倍型和COⅡ基因单倍型分别在系统发育树上明显各自聚为一支,因此,本研究采用的沙果小食心虫、梨小食心虫的COⅠ和COⅡ基因可以用于这2种害虫的分子鉴定之中。
[参考文献]
[1]严善春,刘友樵,李明文.我国果树一新害虫:沙果小食心虫 [J].森林病害通讯,1999(4):15-16.
Yan S C,Liu Y Q,Li M W.Grapholita dimorpha:A new record pest damaging fruit trees of China [J].Forest Pest and Disease,1999(4):15-16.(in Chinese)
[2]Jung C R,Kim Y.Different types of fruit damages of three internal apple feeders diagnosed with mitochondrial molecular markers [J].Journal of Asia-Pacific Entomology,2013,16(2):189-197.
[3]陈梅香,骆有庆,赵春江,等.梨小食心虫研究进展 [J].北方园艺,2009(8):144-147.
Chen M X,Luo Y Q,Zhao C J,et al.Research advance onGrapholitamolestaBusck [J].Northern Horticulture,2009(8):144-147.(in Chinese)
[4]Choi K H,Lee D H,Byun B K,et al.Occurrence ofGrapholitadimorphaKomai (Lepidoptera:Tortricidae),a new insect pest in apple orchards of Korea [J].Korean Journal of Applied Entomology,2009,48(4):417-421.
[5]Jung C R,Jung J K,Kim Y.Effects of different sex pheromone compositions and host plants on the mating behavior of twoGrapholitaspecies[J].Journal of Asia-Pacific Entomology,2013,16(4):507-512.
[6]Komai F.A new species of the genusGrapholitaTreitschke from Japan allied to the oriental fruit moth,Grapholitamolesta(Busck) (Lepidoptera:Tortricidae) [J].Applied Entomology and Zoology,1979,14(2):133-136.
[7]Hada H,Sekine K T.A diagnostic multiplex polymerase chain reaction method to identify Japanese internal apple-feeding Lepidopteran pests:Grapholitamolesta,Grapholitadimorpha(Lepidoptera:Tortricidae),andCarposinasasakii(Lepidoptera:Carposinidae) [J].Applied Entomology and Zoology,2011,46(2):287-291.
[8]Tautz D,Arctander P,Minelli A,et al.DNA points the way ahead in taxonomy [J].Nature,2002,418(6897):479-479.
[9]肖金花,肖晖,黄大卫.生物分类学的新动向:DNA条形编码 [J].动物学报,2004,50(5):852-855.
Xiao J H,Xiao H,Huang D W.DNA barcoding:New approach of biological taxonomy [J].Acta Zoologica Sinica,2004,50(5):852-855.(in Chinese)
[10]莫帮辉,屈莉,韩松,等.DNA条形码识别:Ⅰ.DNA条形码研究进展及应用前景 [J].四川动物,2008,27(2):303-306.
Mo B H,Qu L,Han S,et al.DNA barcoding identification:Ⅰ.Research progress and applied perspective of DNA barcoding [J].Sichuan Journal of Zoology,2008,27(2):303-306.(in Chinese)
[11]Bibb M J,Van Ette R A,Wright C T,et al.Sequence and gene organization of mouse mitochondrial DNA [J].Cell,1981,26:167-180.
[12]牛屹东,李明,魏辅文,等.线粒体DNA用作分子标记的可靠性和研究前景 [J].遗传,2001,23(6):593-598.
Niu Y D,Li M,Wei F W,et al.Reliability of mtDNA as molecular marker and its perspective [J].Hereditas,2001,23(6):593-598.(in Chinese)
[13]常虹,郝德君,肖荣堂,等.基于线粒体COⅠ基因的齿小蠹属昆虫DNA条形码研究 [J].昆虫学报,2012,55(9):1075-1081.
Chang H,Hao D J,Xiao R T,et al.DNA barcoding based on the mitochondrial COⅠ gene sequences forIpsspecies ( Coleoptera: Scolytidae) [J].Acta Entomologica Sinica,2012,55(9):1075-1081.(in Chinese)
[14]游中华,路虹,张宪省,等.入侵害虫西花蓟马及其他8种常见蓟马的分子鉴定 [J].昆虫学报,2007,50(7):720-726.
You Z H,Lu H,Zhang X S,et al.Molecular identification of the introduced western flower thrips,Frankliniellaoccidentals(Pergande) and other eight common thrips species (Thysanoptera: Thripidae) [J].Acta Entomologica Sinica,2007,50(7):720-726.(in Chinese)
[15]Hebert P D N,Penton E H,Burns J M,et al.Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterflyAstraptesfulgerator[J].Proceedings of the National Academy of Sciences of the United States of America,2004a,101(41):14812-14817.
[16]王戎疆,万宏,雷光春,等.利用线粒体COⅡ基因序列对中国尾蛱蝶属系统分化的研究(鳞翅目:蛱蝶科) [J].昆虫学报,2004,47(2):243-247.
Wang R J,Wan H,Lei G C,et al.Phylogenetic analysis ofPolyurain China inferred from mitochondrial COⅡ sequences (Lepidoptera:Nymphalidae) [J].Acta Entomologica Sinica,2004,47(2):243-247.(in Chinese)
[17]杨飞龙,李旭东,闫振天,等.中华按蚊的分子鉴定标准 [J].重庆师范大学学报:自然科学版,2014,31(4):40-44.
Yang F L,Li X D,Yan Z T,et al.The molecular identification markers ofAnophelessinensis[J].Journal of Chongqing Normal University:Natural Science Edition,2014,31(4):40-44.(in Chinese)
[18]Simon C,Fraiti F,Beckenbach A,et al.Evolution,weighting,and phylogenetic utility of mitochondrial gene sequences and a compilation conserved polymerase chain reaction primer [J].Annals of the Entomological Society of America,1994,87:651-701.
[19]Kumar S,Nei M,Dudley J,et al.Mega:A biologist-centtric software for evolutionary analysis of DNA and protein sequence [J].Briefings in Bioinformatics,2008,9(4):299-306.
[20]李青青,李地艳,段焰青,等.DNA条形码在鳞翅目昆虫中的应用 [J].生命科学,2010(4):307-312.
Li Q Q,Li D Y,Duan Y Q,et al.Application of DNA barcoding in lepidopteran insects [J].Chinese Bulletin of Life Sciences,2010(4):307-312.(in Chinese)
[21]Roe A D,Sperling F A H.Patterns of evolution of mitochondrial cytochromecoxidase Ⅰ and Ⅱ DNA and implications for DNA barcoding [J].Molecular Phylogenetics and Evolution,2007,44(1):325-345.
[22]Tautz D,Arctander P,Minelli A,et al.A plea for DNA taxonomy [J].Trends in Ecology & Evolution,2003,18(2):70-74.
[23]Hebert P D N,Cywinska A,Ball S L.Biological identifications through DNA barcodes [J].Proceedings of the Royal Society of London B:Biological Sciences,2003,270(1512):313-321.
[24]Ball S L,Hebert P D N,Burian S K,et al.Biological identifications of mayflies (Ephemeroptera) using DNA barcodes [J].Journal of the North American Benthological Society,2005,24(3):508-524.
[25]Hajibabaei M,Janzen D H,Burns J M,et al.DNA barcodes distinguish species of tropical Lepidoptera [J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(4):968-971.
[26]Aliabadian M,Kaboli M,Nijman V,et al.Molecular identification of birds:Performance of distance-based DNA barcoding in three genes to delimit parapatric species [J].PLoS One,2009,4(1):e4119.
[27]Hebert P D N,Stoeckle M Y,Zemlak T S,et al.Identification of birds through DNA barcodes [J].PLoS Biology,2004b,2(10):e312.
[28]朱振华,叶辉,张智英.基于mtDNA Cytb的六种果实蝇的分子鉴定 [J].昆虫学报,2005,48(3):386-390.
Zhu Z H,Ye H,Zhang Z Y.Molecular identification of sixBactroceraspecies (Diptera:Tephritidae) based on mtDNA [J].Acta Entomologica Sinica,2005,48(3):386-390.(in Chinese)
Molecular identification ofGrapholitadimorphaKomai (Lepidoptera:Tortricidae) andGrapholitamolestaBusck (Lepidoptera:Tortricidae) based on mitochondrial COⅠ and COⅡ genes
WANG Kang,LI Yu-ting,ZHENG Yan,DUAN Xin-le,ZHANG Meng,PENG Xiong,CHEN Mao-hua
(CollegeofPlantProtection,KeyLaboratoryofCropPestIntegratedPestManagementontheLoessPlateauofMinistryofAgriculture,KeyLaboratoryofPlantProtectionResourcesandPestManagementofMinistryofEducation,NorthwestA&FUniversity,Yangling,Shaanxi712100,China)
Abstract:【Objective】 This study aimed to establish a method for identifing Grapholita dimorpha Komai and Grapholita molesta Busck based on mitochondrial COⅠ and COⅡ genes.【Method】 PCR and gene sequencing methods were used to obtain COⅠ and COⅡ sequences from the samples of the two species collected from apple orchards.The acquired COⅠ and COⅡ sequences were verified by BLAST program in GenBank.GeneDoc was also used to analyze the similarity between the obtained sequences.Mega 5.05 was used to analyze the genetic distances between sequences and to construct the phylogenetic trees of COⅠ and COⅡ gene haplotypes,respectively.【Result】 The intraspecific similarities of COⅠ sequences of G.dimorpha and G.molesta were above 99.4% and 99.6%,respectively,whilst the interspecific similarity of COⅠ gene sequences between them was 95.0%-95.6%.The intraspecific similarities of COⅡ sequences of G.dimorpha and G.molesta were above 99.0% and 99.3%,while its interspecific similarity between them was 94.6%-95.5%.A total of 30 variable sites were detected in COⅠ gene sequences from samples of the two species,and 26 were detected in COⅡ sequences.The intraspecific genetic distances were 0.001-0.006 (COⅠ) and 0.001-0.010 (COⅡ),whereas the interspecific genetic distances were 0.046-0.052 (COⅠ) and 0.047-0.057 (COⅡ).The interspecific genetic distance was significantly higher than intraspecific genetic distance.Phylogenetic trees based on COⅠ gene haplotypes and COⅡ gene haplotypes indicated that gene sequences from the same species were clustered in the same branch,whereas gene sequences from different species were clustered in different branches, with the bootstrap value of 100%.【Conclusion】 The COⅠ and COⅡ gene sequences used in this study can be applied in the molecular identification of G.dimorpha and G.molesta.
Key words:Grapholita dimorpha;Grapholita molesta;COⅠ gene;COⅡ gene;haplotypes;molecular identification
DOI:网络出版时间:2016-01-0810:2210.13207/j.cnki.jnwafu.2016.02.022
[收稿日期]2014-06-30
[基金项目]国家自然科学基金项目(31071687);教育部高等学校博士学科点专项科研基金(博导类)项目(20110204110001);西北农林科技大学人才引进项目;国家科技支撑计划项目(2012BAK11B03)
[作者简介]王康(1989-),男,陕西咸阳人,在读硕士,主要从事农业昆虫和害虫防治研究。E-mail:kangwang@nwsuaf.edu.cn[通信作者]陈茂华(1971-),男,湖北武汉人,教授,博士生导师,主要从事昆虫种群遗传学和分子毒理学研究。
[中图分类号]S763.42;Q78
[文献标志码]A
[文章编号]1671-9387(2016)02-0156-09
E-mail:maohua.chen@nwsuaf.edu.cn

