基于网络爬虫和文本挖掘的实体关系研究与实现
谢文彬
(同济大学电子与信息工程学院,上海 201804)
基于网络爬虫和文本挖掘的实体关系研究与实现
谢文彬
(同济大学电子与信息工程学院,上海201804)
摘要:
关键词:
0 引言
随着科技不断进步,越来越多的生物资源在网上发布,PubMed Central(PMC)[1]是一个免费的生物和生命科学文本全文数据库。但是作为国外数据,数据下载速度慢,能够下载的资料并不是完整的PMC数据,而且,仅仅只是下载原文,并不能给许多生物学者提供很多有价值的信息。现在PMC数据库中总计大约拥有380万篇全文数据,但是能够通过FTP下载的文章只占到一半不到。其次,PMC自带的搜索引擎并不能提供很好的实体关系搜索,例如要研究疾病和基因之间的关系。所以我们急需一种快捷有效的方法把生物学者所需的研究数据从网页上下载下来,经过预处理,成为本地可以批量处理的数据,再通过本地的服务器对数据进行快速的搜索抽取工作。
1 系统体系结构
文本挖掘的主要用途是从原本未经处理的文本中提取出未知的知识,但是文本挖掘也是一项非常困难的工作,因为它必须处理那些本来就模糊而且非结构化的文本数据,所以它是一个多学科混杂的领域,涵盖了信息技术、文本分析等技术,而且在网络时代,原始数据的获取主要通过网络途径,所以在实际挖掘过程中,系统分为4个大模块:
1.1收集与实体相关的论文
在PMC中,有官方网页提供的搜索引擎(http:// www.ncbi.nlm.nih.gov/pmc/),通过该搜索引擎,可以先做粗筛选。例如研究基因和疾病的关系,可以在搜索栏中输入“Gene Disease”,结果搜索到大约74万的论文,这样可以去除数据库中大部分与研究对象无关的论文,然后使用网站提供的下载功能抽出这些论文的编号,作为URL的部分地址。
1.2网络爬虫
将上一步收集的网页编号,根据PMC论文自带的论文URL地址,http://www.ncbi.nlm.nih.gov/pmc/articles/ PMC3578923/,其中“3578923”替换成任意待抓取的文章。批量抓取所需要研究的论文。
1.3数据处理与清洗
将从网页上爬取的XML格式的文本数据进行去标记语言,识别论文所具有的编号、题目、摘要、论文主题、作者、引用等信息,存入本地用于文本挖掘。
1.4文本挖掘
在文本挖掘中,预先准备好两种实体的词库,例如基因词库,包括基因的编号,正式名字和同义名的信息。然后将论文切分成句子,搜索两类实体是否同时出现在一个句子里,若有则抽取出来,并认为两个实体间很可能有关系,最后在进行所需研究。
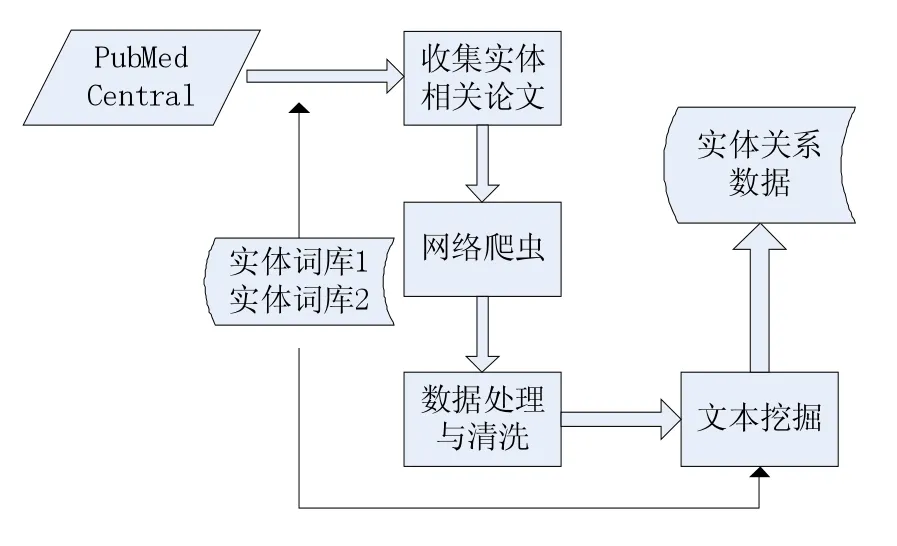
图1 系统基本结构和工作流程
2 关键技术和部分代码
2.1网络爬虫
网络爬虫是捜索引擎抓取系统的重要组成部分。爬虫的主要目的是将互联网上的网页下载到本地形成一个内容的镜像备份。该网络爬虫模块以python为基础语言,调用thread、urllib2和socket三个程序包,编写网络爬虫。
其中thread包的多线程爬取加速爬取速度,并且加入异常处理模块,以下为部分代码:

2.2去标记语言
使用正则表达式,去除无用标记语言,并识别XML网页文件中所需信息,以下为部分代码:

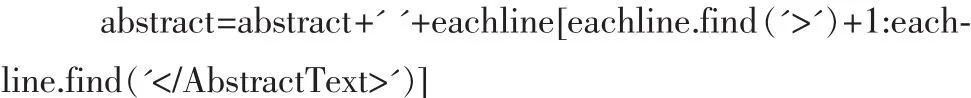
2.3倒排索引
在文本挖掘中,词库以字典作为存储形式,每个疾病对应唯一编号和它的若干个疾病同义名。在查找句子中单词所对应的疾病时,为了加速搜索,使用倒排索引记录疾病词库信息。如表1,字典编号使用疾病的名字,对应的映射是疾病的编号。在检索中,将字典按照名字排序,在使用二分查找对应疾病名字是否存在字典中,并找出对应编号。

表1 疾病倒排索引存储形式
2.4停用词表
停用词表包含了文章中的常用词。例如表示数量的词语,语气词等。这些词不仅可能和研究实体重名,导致严重的检索错误,更会加重我们的检索负担。在实际检索中,系统会先确定单词是否属于停用词表,若属于则不检索该词。
3 结果展示
疾病词库从DiseaseOntology[2]中下载整理,总共8944个不同疾病;基因词库从NCBI(http://www.ncbi. nlm.nih.gov/gene/)中下载整理,总共29521个不同的基因,从PMC中抓取总共74万多篇文献。使用文本挖掘系统检索,结果共有140813条句子同时包含基因和疾病名字。
4 结语
随着网络资源的不断膨胀,社会越来越需要各种自动化的技术来去除有用的信息,收集有价值的信息,并提取信息中的规律造福人类,例如某些基因导致某种疾病的产生。本文基于网络爬虫和文本挖掘的常用技术,为生物科学家对于实体间关系的研究,例如基因和疾病,疾病和药物,药物和蛋白质等各种关系,提供了便利的文本抽取方法。
参考文献:
[1]Coordinators,N. R. "Database resources of the National Center for Biotechnology Information."[J]Nucleic Acids Res,2016 44(D1): D7-D19.
[2]Schriml,LM;Arze,C;Nadendla,S;Chang,YW;Mazaitis,M;Felix,V;Feng,G;Kibbe,WA . Disease Ontology: a Backbone for Disease Semantic Integration.[J]. Nucleic Acids Research 40(Database issue)2012: D940-6.
Research on Entity Relationship Based on Web Crawler and Text Mining
XIE Wen-bin
(School of Electronics and Information Engineering,Tongji University,Shanghai 201803)
Abstract:
Keywords:
随着网络资源的不断膨胀,有关生物文献资源越来越多,生物学家急需各种自动化的技术从海量文献中抽取有价值的信息。基于网络爬虫和文本挖掘的技术,设计研发一个用于挖掘网络上电子版论文中实体关系的系统,并且使用该系统,成功挖掘有关疾病和基因的关系。
网络爬虫;实体;文本挖掘;疾病;基因
文章编号:1007-1423(2016)13-0019-03
DOI:10.3969/j.issn.1007-1423.2016.13.005
作者简介:
谢文彬(1990-),男,江苏苏州人,硕士研究生,研究方向为文本挖掘与关系抽取
收稿日期:2016-03-15修稿日期:2016-04-16
With the continuous increase of web resource,more and more document resource emerges,biologists are urgent to get valuable information from huge document by using a variety of automatics technique. Based on the development of web crawler and text mining,designs a novel system to excavate the entity relationship among electronic papers on the internet and apply successfully such system to catch the relation between disease and gene.
Web Crawler;Entity;Text Mining;Disease;Gene

