乙型肝炎病毒X蛋白相关微小RNA差异表达在乙型肝炎病毒复制中的作用研究
陈 静,陈 磊,吴孟超,石晓祯
·论著·
乙型肝炎病毒X蛋白相关微小RNA差异表达在乙型肝炎病毒复制中的作用研究
陈 静,陈 磊,吴孟超,石晓祯
200437上海市缔达生物科技有限公司(陈静,石晓祯);第二军医大学附属东方肝胆外科医院(陈磊,吴孟超);国际合作信号转导实验室(陈磊)
【摘要】背景乙型肝炎病毒X蛋白(HBX)在乙型肝炎病毒复制及肝癌形成中扮演着重要的角色。研究HBX相关微小RNA(miRNAs)的差异表达有可能为调控乙型肝炎病毒复制、阐明肝癌发展进程及开展预后干预提供重要信息。目的探讨肝癌细胞中HBX相关miRNAs的差异表达及其在乙型肝炎病毒复制中的作用。方法选取2014年1—10月第二军医大学附属东方肝胆外科医院收治的13例肝癌患者的肝癌组织及距离肝癌组织>2 cm癌旁组织的新鲜石蜡包埋标本,采用微阵列技术分析HBX相关miRNAs的差异表达,实时荧光定量反转录聚合酶链式反应(RT-qPCR)法定量分析肝癌组织与癌旁组织中7个与肝脏功能相关的miRNAs及HBX mRNA表达水平,同时分析过表达hsa-miR-373或抑制hsa-miR-22对乙型肝炎病毒复制的影响。结果微阵列技术分析显示,HepG2-X细胞系和HepG2-CAT细胞系中有46种miRNAs差异表达,与HepG2-CAT细胞系相比,HepG2-X细胞系中20种miRNAs表达上调,26种miRNAs表达下调。肝癌组织与癌旁组织中hsa-miR-22、hsa-miR-373、hsa-miR-92、hsa-miR-155及HBX mRNA表达水平比较,差异均有统计学意义(P<0.05);肝癌组织与癌旁组织中hsa-miR-181a、hsa-miR-34a、hsa-let-7i表达水平比较,差异均无统计学意义(P>0.05)。Pearson相关性分析结果显示,hsa-miR-22表达水平与HBX mRNA表达水平呈正相关(r=0.415,P=0.018),hsa-miR-373、hsa-miR-92、hsa-miR-155表达水平与HBX mRNA表达水平呈负相关(r=-0.322,P=0.043;r=-0.369,P=0.028;r=-0.426,P=0.016);hsa-miR-181a、hsa-miR-34a、hsa-let-7i表达水平与HBX mRNA表达水平无直线相关关系(r=0.076,P=0.363;r=0.017,P=0.676;r=0.122,P=0.242)。第2天和第3天hsa-miR-22抑制剂组、hsa-miR-373前体组HepG2.2.15细胞系及HepAD-38细胞系乙型肝炎病毒拷贝数较阴性对照组降低(P<0.05);第2天和第3天hsa-miR-373前体组HepG2.2.15细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组降低(P<0.05)。第2天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组升高,第3天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组降低(P<0.05)。第2天和第3天hsa-miR-373前体组HepG 2.2.15细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组升高(P<0.05)。第2天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组降低,第3天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组升高(P<0.05)。结论HBX相关miRNAs的差异表达可通过靶向相关信号转导通路影响乙型肝炎病毒在肝癌细胞内的复制。
【关键词】肝肿瘤;乙型肝炎病毒X蛋白;微小RNA;病毒复制
陈静,陈磊,吴孟超,等.乙型肝炎病毒X蛋白相关微小RNA差异表达在乙型肝炎病毒复制中的作用研究[J].中国全科医学,2016,19(15):1786-1791.[www.chinagp.net]
Chen J,Chen L,Wu MC,et al.Effect of differential expression of HBX-regulated miRNAs on HBV replication[J].Chinese General Practice,2016,19(15):1786-1791.
微小RNA(microRNA,miRNAs)是一类长度为19~25个核苷酸分子的小RNA,在动、植物体内介导转录后基因沉默,在细胞生长、增殖、分化、凋亡过程中扮演重要的角色[1-2]。研究miRNAs差异表达、miRNAs的调控网络及其对细胞功能的影响,对病毒感染以及肿瘤发生发展具有重要意义[3]。肝癌是一个全球健康问题,肝癌的发生发展与乙型肝炎病毒和丙型肝炎病毒感染密切相关[4]。乙型肝炎病毒X蛋白(HBX)是乙型肝炎病毒基因组HBX基因编码的蛋白,是一种反式激活因子,参与乙型肝炎病毒复制及肝癌进展[5]。因此,鉴别HBX相关miRNAs的差异表达及其潜藏的生物学功能,对研究肝癌形成、针对关键miRNAs开展干预治疗以及调控乙型肝炎病毒复制具有重要的临床意义[6]。本研究利用微阵列技术分析肝癌细胞系,采用实时荧光定量反转录聚合酶链式反应(RT-qPCR)法分析肝癌细胞中HBX相关miRNAs的差异表达,并研究了过表达hsa-miR-373或抑制hsa-miR-22对乙型肝炎病毒复制的影响。
1材料与方法
1.1标本选取2014年1—10月第二军医大学附属东方肝胆外科医院收治的慢性乙型肝炎致肝癌患者13例为研究对象,其中男8例,女5例;年龄35~56岁,平均年龄(43.2±7.2)岁;慢性乙型肝炎病史10年以上,无其他脏器合并症。收集肝癌患者肝癌组织及距离肝癌组织>2 cm癌旁组织的新鲜石蜡包埋标本。
1.2细胞系HepG2-X细胞系(为稳定表达HBX的细胞系)、HepG2-CAT细胞系〔为稳定表达报告基因——细菌氯霉素乙酰转移酶(CAT)的细胞系〕及HepAD-38细胞系[7]由美国托马斯杰斐逊大学Mark A Feitelson教授惠赠,稳定细胞系构建根据文献[5]。HepG2.2.15细胞系购自中国科学院上海生命科学研究院细胞资源中心,并按已报道流程培养[8]。
1.3试剂miRVana miRNA Isolation Kit、miRNA Negative control #1、hsa-miR-373 前体和anti-miR-22购自Ambion(美国);Qiagen Blood Kit购自Qiagen(德国);hsa-miR-22、hsa-miR-181a、hsa-miR-34a、hsa-miR-373、hsa-miR-92、hsa-miR-155、hsa-let-7i和U6的RT-qPCR和 PCR引物由生工生物工程(上海)股份有限公司合成。
1.4HBX免疫组织化学染色切割新鲜石蜡包埋标本并根据文献[9]提供的实验流程用兔抗人anti-HBX抗体x-99多克隆抗体(由国际合作信号转导实验室制备)进行HBX染色。所有肝癌组织及癌旁组织先用10%甲醛溶液固定,然后脱水、石蜡包埋、连续切片(5 μm厚),切片脱蜡、水合,并在枸橼酸盐缓冲液中煮沸修复抗原。1%牛血清清蛋白(BSA)室温封闭30 min,一抗4 ℃孵育过夜后滴加二抗DAKO,37 ℃孵育45 min。二氨基联苯胺(DAB)显色(DAB显色试剂盒购自上海碧云天生物技术有限公司),苏木素复染。在显微镜下观察肝癌组织及癌旁组织切片中HBX的染色强度。所有染色步骤按试剂盒说明书进行。
1.5miRNAs及总RNA分离按miRVana miRNA Isolation Kit的流程抽提HepG2-X细胞系、HepG2-CAT细胞系及13对新鲜肝癌组织/癌旁组织标本中的miRNAs。将细胞悬液或组织匀浆用试剂盒中的Lysis/Binding Buffer裂解,剧烈震荡以破解细胞释放miRNAs;加入1/10体积的miRNA Homogenate Additive蜗旋震荡后冰浴,加入和Lysis/Binding Buffer等体积的苯酚和三氯甲烷溶液,混匀后10 000×g离心5 min,取上层水相与1/3体积的100%乙醇溶液充分混合后,加到结合柱中,10 000×g离心15 s,收集滤液,再添加2/3滤液体积的室温100%乙醇溶液混匀,加入一个新的结合柱中,10 000×g离心15 s后弃去滤液,用miRNA Wash Solution 1、2、3各洗涤1次,最后用95 ℃预热的无RNA酶水或者Elution Solution洗脱。按总RNA抽提液TRIZOL Reagent(Invitrogen,美国)的流程抽提13对新鲜肝癌组织/癌旁组织标本中的总mRNA。将组织匀浆用TRIZOL Reagent裂解,加入三氯甲烷溶液混匀,10 000×g离心5 min后吸取上层含有总RNA的水相置入新EP管,加入异丙醇沉淀总RNA,20 000×g离心15 min用75%乙醇洗涤总RNA沉淀,晾干后用焦碳酸二乙酯(DEPC)水溶解。提纯后的miRNAs及总RNA测定260 nm/280 nm处的吸光度,-80 ℃保存。
本研究要点:
(1)本研究采用微阵列技术分析肝癌细胞中乙型肝炎病毒X蛋白(HBX)相关微小RNA(miRNAs)的差异表达,并初步分析了其中部分miRNAs在乙型肝炎病毒复制中的作用。
(2)本研究发现,肝癌细胞中有46种miRNAs受HBX 影响,其中有20种miRNAs表达上调,26种miRNAs表达下调。
(3)7种可能参与乙型肝炎病毒复制或肝癌发生的miRNAs中,hsa-miR-22、hsa-miR-373、hsa-miR-92、hsa-miR-155表达水平在肝癌组织与癌旁组织中有差异,同时hsa-miR-22表达水平与HBX mRNA表达水平呈正相关,hsa-miR-373、hsa-miR-92、hsa-miR-155表达水平与HBX mRNA表达水平呈负相关。
(4)过表达hsa-miR-373或抑制hsa-miR-22可显著抑制HepG2.2.15细胞系和HepAD-38细胞系中乙型肝炎病毒的复制。
1.6微阵列技术分析HBX相关miRNAs的差异表达HepG2-X细胞系和HepG2-CAT细胞系中抽提得到RNA样品各50 μl,进行配对miRNAs微阵列技术分析(LC Sciences,美国),计算HepG2-X/HepG2-CAT,确定差异表达倍数进行排序。
1.7RT-qPCR法定量分析miRNAs及HBX mRNA表达水平采用RT-qPCR法分析肝癌组织/癌旁组织中miRNAs及HBX mRNA表达水平。基于文献[10],选取7个与肝脏功能相关的miRNAs,包括3个在HepG2-X细胞系中表达上调的miRNAs(hsa-miR-22、hsa-miR-181a、hsa-miR-34a)和4个在HepG2-X细胞系中表达下调的miRNAs(hsa-miR-373、hsa-miR-92、hsa-miR-155、hsa-let-7i)。检测miRNAs时每个RT-qPCR体系中将25 ng miRNAs、miRNA反转录引物、dNTPs、5×RT buffer、RNasin、MMLV酶、DEPC水混合,37 ℃反转录60 min后,85 ℃热灭活5 min;随后加入miRNAs qPCR引物及FastStart Universal SYBR Green Master mix(Rox)(ROCHE,德国),采用ABI 7500 Real-Time定量PCR仪(ABI,美国)进行检测,PCR程序为95 ℃ 预变性3 min,95 ℃变性15 s,60 ℃退火30 s,重复40个循环;以U6作为内参。检测HBX mRNA表达水平时每个RT-qPCR体系中将100 ng mRNA、反转录6nt随机引物、dNTPs、5×RT buffer、RNasin、MMLV酶、DEPC水混合,37 ℃反转录60 min后,85 ℃热灭活5 min;随后加入HBX mRNA qPCR引物及FastStart Universal SYBR Green Master mix(Rox),采用ABI 7500 Real-Time定量PCR仪进行检测,PCR程序为95 ℃预变性3 min,95 ℃变性15 s,60 ℃退火30 s,重复40个循环;以18s rRNA作为内参。miRNAs表达水平用miRNAs与内参U6 Ct值的差值ΔCtmiRNAs〔ΔCtmiRNAs=-(CtmiRNAs-CtU6)〕表示;HBX mRNA表达水平用HBX mRNA与内参18s rRNA Ct值的差值ΔCtHBX mRNA〔ΔCtHBX mRNA=-(CtHBX mRNA-Ct18s rRNA)〕表示。

2结果
2.1HBX调控的miRNAs差异表达微阵列技术分析显示,HepG2-X细胞系和HepG2-CAT细胞系中有46种miRNAs差异表达,与HepG2-CAT细胞系相比,HepG2-X细胞系中20种miRNAs表达上调,26种miRNAs表达下调(见表1、图1)。
2.2RT-qPCR法定量分析miRNAs及HBXmRNA表达水平肝癌组织与癌旁组织中hsa-miR-22、hsa-miR-373、hsa-miR-92、hsa-miR-155及HBXmRNA表达水平比较,差异均有统计学意义(P<0.05);肝癌组织与癌旁组织中hsa-miR-181a、hsa-miR-34a、hsa-let-7i表达水平比较,差异均无统计学意义(P>0.05,见表2)。
2.3相关性分析Pearson相关性分析结果显示,hsa-miR-22表达水平与HBXmRNA表达水平呈正相关(r=0.415,P=0.018),hsa-miR-373、hsa-miR-92、hsa-miR-155表达水平与HBXmRNA表达水平呈负相关(r=-0.322,P=0.043;r=-0.369,P=0.028;r=-0.426,P=0.016);hsa-miR-181a、hsa-miR-34a、hsa-let-7i表达水平与HBXmRNA表达水平无直线相关关系(r=0.076,P=0.363;r=0.017,P=0.676;r=0.122,P=0.242)。
注:miRNAs=微小RNA
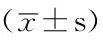
图1 HBX调控的miRNAs差异表达微阵列技术分析图
表1HBX相关miRNAs差异表达微阵列技术分析结果
Table 1Differentially expressed HBX-related miRNAs by microarray analysis

编号探针标识HepG2-X细胞系HepG2-CAT细胞系HepG2-X/HepG2-CATHepG2-X细胞系中miRNAs表达上调 1hsa-miR-542-5p412.654.67-6.21 2hsa-miR-222091.08814.17-1.30 3hsa-miR-6631252.37559.82-1.15 4hsa-miR-63814865.087188.07-1.05 5hsa-miR-575371.21195.98-0.95 6hsa-miR-181a2162.731084.31-0.90 7hsa-miR-5741054.20656.37-0.72 8hsa-miR-148a847.00515.70-0.69 9hsa-miR-99b803.62517.24-0.64 10hsa-miR-34a2287.221593.84-0.55 11hsa-miR-425-5p1531.641048.80-0.55 12hsa-miR-146a1573.181092.16-0.52 13hsa-miR-181b2302.841565.25-0.50 14hsa-miR-10310746.357735.30-0.46 15hsa-miR-1079513.037142.21-0.44 16hsa-miR-2223486.072660.43-0.43 17hsa-miR-2410474.498423.06-0.36 18hsa-miR-19110090.308037.62-0.34 19hsa-miR-935534.524334.91-0.33 20hsa-miR-2158338.6048732.69-0.26HepG2-X细胞系中miRNAs表达下调 1hsa-miR-37336.34184.872.24 2hsa-miR-422b96.69399.792.07 3hsa-miR-7112.72391.411.80 4hsa-miR-30e-3p39.01122.551.69 5hsa-miR-365133.18353.571.38 6hsa-let-7i179.62472.251.36 7hsa-miR-15555.96125.521.29 8hsa-let-7g1232.012950.181.08 9hsa-miR-454-3p625.461180.561.00 10hsa-let-7d487.32915.180.90 11hsa-let-7c438.03768.580.84 12hsa-let-7f1471.042924.930.74 13hsa-miR-148b768.371204.550.69 14hsa-miR-15b3203.725179.950.68 15hsa-miR-15a476.88790.580.68 16hsa-miR-146b975.771552.230.67 17hsa-miR-92b4883.127353.030.51 18hsa-miR-5942168.712918.820.42 19hsa-miR-2157817.8110169.990.42 20hsa-miR-4832011.732654.540.41 21hsa-let-7a2700.143681.230.41 22hsa-miR-20b8008.7110597.250.38 23hsa-miR-9211881.0514904.740.32 24hsa-miR-26b11502.1613990.540.28 25hsa-miR-27b10394.6612018.330.21 26hsa-miR-23a14559.3416142.120.18
注:miRNAs=微小RNA
2.4过表达hsa-miR-373或抑制hsa-miR-22对乙型肝炎病毒复制的影响第2天和第3天阴性对照组、hsa-miR-22抑制剂组、hsa-miR-373前体组HepG2.2.15细胞系及HepAD-38细胞系乙型肝炎病毒拷贝数比较,差异有统计学意义(P<0.05);其中第2天和第3天hsa-miR-22抑制剂组、hsa-miR-373前体组HepG2.2.15细胞系及HepAD-38细胞系乙型肝炎病毒拷贝数较阴性对照组降低,差异有统计学意义(P<0.05);第2天和第3天hsa-miR-373前体组HepG2.2.15细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组降低,差异有统计学意义(P<0.05);第2天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组升高,差异有统计学意义(P<0.05);第3天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝数较hsa-miR-22抑制剂组降低,差异有统计学意义(P<0.05,见表3)。
第2天和第3天hsa-miR-373前体组HepG2.2.15细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组升高,差异有统计学意义(P<0.05)。第2天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组降低,差异有统计学意义(P<0.05);第3天hsa-miR-373前体组HepAD-38细胞系乙型肝炎病毒拷贝抑制率较hsa-miR-22抑制剂组升高,差异有统计学意义(P<0.05,见表4)。
3讨论
据统计,全球范围内约有3亿慢性乙型肝炎病毒感染的患者,慢性乙型肝炎诱发的肝硬化是肝癌发生的主要病因之一[4,11]。而其中乙型肝炎病毒编码的HBX则是导致肝细胞癌变的关键因素[12]。因此,研究HBX相关miRNAs的差异表达将加深对乙型肝炎病毒慢性感染诱发肝癌过程中复杂调控网络的认识,为干预肝癌进展、抗肿瘤复发转移提供新方法、新思路。
Table 3Comparison of HBV replication number of HepG2.2.15 and HepAD-38 cell lines among negative control group,hsa-miR-22 inhibition group and hsa-miR-373 precursor group
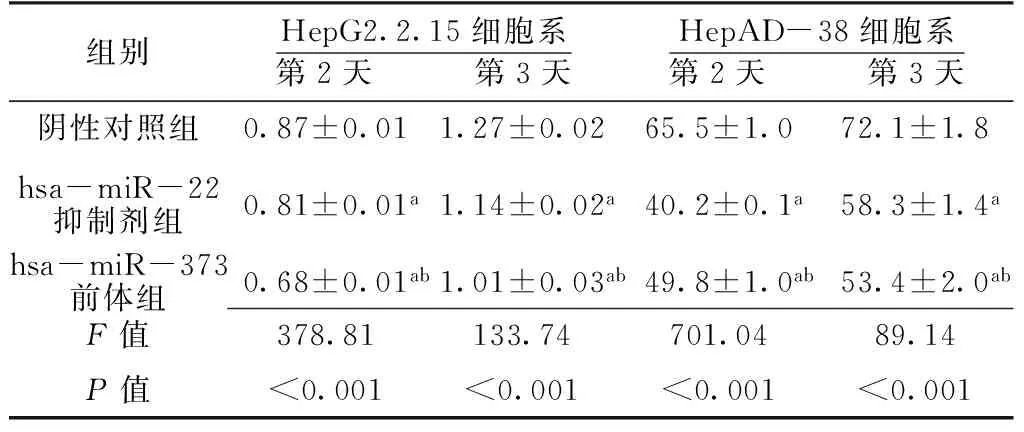
组别HepG2.2.15细胞系第2天 第3天HepAD-38细胞系第2天 第3天阴性对照组0.87±0.011.27±0.0265.5±1.072.1±1.8hsa-miR-22抑制剂组0.81±0.01a1.14±0.02a40.2±0.1a58.3±1.4ahsa-miR-373前体组0.68±0.01ab1.01±0.03ab49.8±1.0ab53.4±2.0abF值378.81133.74701.0489.14P值<0.001<0.001<0.001<0.001
注:与阴性对照组比较,aP<0.05;与hsa-miR-22抑制剂组比较,bP<0.05
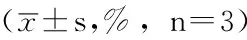
Table 4Comparison of HBV replication inhibition rate of HepG2.2.15 and HepAD-38 cell lines between hsa-miR-22 inhibition group and hsa-miR-373 precursor group
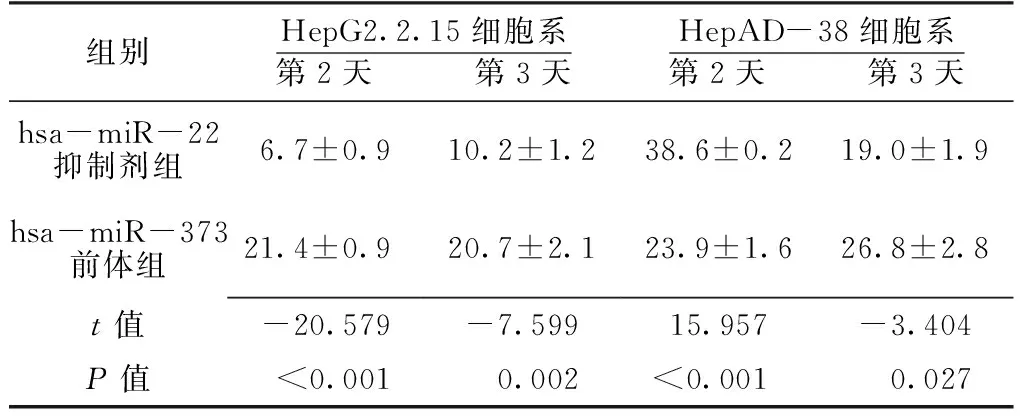
组别HepG2.2.15细胞系第2天 第3天HepAD-38细胞系第2天 第3天hsa-miR-22抑制剂组6.7±0.910.2±1.238.6±0.219.0±1.9hsa-miR-373前体组21.4±0.920.7±2.123.9±1.626.8±2.8t值-20.579-7.59915.957-3.404P值<0.0010.002<0.0010.027
本研究利用微阵列技术分析稳定表达HBX的HepG2-X细胞系及其对照HepG2-CAT细胞系中miRNAs差异表达情况,结果发现46种受HBX调控的miRNAs。随后,对与肝脏功能相关的候选的7个差异表达miRNAs,在13对肝癌组织及癌旁组织中进行了检测,并与HBX表达情况进行了对比,其中有4个miRNAs(hsa-miR-22、hsa-miR-373、hsa-miR-92、hsa-miR-155)与HBX mRNA表达水平相关。进一步研究发现,当引入has-miR-373 前体或has-miR-22抑制剂后HepG2.2.15细胞系(产生低拷贝乙型肝炎病毒)和 HepAD-38细胞系(产生高拷贝乙型肝炎病毒)中乙型肝炎病毒的复制水平均被显著抑制。
上述结果提示,HBX可通过调控miRNAs的表达,在乙型肝炎病毒复制中扮演着非常重要的角色。由于HBX可作为反式激活因子参与调节细胞内基因的转录过程,因此推测HBX有可能通过与不同的转录因子相结合从而分别实现抑制has-miR-373的表达和促进has-miR-22的表达。同时由于不同miRNAs可通过不完全互补配对方式与基因转录本3′非翻译区结合从而抑制基因的表达,因此本研究推测HBX可通过调控has-miR-373和has-miR-22的表达,负向调节不同下游基因的表达,从而实现对乙型肝炎病毒复制的精细调控。
综上所述,HBX相关miRNAs的差异表达可通过靶向相关信号转导通路有效影响乙型肝炎病毒在肝癌细胞内的复制。对于发现的显著影响乙型肝炎病毒复制的has-miR-373和has-miR-22是如何通过干预不同基因的表达来实现对乙型肝炎病毒复制的调控以及其介导的下游信号转导通路之间是否存在交互调控尚需后续深入研究。
作者贡献:陈静提出研究思路,设计研究方案,进行实验实施和数据分析,撰写论文;陈磊完善研究方案,采集样本,数据分析复核,修改论文;吴孟超负责最终版本修订;石晓祯协助采集样本并进行实验。
本文无利益冲突。
参考文献
[1]Yates LA,Norbury CJ,Gilbert RJ.The long and short of microRNA[J].Cell,2013,153(3):516-519.
[2]Lin S,Gregory RI.MicroRNA biogenesis pathways in cancer[J].Nat Rev Cancer,2015,15(6):321-333.
[3]Suzuki HI,Katsura A,Matsuyama H,et al.MicroRNA regulons in tumor microenvironment[J].Oncogene,2015,34(24):3085-3094.
[4]Arzumanyan A,Reis HM,Feitelson MA.Pathogenic mechanisms in HBV-and HCV-associated hepatocellular carcinoma[J].Nat Rev Cancer,2013,13(2):123-135.
[5]Hu L,Chen L,Li L,et al.Hepatitis B virus X protein enhances cisplatin-induced hepatotoxicity via a mechanism involving degradation of Mcl-1[J].J Virol,2011,85(7):3214-3228.
[6]Soifer HS,Rossi JJ,Saetrom P.MicroRNAs in disease and potential therapeutic applications[J].Mol Ther,2007,15(12):2070-2079.
[7]Dews M,Homayouni A,Yu D,et al.Augmentation of tumor angiogenesis by a Myc-activated microRNA cluster[J].Nat Genet,2006,38(9):1060-1065.
[8]Bourne C,Lee S,Venkataiah B,et al.Small-molecule effectors of hepatitis B virus capsid assembly give insight into virus life cycle[J].J Virol,2008,82(20):10262-10270.
[9]Chen L,Hu L,Li L,et al.Dysregulation of β-catenin by hepatitis B virus X protein in HBV-infected human hepatocellular carcinomas[J].Front Med China,2010,4(4):399-411.
[10]Feitelson MA,Lee J.Hepatitis B virus integration,fragile sites,and hepatocarcinogenesis[J].Cancer Lett,2007,252(2):157-170.
[11]Wang HY,Chen L.Tumor microenviroment and hepatocellular carcinoma metastasis[J].J Gastroenterol Hepatol,2013,28(Suppl 1):43-48.
[12]Tian Y,Yang W,Song J,et al.Hepatitis B virus X protein-induced aberrant epigenetic modifications contributing to human hepatocellular carcinoma pathogenesis[J].Mol Cell Biol,2013,33(15):2810-2816.
(本文编辑:陈素芳)
Effect of Differential Expression of HBX-regulated miRNAs on HBV Replication
CHENJing,CHENLei,WUMeng-chao,etal.
ShanghaiDIDABiotechnologyCo.,Ltd,Shanghai200437,China
【Abstract】BackgroundHepatitis B virus X protein(HBX) plays a very important role in HBV replication and hepatocarcinogenesis(HCC).Research on the differential expression of HBX-regulated miRNAs may provide important information for the regulation of HBV replication,the elaboration of the development process of liver cancer and intervention on prognosis.ObjectiveTo explore the differential expression of HBX-regulated miRNAs in HCC cells and its potential role in HBV replication.MethodsWe collected fresh specimens with paraffin embedding of HCC tissue and para-carcinoma tissue >2 cm from HCC tissue from 13 HCC patients who were admitted into Eastern Hepatobiliary Surgery Hospital Affiliated to the Second Military Medical University from January to October in 2014.Microarry technology was used to analyze the differential expression of HBX-regulated miRNAs.Quantitative analysis was conducted to investigate the expression of seven miRNAs relevant with liver function and HBX mRNA in HCC tissue and para-carcinoma tissue by RT-qPCR method,and the influence of over-expression of hsa-miR-373 and inhibiting-expression of hsa-miR-22 on HBV replication was explored.ResultsThe microarray results showed that there were a total of 46 differential expressions of miRNAs in HepG2-X and HepG2-CAT cell lines,and compared with HepG2-CAT cell line,20 miRNAs expressions were higher and 26 miRNAs expressions were lower in HepG2-X cell line.HCC tissue and para-carcinoma tissue were significantly different in the expression levels of hsa-miR-22,hsa-miR-373,hsa-miR-92 and hsa-miR-155 and the expression level of HBX mRNA(P<0.05);HCC tissues and para-carcinoma tissues were not significantly different in the expression levels of hsa-miR-181a,hsa-miR-34a and hsa-let-7i(P>0.05).Pearson correlation analysis showed that the expression level of hsa-miR-22 was postitvely correlated with the expression level of HBX mRNA(r=0.415,P=0.018),and there was negative correlation between the expression level of hsa-miR-373,hsa-miR-92 and hsa-miR-155 and the expression level of HBX mRNA(r=-0.322,P=0.043;r=-0.369,P=0.028;r=-0.426,P=0.016);no linear correlation existed between the expression level of hsa-miR-181a,hsa-miR-34a and hsa-let-7i and the expression level of HBX mRNA(r=0.076,P=0.363;r=0.017,P=0.676;r=0.122,P=0.242).On day 2 and day 3,hsa-miR-22 inhibition group and hsa-miR-373 precursor group were higher than negative control group in HBV replication number of HepG2.2.15 and HepAD-38 cell lines(P<0.05);on day 2 and day 3,hsa-miR-373 precursor group was lower than hsa-miR-22 inhibition group in HBV replication number of HepG2.2.15 cell line(P<0.05).On day 2,hsa-miR-373 precursor group was higher than hsa-miR-22 inhibition group in HBV replication number of HepAD-38 cell line,and on day 3,hsa-miR-373 precursor group was lower than hsa-miR-22 inhibition group in HBV replication number of HepAD-38 cell line(P<0.05).On day 2 and day 3,hsa-miR-373 precursor group was higher than hsa-miR-22 inhibition group in the inhibition rate of HBV replication of HepG2.2.15 cell line(P<0.05).On day 2,hsa-miR-373 precursor group was lower than hsa-miR-22 inhibition group in the inhibition rate of HBV replication of HepAD-38 cell line,and on day 3,hsa-miR-373 precursor group was higher than hsa-miR-22 inhibition group in the inhibition rate of HBV replication of HepAD-38 cell line(P<0.05).ConclusionThe regulation of miRNAs by HBX can influence the replication of HBV in HCC cells by target-related signaling pathway.
【Key words】Liver neoplasms;Hepatitis B virus X protein;Micro-RNAs;Virus replication
基金项目:国家自然科学基金资助项目(91029723)
通信作者:陈静,200437上海市缔达生物科技有限公司;E-mail:dida_bio@163.com
【中图分类号】R 512.62
【文献标识码】A
doi:10.3969/j.issn.1007-9572.2016.15.010
(收稿日期:2015-12-03;修回日期:2016-03-16)

