花生中三个LEA基因的克隆与表达分析
赵小波,张廷婷,闫彩霞,李春娟,王 娟,张 浩,2,石程仁,单世华*
(1.山东省花生研究所,山东 青岛 266100; 2.山东农业大学农学院,山东 泰安 271018)
花生中三个LEA基因的克隆与表达分析
赵小波1,张廷婷1,闫彩霞1,李春娟1,王 娟1,张 浩1,2,石程仁1,单世华1*
(1.山东省花生研究所,山东 青岛 266100; 2.山东农业大学农学院,山东 泰安 271018)
胚胎发生晚期丰富蛋白(LEA)是一类受发育阶段及脱水信号调节的脱水保护蛋白,在响应植物干旱、低温、高盐等逆境胁迫中具有重要功能。本研究在转录组数据分析时,发现3个LEA基因在花生干旱胁迫时大量上调表达。利用RACE技术以及生物信息学方法,发现这三个基因分别属于LEA2、LEA3以及LEA4。通过序列比对进行进化分析,结果表明其均与Arachisduranensis,A.ipaensis相关基因具有极高相似度,印证了栽培花生来源于Arachisduranensis和A.ipaensis的研究结果。通过表达分析发现,正常状态下这三个基因在花生根、茎、叶中均有表达;干旱胁迫状态下,花生根部的LEA基因大量表达,暗示其在花生抗旱机制中发挥重要作用。本研究结果可为筛选新的抗旱花生种质提供理论依据。
花生;胚胎发生晚期丰富蛋白;干旱;转录组
花生(ArachishypogaeaL.)是世界第四大油料作物,也是我国主要的经济和油料作物。花生产业的发展对于增加农民收入和保障国家油料安全具有重要意义。在我国,花生主要种植在占国土面积30.8%的干旱半干旱地区[1],该地区在花生生长季节的降雨量分配不均且年份间波动较大,可出现前期、中期、后期干旱的情况[2]。干旱不仅在很大程度上引起花生产量降低,也会导致黄曲霉抗性下降引起黄曲霉素污染。据估算,我国花生播种面积的70%以上常年受不同程度的干旱威胁,平均减产20%以上,干旱是我国花生生产上影响面最大限制因素[3]。据分析预测,在世界花生生产大国,包括中国与印度,干旱气候出现的频率有逐渐上升的趋势[4]。因此,如何在极端气候条件下保持甚至实现花生产量的增长是一个重要课题,培育抗旱花生品种是解决这一困局的重要手段之一[5]。传统育种方法由于周期较长,效率相对较低因而发展较慢。通过基因工程技术将抗旱基因导入花生,使其增强抗旱能力是相对可行的方法。目前通过基因工程技术从分子水平改良水稻等多种农作物抗旱性已经取得了一定的进展[6]。
众多抗旱相关基因中,LEA(胚胎发生晚期丰富蛋白,Late Embryogenesis Abundant Proteins)基因家族已被大量的研究证实可在多种物种应答逆境胁迫中起到保护作用[7-9]。研究表明,在不同植物物种乃至亚种,LEA各个亚家族基因之间的表达模式具有差异,这暗示了它们功能的歧化,也说明了研究不同物种中LEA基因的必要性[6,10-12]。本研究在干旱胁迫转录组测序数据分析中发现,上调表达最大的前20个基因中,有3个基因属于LEA基因家族,利用RACE技术对这3个基因进行了克隆。同时,对其全长序列进行生物信息学分析、同源性比较及功能验证,从而为下一步的利用工作提供理论基础。
1 材料与方法
1.1 试验材料与处理
供试花生材料为抗旱品种J11,由山东省花生研究所提供,所用材料为当年田间种植样品。样品培养参考Sarkar等的方法[13]。萌发14d后的幼苗采用20% PEG-6000干旱胁迫,分别取处理前(0 h)及处理后6、12、18、24和48h根部组织,二次重复。采集的样品送交北京诺禾致源公司进行转录组测序。同时采集正常状态花生J11的茎、根、叶,液氮速冻后于-80℃保存,用于荧光定量PCR分析。
1.2 叶片总RNA的提取
总RNA提取采用杨晨等[14]方法略有改进。用紫外分光光度计(AA-680,日本岛津)检测RNA浓度并利用琼脂糖电泳检测RNA完整性。
1.3 基因全长的扩增
在转录组测序结果中发现三个LEA类基因片段在干旱胁迫后表达量显著增加,推测其与花生抗旱性有一定关系。根据得到的序列设计RACE引物(表1)。以提取的花生抗旱品种J11根部组织RNA为模板,采用Clontech公司的SMART RACE试剂盒进行RACE克隆。反应条件依据试剂盒说明书进行。
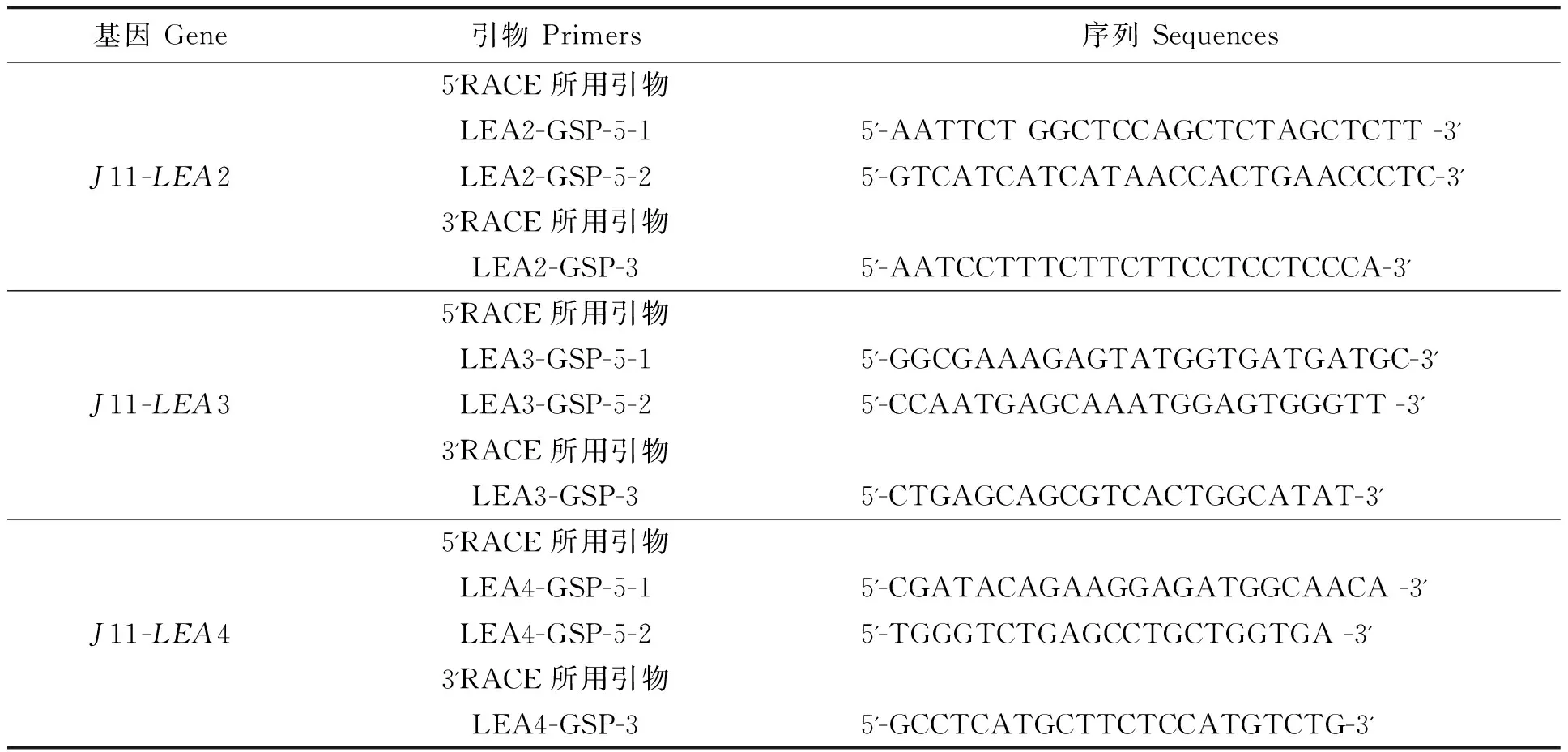
表1 RACE克隆所需引物Table 1 Primers for RACE in this study
反应产生的5' 与3' PCR产物采用琼脂糖凝胶回收试剂盒(天根生化科技)回收纯化。纯化后的产物与T载体(北京全是金生化科技有限公司)连接后转化至感受态E.coliDH5α(大连宝生物)中。LB培养基培养,蓝白斑筛选后随机挑取8个阳性克隆进行扩大培养,菌液PCR扩增预检测是否有插入片段[15]。
1.4 测序与序列比较
测序委托南京金丝瑞生物技术公司完成。通过DNAMAN软件去载体拼接后,得到基因全长。获得的序列数据首先与GenBank中已有序列信息进行比对。然后利用ClustalW软件对测序数据进行人工对齐排序。选择Kimura 2-parameter公式[16]计算遗传距离。并在MEGA5.0中采用邻近法(Neighbor-Joining,NJ)构建系统树,自举值Bootstrap为1000次重复。
1.5 荧光定量RT-PCR分析
根据扩增得到的序列,用 Beacon Designer 7.0设计荧光定量PCR特异引物(表2)。依据前述方法与设置,分别提取花生干旱胁迫的根部,以及正常状态根、茎、叶的RNA。使用7500 FAST荧光定量PCR仪(ABI公司)分析3个LEA基因的相对表达情况。反应条件为 95℃ 10s;95℃ 5s,60℃ 30s,72℃ 10s,40个循环;绘制溶解曲线,温度每10s升高0.5℃。实验设置3次重复。相对表达量的计算采用2-ΔΔCT方法[17],选择Actin 11作为内参基因[18],实验数据采用SPSS12.0分析(SPSS Inc.,Chicago,USA)。

表2 LEA基因表达所用引物Table 2 Primers used for analysis of LEA gene expression by RealTime-qPCR
2 结果与分析
2.1 克隆结果与生物信息学分析
转录组测序结果分析发现,上调表达最大的前20个基因中,有3个属于LEA基因家族。经RACE克隆后,将3个基因在PeanutBase (http://peanutbase.org/)以及NCBI数据库上进行比对,发现分别属于LEA2,LEA3以及LEA4基因,定名为J11-LEA2 (GenBank ID:KX609696),J11-LEA3 (GenBank ID:KX588233) 和J11-LEA4 (GenBank ID:KX579898)。利用NCBI上的ORF Finder分析获得3个基因的全长,发现LEA2基因开放阅读框有624个碱基,编码蛋白质含有207个氨基酸。其中甘氨酸、苏氨酸、赖氨酸为含量前三位的氨基酸,分别为38个,29个以及17个,占比分别为18.36%,14.01%及8.21%。其编码蛋白的分子量大小为21.017kD,等电点为7.51,其二级结构见图1(a)。预测编码蛋白质的跨膜结合区域,发现由内到外、由外到内的跨膜区域均为2个(图2)。LEA3基因开放阅读框有900个碱基,编码蛋白质含有299个氨基酸。其中丙氨酸、赖氨酸、谷氨酸为含量前三位的氨基酸,分别为60个,45个以及40个,占比分别为20.07%,15.05%及13.38%。通过DNAMAN软件预测其分子量大小为32.294kD,等电点为4.93。其二级结构见图1(b)。预测编码蛋白质的跨膜结合区域,发现由内到外、由外到内的跨膜区域分别为15个和10个(图2)。LEA4基因开放阅读框有411个碱基,编码蛋白质含有136个氨基酸。其中丙氨酸、赖氨酸、苏氨酸为含量前三位的氨基酸,分别为23个,16个及14个,占比分别为16.91%,16.76%及10.29%。通过DNAMAN软件预测其分子量的大小为14.241kD,等电点为9.56。其二级结构见图1(c)。由TMpred程序预测该编码蛋白质的跨膜结合区域,发现由内到外、由外到内的跨膜区域均为0个(图2)。
2.2 系统进化分析
利用MEGA软件对三个基因与部分已知序列在核苷酸水平上进行聚类分析(图3),结果表明,落花生属的几个种聚成一类,显示LEA基因在该属内的保守性较强。通过分析发现这三个基因与Arachisduranensis、A.ipaensis相关基因的相似度极高,均达到99%以上。2016年公布的花生基因组测序结果表明,Arachisduranensis与A.ipaensis经过自然杂交,形成了今天栽培花生的祖先。Arachisduranensis的基因组与A亚基因组、A.ipaensis的基因与B亚基因组几乎完全相同[19]。该研究成果也印证了作为栽培花生,J11的LEA基因与Arachisduranensis、A.ipaensis相关基因高相似度这一发现。
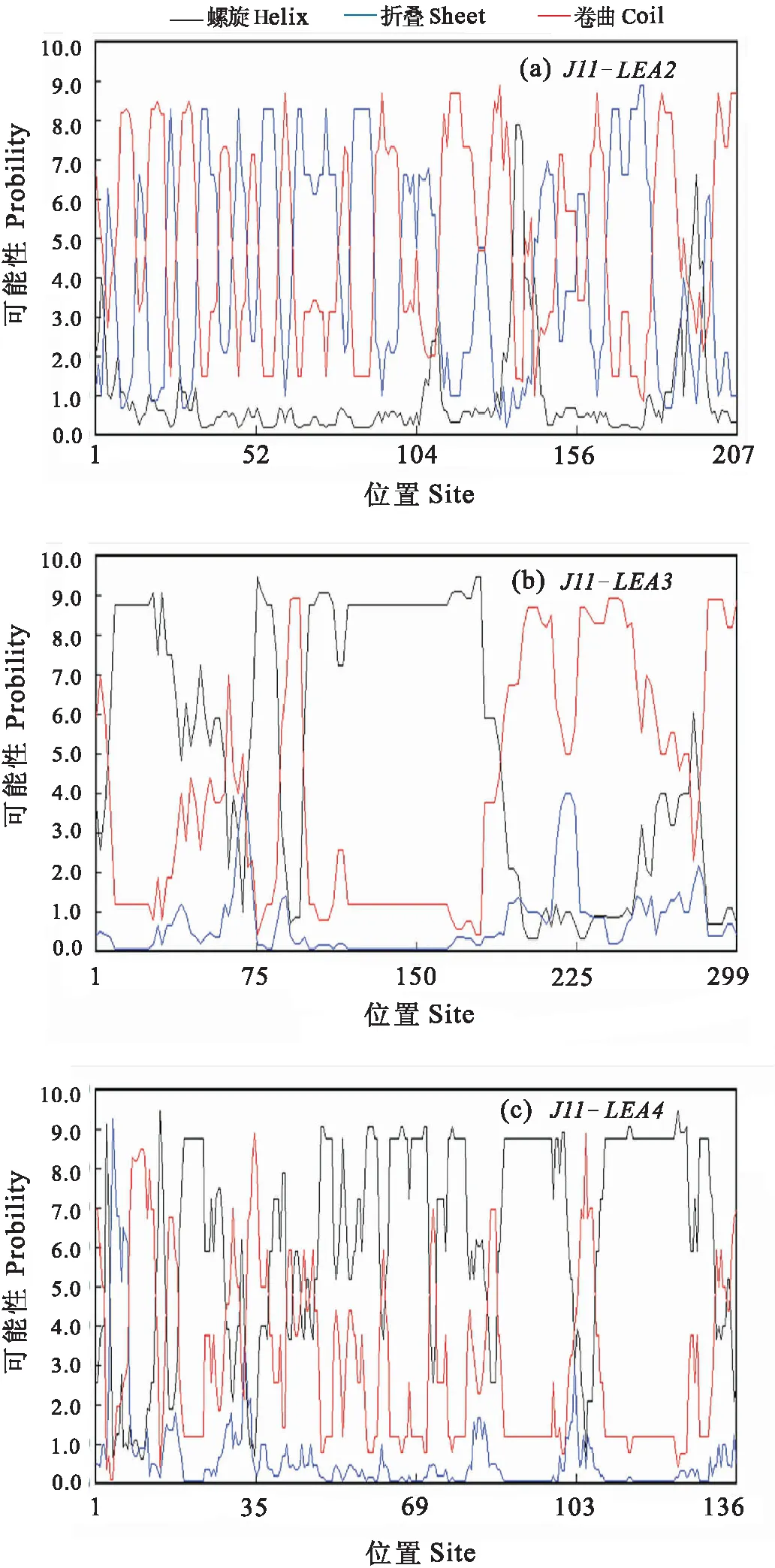
图1 3个LEA基因氨基酸序列的二级结构Fig.1 Secondary structure of three LEA amino acids
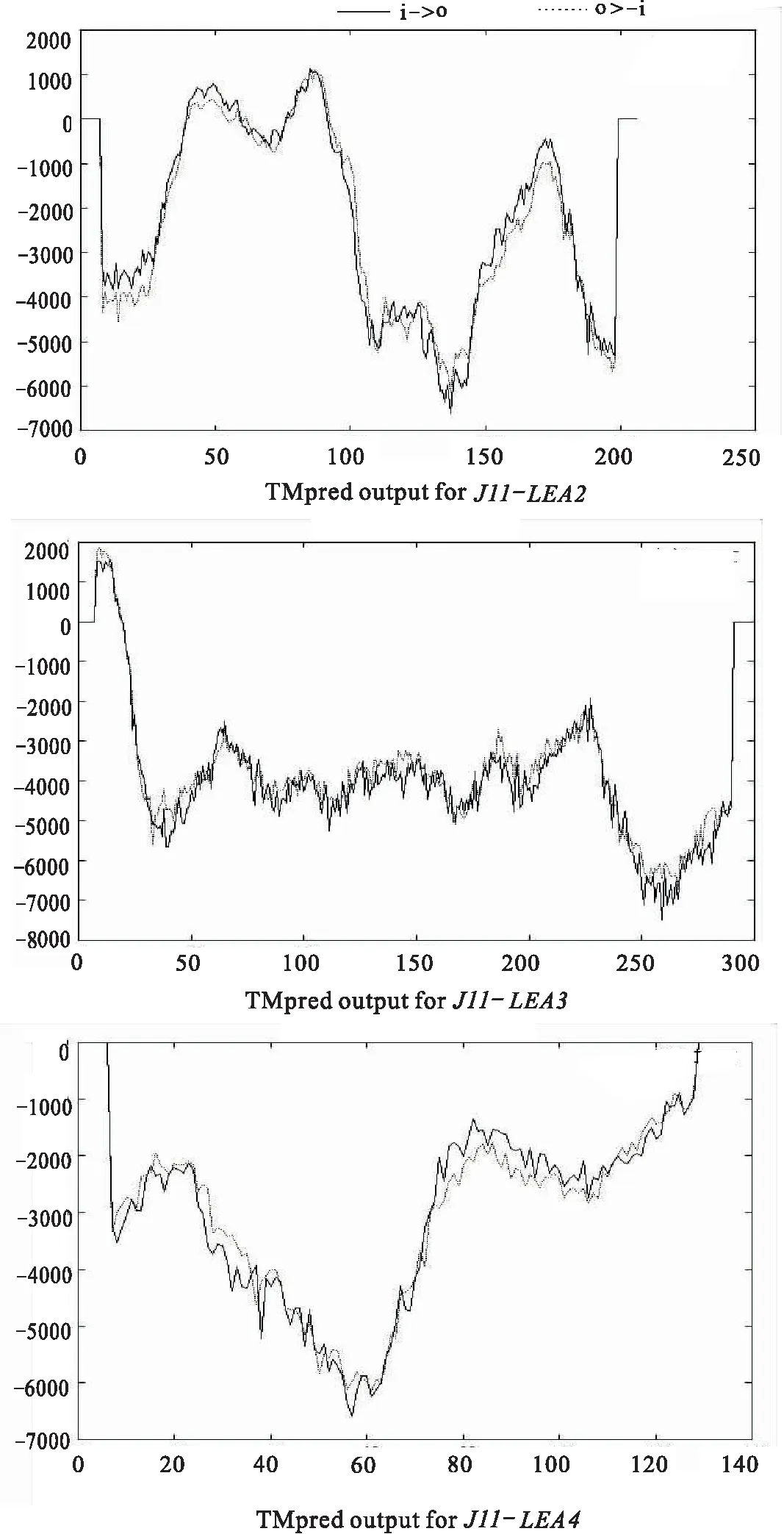
图2 3个LEA基因氨基酸序列的多肽跨膜结合区域Fig.2 Polypeptide transmembrane binding region of three LEA amino acids
2.3 LEA基因表达分析
在正常生长状态下,J11的根、茎及叶片均可检测到这3个基因的表达。其中,叶片中J11-LEA2的相对表达量大于茎部以及根部。J11-LEA3与J11-LEA4的相对表达量均为根部最大(图4)。干旱胁迫处理后,3个LEA基因的相对表达量均有所上升。其中,J11-LEA2的相对表达量在48h时达最大值;24与48h时,J11-LEA3的相对表达量无显著差异(p>0.05),处于平稳表达阶段;J11-LEA4的相对表达量在24h时显著大于48h (p<0.05)(图5)。
3 讨 论
胚胎发生晚期丰富蛋白(LEA)是一类庞大的蛋白家族,现有研究表明,该家族的蛋白可以大量积累以应对诸如干旱、盐碱以及低温等胁迫环境[20-21]。该蛋白家族的积累主要发生在种子胚胎发生后期,但是研究发现拟南芥的一些LEA基因也可在其他组织中表达,例如AtLEA3-7和AtLEA5-3[22]。大多数已知的花生LEA基因也主要在种子胚胎发生后期表达[23]。在研究中,利用从根部提取的RNA进行转录组测序,发现LEA2,LEA3和LEA4均可大量表达应对干旱胁迫;在正常条件下,这三个基因也可在茎、叶部组织表达。在之前的研究中,Su发现鲁花14的LEA4基因在根、茎以及叶中没有表达[23],这一点与本研究结果不一致。不同的研究结果表明即便是同一物种的同一LEA基因亚家族,在不同基因型中的表达模式可能也会有差异。综合荧光定量PCR的实验结果以及其他研究结果,LEA2,LEA3与LEA4基因在花生应答干旱胁迫时可能起到了重要作用。Wang等[24]将小麦的TaLEA3基因通过农杆菌介导转入羊草,在干旱胁迫下转基因植株相对含水量和生长势均优于对照,而丙二醛含量低于对照;Byong等[25]将来自油菜LEA基因转入中国卷心菜,转基因卷心菜在盐胁迫和干旱胁迫条件下植株具有增强的生长能力,表明转LEA基因增强了植物对盐和干旱的耐受性。目前,花生转基因技术体系尚不成熟,花生LEA基因家族的确切功能有待于进一步的验证。另外,其基因表达调控机理尚不清楚,这也将是下一步研究工作的重点。

图3 LEA序列系统进化树Fig.3 Neighbor-Joining (NJ) phylogeny estimated using LEA sequence

图4 正常状态3个基因在不同组织中的表达Fig.4 LEA gene expression of different organs in peanut under normal condition

图5 干旱胁迫3个基因在根中的表达Fig.5 Root LEA gene expression at different time response to drought-stress
[1] 张智猛,戴良香,宋文武,等.干旱处理对花生品种叶片保护酶活性和渗透物质含量的影响[J].作物学报,2013,39(1):133-141.
[2] 孙爱清,张杰道,万勇善,等.花生干旱胁迫响应基因的数字表达谱分析[J].作物学报,2013,39(6):1045-1053.
[3] 苗锦山,王铭伦.水分胁迫对花生生长发育影响的研究进展[J].花生学报,2003,32(增刊):368-371.
[4] Bhatnagar-Mathur P,Rao J S,Vadez V,et al.Transgenic peanut overexpressing theDREB1Atranscription factor has higher yields under drought stress [J].Molecular Breeding,2014,33:327-340.
[5] Hasthanasombut S,Supaibulwatana K,Mii M,et al.Genetic manipulation of Japonica rice using theOsBADH1 gene from Indica rice to improve salinity tolerance [J].Plant Cell,Tissue and Organ Culture,2011,104:9-89.
[6] 裴金玲,杨红兰,李春平,等.转晚期胚胎发生丰富蛋白(LEA)基因棉花及抗旱性分析[J].分子植物育种,2012,10(3):331-337.
[7] Amara I,Odena A,Eliandre Oliveira E,et al.Insights into maize LEA proteins:from proteomics to functional approaches [J].Plant Cell Physiology,2012,53(2):312-329.
[8] Goyal K,Browne J A,Burnell A M,et al.Dehydration-inducedtpsgene transcripts from an anhydrobiotic nematode contain novel spliced leaders and encode atypical GT-20 family proteins [J].Biochimie,2005,87:565-574.
[9] 李剑,赵常玉,张富生,等.LEA蛋白与植物抗逆性[J].植物生理学通讯,2010,46(11):1101-1102.
[10] 李乐,许红亮,杨兴露,等.大豆LEA基因家族全基因组鉴定、分类和表达[J].中国农业科学,2011,44(19):3945-3954.
[11] Welin B V,Olson A,Nylander M,et al.Characterization and differential expression ofdhn/lea/rab-likegenes during cold acclimation and drought stress inArabidopsisthaliana[J].Plant Molecular Biology,1994,26(1):131-144.
[12] Wang X S,Zhu H B,Jin G L,et al.Genome-scale identification and analysis ofLEAgenes in rice (OryzasativaL.)[J].Plant Science,2007,172:414-420.
[13] Sarkar T,Thankappan R,Kumar A,et al.Heterologous expression of theAtDREB1Agene in transgenic peanut-conferred tolerance to drought and salinity stresses [J].PLOS ONE,2014,DOI:10.1371/journal.pone.0110507.
[14] 杨晨,徐凤花,单世华,等.花生子叶RNA提取方法比较与分析[J].山东农业科学,2011(1):1-5.
[15] 赵小波,张廷婷,闫彩霞,等.花生中一个NBS-LRR类基因的克隆和表达分析[J].山东农业科学,2015,47(5):1-5,14.
[16] Kimura M.A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences [J].Journal of Molecular Evolution,1980,16:111-120.
[17] Lane B G,Dunwelll J M,Rag J A,et al.Germin.A protein marker of early plant development,is an oxalate oxidase [J].Journal of Biological Chemistry,1993,68:12239-12242.
[18] Chi X Y,Hu R B,Yang Q L,et al.Validation of reference genes for gene expression studies in peanut by quantitative real-time RT-PCR [J].Molecular Genetics Genomics,2012,287:167-176.
[19] Bertioli D J,Cannon S B,Froenicke L,et al.The genome sequences ofArachisduranensisandArachisipaensis,the diploid ancestors of cultivated peanut [J].Nature Genetics,2016,DOI:10.1038/ng.3517.
[20] Battaglia M,Olvera-Carrillo Y,Garciarrubio A,et al.The enigmatic LEA proteins and other hydrophilins [J].Plant Physiology,2008,148:6-24.
[21] Shimizu T,Kanamori Y,Furuki T,et al.Desiccation-induced structuralization and glass formation of group 3 late embryogenesis abundant protein model peptides [J].Biochemistry,2010,49:1093-1104.
[22] Bies-Ethève N,Gaubier-Comella P,Debures A,et al.Inventory,evolution and expression profiling diversity of the LEA protein gene family inArabidopsisthaliana[J].Plant Molecular Biology,2008,67:107-124.
[23] Su L,Zhao C Z,Bi Y P,et al.Isolation and expression analysis ofLEAgenes in peanut (ArachishypogaeaL.)[J].Journal of Biosciences,2011,36(2):223-228.
[24] Wang L J,Li X F,Chen S Y,et al.Enhanced drought tolerance in transgenicLeymuschinensisplants with constitutively expressed wheatTaLEA3 [J].Biotechnology Letters,2009,31(2):313-319.
[25] Byong J P,Zao C L,Akira K,et al.Genetic improvement of Chinese cabbage for salt and drought tolerance by constitutive expression of aB.napusLEAgene [J].Plant Science,2005,169(3):553-558.
Cloning and Expression Analysis of ThreeLEAGenes in Peanut
ZHAO Xiao-bo1,ZHANG Ting-ting1,YAN Cai-xia1,LI Chun-juan1,WANG Juan1, ZHANG Hao1,2,SHI Cheng-ren1,SHAN Shi-hua1*
(1.ShandongPeanutResearchInstitute,Qingdao266100,China;2.CollegeofAgriculture,ShandongAgriculturalUniversity,Tai'an271018,China)
Late embryogenesis abundant (LEA) family proteins are expressed abundantly at the late stage of embryonic development of plant seeds,and play important roles in responding to various environmental stress,such as drought,low temperature and high salinity.ThreeLEAgenes were highly expressed under drought stress condition in RNA-seq data.Using RACE to isolate and clone the full length of these genes and they were classified toLEA2,LEA3 andLEA4,respectively.LEAbased phylogenetic tree showed that these genes had close relationship withArachisduranensisandA.ipaensis,the diploid ancestors of cultivated peanut.Under normal or drought conditions,these genes could express in vegetative organs and play an important role in peanut responding to drought stress.This study provided theoretical basis for the screening of drought resistant germplasms form peanut.
Arachishypogaea; late embryogenesis abundant protein; drought; RNA-seq
10.14001/j.issn.1002-4093.2016.04.003
2016-08-01
山东省农科院青年基金(2016YQN16);国家国际科技合作专项(2015DFA31190);山东省现代农业产业技术体系花生遗传育种岗位(SDAIT-04-02) ;国家科技支撑计划(2014BAD11B04) ;花生抗逆、广适种质资源创新与利用(山东省农业良种工程)
赵小波(1984-),男,山东青岛人,山东省花生研究所助理研究员,博士,主要从事花生遗传育种研究。
*通讯作者:单世华(1971-),研究员,博士,主要从事花生种质资源与育种研究。E-mail:shhshan@sina.com
S565.2; Q786
A

