菌草种质资源SCoT遗传多样性分析
赵永红 李婵 梅嘉洺

摘 要 利用SCoT分子标记对47个菌草种质资源进行遗传多样性分析。从20个SCoT引物中筛选出10个扩增效果好的引物,对47份菌草种质材料的DNA进行PCR扩增,共获得282个多态片段。结合UPGMA聚类分析方法,算出47份种质材料间的遗传相似系数(SM)在0.77-0.96,所有47份材料被区分开来,在遗传相似系数为0.815时,47份材料可分为5大类群。研究结果表明,SCoT分子标记技术可有效用于菌草资源的遗传多样性分析。
关键词 菌草 ;SCoT分子标记 ;种质资源 ;遗传多样性
中图分类号 S216
Abstract The genetic diversity of 47 Juncao germplasms was analyzed by SCoT markers. Twenty ScoT primers were first used for screening on a subset of four germplasms, of which 10 gave good amplification patterns and were then used for analyzing the DNA of 47 JUNCAO germplasms. A total of 282 polymorphic DNA fragments were scored among the 47 JUNCAO germplasms from the electrophoresis patterns of the 10 selected SCoT primers. By using the NTSYS-pc 2.1 software combined with UPGMA clustering analysis method, the genetic similarity coefficient (SM) was calculated among all accessions and ranged from 0.77 to 0.96. All the 47 accessions were distinguished from each other. The 47 JUNCAO germplasms were clustered into five groups when the genetic similarity coefficient was 0.815. Our results showed that the SCoT markers could be effectively used for analysis of genetic diversity of Juncao germplasms.
Key words Juncao ; ScoT ; Germplasm ; Genetic diversity
菌草(JUNCAO)是指可用于栽培食用菌、药用菌、培养基的野生和人工栽培的草本植物[1],如:芦竹、芦苇、类芦等野生系列,还包括绿洲系列芦竹、巨菌草、象草等人工栽培系列,广义的菌草还包括甘蔗、玉米、高粱等作物。菌草一般丛生、直立,根系发达,产量高,多数菌草适应性广,抗逆性强,适应于各种类型的土壤,酸性粗砂质红壤和轻度盐碱地均能生长。菌草用途广泛,可作为猪、牛、羊等动物的饲料,可用于栽培食用菌,还可做非木材纤维制浆造纸原料,制造燃料乙醇等能源,同时还可以用于防止水土流失等。随着菌草技术的完善和产业不断壮大,关于菌草种质资源的收集、保存、鉴定及利用工作变得越来越迫切和重要。但是,由于可用作菌草的植物种类繁多,使得菌草种质资源在管理上存在一定盲目性和重复性。因而从分子水平上研究菌草种质资源遗传的多样性以及建立菌草种质分子鉴定技术非常重要。
随着DNA分子标记技术以及结构和功能基因组学的飞速发展,标记类型已由随机DNA分子标记(RDMs, random DNA markers)向功能性分子标记(FMs,functional markers)和目的基因分子标记(GTMs,gene targeted markers)发展。RDMs(如AFLP、RAPD、RFLP等)基于基因组中随机多态性位点开发而成,检测的多态性在基因组的位置大多随机分布,因此获得的位点通常与目标性状基因距离较远,这使得它在应用上与其目标有一定的偏差[2-3]。GTMs(如SCoT、SPAR等)和FMs因其本身可能是目的基因的一部分或者与目的基因紧密连锁而受到研究者的重视[4]。
目标起始密码子多态性(SCoT,start codon targeted polymorphism)标记是Collard和Mackill[5]于2009年在水稻上新开发的一种目的基因分子标记。其原理是利用植物基因组中ATG翻译起始位点侧翼保守序列来设计引物,进而扩增产生偏向候选功能基因区显性多态性标记。这种标记不仅能获得与性状联系紧密的目的基因,并能对性状进行跟踪,具有操作简单、多态性高、遗传信息丰富、成本低廉、引物通用性强等优点[4]。经过近几年的迅速发展,该类标记已被成功应用于花生[6]、芒果[7]、枇杷[8]、草莓[9]、苜蓿[10]、甘蔗[11]、高牛鞭草[12]、木瓜[13]、枝稷[14]、鸭茅[15]等多种植物和多个研究领域的相关遗传研究中,在植物种质资源遗传多样性与亲缘关系分析、种质鉴定和指纹图谱构建、基因差异表达和分子图谱构建等方面均取得了阶段性的进展。目前,有关菌草种质资源多样性的研究报道较少,梅嘉洺等[16]在菌草上利用PCR-RFLP技术对46份种质资源PEPC基因进行了多样性分析。本研究在此基础上,采用SCoT分子标记技术对47份菌草种质材料遗传多样性进行较为系统的分析,初步揭示不同菌草种质间亲缘关系,为菌草种质资源管理和利用提供科学依据。
1 材料与方法
1.1 材料
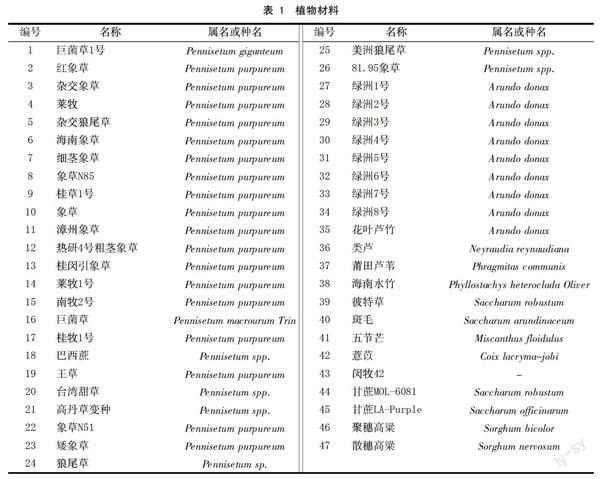
47份菌草材料取自福建农林大学国家菌草工程技术研究中心福州种质资源圃(表1)。采集各品种(系)幼嫩叶片,装入编号袋,保存于-80 ℃冰箱中备用。
1.2 方法
1.2.1 DNA的提取和检测
采用改良的CTAB法提取基因组DNA[17],然后进行1.2 %的琼脂糖凝胶电泳检测DNA样品质量,并在Nanodrop 2000(Thermo Fisher)分光光度计上测定DNA样品浓度。先将母液浓度稀释至200 ng/mL,再取少量配置成浓度20 ng/mL溶液,置于-20 ℃条件下保存备用。
1.2.2 SCoT-PCR反应体系
PCR扩增反应总体积为10 μL。其中包括DNA模板(20 ng/μL)2 μL,引物0.5 μL,2×Taq PCR Starmix(GenStar)5 μL,dd H2O 2.5 μL。引物由上海生工合成。PCR酶试剂购自北京康润诚生物技术有限公司。
PCR扩增反应程序为:94 ℃预变性4 min;94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸1 min,循环35次;随后72 ℃延伸7 min,最后4 ℃恒温保存。
1.3 PCR产物凝胶电泳和成像分析
取5 μL PCR扩增液,在1.2 %琼脂糖、TAE×1、120 V条件下电泳50 min,在凝胶成像仪(伯乐)上观察结果并拍照。
1.4 数据统计与分析
对每个SCoT引物在47份材料上的扩增产物的电泳结果进行人工比对校正,在相同迁移位置,有强带或清晰弱带记“1”,无条带记为“0”,建立原始数据(0,1)矩阵。利用NTSYS-pc 2.1软件中的子程序SIMQUAL对矩阵进行样本之间的相似性系数(SM)计算,然后用子程序SAHN中的非加权类平均法(UPGMA)进行聚类分析,最后用Tree plot绘制树状聚类图。
2 结果与分析
2.1 PCR引物筛选
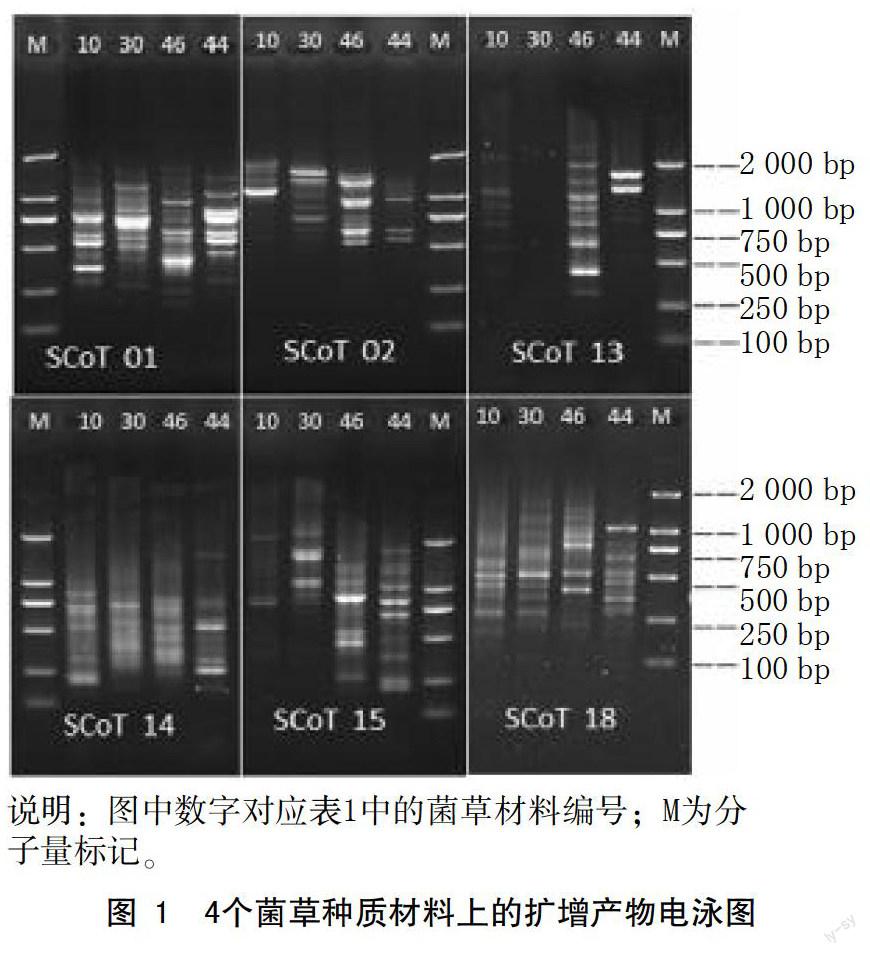
首先,用20个引物对4个菌草材料(象草、绿洲4号、聚穗高粱、甘蔗MOL-6081)进行PCR扩增。结果表明:所有20个引物都能在部分或全部供试材料上扩增出多条强度不同的DNA片段(图1)。其中10个引物扩增结果清晰、均匀(如图1中的SCoT01,SCoT02和SCoT15),因而用来作进一步扩增材料。而剩下的10个引物条带不够清晰(如图1中的SCoT14和SCoT18)或在某个材料上条带较弱甚至无任何条带(如图1中的SCoT13),而未被选择作进一步分析。
2.2 遗传多样性分析
用筛选出的10个引物对47份菌草种质材料进行PCR扩增,对扩增产物进行凝胶电泳。分析结果表明:在47份试验材料上,单个SCoT引物可以给出18-38个多态DNA片段,10个SCoT引物共给出了282个多态片段,平均每个引物给出28.2片段(表2)。引物SCoT06和SCoT07在47份菌草种质材料上的PCR扩增结果见图2。
2.3 菌草种质的聚类分析
47份菌草种质材料的SCot分子标记的UPGMA聚类分析结果见图3。聚类分析表明,供试材料之间的遗传相似系数(SM)分布在0.77-0.96,在遗传相似系数为0.815时,可以将47份菌草种质材料分成I、II、III、IV 、V 5个类群。
第I类群包括23份种质材料,是最大的一个类群,其中包含巨菌草1号、红象草、杂交象草、莱牧、杂交狼尾草、海南象草、象草N85、象草、漳州象草、热研4号粗茎象草、细茎象草、桂草1号、桂闵引象草、巨菌草、桂牧1号、巴西蔗、王草、莱牧1号、南牧2号、台湾甜草、高丹草变种、象草N51、矮象草等,该类群内的23份材料都属于狼尾草属(Pennisetum)植物。在遗传相似系数为0.85时,可以将该类群的23份材料进一步分为3个亚类群:第一亚类群包括巨菌草1号、红象草、杂交象草、莱牧、杂交狼尾草、海南象草、象草N85、象草、漳州象草、热研4号粗茎象草、细茎象草、桂草1号等12份材料;第二亚类群包括桂闵引象草、巨菌草、桂牧1号、巴西蔗、王草、莱牧1号、南牧2号等7份材料;第三亚类群包括台湾甜草、高丹草变种、象草N51、矮象草等4份材料。其中,台湾甜草和高丹草变种之间的遗传距离最相近,遗传相似系数高达0.965;象草N85和象草之间的遗传距离也较为相近,遗传相似系数为0.962。
第II和第III类群各分别包含1份种质。第II类含狼尾草,第III类群含美洲狼尾草,二者与第I大类群的23份材料都属狼尾草属(Pennisetum)植物。
第IV类群包含13份种质材料,其中包括81.95象草(Pennisetum)、海南水竹(Phyllostachys)、斑毛(Saccharum)、高粱(Sorghum)2个、薏苡(Coix)、类芦(Neyraudia)、莆田芦苇(Phragmitas)、五节芒(Miscanthus)、彼特草(Saccharum)、闽牧42、甘蔗(Saccharum)2个共13份种质。在遗传相似系数为0.825时,可以将该类群的13份材料进一步分为2个亚类群:第一个亚类群包括81.95象草、海南水竹、斑毛、高粱2个、薏苡、类芦、莆田芦苇、五节芒9份材料;第二个亚类群包括彼特草、闽牧42、甘蔗2个共4份材料。其中,2个高粱材料之间的遗传距离最相近,遗传相似系数高达0.942;2个甘蔗材料之间的遗传距离也较为相近,遗传相似系数为0.878。
第V类群包括了所有芦竹属(Arundo)的9份材料,其中绿洲8号和花叶芦竹之间的遗传距离最为相近,遗传相似系数高达0.962;绿洲4号和绿洲5号之间的遗传距离也较为相近,遗传相似系数为0.946;而绿洲3号和绿洲6号与其余的7份材料明显区别开来,它们之间的遗传相似系数仅为0.810。
3 讨论与结论
随着DNA分子标记技术及其结构和功能基因组学的飞速发展,分子标记种类越来越多,但是,与传统分子标记相比,SCoT是一种能产生与功能性状遗传连锁的标记,可以对研究目标性状进行有效跟踪,具有操作简单、多态性高、遗传信息丰富、成本低廉、引物通用性强等优点[4],因此,其在相关重要功能基因研究以及分子标记辅助育种中具有十分重要的独特优点,受到众多研究学者的青睐。
种质资源是菌草产业技术发展的重要基础,而遗传多样性是种质资源研究的重要内容。近年来,随着菌草产业的发展,我国已积累了一定的菌草种质资源,但针对菌草种质资源的深入研究仍相对较少,这在一定程度上限制了菌草产业的发展。梅嘉洺等[15]在菌草上利用PCR-RFLP技术对46份种质资源PEPC基因进行了多样性分析,利用12对PEPC特异性引物对供试材料进行PCR扩增,扩增产物再经限制性核酸内切酶Hae III酶切、电泳,在46份材料上共获得176个变异类型,UPGMA聚类分析表明供试材料间遗传相似系数在0.86-1,在遗传相似系数为0.865时,可以将46份材料分为四大类群。本研究在此基础上,采用SCoT新型分子标记技术对47份菌草种质材料遗传多样性进行了较为系统的分析,选出的10个SCoT引物在47份菌草种质材料上共给出了282条多态片段,UPGMA聚类分析表明,供试材料间遗传相似系数在0.77-0.96,在遗传相似系数0.815时,可以将47份材料分为5个类群(图3)。本结果中的第I、IV和V类群和梅嘉洺等[14]聚类图中的第I、II和III类群相对应,但也给出了新的结果内容:(1)先前的研究未能将海南象草和细茎象草、漳州象草和热研4号粗茎象草、台湾甜草和高丹草变种等三对材料区别开来,而本研究利用10个SCoT引物将所有47份供试材料全部区别开来,显示了SCoT标记技术在菌草品系材料鉴定方面应用潜力;(2)先前的研究结果显示狼尾草、美洲狼尾草和81.95象草和其它23份同属狼尾草属(Pennisetum)植物遗传距离较远但被分配到同一个类群中,而本研究中,狼尾草和美洲狼尾草各自形成单独的类群(II和III),81.95象草被分配到了另一个含有甘蔗、高粱等材料的第IV类群中,揭示了狼尾草属的菌草种质材料间存在巨大的遗传多样性;(3)先前的研究结果显示类芦和莆田芦苇形成一个单独的类群,而在本研究中,这2个材料被分配到含有甘蔗、高粱等材料的第IV类群中;(4)目前没有闵牧42这份材料的植物学分类信息,在本研究的聚类分析中,它被分配在彼特草(Saccharum)和2个甘蔗材料(Saccharum)之间,结合先前的聚类分析结果,推断闵牧42应该属于甘蔗属(Saccharum);(5)本研究聚类分析显示,第I、III和V大类群内有明显的亚类群结构。这些结果虽然还不够完善,但为菌草种质资源的鉴定提供了初步的分子依据和技术手段,未来可望将该技术在菌草上进一步推广,通过采用更多的SCoT引物以及分析更多的菌草种质材料,同时还可以结合采用其它分子标记技术,如ISSR[18]、SRAP[19]、iPBS[20]等,以获得更加全面、可靠的聚类分析结果,为菌草种质资源的收集、管理和利用提供参考。
致谢 感谢福建农林大学国家菌草工程技术研究中心刘斌、林辉、吴金寿等老师提供的植物材料。
参考文献
[1] 林占熺. 菌草学(第三版)[M].北京:国家行政学院出版社,2013.
[2] 龙治坚,王 莹,向素琼,等.枇杷属植物分子标记利用的研究进展[J].果树学报,2013,30(3):480-488.
[3] 陆才瑞,喻树迅,于霁雯,等. 功能型分子标记(ISAP)的开发及评价[J].遗传,2008,30(9):1 207-1 216.
[4] 熊发前,唐荣华,陈忠良,等. 目标起始密码子多态性(SCoT):一种基于翻译起始位点的目的基因标记新技术[J].分子植物育种,2009,7(3):635-638.
[5] Collard C Y,Mackill D J.Start codon targeted (SCoT) polymorphism: a simple,novel DNA marker technique for generating gene-targeted markers in plants[J].Plant Mol Biol Rep,2009,27(1): 86-93.
[6] 熊发前,蒋 菁,钟瑞春,等.目标起始密码子多态性(SCoT)分子标记技术在花生属中的应用[J].作物学报,2010,36(12):2 055-2 061.
[7] Luo C, He X H, Chen H, et al.Analysis of diversity and relationship mango cultivars using start codon targeted (SCoT) markers[J].Biochem System Ecol, 2010, 38(6): 1 176-1 184.
[8] 韩国辉,汪卫星,向素琼,等. 多倍体枇杷SCoT分析体系的建立与优化[J].果树学报,2011,28(3):433-437.
[9] 秦国新,何 桥,梁国鲁,等.草莓属植物SCoT分析体系的建立及优化[J].果树学报,2012,29(3):393-397.
[10] 何庆元,王吴斌,杨红燕,等.利用SCoT标记分析不同秋眠型苜蓿的遗传多样性[J].草业学报,2012,21(20):133-140.
[11] 罗 霆,杨海霞,岑华飞,等.SCoT分子标记在割手密遗传图谱构建中的应用[J].植物遗传资源学报,2013,14(4):704-710.
[12] 黄 秀,张新全,张 瑜,等. 高牛鞭草(Hemarthria altissima)及其近缘种种质资源SCoT多样性分析[J].热带作物学报. 2013,34(11):2 192-2 199.
[13] 蔡元保,杨祥燕,陈豪军,等.SRAP结合SCoT标记分析番木瓜种质的遗传多样性[J].植物遗传资源学报,2014,15(2):292-298.
[14] 蒋晓梅,黄琳凯,张新全,等.利用SCoT分析柳枝稷遗传资源的多样性[J].植物遗传资源学报,2014,15(1):89-95.
[15] 蒋林峰,张新全,黄琳凯,等.中国鸭茅主栽品种DNA指纹图谱构建[J].植物遗传资源学报,2014,15(3):604-614.
[16] 梅嘉洺,黄小霞,王 咏,等. 46份菌草种质资源PEPC基因的PCR-RFLP多样性分析[J].热带农业科学,2015,35(11):45-50.
[17] Murray M G, Thompson W F. Rapid isolation of high molecular weight DNA[J]. Nucleic Acids Research,1980,8(19): 4 321-4 325.
[18] Sarwat M. ISSR: A Reliable and Cost-Effective Technique for Detection of DNA Polymorphism[J]. Methods in Molecular Biology,2012,862(7): 103-121.
[19] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical & Applied Genetics, 2001, 103(103): 455-461.
[20] Kalendar R, Antonius K, Smykal P, et al. iPBS: a universal method for DNA fingerprinting and retrotransposon isolation[J]. Theoretical & Applied Genetics, 2010, 121(8): 1 419-1 430.

